![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
R および RjpWiki に関する質問コーナー
過去の記事のアーカイブ
Q&A (初級者コース)/13 の目次
asap (2012-01-12 (木) 18:36:36)
今ある行列irisに、n列の場合分けに応じた新しい列を追加したいのですが、どのようにすればよいでしょうか
新しい列の変数には次のような値を入れたいのですがif (iris$Species[i] == "setosa") { iris$Sp[i] <- 1 } else { iris$Sp[i] <- 0 }
まち (2012-01-11 (水) 21:49:52)
geoRパッケージのvariogで標本バリオグラムの計算を行うと最大距離を持つポイントが省かれてしまいます。
同じデータでgstatパッケージvariogramを使うと省かれません。
パラメータの指定が悪いのでしょうか?
自己相関が無くなる距離なので推定には問題ないと思いますが気になります。
よろしくお願いします。library(geoR) library(gstat) # データの読み込み data(ca20) # geoR vcloud <- variog(ca20) (vcloud) plot(vcloud) # gstat ca20gstat <- cbind(ca20$coords[, "east"], ca20$coords[, "north"], ca20$data) colnames(ca20gstat) <- c("east", "north", "data") ca20gstat <- as.data.frame(ca20gstat) vfgstat <- variogram(data ~ 1, loc = ~east+north, data = ca20gstat, cutoff = vcloud$max.dist, width = vcloud$max.dist / 13) (vfgstat) plot(vfgstat)下記の 13 行目で geoR の場合 $n が 12 点でなく 11 点になってしまいます。
geoRの場合$n $u $v 1 543 43.77376 55.50552 2 1648 131.3213 78.09254 3 2364 218.8688 97.05838 4 2246 306.4163 117.1554 5 2531 393.9638 129.0646 6 2152 481.5114 146.4889 7 1708 569.0589 151.9713 8 1309 656.6064 153.6482 9 694 744.1539 144.7442 10 343 831.7014 146.8878 11 144 919.249 131.8958 12 59 1006.796 139.2288 13 11 1094.344 139.2727gstatの場合
np dist gamma 1 543 60.35719 55.50552 2 1648 132.58886 78.09254 3 2364 220.90992 97.05838 4 2246 305.65494 117.15539 5 2531 391.80321 129.06460 6 2152 482.02453 146.48885 7 1708 568.46999 151.97131 8 1309 652.62969 153.64820 9 694 739.59074 144.74424 10 343 826.14460 146.88776 11 144 913.61963 131.89583 12 59 995.10190 139.22881 13 12 1082.94416 165.16667使用環境
R version 2.12.1 (2010-12-16) Platform: i386-pc-mingw32/i386 (32-bit) gstat_1.0-8 geoR_1.6-35
hiro (2012-01-10 (火) 16:39:38)
R-2.14.1 for Windows(32bit)を使っています.
R-2.13.2までは、以下のサンプルでスライダーを動かすと、グラフはヒストグラムとカーネル密度共に滑らかに表示されます.
しかし、2.14以降、スライダーを動かしている間は一部のグラフが表示されなくなりました.この例ではカーネル密度が時々消えます.
グラフが複雑化すると、目立って見苦しくなります.
複数のWindowsPC(XP/Win7)で試しましたが、100%再現します.
もし解決策を御存知の方がいらっしゃれば、ご教示頂けないでしょうか.
宜しくお願いします.library(tcltk) draw.graph <- function(...) { upper <- as.double(tclvalue(Slider)) hist(x, freq = FALSE, ylim = c(0, upper), col = "cyan", border = "white") lines(den, lwd = 3) } x <- rnorm(200) den <- density(x) tt <- tktoplevel() Slider <- tclVar("0.5") slider <- tkscale(tt, from = 0.1, to = 1, resolution = 0.01, showvalue = TRUE, variable = Slider, orient = "horizontal", command = draw.graph) tkgrid(tklabel(tt, text = "upper"), sticky = "w") tkgrid(slider) tkfocus(tt)
kate (2012-01-06 (金) 17:47:23)
メモリーのエラーがでます。エラー: サイズ 176.4 Mb のベクトルを割り当てることができません > memory.limit() [1] 2047 > memory.size() [1] 817.03上記のエラーを疑問に思うのですが、2047MBの割り当てに対して、現在817MB使用で、あと約1200MB使えるという認識は違うのでしょうか?解決策を教えていただけないでしょうか
Win vista 32bit ,R2.14.1です。
Yasu (2012-01-05 (木) 14:41:54)
データとして(東経,北緯,高さ,値)の3次元空間の座標値と属性値からなるものを解析対象としています。
このデータに対して,東経,北緯,高さから計算される2点間の距離を尺度としてバリオグラムを計算したいのですが,どのようにすれば良いのか分かりません。
東経,北緯から計算される距離を用いたバリオグラムの計算はできました。
> data(meuse) > variogram(log(zinc) ~ x + y + elev, meuse)
manabe (2012-01-05 (木) 14:11:49)
month <- c("1月", "2月", "3月", "4月", "5月", "6月", "7月", "8月", "9月", "10月", "11月", "12月")全ての『月』の文字を取り除くにはどのようにすれば良いのでしょうか?
逆に、1:12を使って上記のベクトルを作るにはどのようにすれば良いのでしょうか?
ぞうに (2012-01-05 (木) 01:54:36)
いくつかのデータセットがあり各変数のlevels()を求めたいのですが,for文を使ってはできないのでしょうか?よろしくお願いします.
例えば,以下のデータセットがありますsex <- c("F", "F", "M", "M", "M") height <- c(158, 162, 177, 173, 166) weight <- c(50, 50, 60, 60, 70) ( x <- data.frame(SEX=sex, HEIGHT=height, WEIGHT=weight) )求めたいことは以下の事なのですが,
levels(factor(x$SEX)) levels(factor(x$HEIGHT)) levels(factor(x$WEIGHT))イメージとしては以下のような形で求めたいのですが,うまくいきません.for文を使ってできないでしょうか?
colname <- names(x) for(i in 1:3) { levels(factor(x$colname[i])) }以下,参考にしましたが解決できませんでした
http://q.hatena.ne.jp/1316156944
- x[, colname[i]]とすればいいのです。もっといえばx[, i]でOK。そもそもforを使わずにlapply(lapply(x,factor),levels)でもOKでは? -- 2012-01-05 (木) 02:03:17
- 解決しました.特に後者が参考になりました.どうもありがとうございました -- ぞうに 2012-01-05 (木) 03:10:50
- lapply を二重に使うのは初めてみました。sapply(x, function(z) levels(factor(z))) の方がわかりやすいかな。 -- 2012-01-05 (木) 10:23:55
Aizawa (2012-01-03 (火) 00:30:20)
functionの使用方法が理解できず悩んでいます。
abc <- function()(print("ABC")) と関数定義をすれば、> abcと入力した時点でprint("ABC")が実行されて"ABC"と返ってきそうな気がします。しかし、実際には、function()(print("ABC"))、と返ってきます。これだと、abcにfunction以下の文字式が代入されているだけに想えます。print文が関数として登録されているのではないようでしょうか。何か、関数定義に関して基本的なことを勘違いしているように想われます。全く初歩的な質問で恐縮ですが、どなたか疑問を解いて頂けると嬉しいです。
> abc <- function() (print("ABC"))
> abc()
[1] "ABC"
[1] "ABC"
> abc <- function() {print("ABC")}
> abc()
[1] "ABC"
> abc <- function() print("ABC")
> abc()
[1] "ABC"> (a <- 999) [1] 999 > print(a <- 999) [1] 999
Q&A (初級者コース)/13 http://www.okada.jp.org/RWiki/?%A3%D1%A1%F5%A3%C1%20%28%BD%E9%B5%E9%BC%D4%A5%B3%A1%BC%A5%B9%29%2F13 [ トップ | Tips紹介 | 中級Q&A | 初級Q&A | R掲示板 | 日本語化掲示板 | リンク集 ] [ リロード ] [ 新規 | 編集 | 凍結 | 差分 | ファイル添付 ] [ 一覧 | 検索 | 単語検索 | 最終更新 | バックアップ | ヘルプ ]やり損なった不細工な投稿も編集できる。 -- 河童の屁は,河童にあらず,屁である。 2012-01-08 (日) 23:05:43
MasHARA (2011-12-30 (金) 20:55:44)
Rのroundで小数第2位を丸めるために、round(1.05,1) を実行すると、日本の四捨五入とは異なり、1.1ではなく、1.0になります。
これは、切り上げ後の値の末尾の数字が偶数になるようにする、「JIS Z 8401」の2.c)で決められている丸め方のようですが、Rのプログラミングの練習を兼ねて、自分で四捨五入のための関数を作ることにしました。 正の数のみを扱うものとして、以下のような関数を作りました。> gonyu <- function(x, digits = 0) { > ceiling(x * 10 ^ digits) * 10 ^ (-digits) - + round(ceiling(x * 10 ^ digits) * + 10 ^ (-digits) - x, digits) > }作業環境は、Windows XP SP3で、version 2.13.0のRを使用しています。
この関数を試したところ、ほとんどの場合は問題なく四捨五入になりますが、時々、以下のように四捨五入にならない場合が発生しました。
例えば、関数の定義後、次の式を評価すると、> gonyu(1.45, 1)返ってきた値は、1.4 でした。
> gonyu(2.45,1)であれば、ちゃんと 2.5 が帰ってきます。そこで、問題の発生した
> x <- 1.45 > digits <- 1としてから、関数の中を一つ一つ見ていきました、すると、
> ceiling(x * 10 ^ digits) * 10 ^ (-digits) - xが 0.05 を返すものの、
> round(ceiling(x * 10 ^ digits) * 10 ^ (-digits) - x, digits)は、なんと 0.1 を返したのです。
> round(ceiling(1.45 * 10 ^ 1) * 10 ^ (-1) - 1.45, 1)も、なぜか0.1を返してきます。
> round(0.05, 1)を実行すれば、ちゃんと、0 (ゼロ)を返してきます。
> ceiling(1.45 * 10 ^ 1) * 10 ^ (-1) - 1.45は 0.05 を返してくるが、表示されていない桁で誤差を含むのでは、と思い、
> ceiling(1.45 * 10 ^ 1) * 10 ^ (-1) - 1.45 - 0.05を実行すると、返された値は、4.163336e-17 でした。
後ろにある引き算、2つをカッコでまとめて、> ceiling(1.45 * 10 ^ 1) * 10 ^ (-1) - (1.45 + 0.05)とすると、返される数字は、ちゃんと 0 です。
一体、この表示されない誤差は生じたり、生じなかったりするのでしょう。御存じの方が居られれば、御教授のほど、御願い申し上げます。
> func <- function(x, n)
+ {
+ a <- x * 10 ^ (n + 1)
+ b <- a - floor(a / 10 ^ n) * 10 ^ n
+ return(round(a + 5 * (b >= 5), -1) / 10 ^ (n + 1))
+ }
> func(1.45, 1)
[1] 1.5
> func(1.449999999999999, 1)
[1] 1.4
> func(2.5, 1)
[1] 2.5
> func(2.449999999999999, 1)
[1] 2.4takayuki (2011-12-27 (火) 20:39:17)
3次元の散布図を書くのに,scatterplot3dを使用しています。Z軸についてグラフを回転させたいのですがそのようなことは可能でしょうか。もし可能であればご教授お願い致します。
Montecarlo (2011-12-26 (月) 19:35:10)
現在では以下のマウス操作またはショートカットにてコンソール画面を消去することが出来る事は確認しています。「編集」→「コンソール画面を消去」 または 「Ctrl」+「L」この作業をスクリプトの命令から再現することは出来ますでしょうか。
ご存じの方がいらっしゃいましたら、よろしくお願い致します。
(2011-12-22 (木) 17:40:53)
回帰解析をしたいのですが、plot(x,y) を見ていたら、ガンマ分布の pdf を当てはめてみたくなりました。2つパラメータがありますが、それぞれを最小二乗法で求めるにはどうしたらいいのですか?
さらに、例えば勝手に自分が作った関数のパラメータを使うことも可能ですか?でっちあげの例ですが、y <- function(x,a,b){ log(a*x + x^-(a/(2*b)) }として、x と y が与えられた時に、a と b を推定したいということです。
よろしくお願いします。
T (2011-12-21 (水) 20:02:30)
超初心者質問で大変恐縮なのですが、ヘルプを読んでも解決せず、質問します。
例えば、matplot(outer(1:5, 1:3), type="b", pch=1:3, lty=1:3) legend("topleft", LETTERS[1:3], pch=1:3, lty=1:3, col=1:3, seg.len=4)とすると、左上に凡例が表示されますが、線にシンボルが上書きされた type="o" のような表示になります。
グラフは type="b" で描いていますので、凡例も同様にしたいのですが、legend() のヘルプを読む限りは、type="b" と同じ表現になるオプションはないようです。
もちろん、legend(..., type="b") とすると、エラーになります。
上記 2 行の出力画像を貼った方がよければ貼ります。
ヘルプの見落としのような気もいたしますが、よろしくお願いします。
matplot(outer(1:5, 1:3), type="b", pch=1:3, lty=1:3)
a <- legend("topleft", LETTERS[1:3], pch=1:3, lty=1:3,
col=1:3, seg.len=4, plot=FALSE)
i <- par("cxy")
sapply(1:3, function(j) {
with(a, {
segments(rect$left+i[1]/2, text$y[j], rect$left+i[1]*1.9,
text$y[j], col=j, lty=j)
segments(rect$left+i[1]*3.1, text$y[j], rect$left+i[1]*4.5,
text$y[j], col=j, lty=j)})
})
points(rep(a$rect$left+i[1]*2.5, 3), a$text$y, pch=1:3, col=1:3)
text(a$text$x, a$text$y, LETTERS[1:3])
with(a$rect, lines(c(left+w, left+w, left), c(top, top-h, top-h)))Montecarlo (2011-12-21 (水) 18:10:04)
文字列を表示する「cat」関数があると思いますが、大文字小文字のアルファベットを調べたところ、以下3個の文字が何を表しているかGoogle等で調べてみても分かりませんでした。
「\b」「\r」「\v」
実行コードcat("\a") #音が鳴る cat("\b") #不明 cat("\f") #フィード文字(半角スペース?) cat("\n") #改行 cat("\r") #不明 cat("\t") #タブ cat("\v") #不明(半角スペース?)この3個特殊文字の意味をご回答頂ければ幸いです。よろしくお願い致します。
いぬ (2011-12-21 (水) 10:18:21)
成績が勉強時間の2乗に比例しているのか3乗に比例するのか調べたいです。
式としては
y=a0+a1*x+a2*x^2
y=a0+a1*x+a2*x^2+a3*x^3
という式です。
手元の資料からは決定係数と調整済みの決定係数が1に近い方がいい、特に調整済みの決定係数が近いほうがいいと読み取れます。
また、t検定、F検定、AICも見たほうがいいと思うのです。
どれを1番優先してみればいいのでしょうか?
それとも違うものを見たほうがいいのでしょうか?
どなたか教えてください。
あとRでanovaの結果の見方がわかりませんので教えていただきたいです。
ほし (2011-12-21 (水) 00:13:46)
初めて投稿させていただきます。
パッケージVeganのRDAを用いた解析・解釈に苦戦しております。
20以上のデータを用いて解析を行い、plotで表にしました。
しかし、表には4つ程のデータ名しか表示されません。
また、summaryで詳細な情報を見ても、表示されたデータ名についてしか書かれていません。
これは、表示されなかったデータは取るに足らないデータであったということなのでしょうか?
初歩的な質問で恐縮ですが、どなたかアドバイスの程よろしくお願いいたします。
> plotで表にしました。
plotで表にするとはどういうことでしょうか。グラフではないのですか?
rda関数のExamplesを実行してみても表にはなりませんが。
> データ名 というのは具体的に何を指しているのでしょうか。 -- Iona 2011-12-21 (水) 06:17:14
> test <- read.csv("sample.csv", header=TRUE, row.names="x")
> testa <- read.csv("sample.csv", header=TRUE, row.names="x")
> testb.rda <- rda(test~.testa, nangteng, scale=TRUE)
> testb.rda
ここで、次に表示される
Call:にはtestbのデータ名(エクセル第二行目からの第一列です。サンプル名といった方がよかったかもしれません。。)はすべて表示されますが、
> plot(testb.rda)によってグラフ(表ではなくグラフでした。申し訳ありません。)を表示したところ、testbのデータだけ質問のようになってしまいます。
rda(x ~ z$y)を
rda(x ~ y, data = z)と書けるというものです。
dune.Manure <- rda(dune ~ Manure, data = dune.env) dune.Manureでは、dune.envの行名は表示されませんが、summary()の間違いではないですか? でも、一度目の投稿によるとsummary()では表示されないんですよね? -- Iona 2011-12-22 (木) 00:51:45
S (2011-12-20 (火) 18:14:37)
表を作って平均をとろうとすると、次のような警告がでます。mean(<data.frame>) is deprecatedR2.13では出なかったのですが、R2.14で出るようになったと思います。これは何でしょうか?
> mean(iris[,1:4])
Sepal.Length Sepal.Width Petal.Length Petal.Width
5.843333 3.057333 3.758000 1.199333
警告メッセージ:
mean(<data.frame>) is deprecated.
Use colMeans() or sapply(*, mean) instead. # これからは,こうしてね!という示唆
> colMeans(iris[,1:4]) # つまり,こんな風にとか
Sepal.Length Sepal.Width Petal.Length Petal.Width
5.843333 3.057333 3.758000 1.199333
> sapply(iris[,1:4], mean) # こんな風にとか
Sepal.Length Sepal.Width Petal.Length Petal.Width
5.843333 3.057333 3.758000 1.199333
愚かなるユーザは,「エラーメッセージを出して,正解を出す」のなら,「エラーメッセージを出さずに正解を出せばいいじゃないか」と思うのです。お偉いさんは,何を考えているんだろうか。ストイックなんでしょうなぁ。でも,愚かなる衆生はそんなのに従わされなくても言いと思うのだけど。> mean.data.frame
function (x, ...)
{
msg <- "mean(<data.frame>) is deprecated.\n Use colMeans() or
sapply(*, mean) instead."
warning(paste(msg, collapse = ""), call. = FALSE, domain = NA)
sapply(X = x, FUN = mean, ...)
}
となっているわけだけど,
> mean.data.frame
function (x, ...)
{
sapply(X = x, FUN = mean, ...)
}
にすれば良いだけだろうと思うのだ!カス (2011-12-20 (火) 14:03:20)
offset項を入れた時の推定結果を図示するには、どのようにすればよいのでしょうか?
二項分布でリンク関数を"cloglog"にした場合と、ポアソン分布(リンク関数はデフォルトの"log")の場合についてご教授いただけないでしょうか?
例えば、二項分布でoffsetなしの場合、bi <- glm(y ~ x, family = binomial(link = "cloglog")) lines(x, 1-exp(-exp(b + a*x)))とすれば良いと思うのですが、二項分布でoffsetありの場合
bioff <- glm(x ~ y + offset(log(z)), family = binomial(link ="cloglog"))この推定結果の図示はどのようにすればよいでしょうか?
また、同様にポアソン分布でオフセットなしの場合、po <- glm(y ~ x, family = poisson) lines(x, exp(b+ax))とすればよいと思うのですが、
pooff <- glm(y ~ x + offset(log(z)), family = poisson)の場合はどうでしょうか? よろしくお願い致します。
matak (2011-12-15 (木) 12:15:41)
CSVファイルを例えば、> data <- read.csv("read_sample.csv", header=TRUE, row.names=1)という風に読み込んだ時、CSVファイルの一番左上のセルだけ読み込まれないですよねぇ。
結果、読み込んだ行列で何らかの演算を行って、書き込んだCSVファイルも一番左上のセルが空白になってしまいます。
どうすれば一番左上のセルも読み込み&書き込みできるでしょうか。
読み込むCSVファイルの1行目および1列目は文字列で、第2行第2列目以降から数値データなので、header=TRUE, row.names=1で読み込んだ方が都合がいいのですが。
data <- read.csv("sample.csv")
data[, -1] <- function(data[, -1])
write.csv(data)Montecarlo (2011-12-12 (月) 21:25:13)
初の投稿で、なにぶん至らない点があるかと思いますがよろしくお願いします。
使用環境は以下です。R version 2.14.0 (2011-10-31) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base質問になりますが、行列の行に対してcumsumを適用することは出来るでしょうか?
> x <- c(1, 1, 1, 0, 0, 0, 0, 1, 1, 1, 1, 1, 0, 1, 1, 1, 1, 0, 1, 0, 1, 0, 0, 1, 0, 1, 1, 0, 1, 0, 0, 0, 0, 1, 1, 1, 0, 1, 1, 0, 0, 1, 1, 0, 0, 1, 1, 0, 0, 1, 0, 1, 0, 1, 1, 0, 1, 0, 0, 1, 1, 0, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 0, 0, 1, 0, 0, 0, 1, 1, 1, 0, 1, 0, 0, 1, 1, 0, 0, 1, 1, 0, 0, 0, 0, 1, 1, 1, 0, 0) > y <- matrix(x, nrow=10, ncol=10, byrow=TRUE)を実行すると、私の意図通りに10*10の行列ができあがります。
ここからが詰まっているところです。
この「y」の行に対するcumsumを適用したいと思っています。
列に対するcumsumは> apply(y, 2, cumsum)によって計算出来ていることは確認しています。
ですが> apply(y, 1, cumsum)ではうまくいかないのです。(結果はapply(y, 2, cumsum)に似ていますが違った答えが返ってきます。)
質問事項としてまとめると以下の点となります。
1.行に対してcumsumを適用することは可能か(cumファミリー全般)
2.列はうまくいくのに行でうまくいかない事からすると、cumsumの挙動はどのようになっているのか
以上2点について分かる方がいらっしゃいましたらお願い致します。
> (y <- matrix(1:12, 3, 4))
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12
> apply(y, 1, cumsum)
[,1] [,2] [,3]
[1,] 1 2 3
[2,] 5 7 9
[3,] 12 15 18
[4,] 22 26 30
> t(apply(y, 1, cumsum))
[,1] [,2] [,3] [,4]
[1,] 1 5 12 22
[2,] 2 7 15 26
[3,] 3 9 18 30
つまりのところ,t(apply(y, 1, cumsum))でよいでしょう。 -- 河童の屁は,河童にあらず,屁である。 2011-12-12 (月) 21:48:54Alin (2011-12-12 (月) 17:05:20)
C言語のint型、4バイト長のバイナリデータ(sample.bin)をreadBin関数を使って読み込もうとしているのですが、> conn <- file("sample.bin", "rb") > readBin(conn, integer(), n=100*100, size=4)のようにして読み込むと正常な値が読み込まれず(100*100はデータの要素数です)、
> readBin(conn, integer(), n=100*100, size=2)のようにして、sizeを2に指定すると、正常な値が読み込まれます。
これはRで扱うデータ型(?)の基本的な概念が、C言語等と異なっているからなのでしょうか?
vivaTS (2011-12-12 (月) 13:32:48)
R64 2.14.0 (Lion)を使用しています。
ccfで計算される2変数の時系列相関のp値, 95%信頼区間を求めるには,どのような方法があるのでしょうか。
波 (2011-12-06 (火) 17:13:05)
お世話になります。
CentOSにR 2.13.1をインストールし、プロキシサーバー経由でパッケージのアップデートをしようとしています。
Rのインストールディレクトリに以下のコマンドを記述した.Rprofileを保存しているのですが、update.packeges()で「HTTPステータスは407 Proxy Authentication Requiredです」といわれて接続できません。options(CRAN="http://cran.md.tsukuba.ac.jp/bin/windows/contrib/2.13") Sys.setenv("http_proxy"="http://(アドレス):(ポート番号)")また、.Rprofileを削除してRの起動直後の画面に上記のコマンドを入力しても接続できませんでした。
Windows XPのR 2.13.1ではマイドキュメントに上記の.Rprofileを保存することでアップデートが可能なことを確認しています。
Linuxの場合はWindowsと異なる設定が必要なのでしょうか?対応策をご存知であれば教えてください。
宜しくお願いします。
K (2011-12-06 (火) 10:29:04)
こんにちは。WinXP R 2.13.2を使用しています。
文字列ベクトルA,B,C・・・があり、これらに共通する要素を抽出したいと考えています。
例えば以下のような場合は"aaa"が共通しているので、"aaa"を抽出したいということです。A <- c("aaa","aaaa","aaaaa") B <- c("bbb","aaa","bbbbb") C <- c("aaa","ccc","cccc")実際には文字列ベクトルとその要素は数百個あります。apply()を使えばスマートに記述できそうな気はするのですが、どうにもできませんでした。
どなたか御助言を宜しくお願いします。
system.time({
### テストデータを作る
### 要素数 n=500 個のリスト
### 各要素は 503 個の要素を持つ文字列ベクトル
### テストのため最後の 3 つ, foo, bar, baz は全ベクトルに含まれる
### 各要素は 1 〜 5 文字からなる
set.seed(1234567)
n <- 500
k <- 500
d <- vector("list", n)
for (i in 1:n) {
str <- character(k)
for (j in seq_along(str)) {
str[j] <- paste(sample(letters,
sample(5, 1), replace=TRUE), sep="", collapse="")
}
d[[i]] <- c(str, "foo", "bar", "baz")
}
})
system.time({
### 重複する文字列の抽出
ans <- intersect(d[[1]], d[[2]])
for (i in 3:n) {
ans <- intersect(ans, d[[i]])
}
print(ans)
})
実行結果は
テストデータの作成にかかった時間
ユーザ システム 経過
9.297 0.257 9.508
共通要素
[1] "foo" "bar" "baz"
共通要素を抽出するのにかかった時間
ユーザ システム 経過
0.049 0.001 0.055ひろ (2011-12-05 (月) 12:55:27)
ヘッダに括弧が含まれるCSVから正しくヘッダを取得したいのですが,方法がわからずに困っております.
下記例ではファイル中のヘッダは「ほげ(ほげ)」なのですが,read.csvで読み込むと「ほげ.ほげ.」と括弧→.となり困っております.
もし改善方法をご存知でしたらご教示いただければ幸いです.
環境:win7 64bit + R 2.14.0 64bit
調べたこと:?read.csvや?read.tableの結果からparenthes,bracket(丸かっこ)の単語がないか.googleで本サイト(site:)指定/指定なしに対して,「括弧 R言語 ヘッダ名or列名」で調べてみました.
testCSV.csvの中身は下記通りです.ほげ(ほげ) 1 2 3
> read.csv("input/testCSV.csv", header=TRUE) ほげ.ほげ. 1 1 2 2 3 3
笹川よつ斗 (2011-12-05 (月) 12:42:13)
例えば、x <- data.frame( matrix(rnorm(500), ncol=10) ) names(x) <- c( sample(LETTERS[1:26], 9, FALSE), 'y' )というデータがあります。
column の名前の 'y' 以外を入力せずに lm() を使うことは可能でしょうか?
大きなデータフレームから column をサンプルして繰り返し解析する、ということを試しています。 for loop の中で応用することになります。 今のところ以下のようにやっていますが、もう少し簡潔な方法があるものかと思い投稿しました。f <- paste( 'y ~', paste(names(x)[1:9], collapse='+') ) lm( as.formula(f), data=x )
> x <- data.frame( matrix(rnorm(500),ncol=10) )
> lmtmp <- lm(X10 ~ ., data=x)
> step(lmtmp)
Start: AIC=-3.58
X10 ~ X1 + X2 + X3 + X4 + X5 + X6 + X7 + X8 + X9
Df Sum of Sq RSS AIC
- X7 1 0.0288 31.227 -5.5375
- X1 1 0.0427 31.241 -5.5154
- X2 1 0.0650 31.263 -5.4796
- X9 1 0.0659 31.264 -5.4783
- X4 1 0.0936 31.291 -5.4340
- X5 1 0.1595 31.357 -5.3287
- X3 1 0.4099 31.608 -4.9310
<none> 31.198 -3.5837
- X6 1 1.3648 32.563 -3.4429
- X8 1 8.1977 39.395 6.0814
(途中省略)
Step: AIC=-16.17 ### AIC基準で最終的に選ばれたモデル
X10 ~ X8
Df Sum of Sq RSS AIC
<none> 33.405 -16.1657
- X8 1 7.3309 40.736 -8.2455
Call:
lm(formula = X10 ~ X8, data = x)
Coefficients:
(Intercept) X8
-0.04151 0.39186> x <- data.frame(matrix(rnorm(10000),ncol=100))
> Formula <- unique(sapply(seq(10^4),FUN=function(i) paste("X100 ~ ",
paste(sort(sample(names(x)[-100],9)),collapse="+"))))
> res <- lapply(seq(10^4), FUN=function(i) lm(Formula[i],data=x))
> Formula[9999]; res[[9999]]
[1] "X100 ~ X20+X25+X30+X41+X55+X6+X68+X80+X99"
Call:
lm(formula = Formula[i], data = x)
Coefficients:
(Intercept) X20 X25 X30 X41 X55
0.054830 0.020898 0.036853 0.025012 -0.007347 -0.138181
X6 X68 X80 X99
0.013766 0.071437 -0.010897 0.083487
斉藤正 (2011-12-02 (金) 12:26:18)
これまで scatterplot3 を使って、計算結果を3次元表示していました。Rのバージョンを2.14.0に上げたら、> library(scatterplot3d) 以下にエラー library(scatterplot3d) : '‘scatterplot3d’' という名前のパッケージはありません : 関数 "scatterplot3d" を見つけることができませんでしたというエラーメッセージがでて、図が描けなくなりました。対処法をお教えください。
@garuby (2011-11-30 (水) 18:58:49)
すでに重回帰モデルやSVMモデルを作成しており、このモデルを使って新しいサンプルの目的変数を予測することを現在Rscriptで行なっています。この方法の場合、毎回重回帰モデルなどを保存したファイルをloadして、目的変数を予測させているので、いつも計算するのに時間がかかります。
計算する量は、サンプル数が1つしかない場合もありますし、10万を超える場合もあります。複数の人が利用するため頻度も多いです。
そこで、Rserveを使って最初にモデルをloadして予測ジョブごとでわざわざloadすることを避けられないか検討しています。ヘルプを見ますと、セッション番号を共有すれば何とかなりそうではないかと思っています。が、まったく分からず立ち往生しています。
このようなことはRserveで実現できるのでしょうか?あるいは他の方法で解決するのでしょうか?
さ (2011-11-29 (火) 19:09:19)
・Rでlm関数を使って分析をしました。summaryで結果を出したのですがEstimateの所の数字をどのようにとらえていいのかがわかりません。*の数も同じです。この数字や*は大きかったり、多いほうが相関関係があるのでしょうか?
・もうひとつstep関数も用いました。これはlm関数よりも詳しい値が出ると考えてよいのでしょうか?
・最後に題とは少し変わるのですが、目的変数が見付からないときにはなしでそれぞれの相関をもとめる(lm関数とstep関数を用いて)ことは可能でしょうか?例えば県ごとの脳梗塞の死亡率と癌の死亡率と糖尿病の死亡率しか出ていない場合です。県を目的変数にしようとしましたが、数字でないせいかエラーになりました。
どなたか教えてください。よろしくお願いします。
yyy (2011-11-29 (火) 15:30:47)
お世話になります.
例えば「3*x^2-4*x*y+3*x+6*y^2+8*y」のような2変数の最適化を行うにはどうすればよいのでしょうか.
uniroot()は1変数でしか使えませんよね?
どなたかご教示下さい.宜しくお願いします.
> f <- function(p) 3*p[1]^2-4*p[1]*p[2]+3*p[1]+6*p[2]^2+8*p[2] > nlm(f, c(1,1)) $minimum [1] -6.107143 $estimate [1] -1.214287 -1.071429 $gradient [1] 6.422047e-07 4.269174e-07 $code [1] 1 $iterations [1] 3
zoo (2011-11-29 (火) 11:55:26)
お世話になります。
・集合xと集合yがある
・サイズは x > y で、yはxに全て含まれる
・xに含まれるyをyの順番を維持したままxから抜き出したい
という処理をしようとしています。
たとえばx <- c("A","B","C","D","E") y <- c("D","E","B") x[x %in% y]このように入力すると「"B" "D" "E"」が得られますが、yの順番である「"D" "E""B"」として出力するにはどうすればよいでしょうか?
どなたかご教示ください。宜しくお願いします。
> x <- c("B","A","B","D","C","D","E")
> y <- c("D","E","B")
> unlist(sapply(y, function(ch) x[x==ch]))
D1 D2 E B1 B2
"D" "D" "E" "B" "B"> (x <- data.frame(str.x=LETTERS[1:5], num.x=1:5))
str.x num.x
1 A 1
2 B 2
3 C 3
4 D 4
5 E 5
> (y <- data.frame(str.y=c("D", "E", "B"), num.y=c(1,3,6)))
str.y num.y
1 D 1
2 E 3
3 B 6
> x[sapply(y$str.y, grep, x$str.x),]
str.x num.x
4 D 4
5 E 5
2 B 2m (2011-11-29 (火) 02:23:48)
Mac OS X(10.6.8)でR2.14.0を使用しています。plotがデフォルトの画面上(Quartz)では問題無いのですが、png()では文字化けしてしまいます。png("hoge.png", width=640, height=640, unit="px") plot(1,1) text(1,1,"ほげ") dev.off()こうすると「ほげ」部分が□□になってしまうのですが、何か見落としているためでしょうか?
ターミナルから「R」と入力して起動した場合も、Rのアプリケーション上の「Rコンソール」でも同じ結果になります。
よろしくお願いいたします。
png("test.png")
par(family="HiraMaruProN-W4")
hist(rnorm(10000), main="日本語を使う")
dev.off()shannon (2011-11-28 (月) 00:13:55)
既出でしたらすいません。
Rではデータ型がinteger(整数),numerical(実数),complex(複素数),character(文字)に分類されるとのことですが、
C言語などで使われるfloat(32bit),double(64bit)でビット幅を固定して、データを書き込むことはできないのでしょうか?
writeBin関数ではそのようなオプションがないように思うのですが、
書きこむ前のデータのビット幅を何らかの方法で固定して、その後writeBin関数で書き込む、という流れになるのでしょうか?
分かりにくい質問かもしれませんが、よろしくお願い致します。
> object.size(1L) 32 bytes > str(1L) int 1 > object.size(as.single(1L)) 120 bytes > str(as.single(1L)) atomic [1:1] 1 - attr(*, "Csingle")= logi TRUE > object.size(as.double(1L)) 32 bytes > str(as.double(1L)) num 1
QDY (2011-11-24 (木) 22:47:47)
最新バージョンのRには並列処理用のパッケージ parallel が同梱されています。特にマルチコアのパソコンでは便利だと思いますが、並列処理したい関数が(その関数にとっての)外部変数を含む場合はエラーになるようです。おそらく子プロセスが親プロセスの実行環境を知らないからだと思いますが(?). 関数の実行環境(外部変数)をすべての子プロセスで共有する簡単なおまじないがあるのでしょうか。(Linux, R2.14.0)> library(parallel) > cl <- makeCluster(rep("localhost", 4), type="SOCK") > y <- 3 > tmp <- function(x) x+y > clusterApply(cl, 1:4, tmp) 以下にエラー checkForRemoteErrors(val) : 4 nodes produced errors; first error: オブジェクト 'y' がありません > stopCluster(cl)
> library(parallel)
> cl <- makeCluster(rep("localhost", 4), type="SOCK") # 4つのコアで実行
> y <- 3
> tmp <- function(x) x+y
# すべての実行時環境オブジェクトを子プロセスにエクスポート。
# この例では clusterExport(cl,"y") で十分
> clusterExport(cl, ls())
> clusterApply(cl, 1:4, tmp)
[[1]]
[1] 4
[[2]]
[1] 5
[[3]]
[1] 6
[[4]]
[1] 7
> stopCluster(cl)
バイソン (2011-11-22 (火) 13:20:05)
Linux(CentOS5.5)上で、R version 2.11.1を使用しています。a = 1 cat(a,"\n")上のような内容のRファイル(test.r)を作成して、
> R --vanilla --quiet < test.rのようにして実行すると、コンソール上には実行コマンドとcat関数による出力の両方が表示されてしまいます。
=====コンソール表示====== > a = 1 > cat(a) 1 =========================cat関数で出力する値だけをコンソール画面に表示させたいのですが、R --helpを見てもそのようなオプションが無いように思います。
sink関数を使って、出力先を別ファイルに変更させて確認するという方法もあるとは思うのですが、 出力させたい部分で一回一回sink関数で囲うのが面倒なので、
できればRコマンドのコンソール出力を抑えて、cat関数等で出力させる値(上の例では1)のみをコンソールに表示できないものかと考えているのですが、良い方法はありますでしょうか?
> R --vanilla --slave < test.rのようにしても同じように出力が抑えられました。-- バイソン 2011-11-24 (木) 04:56:01
まあくん (2011-11-22 (火) 09:05:29)
タイトルの通りなんですが、関数persp()で軸のタイトル(xlab,ylab,zlab)が、角度によってお互いが重なってしまい、見にくくなってしまうので、文字サイズの変更もしくは、表示位置の微調整ができないでしょうか?
小田ちん (2011-11-19 (土) 00:51:34)
ggplot2を使いたいのですが、plyrのロードで失敗します。
MacのR64を使っています。どうすれば使えるようになるのでしょうか?
よろしくお願いいたします。> library(ggplot2) 要求されたパッケージ reshape をロード中です 要求されたパッケージ plyr をロード中です Error in dyn.load(file, DLLpath = DLLpath, ...) : 共有ライブラリ '/Library/Frameworks/R.framework/Versions/2.14/ Resources/library/plyr/>libs/x86_64/plyr.so' を読み込めません: dlopen(/Library/Frameworks/R.framework/Versions/2.14/Resources/ library/plyr/libs/x86_64/plyr.so, 6): Library not loaded: /Library/Frameworks/R.framework/Versions/2.13/Resources/lib/libR.dylib Referenced from: /Library/Frameworks/R.framework/Versions/ 2.14/Resources/library/plyr/libs/x86_64/plyr.so Reason: image not found エラー: パッケージ '‘plyr’' をロードできませんでした
T.I. (2011-11-16 (水) 07:11:07)
Rの初心者です。グラフの描き方に苦戦しております。
Boxプロットで、項目軸(X軸)を数値軸にしてグラフを描きたいのですが、方法をご存知の方がおられましたら、教えていただけないでしょうか?
set.seed(100) ; x <- sample(c(1,2,5,7), 200, TRUE) ; y <- rnorm(200) boxplot(y~x, at=sort(unique(x)), xlim=c(0,8))
yasushi (2011-11-15 (火) 18:56:52)
R初心者です。
次の2つの実行結果は同じになると思ったのですが,なりません(2番目の数値がおかしい)。> x <- matrix(c(1:4), nrow=2, ncol=2, byrow=T) > (x/apply(x, 1, sum)) y <- matrix(c(1:4), nrow=2, ncol=2, byrow=F) t(y/apply(y, 2, sum))2番目のものを,colSumsにしてみても同様の結果でした。R 2.14.0でもR 2.2.0でも同様の結果でした(Window XP上で)。
どういうことなのでしょうか?
> (x <- matrix(c(1:4), nrow=2, ncol=2, byrow=T))
[,1] [,2]
[1,] 1 2
[2,] 3 4
> apply(x, 1, sum)
[1] 3 7
> x/apply(x, 1, sum)
[,1] [,2]
[1,] 0.3333333 0.6666667
[2,] 0.4285714 0.5714286
> (y <- matrix(c(1:4), nrow=2, ncol=2, byrow=F))
[,1] [,2]
[1,] 1 3
[2,] 2 4
> t(y/apply(y, 2, sum))
[,1] [,2]
[1,] 0.3333333 0.2857143
[2,] 1.0000000 0.5714286
> apply(y, 2, sum)
[1] 3 7
> y/apply(y, 2, sum)
[,1] [,2]
[1,] 0.3333333 1.0000000
[2,] 0.2857143 0.5714286
> t(y/apply(y, 2, sum))
[,1] [,2]
[1,] 0.3333333 0.2857143
[2,] 1.0000000 0.5714286
バグでも何でもない,当たり前の実行結果です。(このような,単純な計算結果にバグはないと思うのが普通です)。不審に思えば,デバッグ(途中結果の詳細を表示させて,何がどのようになっているかを理解する)する。> t(y)/apply(y, 2, sum)
[,1] [,2]
[1,] 0.3333333 0.6666667
[2,] 0.4285714 0.5714286
ということになりますね。> x <- matrix(c(1:4), nrow=2, ncol=2, byrow=T)
> x/apply(x, 1, sum)
[,1] [,2]
[1,] 0.3333333 0.6666667
[2,] 0.4285714 0.5714286
> prop.table(x, 1)
[,1] [,2]
[1,] 0.3333333 0.6666667
[2,] 0.4285714 0.5714286
まあ,そんな関数があることなんか知らなくてもどうってことはないです。 -- 河童の屁は,河童にあらず,屁である。 2011-11-15 (火) 21:21:41前田 (2011-11-14 (月) 22:45:36)
R初心者のものです。
大学の卒業研究でポンドの解析を行っています。
ARIMAモデルでの解析結果は出せるのですが
解析結果の詳細を表示させたいのですが
どなたかお知恵を貸していただけませんでしょうか?
パワースペクトルなどの結果も見れたらと思います。
宜しくお願い致します。
(x <- stats::arima(lh, c(2, 0, 1)))ここで表示されるものでは不十分ということでしょうか。
みのむし (2011-10-27 (木) 13:26:24)
Rの初心者です。簡単な質問で大変申し訳ないのです。
ある関数の、2階の導関数をもとめ、それを他の計算に使いたいと思っています。f <- deriv(~x^2*y, c("x", "y"), function(x, y) { }, hessian=TRUE)と書くと、4種類の2階の導関数を計算してくれるのはわかるのですが、個別の2階の導関数をどう選択して良いのかわかりません。たとえば、この場合、xとyで1回ずつ微分したものはどう選択すればよいのでしょう?
attr[f, "hessian"][x, y]こんな風にかいてみたのですが、だめなようです。ご存知の方教えていただけませんか?
> f12 <- function(x, y) attr(f(x, y), "hessian") > f12(1, 2) [1] 2 > f12(4, 3) [1] 8
nkoji (2011-10-08 (土) 14:45:51)
ベクトルのある値の連続と各連続における順番を取得したいです
例えば,c("a","a","a","b","c","a","b","a","a")に対してc(1,2,3,0,0,1,0,1,2)を得たいのです。今はこんなコードでやっています。A <- c("a","a","a","b","c","a","b","a","a") B <- rep(0,length(A)) A.str <- paste(A, collapse="") #開始位置と長さの取得 C <- gregexpr("a+", A.str) #C.mat[1,] 開始位置 C.mat[2, ] 長さ C.mat <- rbind(C[[1]], attr(C[[1]], "match.length")) B[A=="a"] <- unlist(apply(C.mat, 2, function(x){c(1:x[2])}))ベクトルの要素を一つずつ比較するよりは速いと思うのですが,上よりもっと速くなるコードがあれば教えてほしいです。
> my.func <- function(A)
+ {
+ B <- integer(length(A))
+ C <- rle(A == "a")
+ B[A == "a"] <- unlist(lapply(C$lengths[C$values], function(x) 1:x))
+ B
+ }
> your.func <- function(A)
+ {
+ B <- rep(0, length(A))
+ A.str <- paste(A, collapse="")
+ # 開始位置と長さの取得
+ C <- gregexpr("a+", A.str)
+ # C.mat[1,] 開始位置 C.mat[2, ] 長さ
+ C.mat <- rbind(C[[1]], attr(C[[1]], "match.length"))
+ B[A == "a"] <- unlist(apply(C.mat, 2, function(x) {c(1:x[2])}))
+ B
+ }
> a <- c("a","a","a","b","c","a","b","a","a")
> my.func(a)
[1] 1 2 3 0 0 1 0 1 2
> your.func(a)
[1] 1 2 3 0 0 1 0 1 2
> a <- sample(letters[1:5], 1000000, replace=TRUE)
> system.time(my.func(a))
ユーザ システム 経過
0.615 0.035 0.646
> system.time(your.func(a))
ユーザ システム 経過
2.448 0.040 2.453End.index.all <- cumsum(C$lengths) Start.index.all <- End.index.all - (C$lengths - 1) End.index <- End.index.all[C$values] Start.index <- Start.index.all[C$values]
my2.func <- function(A)
{
B <- A == "a"
C <- rle(B)
B[B] <- unlist(lapply(C$lengths[C$values], function(x) 1:x))
B
}もうり (2011-10-06 (木) 08:38:06)
> A <- read.table("matrix.dat") > B <- matrix(A[1,], 3, 3) > kappa(B, exact=TRUE)としたところ,
Error in svd(z, nu = 0, nv = 0) : infinite or missing values in 'x'というエラーが出力されました.
matrix.dat は1000行9列スペース区切りで実数を並べたテキストファイルです.
そこで,svd 関数の定義を見たところ,if (any(!is.finite(x))) stop("infinite or missing values in 'x'")との記述がありました.そこで確認のため,
> any(!is.finite(B))としたところ結果はTRUE.
ここまでは納得できなくもないのですが,> kappa(A, exact=TRUE)はエラーが出ず,しかも,
> any(!is.finite(A))の結果はTRUEです.
> A <- matrix(scan("matrix.dat"), ncol=9)と読み込むことによって,
> any(!is.finite(A)) > any(!is.finite(B))が両方FALSEとなったため一応解決はしましたが,すっきりしません.
原因に心当たりがある方がいらっしゃったらお教えいただければと思います.
最後に実行環境について記述しておきます.$ uname -sr Linux 2.6.18-92.el5 $ R --version R version 2.10.0 (2009-10-26)
怪獣 (2011-09-01 (木) 12:21:54)
よろしくお願いします。初級Q&A(10)のところで,LSMEANについてのやりとりがされていました。私のデータでLSMEANを得ることはできたのですが,グループの変数が3つあり,多重比較をしなければいけません。教えて頂きたいことは,LSMEANをベースにした3つの群のそれぞれのsdの求め方と二次データを使った多重比較の方法です。被験者数はすべて同じ数です。どうぞよろしくお願いいたします。
さとう (2011-08-29 (月) 18:16:59)
Rでcsv形式のデータからXMLへ変換しています。日本語のタグやデータも扱いたいのですが,以下のコードを実行すればわかるように,日本語が文字化けします。
(タグは文字化けしませんが,データの値は文字化けします。)
これを防ぐ方法はあるでしょうか?
library(XML) data <- c("変数1の値です。","value of variable 2.") xml <- xmlTree() xml$addTag('データセット', close=FALSE) xml$addTag("データ", close=FALSE) xml$addTag("変数1", data[1]) xml$addTag("変数2", data[2]) xml$closeTag();xml$closeTag();xml$closeTag() cat(saveXML(xml))
> cat(saveXML(xml,encoding = "UTF-8"))
<?xml version="1.0"?>
<データセット>
<データ>
<変数1>変数1の値です。</変数1>
<変数2>value of variable 2.</変数2>
</データ>
</データセット>R初心者 (2011-07-25 (月) 17:59:00)
今大学でRを使って研究をしているものですが初心者なので苦戦しています
今、等間隔でない座標値がいくつかあって線で結ばれたデータがあります
このデータを等間隔に(例えば2m間隔とか5m間隔とか)プロットさせるようなプログラミングを作成したいのですがどのようにやったらいいのでしょうか
簡単な質問でしたらすみません。何分初心者なもので・・・
ご指導よろしくお願いします。
kero_10625 (2011-06-22 (水) 12:26:37)
節電対策などで、建物のエネルギー解析が必要になっています。ビル管理システムにExcelで記録された数百から一万程度の多数のファイルの処理を行っております。
Excelの単一のセル、1列あるいは1行だけを読み込む必要が出てきました。
RODBCを用いて、sqlQuery を使うとデータフレームで値を返すためか、空欄のセルがあるとエラーが発生します。
xlsReadWriteパッケージのread.xls()関数を使うと可能なのですが、xlsx形式に未対応であり基本的にはシェアウエアであり将来の対応に不安が残ります。
建築技術者にRを使ってもらおうと考えていますので、Pearlなどあまり追加のソフトのインストールは避けたいです。
何か良い方法をご存じでないでしょうか。
ktr (2011-06-21 (火) 21:30:21)
Rで,ある関数を展開し,中身を見ると.CとしてC言語で書かれた関数が使用されていました.Rの関数は関数名をタイプすれば中身を見ることが出来ますが,このように他言語で書かれた関数をRで展開することは可能なのでしょうか?
竹澤 (2011-05-31 (火) 16:27:12)
t.test()の機能を確認するために以下のようなRプログラムを試しました。function() { nt <- 10000 pt <- 0 for(kk in 1:nt){ set.seed(12+kk*144) xx1 <- abs(rnorm(50, mean = 0, sd = 1)) xx2 <- abs(rnorm(50, mean = 0, sd = 1)) xx <- c(xx1, - xx2) ttest1 <- t.test(xx, mu = 0) pp <- ttest1$p.value if (pp < 0.05){ pt <- pt +1 } } pt <- pt/nt print(pt) }正規分布に従うデータを作成する方法がややひねくれています。結果は、0.05に近くなるはずなのに0.0018になりました。正規分布に従うデータを作成するときに普通の方法を使うと0.0517になりました。このような違いが生じるのはなぜでしょうか。
miya (2011-05-02 (月) 10:11:04)
polr関数を用いて,順序ロジットモデルを推計しています.
SUMMARY()を利用すると,推計結果が表示されます.
その際,AIC指標が表示されますが,尤度比は表示されません.
初期尤度,最終尤度,ならびに尤度比を求めたいのですが,polr関数にどのようなオプションを付ければよいのか,教えていただけると助かります.
もしかすると,定義式にしたがって,自分で計算式を作成して計算するしか方法がないのでしょうか.
ウルトラ初心者 (2011-04-18 (月) 15:33:39)
BioconductorのDESeqというパッッケージを使ってます。ほとんどコピペ程度でしか使えてません。plot(res$baseMeanA, res$baseMeanB, pch=20, cex=.1, col = ifelse( res$padj < .1, "red", "black" ))というコマンドで散布図がマニュアルに載っていて、そのままコピペしてかけていました。
resという表のpadjカラムの値がが0.1より小さければ赤、それ以外を黒という風にプロットされています。
さて、別のパッケージBaySeqで統計解析しか結果もこの散布図に反映させたいと考えました。
そこで、一つカラムを増やしてanalysisとなづけ、DESeqで有意なデータを1、BaySeqで有意なデータを2、DESeqでもBaySeqでも有意なデータを3、それ以外を0というように入力しました。
そこで、analysisカラムをみて1ならば赤、2ならば青、3ならば緑、0ならば黒というように上のplotコマンドを改良したいのです。
たぶん、ifを重ねて使っていけばいけそうなのですが、どのようにググってよい物かわかりません。
全くの丸投げ質問で恐縮ですが、よろしくお願いします。
さと (2011-04-17 (日) 05:21:20)
現在、クアッドコアのCPUを搭載したWINDOWSマシンにRをインストールして利用しています。
そのうえで、for文を使って繰り返し計算を行っていますが、非常に時間がかかっております。
Rを立ち上げて計算させても一つのコアしか使わないので、マルチコアの資源を有効に使えておりません。
仕方がないので、Rを4つ一度に立ち上げて、for文の繰り返しを, 1:25, 26:50, 51:75, 76:100というように分けて実行しています。
これで計算時間は短くなるのですが、もっとスマートにやる方法をご教授いただければ幸いです。
UNIX環境で使える multicore というパッケージは見つけたのですが、WINDOWSで使える方法を見つけられませんでした。
こちらの環境ですが、
OS: Windows 7 Professional, Service Pack 1
プロセッサ: Intel(R) Core(TM) i7 CPU M620 @ 2.67GHz
実装メモリ(RAM): 8.00GB
システムの種類:64ビット オペレーティングシステム
なにとぞよろしくお願いいたします。
Tetsu (2011-01-27 (木) 22:59:49)
現在、ts.plotを使用して、時系列データの作図をしております。
その際に、1つの点を黒丸、もう一方の点を白抜き三角でプロットを行っております。
三角が手前に来るようにデータ系列の指定の順番を変えたり、par(plot=new)を使用して重ね書きをしてみたのですがうまくいきません。
結局、三角の中身が白で塗りつぶされていないのが問題だと思うのですが、pchで指定可能な白三角と黒丸を一通り試したところ、全て同様の図が表示されてしまいました。
このような問題を解決する方法は無いのでしょうか?
あと、一点質問があります。
作図をする際に見易くするために、軸に主目盛りと副目盛りをつけようとしております。
このような場合はaxis()を2回使用して、目盛りの長さが異なる軸を重ねて描く以外に方法はないのでしょうか?
教えていただけないでしょうか?
よろしくお願いいたします。
Natsu (2011-01-26 (水) 00:02:56)
Rをインストールして、Rコマンダーを使いたいのですが、パッケージからRコマンダーを選ぶと、以下のように出てしまいます。install.packages(NULL, .libPaths()[1L], dependencies = NA, type = type) 中で警告がありました: 'lib = "C:/PROGRA~1/R/R-2.12.1/library"' は書き込み可能ではありません 以下にエラー sprintf(msg, userdir) : <83>シ<e3>Ν縺吶k縺溘a縺ォ蛟倶ココ逧<84>縺ェ繝ゥ繧、繝悶Λ繝ェ '%s' 繧剃ス懊j縺溘>縺ョ縺ァ縺吶°<ef>シ<9f> に不正なマルチバイト文字がありますどのようにすればインストールできるのでしょうか?
初歩的な質問で申し訳ないのですが、教えてください。
S.K (2011-01-21 (金) 23:19:26)
GAMを用いてデータ解析をしているのですが、変数選択の方法が分からなくて
困っています。web上にはstep.gamというものがあるそうなのですが、どのパッケージに入っているかもわからない状態です。何かご意見を頂けないでしょうか。よろしくお願い致します。
R初心者 (2011-01-10 (月) 09:58:15)
R初心者です。
JavaからRserveを利用して、Rを起動しようとしてます。
そこである程度同時実行をさせて、最終的に200件ほどの要求をRserveになげるようにしています。そこで、問題なのですが、Rserveからコネクションエラーが数十件返ってきます。そのコネクションエラー数は毎回毎回まちまちで数も特定できません。私が調べた限りでは、Rserveでコネクションの制限を設定している部分がないことから現状なぜこのようになるか不明な状態です。
そこでもしこのような現状に対する情報をお持ちでしたら、教えていただけないでしょうか。よろしくお願いいたします。
H.A (2010-12-13 (月) 21:07:38)
はじめまして
大学で企業財務を勉強しているものです。
さて、初歩的な質問で恐縮なのですが、Rでパネル分析を行った際に、企業固定効果の係数を出力することはできないのでしょうか?
各企業ごとの企業固定効果を分析上使用する必要があるため、それらをぜひとも抽出したいのです。
もしコードなどをご存じのかたがいましたらご教授お願いします。
XC (2010-12-11 (土) 12:04:28)
はじめまして
Rを調べ出しの初心者です
MATLAB系のスクリプトをRで動くようにする変換プログラムを作っているのですが、元のスクリプト群で多用されている、スカラを返す補間関数がマニュアルを探しても見付かりませんでした。
例えば、エンジン回転数ー発生トルクの2次元のマップ(回転数1000毎とか)、rpm_tqがあったとして、任意の、例えば2850回転の時の発生トルクであれば、
interp1()を使用して、
tq = interp1(rpm_tq(:,1), rpm_tq(:,2), 2850)
(rpm_tq(:,1)、 rpm_tq(:,2)は、回転数とトルクのベクタになります)
の様になっているので、Rで
tq <- interp1(rpm_tq[1], rpm_tq[2], 2850)の様に書けると嬉しいのですが
皆様のご教示をいただけましたら幸甚です。
arima (2010-10-31 (日) 15:09:12)
直線的なトレンドのある時系列に対してarima(時系列,order=(1,1,0))とすると、AR部分の係数が出力されるのみで定数項が出力されません。
これにpredictを施しても、定数項がないため、将来の値が一定となってしまいます。
定数項を出力させるためにはどうすればよいのでしょうか。
宜しくお願い致します。
風の歌を聴け (2010-10-30 (土) 10:23:53)
いつも参考にさせていただいております。
splancsのK関数を求めるkhat関数について質問させていただきます。
ランダム分布のsimulation envelopeの上下限を求めるシミュレーション回数(nsim)はどのように決めるのでしょうか?19回や99回などが多く使われているようですが、回数はどのように決めるのでしょうか?また、回数が多いほうが上下限の幅が狭くなるのでしょうか?谷村晋さんの新刊「空間地理データ分析」(共立出版)を拝見しましたがnsim=99の例がのっていました。
よろしくお願いいたします。
ST (2010-10-29 (金) 23:46:16)
クラスター分析での項目名についてです。
Rコマンダーを使用してデータを読み込んでいます。
クラスター分析を行い、樹形図を表示させた場合、各項目が項目名にならず、1からの連番になってしまいます。(項目名がAA,BB,CCでも1,2,3になるということです。)
どのようにすれば項目名が表示されるようになるのでしょうか?
よろしくお願いします。
TKです (2010-09-16 (木) 17:54:37)
リッジトレースをRで実行したいのですが、どのようにすればいいですか?
プログラムを作るしかないですか?
リッジ回帰のコマンドは一応あるのですが、実行してはみたものの、あまり使い道がないと思うのですが。
atsuo (2010-07-26 (月) 18:01:11)
�FEARのdeaでは、CCRとBCCとNIRSの結果は出ます。さらに、NDRSの結果を出すことは可能でしょうか?
�deaの分析を行うためにFEARのdeaを改良したいと考えた場合、元のソースプログラムを手に入れる方法はありますか。(もしかしたら、基本的な質問かもしれません。申し訳ありません。Rを最近始めたばかりです。なんとかヒントをいただけませんでしょうか。よろしくお願いします。)
kazuaki (2010-06-03 (木) 01:11:15)
目的関数 : x[1]^2+x[2]^2+...+x[n]^2
制約条件 : Ax=b , x>=c
xとcはn次元ベクトル,bはm次元ベクトル,行列Aはn*m行列。
xが決定変数。
上記の最適化問題(最小化問題)を解きたいのですが、適切な手法やパッケージが特定できずにおります。
shigeru (2010-04-09 (金) 06:35:07)
2元配置分散分析を行う前の等分散性の検定についてお伺いします。たとえば、要因A(2水準)と要因B(3水準)で繰り返しのある2元配置実験データが
y A B
y1 A1 B1
y2 A1 B2
y3 A1 B3
・・・
のような形で得られているときに、
bartlett.test(y ~ A * B)
とすると、自由度はdf=1となり、また
bartlett.test(y ~ B * A)
とすると、df=2となり、最初に出てくる要因しか評価されていないようです。AとBの組み合わせ要因(この場合はdf=2×3-1=5)に対して等分散性の検定をするにはどうすればよいのでしょうか。AとBの組み合わせからCという要因を新たにつくって対処することもできるでしょうが、その場合でも少し面倒になるような気がします。簡単に対処する方法はないのでしょうか。ご教示頂ければ大変助かります。
> set.seed(20100409)
> d <- data.frame(y=rnorm(24),
+ A=gl(2, 12, labels=c("A1", "A2")),
+ B=gl(3, 4, 12, labels=c("B1", "B2", "B3")))
> d$temp <- as.integer(d$A)*10+as.integer(d$B) # 策を弄する
> d$C <- factor(d$temp, # 新たに factor 変数を作る
+ labels=c("A1B1", "A1B2", "A1B3", "A2B1", "A2B2", "A2B3"))
> d
y A B temp C
1 0.807748961 A1 B1 11 A1B1
2 0.513417877 A1 B1 11 A1B1
3 -0.156009578 A1 B1 11 A1B1
4 -0.192902834 A1 B1 11 A1B1
5 0.043651547 A1 B2 12 A1B2
6 -1.530954060 A1 B2 12 A1B2
7 -1.984433103 A1 B2 12 A1B2
8 0.416585172 A1 B2 12 A1B2
9 -0.001342267 A1 B3 13 A1B3
10 0.364838567 A1 B3 13 A1B3
11 1.182524866 A1 B3 13 A1B3
12 -1.087637431 A1 B3 13 A1B3
13 -1.552197067 A2 B1 21 A2B1
14 0.323540547 A2 B1 21 A2B1
15 -0.635204370 A2 B1 21 A2B1
16 -2.889526450 A2 B1 21 A2B1
17 0.223709445 A2 B2 22 A2B2
18 0.118526662 A2 B2 22 A2B2
19 0.172598117 A2 B2 22 A2B2
20 1.846951151 A2 B2 22 A2B2
21 0.673207028 A2 B3 23 A2B3
22 1.080020322 A2 B3 23 A2B3
23 1.383334771 A2 B3 23 A2B3
24 -0.260967601 A2 B3 23 A2B3
> bartlett.test(y~C, d)
Bartlett test of homogeneity of variances
data: y by C
Bartlett's K-squared = 3.125, df = 5, p-value = 0.6807
今の例では,以下の方でもよい
> set.seed(20100409)
> d <- data.frame(y=rnorm(24),
+ A=gl(2, 12, labels=c("A1", "A2")),
+ B=gl(3, 4, 12, labels=c("B1", "B2", "B3")))
> d$C <- factor(paste(d$A, d$B, sep=""), # 今の例ではこの方がよいかな?
+ labels=c("A1B1", "A1B2", "A1B3", "A2B1", "A2B2", "A2B3"))
> d
y A B C
1 0.807748961 A1 B1 A1B1
2 0.513417877 A1 B1 A1B1
途中省略
23 1.383334771 A2 B3 A2B3
24 -0.260967601 A2 B3 A2B3
> bartlett.test(y~C, d)
Bartlett test of homogeneity of variances
data: y by C
Bartlett's K-squared = 3.125, df = 5, p-value = 0.6807ohgi (2010-03-20 (土) 10:46:20)
仰木と申します.
Rで時系列グラフをplot()を使って描画していますが,実験最中に取れたデータ(約数十万行分)を描画して,必要そうな部分,注目したい部分を拡大して描画したいとき,Scilabではマウス右クリックでエリア指定の拡大ができるのですが,Scilabではあいにく今回の実験データが最大メモリ許容量を超えてしまうため,Rで同じことができないかと思っています.Rでデータの読み込みと全体描画はできるのですが,グラフ画面上で拡大したい部分をマウス操作で拡大するようなことはできないでしょうか?
もちろん,描画する範囲をスクリプトで描けば再描画することはわかっていますが,次々にその時刻を計算して拡大し,閲覧する作業が必要なので出来ればグラフ画面上で出来ないか,と思っています.
「できない」という答えでもよいので教えていただけませんでしょうか.
あんどう (2010-03-14 (日) 20:57:00)
Help にある .rds, .rdb, .rdx はどのように開くのですか。
boston (2010-03-08 (月) 05:47:02)
test.R はcat("Hello World")だが、test.Routに出力されない
R --vanilla < test.R > test.Routは出力される BATCHの書き方に問題がありますか
初心者 (2010-02-15 (月) 02:52:47)
パッケージSOMの、関数somの学習回数は何回なんでしょうか?
あんどう (2010-02-07 (日) 12:28:45)
lmの処理を調べようとtest_lma.Rを作成し実行すと、match.call()で以下にエラー match.call() : 'match.call' がその中から呼び出されたクロージャを見付けることができません。となります。 プログラムの冒頭は以下のとおりで
formula <- y ~ 1 + x xx <- c(1.386294, 2.079442 ,2.772589, 3.465736) yy <- c(0.06254298 ,0.71444544, 1.27103337, 1.28339677) ww <- c( 1.0000000 ,0.5000000, 0.3333333, 0.2500000) data <- data.frame(x = xx, y = yy, w = ww) subset <- 0 weights <- ww na.action <- 0 method <- "qr" model <- TRUE x <- FALSE y <- FALSE qr <- TRUE singular.ok <- TRUE contrasts <- NULL offset <- 0 ret.x <- x ret.y <- y cl <- match.call() mf <- match.call(expand.dots = FALSE)これ以降はlmのままです。
目的は余分な処理を削除したいのです。
> cl <- match.call() エラー: match.call() was called from outside a function > mf <- match.call(expand.dots = FALSE) エラー: match.call() was called from outside a functionエラーメッセージの言わんとしていることは,「関数の外で match.call なんかやってもだめだよ〜」ということかな?
あんどう (2010-02-06 (土) 08:50:29)
libraryに関数を追加する方法を教えてください。
新たな関数を作り既存の関数をリンクさせたいのですが。
.Rをつくり cl <- match.call()を組み込んだのですがエラーになります。新たな関数をlibraryに追加できればエラーがなくなると考えています。
こうや (2010-01-18 (月) 11:13:26)
それぞれの説明変数に対する回帰係数が応答変数のカテゴリーごとに出ると思うんですが、それらの係数がどのカテゴリーと対応しているかがよく分かりません。
教えていただけないでしょうか。
吉川 (2010-01-14 (木) 17:37:24)
Rで極値統計をやっております。
extRemes, ismev を使ってGEVの推定を行っています。
gev.fit(x)を使って推定を行うと、
グンベル分布・ワイブル分布・フレシェ分布の中から推定されます。
これを、グンベル分布のみで推定したい場合にはどのようにしたらよいでしょうか。
ご存知の方がいらっしゃいましたら、よろしくお願いいたします!
genri (2010-01-03 (日) 13:51:31)
どう計算しているのか原理をお教えください。
この関数はdist構造のデータを使って計算します、たとえば距離マトリクスhogeにたいして
hclust( as.dist(hoge), method="ward")
で結果を返します。
Ward'sの方法では、クラスター間の距離はクラスターの統合によって増加した分散によって定義されています。そこで、距離マトリクスを算出した最初のデータがないと距離が算出できないのではないかと思うのです。
どうやって距離マトリクスからwardの距離を算出しているのでしょう?
a<-rnorm(10000, sd=2, mean=10) b<-rnorm(10000, sd=1, mean=0) var(a)+var(b) var(a-b)同じくらいの値が出てくるはず。
zhang (2009-12-07 (月) 10:25:06)
おはようございます。張@弘前大学です。R初心者で何か間違ったらご指摘ください。
今研究で,リンゴの形状推定を行っています。リンゴのお尻のくぼみを三次元計測して,2次元正規分布関数として推定しようと考えています。
R初心者で,色々調べたら,kde2d()やbked2d()でできるらしいが,しかし,最終的に出力してほしいのは中心値(μ1,μ2)と標準偏差(σ1,σ2)です。これでできるでしょうか?
ほかに何か方法がありませんか?宜しくお願いします。
Shigeru (2009-11-21 (土) 17:17:18)
等分散性の検定にコクランのC検定を使った文献をよくみかけますが、どのような方法なのでしょうか。またRでは実行可能でしょうか。お教えいただけるとありがたいです。
Cochran's C test A test, introduced by Cochran in a 1941 paper, for equality of variance in m normal populations having unknown means and having sample variances denoted by . Let be the ...などという説明があったりしますけど,少なくと r-project.org のトピックには上がっていないようです。 -- 河童の屁 2009-11-21 (土) 20:54:36
b.b. (2009-11-04 (水) 10:50:45)
質問です。
スプライン平滑化を行ってグラフを得たのですが、
xがある値の時の平滑化されたyの値を出力するには
どうしたらいいのでしょうか?
koba (2009-09-03 (木) 12:51:05)
線形不等式制約付きの最適化関数constrOptimにてhessianでヘッセ行列を表示しようとしてもエラーが出てしまいます。
そもそも存在しないのでしょうか?
dtak (2009-08-13 (木) 07:06:37)
はじめまして、dtak と申します。
R (version 2.9.1 i386-apple-darwin8.11.1) でのMap関数(mapplyのラッパー関数)の振る舞いについて少し気になることがありましたので、投稿いたします。
Mapを用いて関数のリストを作ろうとしたのですが、Mapの引数fの内部で作られるクロージャでのみ評価されるfの引数が、最後の要素の値で上書きされてしまいました(なんだかややこしいですね、以下の単純化したコードをみてください)。> Map (function (x) x(), Map (function (y) {function () y}, c(1, 2, 3))) ## list(1, 2, 3) が返ることを期待 [[1]] [1] 3 [[2]] [1] 3 [[3]] [1] 3 >あらかじめ評価してしまうことで回避することはできるのですが、
> Map (function (x) x(), Map (function (y) {y;function () y}, c(1, 2, 3))) [[1]] [1] 1 [[2]] [1] 2 [[3]] [1] 3 >(遅延評価が行われるのであっても)少し奇妙な印象を受けました。これはこのような挙動をするものと考えるべきなのでしょうか、それとも不具合と考えるべきなのでしょうか?
ご存知の方がおられましたら、よろしくお願いいたします。
fall (2009-07-07 (火) 13:28:37)
パッケージMASSのplorを用いて順序ロジスティックを行おうと考えております。
ここで私が困っていることが以下のような内容であります。
(目的変数にhelpでは”factor”を指定しなければならないと書かれており、数値を目的変数に入れるとエラーになってしまいます。目的変数を1,2,3など順序のある数値として解析をおこなうためのパッケージやplorの設定方法があるのであれば、教えて頂きたいと思います。)
どうぞよろしくお願いいたします。
かわお (2009-06-23 (火) 23:14:25)
非線形の連立方程式を解くプログラミングを教えて下さい。
例えば、
x^2+y^2=5, 2×x^2+y=4の解x,yを解く方法が知りたいです。
よろしくお願いします。
たけぼう (2009-06-14 (日) 01:53:04)
R超初心者です。
ある半導体関連装置のサポートエンジニアをやっております。担当している装置はPC上で実行されるプログラムに従って動作するようなシステムです。
担当している装置が出力する日付、時間を含んだエラーログを見る機会が多く、その解析にRを利用できないかと考え入門しました。 担当している装置はログファイルの出力が「コマンド実行履歴」と「エラーログ」に分かれて記録されているので、「エラーが発生したとき、その装置は何をしていたか?」を調べるのに毎回エクセルで悪戦苦闘していましたが、この部分をRで一発で視覚的に表示できないかな?、と考えています。ログのフォーマットは次の通りです(csv)。
[エラーログ]
YYYY/MM/DD, HH:MM:SS, ALARM1
YYYY/MM/DD, HH:MM:SS, ALARM2:[コマンド実行履歴]
YYYY/MM/DD, HH:MM:SS, COMMAND1
YYYY/MM/DD, HH:MM:SS, COMMAND2:「上記二つの時系列ファイルから、両者の因果関係を視覚的に表示したい」
というのがやりたいことなのですが、Rでできそうでしょうか?
ヒント・アイディアだけでも良いので、何か閃いた方はご教示下さいませ。
路地裏の少年 (2009-05-25 (月) 22:57:16)
低水準作図の繰り返しコマンドについて教えてください。
データは以下のようになっています。
緯度 経度 年
35 156 2000
38 165 2000
37 150 2001
38 148 2001・・・・
年毎の緯度経度のプロット図(20年分、各図の題名にはその年を充てたいと思っています)を書かせたいと思っています。
年ごとに抽出すれば、書けるのですが、20回繰り返せねばなりません。
初歩的な質問で恐縮ですが、よろしくお願いします。
df<-data.frame(lat=c(35,38,37,38),
long=c(156,165,150,148),
year=c(2000,2000,2001,2001)
)
cat(deparse(df),fill=T)
library (maps);
par(ask=T)
lapply(unique(df$year),function(y)
{
cat(y,fill=T)
longlat<-df[df$year==y,c("long","lat")]
plot (longlat,
xlim = c(135, 155),
ylim = c(32, 50),
axes = F, main = y, pch = 20)
map ("world2", xlim = c(135, 155), ylim = c(32, 50), add = T)
map.axes()
dev.copy(pdf, file = paste(y,".pdf",sep=""))
dev.off()
}
)
cat(deparse(df),fill=T)とかすると, 編集した小さなデータの切り出しが簡単でしょ?ま (2009-04-16 (木) 19:13:38)
非線形最小二乗法を実施する関数として,nlsがありますが,このポアソン分布版はありませんでしょうか?教えていただければ幸いです。
x <- c(1:10) y <- c(10,10,9,8,5,3,2,1,0,0) plot(y~x)
K.K (2009-01-27 (火) 16:15:38)
クラスター分析で、plotされた樹形図の細かい統合部分をsplancsのzoomで見ようとしましたが、樹形図が表示されないまま拡大されてしまいます。
plotされた樹形図をzoomで細かく表示させる方法はありませんか?
y.t (2009-01-15 (木) 18:10:28)
MASSパッケージにある順序ロジットの関数 polr() に 定数項を入れたいのですがどうすればよいでしょうか?
yuki (2008-12-16 (火) 01:49:28)
WinBUGSではDICは簡単に計算できるコマンドがあるようですが,BRugsでもそのようなコマンドはあるのでしょうか?
大津起夫 (2008-12-05 (金) 17:33:15)
R-2.8.0 を CentOS5.2 x64上でコンパイルし、make check したところ, tests/Examples/methods-Ex.Rの実行でエラーが生じます。 173行目あたりの setMehods("f","B0", function(x) c(x@b0^2, callNextMethod()))) 実行時にsprintf実行例外が発生してしまいます。これは他の版などで生じていないのでしょうか? また、すでに知られたバグでしょうか?
エラーログ> f <- function(x) class(x) > > setMethod("f", "B0", function(x) c(x@b0^2, callNextMethod())) 以下にエラー sprintf(gettext(fmt, domain = domain), ...) : 引数が少なすぎます Calls: setMethod -> message -> gettextf -> sprintf 実行が停止されましたOSは gcc 4.1.2環境ですが、コンパイルはIntel C++ 11.0 +Intel ifort 11.0+MKL 10.0でおこなっています。
下記が、configure.siteの指定、および ./configure の引数です(インストールマニュアルにある B.Ripley先生となかまさんの指定にしたがっていますが、config.siteのパラメータは -mpを-ieee-fpにしました)。
上記の命令は2回目以降はエラーを起こしません。上記の箇所の直前に try(setMethods(....)) )のように同様の命令を挿入すると、テストをパスします。
また,CentOS5.2のパッケージ compat-libstdc++33-3.2.3-61 から/usr/lib64/libstdc++.so.5をインストールしています。
--- config.site ---- CFLAFGS="-g -O2 -wd188 -ip -std=c99" F77=ifort FFLAGS="-ieee-fp -g -O3" CXX=icpc CXXFLAGS="-g -O3 -ieee-fp" FC=ifort FCFLAGS="-g -O3 -ieee-fp" ICC_LIBS=/opt/intel/Compiler/11.0/074/lib/intel64 IFC_LIBS=/opt/intel/Compiler/11.0/074/lib/intel64 LDFLAGS="-L$ICC_LIBS -L$IFC_LIBS -L/usr/local/lib64" SHLIB_CXXLD=icpc ----- configure 実行引数 ----- OMP_NUM_THREADS=8 MKL_LIB_PATH=/opt/intel/Compiler/11.0/074/mkl/lib/em64t MKL=" -L${MKL_LIB_PATH} \ -Wl,--start-group \ ${MKL_LIB_PATH}/libmkl_intel_lp64.a \ ${MKL_LIB_PATH}/libmkl_intel_thread.a \ ${MKL_LIB_PATH}/libmkl_core.a \ -Wl,--end-group \ -liomp5 -lguide -lpthread -lgomp" ./configure --with-lapack="$MKL" --with-blas="$MKL"リストの終わり
patch < methods.patchのパス名とする。R-ja.poが修正される。
(cd po; make remove-potcdate.sed; make en@quot.insert-header) (cd po; make methods.pkg-update)8) make check を 実行すると問題なくとおる。
Rcommander利用者 (2008-09-02 (火) 10:45:00)
線形モデルでの変量効果(ランダム効果)・混合効果の解析方法を教えてください。変数の指定方法などわかりません。よろしくおねがいします。
むかしちゃん (2008-06-25 (水) 22:20:15)
統計用語にJACKNIFE という物騒な用語をインターネットで見つけましたが
どんなところでどんな風に使うのですか?
気になった人 (2008-05-20 (火) 09:32:44)
Rの魅力の一つは、各関数が比較的詳しい参考実行コードを持っており、関数の使い方がわかることです。もう一つ豊富な組み込みデータセットがあり、関数参考実行コードはしばしばそれを用いることで、単に参考のための参考に止まらない迫真性を与えることができます。
ところで逆ができるでしょうか。つまり、ある組み込みデータセット(例えば co2)を例示用コードで参照している関数を一覧することです。
例えば Linux なら端末から次のような命令を実行すればそれらしいことができますが、Rには元々そうした機能は無い?
$ grep -r "co2" /usr/lib/R/* | grep "/help"
NP (2008-04-22 (火) 14:27:46)
Rを用いてCumulative incidence curveを書くところまではできたのですが、tick marksを打ち切りとなったものの観察期間を示すためにつけろといわれました。どのようにすればできるでしょうか。教えてください。よろしくお願いいたします。
for (j in 1:nrow(x$est)) {
temp <- as.integer(unlist(strsplit(rownames(x$est)[j], " ")))
temp <- ftime[fstatus == 0 & group == temp[1]]
temp2 <- timepoints(fit, temp)
mapply(function(x, y) segments(x, y, x, y+0.02, col=col[j]),
sort(temp), temp2$est[j,])
}#ref(): File not found: "fig.png" at page "Q&A (初級者コース)/13"
legend("topleft", legend = rownames(x$est), x.intersp = 2,
bty = "n", xjust = 1, col = col, lty = lty, lwd = lwd)
for (j in 1:nrow(x$est)) {
temp <- as.integer(unlist(strsplit(rownames(x$est)[j], " ")))
temp <- ftime[fstatus == 0 & group == temp[1]]
temp2 <- timepoints(fit, temp)
mapply(function(x, y) segments(x, y, x, y+0.02, col=col[j]),
sort(temp), temp2$est[j,])
}
out <- list(test = tests, est = tfit$est, se = sqrt(tfit$var))> ( x <- read.csv("bmt.csv", header=TRUE, sep=";") )
dis ftime status
1 0 13 2
2 0 1 1
3 0 72 0
4 0 7 2
途中省略
34 1 32 0
35 0 12 1
で,プログラムはちゃんと挿入できていますね。> CumIncidence(x$ftime, x$status, x$dis)
+-------------------------------------------------------------------+
| Cumulative incidence function estimates from competing risks data |
+-------------------------------------------------------------------+
Test equality across groups:
Statistic p-value df
1 1.302 0.253915 1
2 7.082 0.007785 1
Estimates at time points:
0 10 20 30 40 50 60 70
0 1 0.05882 0.1176 0.1765 0.1765 0.1765 0.1765 0.1765 0.1765
途中省略
0 2 0.05882 0.12631 0.12667 0.12077 0.12077 0.12077 0.12077 0.12077
1 2 0.00000 0.11558 0.11558 0.11558 0.11558 0.11558 0.11558 0.11558
以上で終了し,tick mark のついている図が描かれており,warning なんて出ないのですけど。
それと,今更なんですが,「グラフ自体はお示しいただいたのと同じものがかけるのですが」って,上に掲示した図から tick mark を除いたものということでいいのですね?UO (2008-04-09 (水) 11:48:43)
皆さんに相談があります。
RでAutoregressive Conditional Durationモデルを使いたいのですが。
可能でしょうか?
ご存知の方がいらっしゃっいましたら、教えてください。
rowhelp (2008-04-07 (月) 10:57:23)
はじめまして、現在、Rを使用し遺伝子解析を始めたばかりです。
データフレーム内のある特定の列に含まれる文字列をrownameに変換したいと思います。
どのような作業を行うのが一番よろしいでしょうか。
ご教授をお願いいたします。
> DF <- data.frame(A=letters[1:4], B=runif(4), C=rnorm(4))
> DF
A B C
1 a 0.1429617 0.8767259
2 b 0.7656706 0.2077312
3 c 0.5311202 -0.6043531
4 d 0.9375921 1.0804659
> rownames(DF) <- DF[,1]
> DF
A B C
a a 0.1429617 0.8767259
b b 0.7656706 0.2077312
c c 0.5311202 -0.6043531
d d 0.9375921 1.0804659
> DF <- DF[,-1]
> DF
B C
a 0.1429617 0.8767259
b 0.7656706 0.2077312
c 0.5311202 -0.6043531
d 0.9375921 1.0804659> DF.new <- data.frame(DF, row.names="A")
> DF.new
B C
a 0.79124741 1.2432360
b 0.03667521 -1.0248516
c 0.78015687 0.1808611
d 0.30211288 0.2649450yuta (2008-04-03 (木) 16:43:46)
こんにちは。いつも参考にさせていただいています。
以下に示すような折れ線グラフと縦線プロット(plot()のtype="h")の混在したグラフがあります。このグラフに凡例を付けたいのですが、図にもあるように、legend()を普通に使うだけではラインはすべて横線になってしまいます。(plotのスクリプト省略) par(xpd=NA) legend(locator(1),c("a-pro","a-height","b-pro", "b-height"),col=c("black","black","Royalblue","Royalblue"), lwd=c(2,3,1,1.5),pch=c(1,NA,1,NA))凡例中で縦線を使う方法はあるのでしょうか。お尋ねいたします。
#ref(): File not found: "QA.png" at page "Q&A (初級者コース)/13"
legend(locator(1),c("a=pro","a-height","b-pro",
"b-height"),col=c("black","black","Royalblue","Royalblue"),
lty=c(1,0,1,0),lwd=c(2,0,1,0),pch=c(1,15,1,15))
#ref(): File not found: "QA2.png" at page "Q&A (初級者コース)/13"
たいち (2008-02-12 (火) 18:20:13)
こんにちは。こんなデータがあるとします(実際はもっと長く,同じschoolに属するデータの個数は一定ではありません)。school y x 1 1 4 1 2 1 3 2 3 1 2 3 4 5 4 2 5 5 5 3 6 5 3 4 7 8 4 3 8 8 4 3 9 8 1 2ここから,同じschoolに属する人のxとyだけをランダムに抽出したいのですが,どのようにすればいいのでしょうか?
よろしくお願いします。
> A <- data.frame(school=sample(1:3, 10, 1),x =sample(1:4,10,1), y=sample(10:14,10,1)) > A school x y 1 2 1 12 2 1 4 10 3 1 1 12 4 1 1 13 5 1 1 14 6 3 1 12 7 3 4 12 8 2 4 13 9 3 3 13 10 2 1 11 > A1 <- A[A$school==1,,] > A1 school x y 2 1 4 10 3 1 1 12 4 1 1 13 5 1 1 14 > A1[sample(seq(ncol(A1)),3),,] # school=1 から3ケースを非復元ランダム抽出 school x y 2 1 4 10 4 1 1 13 3 1 1 12 > A1[order(sample(seq(ncol(A1)),3)),,] # 行番号を整列させたければ school x y 2 1 4 10 3 1 1 12 4 1 1 13
使用歴だけは長い人 (2008-02-06 (水) 14:10:12)
ルート権限の無いシステムに個人的にパッケージをインストールしようとしています。ほとんどのパッケージは問題なく個人ディレクトリにインストールできるのですが、(これまた個人的にインストールした)外部ライブラリへのラッパーであるパッケージをインストールしようとすると、(当然でしょうが) /usr/local/lib を参照しようとしてエラーになります。こうしたとき必要なライブラリ位置をRに教えてあげるにはどうすればよいのでしょうか。ちなみに問題のパッケージは gsl でこれは GNU GSL ライブラリーへのラッパーです。OS は Linux, R 2.6.1 を使用しています。GSL ライブラリは個人ディレクトリにインストール済みです。管理者に GSL をインストールしてもらえば済むことでしょうが、同様の問題が今後も起きそうで、何とか個人的にインストール出きると便利なのですが。うまい方法があれば教えてください。パッケージのインストールにはいつも R CMD INSTALL xxx.tar.gz としています。
> install.packages("hoge",lib="~/R/library",
configure.args = "CPPFLAGS = -I~/include LDFLAGS = -L~/lib")
とすると(configure.argsの内容はあくまで例ですので、自分の指定したいオプションに書き換えてください) 、どうなりますか?どこにインストールしたかは、
> library()で分かります。~/R/libraryに入れたpackageを使うには、.RenvironにR_LIBS=~/R/libraryを足す必要があるかも。現在のパスは、
> .libPaths() [1] "~/R/library" "/usr/local/lib/R/library"これで確認できます。-- 谷村 2008-02-12 (火) 20:59:56
* Installing * source packages 'gsl'... checking for gcc... gcc checking for C compiler dafault output... configure: error C compiler cannot create executables
$ tar xzf gsl_1.8-8.tar.gz && cd $(tar tzf gsl_1.8-8.tar.gz |head -n1) $ pwd /tmp/gsl $ ./configure LDFLAGS="/opt/local/lib" CPPFLAGS="-I/opt/local/include" checking for gcc... gcc checking for C compiler default output... configure: error: C compiler cannot create executables See `config.log' for more details.正しいオプションは下記です。
$ ./configure LDFLAGS="-L/opt/local/lib" CPPFLAGS="-I/opt/local/include" checking for gcc... gcc checking for C compiler default output... a.out checking whether the C compiler works... yes [以下略]参考になりますでしょうか。
大学生K (2008-01-24 (木) 04:45:22)
Rでパワーアナリシスをできる検定方法は、
t-test,anova,propのほかにはないのでしょうか?
個人的にノンパラメトリックを含め、いろいろな検定手法に対する、パワーアナリシスを行う必要がありまして・・・ご教授ください。
田島 (2008-01-18 (金) 22:06:14)
はじめまして。田島と申します。突然のお尋ねで恐縮ですがよろしくお願いいたします。
WindowsXpにてR-2.6.1+MinGWにて下記のCコードをコンパイルしました。コンパイルに関するエラーは出ませんので、includeの問題はなさそうですが、seq[i]という入力ベクトルにどんな値が入っていても返り値であるsfというベクトルのすべての要素に0が返ります。ためしに、すべての返り値にsin(1.0)という固定値を代入するようにしても0が返るはずはないのですが、すべて0と返ります。私だけの環境で生じている可能性もあろうかと思いますので、初心者Q&Aがふさわしいかもしれませんが、なぜか書き込みができませんでしたので、こちらに投稿させていただいております。
その他の四則演算をするコードでは、期待した結果が返ります。
あまりコンパイルするような作業の経験はないのですが、なんかのパスが悪いのかと思いますが、お恥ずかしいことにかなり検索などもしましたがどこに問題があるか見当がつかない状況です。三角関数を使いたいだけということですので、四則演算で計算するアルゴリズムをつかって計算すればいいのですが、本来できることができていないのであれば、非常に気持ち悪いことですし、今後のために理解を深めたいということもありまして、お尋ねしているしだいです。
もし、ほかにお尋ねすべき適切なセッションがあれば、ご指導いただければ幸いです。どうぞよろしくお願いいたします。
test1.c#include <math.h> void test1(double *seq, int *seqn, double *sf) { int i; for(i=0;i<=*seqn-1;i++){ sf[i] = sin(seq[i]); } }やったこと
Rcmd SHLIB test1.cR上でやったこと
dyn.load("test1.dll") a<-seq(0,pi,length=10) .C("test1",as.double(a),length(a),sf=double(length(a)))$sf
こはく (2008-01-12 (土) 22:14:03)
繰り返しのない2元配置(乱塊法または反復測定一元配置(対応のある一元配置?))で分析できるデータがあります。
これをボンフェローニの補正をおこなって多重比較するために、たとえば
a <- rep(1:3, each=4)
b <- rep(1:4, 3)
x <- c(7, 8, 5, 8, 1, 14, 16, 11, 7, 7, 6, 8)
pairwise.t.test(x,a,p.adjust.method="bonferroni")
pairwise.t.test(x,b,p.adjust.method="bonferroni")
ってかんがえたのですが、Rをはじめたばかりでよく分かりません。
ご意見いただけるとうれしいです。
atte (2007-12-01 (土) 11:05:31)
scatterplot3dのグラフを回転させたいのですが、angleではxy方向しか回転できません。xz、yz方向に回転する方法はないのでしょうか。
PC1 PC2 PC3 a -0.2138188 -0.09690295 -0.056538208 b -0.1983966 -0.06068939 0.108130244 c -0.3581248 -0.04182101 0.157024270 d -0.2755906 -0.07243737 0.004878913 e -0.4614072 -0.02512825 0.006534084 f -0.3580950 -0.11927931 0.036777825 g 0.2977400 0.60951743 0.180188932 h 0.3043281 0.62594929 0.290022187 i 0.1160385 -0.55632764 0.073677929 j 0.7941420 -0.39650995 0.289428417 k 0.4994019 -0.38653550 -0.234907993 l 0.3296465 0.32195986 -0.691875477 m -0.2982350 -0.06277819 -0.159138712 n -0.1776291 0.26098297 -0.004202413で、次のようにrglを試してみました。
pcalabel<-row.names(pca3) #各プロットにつけたいラベルの名前。
rgl.bg(color=c("white","black"))
rgl.lines(c(-1,1),0,0,color="black")
rgl.lines(0,c(-1,1),0,color="black")
rgl.lines(0,0,c(-1,1),color="black")
rgl.texts(c(1,0,0),c(0,1,0),c(0,0,1),
text=c("PC1","PC2","PC3"),color="blue")
rgl.points(pca3,color=heat.colors(1000),size=2)
rgl.texts(pca3[,1],pca3[,2],pca3[,3],pcalabel,adj = 1) #ここでラベル付けにトライ
どこかplotのtype="h"をrgl.pointsのところに入れたらエラーが出たので、省きました。rp.plot3d {rpanel} R Documentation
Interactive display of a plot of three variables
Description
This function produces a scatterplot of three variables, using the rgl package
for three-dimensional display.
Usage
rp.plot3d(x, y, z, xlab = NA, ylab = NA, zlab = NA,
axes = TRUE, type = "p", size = 3, col = "red",
xlim = NA, ylim = NA, zlim = NA, ...)
Arguments
x,y,z vectors of observed values.
xlab a character variable used for the first axis label.
ylab a character variable used for the second axis label.
zlab a character variable used for the third axis label.
axes a logical variable determining whether the axes are shown.
type a character variable controlling the type of plotting.
If the value is set to "n", the points are not plotted.
size the size of the plotted points.
col the colour of the plotted points.
xlim the plotting range for the first variable.
ylim the plotting range for the second variable.
zlim the plotting range for the third variable.
... other rgl parameters which control the appearance of the plotted points.
Details
The plot is produced by appropriate calls to the rgl package. This allows
interactive control of the viewing position. Other objects may subsequently be
added to the plot by using rgl functions and data which are centred and scaled
by the returned values indicated below.library(rgl)
open3d()
pcalabel<-row.names(pca3) #各プロットにつけたいラベルの名前。
rgl.bg(color=c("white","black"))
rgl.lines(c(-1,1),0,0,color="black")
rgl.lines(0,c(-1,1),0,color="black")
rgl.lines(0,0,c(-1,1),color="black")
rgl.texts(c(1,0,0),c(0,1,0),c(0,0,1),
text=c("PC1","PC2","PC3"),color="blue")
rgl.points(pca3,color=heat.colors(1000),size=2)
text3d(pca3[,1],pca3[,2],pca3[,3],text=pcalabel,adj =1) #text3d()を利用似非R使い (2007-11-30 (金) 16:50:43)
初めて投稿させていただきます。
Rを用いた統計の教科書の中で、display()とあるのですが、その通り書き込んでもエラーが出ます。display()は削除されたか、別の関数に置き換わったのでしょうか?
SAStoR (2007-11-19 (月) 16:23:14)
初めて投稿させていただきます.
barplot()を使って棒グラフを作成したいのですが,長いラベル(全角16文字程度)がついています.dotchartのようにラベルを横に表示させるにはどうすればよいでしょうか.棒グラフの場合,horiz=Tを指定してもラベルは垂直方向になってしまうため,全て表示がされず困っています.
”barplot(),text(),names,長いラベル”の組み合わせで検索してみましたが,うまく情報を見つけられませんでした.
どうぞよろしくお願いいたします.A1 <- c(9,8,19,8,2,23,2,10) A1label<-c("AAAAAAA","BBBBBBBBBBBBBBBBBBBBBBB","CCCCCCCCCCCCCC", "DDDDDDDDDDDD","EEEEEEEEEEEEEEEEEE","FFFFFFFFFFFFFFFFFF", "GGGGGGGGG","HHHH") barplot(A1,names=A1label,horiz=T)
Saito (2007-11-13 (火) 15:55:26)
皆様、的確なご指摘ありがとうございました。
それぞれのコメントにお答えする前に、まずマルチポストに関してお詫び申し上げなければなりません。その件に関しては非常に軽率であり、マナーに欠けた行為であったと反省しております。統計学関連なんでも掲示板に投稿したあとに自分の質問がその掲示板に不適切だと判断してこちらに投稿したのですが、もう少しインターネット上のルールを勉強してから投稿するべきでした。さらに言葉を重ねますと、統計学関連なんでもありの掲示板に書き込みをしたあと確認を怠り、こちらの掲示板で皆様の非難の声を聞いてから統計学なんでもありの掲示板にコメントがなされているのを知った次第です。皆様に不快な思いをさせてしまい申し訳ありませんでした。
また、中級者用掲示板にこのような初心者の質問をしてしまったこともお詫びいたします。私の全くの勘違いなのですが、昨日の時点では初級者用掲示板がスパム防止のため封鎖した、というようなことが書いてあったので。しかし今はそんな文はどこにも記載されておらず、完全に私の勘違いでした。皆様にご迷惑をかけてしまい、申し訳ありませんでした。お詫びの言葉にもなっていないかもしれませんが、なにとぞご容赦ください。
>簡単でいいですから疑問点が再現できるような例(もしくは組込みデータを使った例)をつけてください。
質問する側として当然の配慮が欠けていました。申し訳ありません。私が扱っているデータは以下のような感じです。すみません何度も試したのですが、うまく表示できませんでした。> x StandardLength Feed Area 1 100 10 N 2 100 11 N 3 100 12 N 4 100 13 N 5 100 14 N 6 100 15 N 7 100 16 N 8 100 17 N 9 100 18 N 10 100 19 N 11 100 20 N 12 100 10 N 13 150 21 W 14 150 22 W 15 150 23 W 16 150 24 W 17 150 25 W 18 150 26 W 19 150 27 W 20 150 28 W 21 150 29 W 22 200 31 S 23 200 32 S 24 200 33 S 25 200 34 S 26 200 35 S 27 200 36 S 28 200 37 S 29 200 38 S 30 200 39 S > result=lm(StandardLength~Feed+Area) #StandardLengthを独立変数、 FeedとAreaを従属変数として重回帰 > summary(result) Call: lm(formula = StandardLength ~ Feed + Area) Residuals: Min 1Q Median 3Q Max -4.422e-14 -1.798e-15 1.179e-15 4.160e-15 9.431e-15 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 1.000e+02 9.435e-15 1.060e+16 < 2e-16 *** Feed 2.209e-15 6.171e-16 3.579e+00 0.00139 ** AreaS 1.000e+02 1.332e-14 7.506e+15 < 2e-16 *** #問題2の該当箇所。 AreaW 5.000e+01 7.750e-15 6.452e+15 < 2e-16 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 9.815e-15 on 26 degrees of freedom Multiple R-Squared: 1, Adjusted R-squared: 1 F-statistic: 1.791e+32 on 3 and 26 DF, p-value: < 2.2e-16 > sd(Feed) #問題3の該当箇所。 今となっては恥ずかしいかぎりです。 [1] 9.123986 > sd(AreaS) 以下にエラーsd(AreaS) : オブジェクト "AreaS" は存在しません #問題1の該当箇所。 sd()が計算されないため、 前に投稿したやり方では標準化できませんでした。>result() とは? >summary()のことだと,思いますよ(^_^)
ご指摘のとおりです。Summary(result)の間違いでした。申し訳ありません。
>問題3はあなたの理解通りですが、なぜ両者が同じだと思ったのですか(そもそも sd() で何を計算したのかそこがわからぬ)
単純に標準誤差という言葉で表されるので同じ意味なのかな、と。
>稲垣宣生著「数理統計学改訂版」定理12.5(正規誤差仮定の下での回帰係数の分布)を見てください。
ご紹介ありがとうございます。また私の書いた問題3の中で、「その違いを述べている参考書は見当たりませんでした」とありますが、これは統計学的にではなく、Rの参考書の話です。誤解があったようで失礼致しました。
>sd() で,従属変数の標準偏差を調べたのでは?推測に過ぎませんが。
推測のとおりです。説明不足、配慮不足でした。申し訳ありません。
>問題1は,そもそも,変数の単位に明確な意味があるケースなのですから,標準偏回帰係数を算出する必要性が薄い気がします。
ご指摘のとおりです。カテゴリカルデータの傾きを知ったところで意味がないのは承知しているのですが、以前読んだ論文にカテゴリカルデータにも関わらず、GAM Coefficientというものを算出しているものがあったので…。現在本当にその意味(Coefficientを回帰係数と訳すこと)であっているのか読み直しているところです。
>他のソフトウェアでは,おそらく,カテゴリカルデータを0, 1で表現して,連続変量と同様の算出法をしていると思いますので,ご確認して,ご報告頂けると幸いです。
>問題2は,? contrasts を読むとわかるかと思います。
>問題3は,すでにご紹介された教科書を参照なさると解決するかと思います。
丁寧なご回答ありがとうございました。自分の勉強不足を実感しました。少し勉強してみます。稚拙なコメントしか返せず申し訳ありません。
>推測の必要はないので以下のように答えておきます。…
わかりやすい説明ありがとうございました。返す言葉もありません。もう一度勉強させてください。
この掲示板をマナーを守って利用している方々にご迷惑をおかけしたことを重ねてお詫び申し上げます。
StandardLength の予測値 = 100 + 0*Feed + 0*AreaN + 50*AreaW + 100*AreaSというもの。Feed にかかる係数は,実際は 2.209e-15,AreaNに掛かっている係数0は,ベースラインを示すので本当の0。
StandardLength の予測値バージョンII = 150 + 0*Feed - 50*AreaN + 0*AreaW + 50*AreaSそういうこと。-- 2007-11-13 (火) 17:13:48
Saito (2007-11-12 (月) 15:39:20)
初めて投稿させていただきます。
現在、統計ソフトRを使って卒業論文用のデータを解析しているのですが、重回帰をしているときに3つほど問題が出てきてしまいました。もしどなたか解決法をご存知でしたら、ご教授のほどよろしくお願いします。
問題1.カテゴリカル型データの標準化偏回帰係数が計算されない。
私は連続型とカテゴリカル型を同時に重回帰にかけているのですが、連続型のほうは
res <- lm(y~x1+x2+x3)
sdd <- c(0,sd(x1),sd(x2),sd(x3))
stb <- coef(res)*sdd/sd(y)
のような形でできたのですが、カテゴリカル型データはできません。しかし種々の参考書を見ると、カテゴリカル型データでも標準化偏回帰係数が出力されており、途方に暮れております。
問題2.カテゴリカル型データで一つの項目に複数の場合があるとき、うち一つが表示されない。
正確には表示されないことが問題なのではなくて、計算できないことが問題です。
例えば、
Yield=fertileのような関係を調べたいときに、カテゴリカル型データであるfertileの中にfertile1,fertile2,fertile3があったとします。これをそのまま重回帰にかけると、fertile2,fertile3の偏回帰係数やp値は表示されるのですが、fertile1は表示されません。Minitabの場合でしたら普通、fertile1の偏回帰係数はfertile1,fertile2,fertile3を足して、0になるように計算できる(らしい)のですが、Rはどうもそうではないような気がします。おそらく基準点のとり方が違うと思うのですが、どなたかRでの基準点をご存知でしたら教えていただけないでしょうか。
問題3.重回帰を行った際に、result()で出力される結果に含まれるStd.Errorと、sd()で出力される結果が違う。
おそらく前者が傾きに対する標準誤差の計算結果で、後者がデータそのものの標準誤差ではないか、という予測はしているのですが私の調べた限り、その違いを述べている参考書は見当たりませんでした。データがカテゴリカル型であった場合には、sd()では出力されないのに、Std.Errorでは出力されているので、傾きに対する標準誤差なのかな、と。せめて傾きに対する標準誤差の計算方法がわかれば検算できるのですが、それもわからなかったため質問させていただきました。
以上の3つです。もしどなたか解決法をご存知でしたら、ご教授のほどよろしくお願いします。
星野 (2007-11-01 (木) 14:42:36)
ヒストグラムのソースなのですが、# 2 群のヒストグラム hist2 <- function( x1, # 第一群のデータ x2, # 第二群のデータ brks=NULL, # 階級分割点 ...) # barplot に引き渡す任意の引数 { if (is.null(brks)) { # 階級分割点が与えられないときには,適切に設定 brks <- hist(c(x1, x2), right=FALSE, plot=FALSE)$breaks } c1 <- hist(x1, breaks=brks, right=FALSE, plot=FALSE)$counts # 度数1 c2 <- hist(x2, breaks=brks, right=FALSE, plot=FALSE)$counts # 度数2 barplot(rbind(c1, c2), beside=TRUE, space=c(0, 0.2), # 棒を並べて描く names.arg=brks[-length(c1)], # 横軸の目盛りラベル等 axisnames=TRUE, axis.lty=1, ...) }というソースを用いているのですが、このソースだとx1、x2が数値でないといけないというエラーがでてきてしまいます。また、Rに関係する本でヒストグラムについてみても同じように数値の例ばかりです...。これだと、1000個のデータをヒストグラムにする場合に入力が無理ではないでしょうか?データフレームからベクトルにして、ヒストグラムを描くような方法はないのでしょうか?よろしくお願いします。
data(USArrests) USArrests str(USArrests) x1<-USArrests$Murder x2<-USArrests$Rape str(x1) str(x2) hist2(x1,x2)
if (is.data.frame(x1)) x1 <- as.matrix(x1) if (is.data.frame(x2)) x2 <- as.matrix(x2)
x <- data.frame(matrix(1:12,ncol=3,dimnames=list(1:4,c("AAA","BBB","CCC"))))
x$"AAA"
x$AAA
x$AA
x$A
どれも同じ結果になるので、とっても幸せになれますね -- akira 2007-11-04 (日) 00:48:04> x <- data.frame(matrix(1:12,ncol=3,dimnames=list(1:4,c("AAA","BBB","CCC"))))
> x["AAA"]
AAA
1 1
2 2
3 3
4 4
> x[,"A"]
NULL> x[[1]] # 成分をベクトルとして取り出す「古風な」しかた [1] 1 2 3 4 > x[["AAA"]] [1] 1 2 3 4 > x[["A"]] # 二重鈎括弧演算子も補完機能を持つことも今回初めて知った [1] 1 2 3 4
R : Copyright 1997, Robert Gentleman and Ross Ihaka
Version 0.49 Beta (April 23, 1997)
R is free software and comes with ABSOLUTELY NO WARRANTY.
You are welcome to redistribute it under certain conditions.
Type "license()" for details.
[Previously saved workspace restored]
Error: workspace file corrupted -- no data loaded
Error in device("X11", as.character(c(display[1], paper)), as.double(c(width, : unable to start device X11
> x <- data.frame(matrix(1:12,ncol=3,dimnames=list(1:4,c("AAA","BBB","CCC"))))
> x$"AAA"
[1] 1 2 3 4
> x$AAA
[1] 1 2 3 4
> x$AA
[1] 1 2 3 4
> x$A
[1] 1 2 3 4青木.学生 (2007-10-31 (水) 14:17:21)
gstatでクリギングを行いたいのですが、予測グリッドデータの作成方法がわからなくて難航しています。
gstatマニュアルを読んで「RのGRASSパッケージにおいて国際横メルカトール・グリッドに変換した」ということはわかったのですが、具体的な方法がわからずにいます。
どなたかご存知の方がいらっしゃればご教示願えないでしょうか?
ちなみに現在The R BookについているR Ver1.9.0を使っています。
> xy <- expand.grid(1:100, 1:100) # これが予測位置グリッド座標
> names(xy) <- c("x", "y")
> g.dummy <- gstat(formula = z ~ 1, locations = ~x + y, dummy = TRUE,
beta = 0, model = vgm(1, "Exp", 15), nmax = 20)
> yy <- predict(g.dummy, newdata = xy, nsim = 4)緑茶 (2007-10-16 (火) 15:41:17)
RでARFIMAXも推定することはできるのでしょうか?
出来るのであれば、よかったらやり方を教えてください。
等々力渓谷 (2007-09-24 (月) 04:43:47)
CPUよりも浮動小数点計算が高速で並列処理に向いていると言われる、最近のGPU(Graphics Processing Unit)ですが*1、Rでもこれを非グラフィックの数値計算に利用するプロジェクトはあるのでしょうか?
堀内 (2007-09-02 (日) 11:46:25)
実験計画を作成するにあたり、検出力80%以上となるようにサンプル数を設定したいと考えておりますが、その際比較する2群のサンプル数を同数ではなく、1:9もしくは2:8程度としたいと考えております。
他のソフト(SAS、Power and precisionなど)ではこのような設定での算定ができるようですが、power.prop.testに関するマニュアル等を見る限りではやり方が見当たりません。
対応方法をご存知の方がいらっしゃればご教示願えないでしょうか?
(特別な引数設定の仕方がある!他の関数でできる!アドインを入れてこのようにコーディングすればできる!など)
片方のサンプルサイズNc,もう一方のサンプルサイズNt,その比r(=Nc/Nt)
Pc, Pt は属性を持つものの割合,Qc=1-Pc, Qt=1-Pt
P =(r Pc + Pt) / (r+1)
Zα=qnorm(α/2, lower.tail=FALSE)
Zβ=qnorm(β)
Nt ={Zα*sqrt((r+1) P Q)+Zβ*sqrt(r Pt Qt +Pc Qc)}^2 /(r(Pt-Pc)^2)
まちがいなく伝わったかどうか分からないので,浜島先生の本25-26ページを参照してください -- 2007-09-04 (火) 09:59:44> library(Hmisc)
> foo <- function(N) {
power <- bpower(p1=P1, p2=P2,
n1=N, n2=ceiling(Nratio*N), alpha=Alpha)
if (power >= Power) { # 要求パワーを越えた時点でストップ
cat(N, ceiling(Nratio*N), power, fill=TRUE); stop()}
}
> P1 <- 0.3; P2 <- 0.5; Nratio <- 0.2; Alpha=0.05; Power = 0.8
> sapply(100:10000, foo)
266 54 0.8003697
以下にエラーFUN(100:10000[[167L]], ...) : # これはループを中断したことによる注意で無視可能
> P1 <- 0.3; P2 <- 0.5; Nratio <- 0.3; Alpha=0.05; Power = 0.8
> sapply(100:10000, foo)
195 59 0.8002799
以下にエラーFUN(100:10000[[96L]], ...) :
> P1 <- 0.3; P2 <- 0.35; Nratio <- 0.3; Alpha=0.05; Power = 0.8
> sapply(100:10000, foo)
2954 887 0.8002141
以下にエラーFUN(100:10000[[2855L]], ...) :
> P1 <- 0.3; P2 <- 0.35; Nratio <- 1; Alpha=0.05; Power = 0.8
> sapply(100:10000, foo)
1377 1377 0.8002008
以下にエラーFUN(100:10000[[1278L]], ...) :power.prop.test2 <- function( p1=NULL, p2=NULL, n1=NULL, r=NULL, sig.level=0.05, power=NULL)
{
if (sum(sapply(list(p1, p2, n1, r, sig.level, power), is.null)) != 1) {
stop("p1, p2, n1, r, sig.level, power のどれか一つだけを NULL とする")
}
power.function <- quote(bpower(p1=p1, p2=p2, n1=n1, n2=ceiling(n1*r), alpha=sig.level))
if (is.null(power)) {
power <- eval(power.function)
}
else if (is.null(n1)) {
n1 <- uniroot(function(n1) eval(power.function)-power, c(1, 1e7))$root
}
else if (is.null(p1)) {
p1 <- uniroot(function(p1) eval(power.function)-power, c(1e-5, 1))$root
}
else if (is.null(p2)) {
p1 <- uniroot(function(p2) eval(power.function)-power, c(1e-5, 1))$root
}
else if (is.null(r)) {
r <- uniroot(function(r) eval(power.function)-power, c(1e-5, 1000))$root
}
else if (is.null(sig.level)) {
sig.level <- uniroot(function(sig.level) eval(power.function)-power, c(1e-5, 0.99999))$root
}
else {
stop("internal error")
}
METHOD <- "Power calculation of the tow-sample proportion test."
structure(list(p1=p1, p2=p2, n1=ceiling(n1), n2=ceiling(n1*r), r=r,
sig.level=sig.level,power=power, alternative="two.sided",
method=METHOD), class="power.htest")
}
> power.prop.test2(p1=P1, p2=P2, n1=NULL, r=0.2, power=0.8)
Power calculation of the tow-sample proportion test.
p1 = 0.3
p2 = 0.5
n1 = 266
n2 = 54
r = 0.2
sig.level = 0.05
power = 0.8
alternative = two.sided
> power.prop.test2(p1=P1, p2=P2, n1=NULL, r=1, power=0.8)
Power calculation of the tow-sample proportion test.
p1 = 0.3
p2 = 0.5
n1 = 93
n2 = 93
r = 1
sig.level = 0.05
power = 0.8
alternative = two.sided
> power.prop.test2(p1=P1, p2=P2, n1=266, r=NULL, power=0.8)
Power calculation of the tow-sample proportion test.
p1 = 0.3
p2 = 0.5
n1 = 266
n2 = 54
r = 0.1992972
sig.level = 0.05
power = 0.8
alternative = two.sided
> power.prop.test2(p1=P1, p2=P2, n1=266, r=0.2, power=NULL)
Power calculation of the tow-sample proportion test.
p1 = 0.3
p2 = 0.5
n1 = 266
n2 = 54
r = 0.2
sig.level = 0.05
power = 0.8003697
alternative = two.sided## 二項分布 B(n,p),B(m,p) に従う独立標本 N 個を有意水準 1-alpha で
## 両側検定したとき棄却される割合(標本検出力)を計算する関数
> foo <- function(N,n,p,m,q, alpha) {
X <- rbinom(N,n,p) # B(n.p) に従う標本N個
Y <- rbinom(N,m,q) # B(m,q) に従う標本N個
phat <- (X+Y)/(n+m) # 帰無仮説p=qの下での共通比率の推定値
S <- sqrt((1/n+1/m)*phat*(1-phat)) # 帰無仮説の下での(X/n-Y/m)の標準偏差推定値
z <- qnorm(1-alpha/2) # 両側検定棄却限界値
mean(abs(X/n - Y/m)/S > z) # N 回の検定で棄却される標本比率
}
> bar <- function(n) { # m=0.2*n のケース
x <- foo(2e6, n, p, ceiling(0.2*n), q, alpha)
cat(k, ceiling(0.2*n), x, fill=TRUE)
}
# 実行例
> p=0.3; q=0.5; alpha=0.05; beta=0.8
> set.seed(1); sapply(260:300, bar)
260 52 0.7882775
261 53 0.793533
262 53 0.795987
263 53 0.791743
264 53 0.7943355
265 53 0.797369
266 54 0.802263 # 標本検出力が初めて80%を越える標本数(bpower関数の場合,266 54 0.8003697,とほぼ一致)
267 54 0.799412
268 54 0.801248
269 54 0.8035085
.................Blueblink (2007-08-22 (水) 01:18:17)
スプラインを用いたセミパラメトリック回帰をRで行いたいと考えております。まず,mgcvライブラリのgam関数を用いることを試みました。
こちらやこちらの文献では,gam関数のsオブジェクトを,平滑化スプラインを表すために用いています。例えば次のスクリプトで,変量Ozoneを3つの平滑化スプライン関数の和で説明する,加法モデルを求めることができるものとしています。>library(mgcv); data(airquality) >airq.gam<-gam(Ozone~s(Solar.R)+s(Wind)+s(Temp),data=airquality) >summary(airq.gam)ところが,help(gam)で見られるヘルプによると,mgcvのgamにおけるノンパラメトリック項(smooth term)には,罰則付きの回帰スプライン(Penalized Regression Spline)もしくは自由度を固定した回帰スプラインを用いると書いてあります。私の理解では,平滑化スプラインは共変量の数と同数の節点を用いますが,罰則付き回帰スプラインは計算負荷低減のため節点の数を抑えるもので,両者は異なります。
私は罰則付きの回帰スプラインの理論が良くわからないので,できればスプラインとして平滑化スプラインを用いたいのですが,本当にmgcvライブラリのgam関数で平滑化スプラインを用いることができるのでしょうか?
ご存知の方がいらっしゃいましたら,ご教授頂ければ幸いです。
どうぞよろしくお願いいたします。
有吉 (2007-07-11 (水) 17:15:20)
1万×3千〜4千ぐらいのサイズの行列(各要素は非負の実数)の特異値分解をしたいと思っています。
WindowsXPでR2.4を使って、
options("object.size"=160e+006)
memory.limit( size = 3072)
と設定してsvd()したのですが、メモリが足りなくなってできませんでした。
それで、転置行列とかけ合わせて3千〜4千×3千〜4千の対称行列にして固有値分解することにしたら、eigen()はできました。でも、そこでメモリ(ヒープ?)が足りなくなって、その後は行列演算ができなくなりました。
そこでいったんquit()してRを起動しなおして、行列演算を1つして、quit()して、起動しなおして、...の繰り返しになって作業が進みません。
上記のようなサイズの行列の特異値分解や固有値分解をして、さらに行列演算を続けていくにはどうしたらいいか教えていただけないでしょうか?
# 64bitOSで、64bitモードでmakeしたRを使えば、それで解決?
岩田 (2007-07-08 (日) 18:33:35)
はじめまして。最近 R を使い始めた、岩田と申します。RjpWiki の皆様の努力のおかげで、R に関する情報が手軽に手にはいることを、感謝しています。
さて、qcc を使って X-Bar 管理図を作成したいと思っています。元となるデータは Excel で一つの列に納めて作成し、csv として書き出したものを、read.csv で R に読み込ませました。
X-Bar 管理図を作成するには、このデータをサブグループに分けなくてはなりませんが、qcc にそのような機能はありますか? それとも、事前に複数列にまたがるテーブルに整形しなくてはならなかったのですか? たとえばサブグループサイズが5の場合、R または Excel 上で簡単に複数列にまたがるテーブルに整形しなおす方法はありますか? 皆様のお知恵を拝借できればと思います。
17 5 8 26 15 26 15 26 5 0 26 ...これを、5つごとのサブグループに区切って、X-Bar 管理図を作るのがねらいです。グループの区切り方は、具体的には、
17 サブグループ1 5 サブグループ1 8 サブグループ1 26 サブグループ1 15 サブグループ1 26 サブグループ2 15 サブグループ2 26 サブグループ2 5 サブグループ2 0 サブグループ2 26 サブグループ3 ...とする予定です。 qcc に自動で区切る機能があれば、それが一番手っ取り早いと思いますが、もしない場合は、データを整形して R または qcc に渡してあげなくてはならないだろうと予想しています。事前にデータを整形する場合のテーブルは、
サブグループ1 17, 5, 8, 26, 15 サブグループ2 26, 15, 26, 5, 0 サブグループ3 26, ...としなくてはならないようです。 以上で、私がやりたいことが伝わりましたでしょうか? 引き続きアドバイスをよろしくお願いいたします。 -- 岩田 2007-07-08 (日) 20:31:50
> x
[1] 1 14 19 5 8 4 8 10 1 4 16 7 3 18 8 20 4 1 8 12 7 8 3 12 10
[26] 19 5 15 8 9
> xx <- matrix(x, 6, 5, byrow=TRUE)
> xx
[,1] [,2] [,3] [,4] [,5]
[1,] 1 14 19 5 8
[2,] 4 8 10 1 4
[3,] 16 7 3 18 8
[4,] 20 4 1 8 12
[5,] 7 8 3 12 10
[6,] 19 5 15 8 9
> as.data.frame(xx)
V1 V2 V3 V4 V5
1 1 14 19 5 8
2 4 8 10 1 4
3 16 7 3 18 8
4 20 4 1 8 12
5 7 8 3 12 10
6 19 5 15 8 9x <- rnorm(1:20)
y <- c(rep(1, 4), rep(2, 6), rep(3, 10))
z <- data.frame("dat"=x, "sub.g"=as.factor(y))
とか -- 2007-07-09 (月) 17:10:02Ovation (2007-06-12 (火) 11:11:51)
Rでwavelet変換をやっていますが、コンター図を描こうとしたら、> filled.contour(Time,Frequency,t(z),nlevel=50,color.palette=topo.colors) 以下にエラーdiff(x) : 既定引数の再帰的な参照ですというエラーが出ました。
原因が分かる方、ご教授いただけると幸いです。
kd (2007-06-12 (火) 10:17:56)
NAN値もcプログラムに渡す方法はあるでしょうか?
試しに下記のcプログラムを書いて実験を行いますと,.C() のところでエラーを出すようで,
NAN値を含むデータはcプログラムに渡らないようです.#include <R.h> void countnan(double *x, int *n, int *count) { int i; for (i=0,*count=0;i<*n;i++) if (ISNAN(x[i])) (*count)++; } > countnan <- function(x){ n=length(x); .C("countnan",arg1=as.double(x), arg2=numeric(n),arg3=numeric(1))$arg3 } > x<-c(1, NA , 3 , 4) > countnan(x) 以下にエラーcountnan(x) : 外部関数の呼び出し(引数 1) 中に NA/NaN/Inf があります
> countnan <- function(x){
.C("countnan",arg1=as.double(x),
arg2=length(x),arg3=integer(1),NAOK=TRUE)$arg3 }
> countnan(c(1,2,NA,4))
[1] 1
cソースは
#include <R.h>
void countnan(double *x, int *n, int *count)
{
int i;
for (i=0,*count=0;i<*n;i++)
if (ISNAN(x[i])) (*count)++;
(*count)--;
if (ISNAN(R_NaReal)) (*count)++;
}パックマン (2007-06-07 (木) 16:28:32)
R で Windows CSSを利用している例はあるのでしょうか?
とも (2007-06-06 (水) 16:49:44)
クラスタリング結果のデンドログラムを解析して、ノードごとの高さやリーフの情報を得る関数を書いています。
ノードやリーフの情報をベクトルとして取得する方法を教えてください。例えば、("leaf","leaf","node","node")のような形で返してほしいです。
dendrapplyという関数を参考にして書いたコードを示しました。
ノードやリーフの情報を再帰的に取得して表示することはできます。
dendrogramでRjpWikiやGoogleを検索してみましたが分かりませんでした。
引数の参照渡しの方法が分かるとできると思います。
dendrapplyはもとのデンドログラムのグラフ構造と同じ構造を返してくるので使えませんでした。DNapplyTest <- function(d) { if (!is.leaf(d)) { for (j in seq_along(d)) Recall(d[[j]]) cat("node:",attr(d,"height"),"?n") }else{ cat("leaf:",labels(d),"?n") } } > DNapplyTest(as.dendrogram(hclust(dist(USArrests)))) leaf:Florida leaf:North Carolina node:38.52791 ... node:87.32634 node:168.6114 node:293.6228
## 文字列が長すぎるので適宜改行しています
> dhc <- as.dendrogram(hclust(dist(USArrests)))
> x <- capture.output(dendrapply(dhc, function(n) {cat(labels(n),"?n"); cat(attr(n,"height"),"?n")}))
> xx <- x[1:198] # 手作業で末尾につく無意味な部分を削除
> xx[5:20]
[5] "Florida North Carolina "
[6] "38.52791 "
[7] "Florida "
[8] "0 "
[9] "North Carolina "
[10] "0 "
[11] "Delaware Alabama Louisiana Alaska Mississippi South Carolina Maryland
Arizona New Mexico California Illinois New York Michigan Nevada "
[12] "64.99362 "
[13] "Delaware Alabama Louisiana Alaska Mississippi South Carolina "
[14] "48.72515 " lines(c(apx[c1], hc$height[i], hc$height[i], apx[c2]),
c(apy[c1], apy[c1], apy[c2], apy[c2]))f<-function(X){
ret<-NULL
f<-function(x){
if(is.leaf(x)){
attr(x,"label")
}else{
r<-c(sapply(x,f),recursive=T)
ret<<-rbind(ret,list(leafs=r,size=attr(x,"members"),height=attr(x,"height")))
r
}
}
f(X)
ret
}
r<-f(as.dendrogram(hclust(dist(USArrests), "ave")))関数定義
dend2nodelist <- function(hc) {
GetElement <- function(i, j) {
if (i < 0) {
if (j < 0) return(c(hc$labels[-i], hc$labels[-j]))
else return(c(hc$labels[-i], GetElement(hc$merge[j,1], hc$merge[j,2])))
} else {
if (j < 0) return(c(GetElement(hc$merge[i,1], hc$merge[i,2]), hc$labels[-j]))
else return(c(GetElement(hc$merge[i,1], hc$merge[i,2]),
GetElement(hc$merge[j,1], hc$merge[j,2])))
}
}
n <- length(hc$height)
leafs <- NULL
size <- integer(n)
for (i in 1:n) {
labels <- GetElement(hc$merge[i,1], hc$merge[i,2])
size[i] <- length(labels)
leafs[i] <- list(labels)
}
return(cbind(leafs=leafs, size=size, height=hc$height))
}
実行例
> hc <- hclust(dist(USArrests[1:10,]))
> result <- dend2nodelist(hc)
> result
leafs size height
[1,] Character,2 2 16.80625
[2,] Character,2 2 23.19418
[3,] Character,2 2 25.09303
[4,] Character,3 3 36.73486
[5,] Character,3 3 45.18296
[6,] Character,3 3 60.98073
[7,] Character,6 6 77.19741
[8,] Character,9 9 148.7357
[9,] Character,10 10 226.303
> result[1,]
$leafs
[1] "Alabama" "Delaware"
$size
[1] 2
$height
[1] 16.80625
> result[5,]
$leafs
[1] "Alaska" "Alabama" "Delaware"
$size
[1] 3
$height
[1] 45.18296
> result[9,]
$leafs
[1] "Connecticut" "Florida" "Arizona" "California"
[5] "Arkansas" "Colorado" "Georgia" "Alaska"
[9] "Alabama" "Delaware"
$size
[1] 10
$height
[1] 226.303dend2nodelist <- function(hc) {
temp <- function(hc, j) GetElement(hc$merge[j,1], hc$merge[j,2])
temp2 <- function(hc, j) if (j < 0) hc$labels[-j] else temp(hc, j)
GetElement <- function(i, j) if (i < 0) c(hc$labels[-i], temp2(hc, j))
else c(temp(hc,i), temp2(hc, j))
leafs <- size <- integer(n <- length(hc$height))
for (i in 1:n) {size[i] <- length(labels <- temp(hc, i)); leafs[i] <- list(labels) }
return(cbind(leafs=leafs, size=size, height=hc$height))
}kd (2007-06-02 (土) 21:07:02)
wavelets パッケージを調べてみたのですが,「不等間隔な x_i とそれに対する y_i 」というデータ系列を与えて処理できるものが見当たらないようです. このようなパッケージはどこかにあるでしょうか?
たく (2007-04-25 (水) 11:33:54)
glmmML でモデルを作成した際、info = といった警告が出ることがあります。
モデルは作成されている用なのですが、このモデルは採用してもいいのでしょうか?
また、この際の AIC なども信頼できるのでしょうか?
どなたかご教授いただけますでしょうか?
info: From hessian inversion. Should be 0. If not, no variances
could be estimated. You could try fixing sigma at the
estimated value and rerun.
作成されたモデルやAICの信頼性をどのように解釈すればいいのかがわかりません。> library(glmmML)
> data(anorexia, package="MASS")
> id <- rep(1:nrow(anorexia))
> class <- c(rep(1,10), rep(0,62))
> glmmML(class ~ Prewt, data=anorexia, cluster=id, family=binomial)
info = 3
Call: glmmML(formula = class ~ Prewt, family = binomial, data = anorexia, cluster = id)
coef se(coef) z Pr(>|z|)
(Intercept) -12.124711 NA NA NA
Prewt -0.001919 NA NA NA
Standard deviation in mixing distribution: 48.71
Std. Error: NA
Residual deviance: 31.01 on 69 degrees of freedom AIC: 37.01
Warning message:
Hessian non-positive definite. No variance!
>
よろしくお願いいたします。-- たく 2007-04-25 (水) 16:06:18> glmmML(class ~ Prewt, data=anorexia, cluster=id, family=binomial,
fix.sigma=TRUE)
Call: glmmML(formula = class ~ Prewt, family = binomial, data = anorexia,
cluster = id, fix.sigma = TRUE)
coef se(coef) z Pr(>|z|)
(Intercept) -1.772713 5.59883 -0.31662 0.752
Prewt -0.001720 0.06783 -0.02536 0.980
Standard deviation in mixing distribution: 0
Std. Error: 0
Residual deviance: 57.99 on 69 degrees of freedom AIC: 61.99
あるいは
> glmmML(class ~ Prewt, data=anorexia, cluster=id, family=binomial,
start.sigma=1e-4)
Call: glmmML(formula = class ~ Prewt, family = binomial, data = anorexia,
cluster = id, start.sigma = 1e-04)
coef se(coef) z Pr(>|z|)
(Intercept) -1.686092 5.46507 -0.30852 0.758
Prewt -0.001680 0.06621 -0.02538 0.980
Standard deviation in mixing distribution: 0.0002467
Std. Error: 406.6
Residual deviance: 58.02 on 69 degrees of freedom AIC: 64.02
結局の所,
The optimization may not converge with the default value of start.sigma. In that case, try different start values for sigma. If still no convergence, consider the possibility to fix the value of sigma at several values and study the profile likelihood.
たつや (2007-01-25 (木) 16:34:54)
重回帰分析して出た結果が英語で意味が読み取れませんでした。簡単な例を挙げて重回帰分析を行いましたので解説をお願いします。
質問はまず、この2つの違いについて。F=lm(Z~X) G=lm(Z~X-1)つぎにsummaryで表示された内容がわかりません。できれば詳しい説明をお願いします。 自分で行う回帰分析、もしくはExcelの回帰分析は多少理解しているつもりなので、そっちと関連させて解説してもらえれば幸いです。
Z=c(1,2,3,4,5,6,7,8,9,10) X=c(1,3,4,2,5,6,7,8,10,10) F=lm(Z~X) G=lm(Z~X-1) summary(F) Call: lm(formula = Z ~ X) Residuals: Min 1Q Median 3Q Max 1.11283 -0.47400 0.09181 0.27600 1.80531 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 0.35841 0.59297 0.604 0.562 X 0.91814 0.09329 9.842 9.56e-06 *** Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 0.887 on 8 degrees of freedom Multiple R-Squared: 0.9237, Adjusted R-squared: 0.9142 F-statistic: 96.86 on 1 and 8 DF, p-value: 9.561e-06 summary(G) Call: lm(formula = Z ~ X - 1) Residuals: Min 1Q Median 3Q Max 0.9035 -0.5006 0.1770 0.2494 2.0644 Coefficients: Estimate Std. Error t value Pr(>|t|) X 0.96782 0.04255 22.75 2.91e-09 *** Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 0.8552 on 9 degrees of freedom Multiple R-Squared: 0.9829, Adjusted R-squared: 0.981 F-statistic: 517.5 on 1 and 9 DF, p-value: 2.909e-09
- Excelで実行して,内容を比較してみたら? -- 2007-01-25 (木) 16:48:51
- 何がわからないのか?というか何が知りたいのか? -- 2007-01-25 (木) 17:34:36
- 何でも聞けばよいというものではない。 -- 2007-01-25 (木) 17:35:28
- > 結果が英語で意味が読み取れません --- なのでしょうね。 -- 2007-01-25 (木) 17:38:19
- 「重回帰分析」でキーワード検索、既に幾つかあるR本を見る、それでもわからなければ、改めて聞く。 -- 2007-01-25 (木) 17:42:33
- Estimate Std. Error t value Pr(>|t|) の意味を教えてほしいです。 -- 2007-01-25 (木) 18:10:30
- あと、F=lm(Z~X) とG=lm(Z~X-1)の違いが意味がわからないからできない…。 -- 2007-01-25 (木) 18:12:25
- すみません。お願いします。 -- 2007-01-25 (木) 18:13:38
- いっこめは係数の推定値、標準誤差、t統計量、p値。二個目は切片項を含むか含まないか。excelの結果と比べれば判ると思うんだけど。 -- 2007-01-25 (木) 18:19:16
- 1つ目の方は比較すればわかりました。すみません。 -- たつや 2007-01-25 (木) 18:29:11
- 返答ありがとうございました。だいたい理解できました。あとは自分で考えます。 -- たつや 2007-01-25 (木) 18:32:45
佐藤 (2007-01-15 (月) 16:57:21)
> attach(learning) > learning.glm<-glm(def~lastprice+volume+size+rtn+next1rtn+lsize+epr1 +greps1+dy+bpr+wcr+rrl+rrs+cfpr+roe+spr+ebitda+igr +sgrowth+esr1+lev+cgrowth+vol+roa1,family=binomial) > summary(learning.glm) > detach(learning) > test<-read.csv("property200006.csv",header=TRUE) > attach(test)ある(learning)データから回帰分析して得られた回帰係数を用いて、違う(test)データを回帰分析しようと考えています。
現状は回帰係数を自分で入力しているのですが、自分で入力することなく回帰分析することはできないでしょうか。
よろしくお願いします。
GEO (2007-01-06 (土) 18:34:20)
はじめまして。
結果率(果実/花)を従属変数、各花形質を独立変数としてロジスティック重回帰を行おうとしております。この際に、関数step()を用いて、変数選択しようと試みたのですが、以下のようなエラーが出てうまくいきません。どのような原因でこのようなエラーが出るのか、また、このまま解析を進めても良いのか、教えていただけないでしょうか?> h0 <- read.delim("c:/2000R2.txt") > fs <- h0$fru/h0$flo > result <- glm(fs~h0$ftl+h0$ptl+h0$ptw+h0$sta+h0$pis+h0$blt+h0$wit, weights=h0$flo) > result2 <- step(result) Start: AIC= -2819.3 fs ~ h0$ftl + h0$ptl + h0$ptw + h0$sta + h0$pis + h0$blt + h0$wit Df Deviance AIC - h0$wit 1 22.05 -2821.19 - h0$ptw 1 22.05 -2821.16 - h0$sta 1 22.06 -2821.13 - h0$blt 1 22.12 -2820.58 - h0$ptl 1 22.14 -2820.44 <none> 22.04 -2819.30 - h0$pis 1 22.33 -2818.70 - h0$ftl 1 23.20 -2811.30 Error in step(result) : number of rows in use has changed: remove missing values?また、remove missing values? という問いかけが出てきますが、これに対しての答え方が良くわかりません。。どのようにすれば、欠損値を除いて、解析を先に進めることができるのでしょうか?
よろしくお願いいたします。
秋田県民A (2006-12-17 (日) 17:43:33)
最近時系列分析を行うためにRを使い始めた初心者です。
まず定常性を確認するためにadf.test()関数を用いているのですが、
?定数項あり、トレンドあり ?定数項あり、トレンドなし
?定数項なし、トレンドあり ?定数項なし、トレンドなし
などの各状況に応じた使い方が分かりません。そのままadf.test()を使った場合にはどの場合の検定になるのでしょうか?
どなたかお分かりになる方がいらっしゃいましたら、教えてください。
やまだ (2006-11-21 (火) 11:14:18)
初めて書き込ませていただきます。どうぞよろしくお願いします。
今までPowerBookG4でMac版を動かして使っていたのですが、今回先月発売になったCore2DuoのMacBook Proを新しく購入したのでインストールしてみたところ、起動時に以下のコメントが赤字で表示されるようになりました。「2006-11-21 10:48:31.215 R[261] CFLog (21): dyld returns 2 when trying to load /Users/(ユーザ名) /Library/ScriptingAdditions/YouHelper.osax/Contents/MacOS/YouHelper」このときインストールしていたのがバージョン2.3.1でして、CRANを調べたらユニバーサル版の最新版として2.4がありましたので、それを再度インストールしてみましたが状況は同様で毎回起動時に表示されます。一応試しに使ってみましたが、問題なく動いてはいるようなんですが。。。ちょっと気持ち悪いので。
もしこの状況に見覚えのある方等おられましたらコメントいただけるとありがたいです。 どうぞよろしくお願いします。
lostway (2006-11-12 (日) 01:58:50)
Rでwavelet解析やっている方いますでしょうか?
イシ (2006-11-10 (金) 15:33:30)
cgiスクリプトからRを起動し、グラフを作成したいのですが、Rのjpegデバイスを開く段階でエラーが出てしまいます。対処法ありますか?
R-Version 2.3.1#### エラーメッセージ #####
Error in X11(paste("jpeg::", quality, ":", filename, sep = ""), width, : unable to start device JPEG In addition: Warning message: unable to open connection to X11 display '' Execution halted
服部 (2006-11-02 (木) 14:06:43)
ある目的変数(比率)を説明する変数を特定するときに、glmを用いて解析をしたいと思っています。
その際説明変数に割合の変数含めたいのですが、どのようにしたらよいのでしょう?
現在以下のようにglm関数を使っています、訂正すべき場所があったらそこも指摘していただけると幸いです。> a<-adult/colonysize > yy<-glm(cbind(x,y)~a+b+c+d,binomial)
アール (2006-10-27 (金) 16:26:24)
R 2.4.0 からは "?.foo" のような無意味なエスケープシークエンスに warning を出すようになりました。それはそれでいいのですが,これを考えていてちょっとした疑問が出てきました。
"foo.bar.baz" のような正規表現で,最初のピリオドは文字通りピリオド(他の言語の正規表現では"?.")二番目のピリオドは何でもよい一文字を表す正規表現"."なんかは,書きようがないんじゃないかと。fixed 引数も,全てに適用されるものなので無力だしね。> sub("foo?.bar.baz", "match", c("foo.bar.baz", "foo.bar,baz", "foo,bar.baz", "foo,bar,baz")) [1] "match" "match" "match" "match" Warning messages: 1: '?.' is an unrecognized escape in a character string 2: '?.' is an unrecognized escape in a character string > sub("foo.bar.baz", "match", c("foo.bar.baz", "foo.bar,baz", "foo,bar.baz", "foo,bar,baz")) [1] "match" "match" "match" "match"
> sub("foo??.bar.baz", "match", c("foo.bar.baz", "foo.bar,baz", "foo,bar.baz", "foo,bar,baz"))
[1] "match" "match" "foo,bar.baz" "foo,bar,baz"青木繁伸 (2006-10-19 (木) 15:41:02)
お絵かきソフトでよくある「ペイント缶」の機能を果たす関数がないのかなあと。
曲線にこだわるわけではない(曲線だって,グラフに描くときには折れ線)ので,polygon関数でも良いわけだが,点を与えてポリゴンを描いて内側を塗りつぶすのではなくて,経過は問わず既にできている閉局面で囲まれている領域を塗りつぶす関数が欲しいということ。#ref(): File not found: "ex.png" at page "Q&A (初級者コース)/13"
しも (2006-10-02 (月) 21:40:16)
マニュアル等で調べても、解決法が見つかりませんでしたので質問します。
関数を作成し、計算をさせています。計算途中で、以下の警告が出て計算がストップします。以下にエラーodbcQuery(channel, query, rows_at_time) : Calloc がメモリー (263168 of 1) を割りあてられませんでした 追加情報: Warning message: Reached total allocation of 1015Mb: see help(memory.size)使用するメモリーサイズを"--max-mem-size=2G"を使用して拡大しても、計算が止まる状況です。
関数内では「rm()」を使用し、こまめにオブジェクトは削除しています。
Windows タスク マネージャのPF使用量を見るとrm()を使用していても使用量がどんどん積み重なり、ある一定値で計算が止まっり、計算が止まっても使用量は減りません。使用量を減らす唯一の方法はq()でRを閉じる方法しかありませんでした。
メモリーを上手に割り当てるようにするにはどうしたらよろしいでしょうか?
SS (2006-09-28 (木) 14:53:42)
2パラメータの最尤推定を行う際、optim()を使って尤度関数を最大化する値を探そうとしたら、「以下にエラーchol(M) : 次数 1 の主対角行列が正定値ではありません」というエラーメッセージを得ました。nlm()でも同様のエラーメッセージを得ました。
optimの場合、optionとして"Nelder-Mead", "BFGS", "CG", "L-BFGS-B", "SANN"の全てを試しましたが、同様のエラーメッセージが出ました。
2パラメータなので、尤度関数をプロットしてみた感じだと、山頂はある様子でした。初期値も、プロットした尤度関数の山頂付近に設定しました。
このエラーメッセージは、どういう状況のときに出力されるのか、ご存知の方がいらっしゃったら、教えていただけるとありがたいです。
> m <- structure(c(1, 0.1, 0.8, 0.1, 1, -0.7, 0.8, -0.7, 1),
+ .Dim = c(3, 3))
> m
[,1] [,2] [,3]
[1,] 1.0 0.1 0.8
[2,] 0.1 1.0 -0.7
[3,] 0.8 -0.7 1.0
> chol(m)
以下にエラーchol(m) : 次数 3 の主対角行列が正定値ではありません
> eigen(m)
$values
[1] 2.0147034 1.0990993 -0.1138027 # 3つめの固有値がマイナス
$vectors
[,1] [,2] [,3]
[1,] -0.5262037 -0.65755945 0.5391894
[2,] 0.4471271 -0.75329549 -0.4823104
[3,] -0.7233167 0.01270732 -0.6903995
> m <- matrix(-1, 1,1) # 次数1というと,1行1列の行列ですが。。。?
> chol(m)
以下にエラーchol(m) : 次数 1 の主対角行列が正定値ではありませんnonami (2006-09-14 (木) 19:45:14)
散布図を作っています。
x, yのデータ以外に第3のデータがありまして、それが0〜100まであります。
rainbowをの一部を使って、0なら青、100なら赤その間は緑から黄色、オレンジというようになんとなくサーモグラフィっぽい感じで色をつけています。
plot自体はうまく行ったと思うのですが、legendを付けようとしたときに
はたと困ってしまいました。青→緑→黄色→オレンジ→赤の順に数値が高くなるよというlegendは作成できるのでしょうか?
cols=rainbow(101)
x <- rnorm(1000)
y <- rnorm(1000)*200
z <- sample(101, 1000, replace=TRUE)
plot(x, y, col=cols[z], type="p", pch=20)
for (i in 1:101) rect(1.5, 502-2*i, 2.5, 500-2*i,col=cols[i], border=cols[i])
text(2.5, seq(502, 300, length=3), c("lo", "med", "hi"), pos=4)
定数で決め打ちしているところを引数で渡す位置情報に基づく計算できめるなど,汎用化して関数にすればよい。
こんな散布図を描いているんでしょうか?
#ref(): File not found: "scatter.png" at page "Q&A (初級者コース)/13"
TH (2006-08-13 (日) 18:39:13)
Rで安定分布に従う擬似乱数を発生するパッケージ等をご存知の方はご教示ください。
はるか (2011-11-11 (金) 11:56:20)
R初心者です。先日は疑問に対して丁寧にお答えいただいてありがとうございました。
その後、tune.svmを使おうと先日からお世話になっているサイトで丁寧に解説があったので、下記のように同様に実行してみたところgammaRange <- 10^(-5:5) costRange <- 10^(-2:2) t <- tune.svm(Species ~ ., data = iris, gamma=gammaRange, cost=costRange,tunecontrol = tune.control(sampling="cross", cross=8)) 以下にエラー sample(n) : .Random.seed は整数ベクトルではなくタイプ 'list' でと出てしまい実行することができませんでした。.Random.seedの設定?など特にしていないのですが、何か特別な設定等必要なのでしょか?webを検索したのですが、同じような点で悩んでいる例がなかったので、質問させていただきました。
環境は、OS WindowsXp, R version 2.12.1です。お手数ですが、アドバイスいただけると大変助かります。よろしくお願い致します。
mat (2011-11-10 (木) 13:03:16)
あるスクリプトを開いてそれを実行する際に、作業ディレクトリを(スクリプト内で動的に)そのスクリプトファイルが置かれている場所に変更することは出来ないのでしょうか?
KT9 (2011-11-09 (水) 09:45:58)
医学系の研究者です。
JPEG形式の超音波画像があります。これをRで取り込んで、ある一点の(x,y)座標、2点間の距離、面積などを計測する方法はあるのでしょうか。
(x,y)さえ打ち出せれば、後は自分で別個に組んだプログラムで、距離、面積、角度などは簡単に測れると考えています。
以前他の研究者がMathLabでは、JPEGで取り込んだ画像上で、様々な処理を行っているのを見ました。
宜しくお願い致します。
diereinevernunft (2011-11-03 (木) 00:17:54)
R初心者です。
Rを2.13.0から最新バージョン(2.14.0)に更新し、Rコマンダーも新たに1.7-0をインストールし直したのですが、データのインポートでCSVファイルからデータを読み込もうとすると、Dataset <- read.table("C:/Users/Documents/ワークショップ/Rデータ処理用/(ここで強制改行) 修論素材/加工後/R用/111024/tsushin_growth.csv", header=TRUE, sep=",", na.strings="NA", dec=".", strip.white=TRUE)が実行されたあと、メッセージ欄に
[2] エラー: NAと表示され、データがインポートできません。
ちなみに、CSVファイルをtxtファイルに変えるとインポートできるようなので、現在はそれで対応しています。
googleで検索してみたのですが、現状では有効な対策を見つけることができませんでした。OS Windows7(64bit) R version 2.14.0 Rcmdr version 1.7-0
はるか (2011-11-02 (水) 15:38:48)
R初心者です。svmに興味があって、googleを検索したところ、丁寧な解説があったのでこのWebに沿って、同じようにやるとTotal Accuracy:94 Single Accuracies: 90 96 96と同じように結果が再現され正答率がでたところまではよかったのですが、いざ自分の持っているデータでやって結果を見ようとすると...
model <- svm(judge~., data=dataset, corss=3) summary(model) Parameters: SVM-Type: eps-regression SVM-Kernel: radial cost: 1 gamma: 0.01190476 epsilon: 0.1 Number of Support Vectors: 51 3-fold cross-validation on training data: Total Mean Squared Error: 0.6049732 Squared Correlation Coefficient: 0.06871624 Mean Squared Errors: 0.4767778 0.5592772 0.7788645と出てしまい、Accurancyが出てきません。
何がいけないのか分からず、困っています。
OS WindowsXp, R version 2.12.1でやっています。
扱ったデータから解を求められないということなのでしょうか?
それとも何か別に問題点があるのでしょうか。
周りにアドバイスを求める人がおらず困っています。
お手数ですが、アドバイスいただけると大変助かります。よろしくお願い致します。
みのむし (2011-10-30 (日) 16:58:40)
R初心者です。導関数を求めるのに、deriv()を使っています。直接、カッコ内に関数を打ち込まないといけないようなのですが、そのほかの方法はないのでしょうか?
たとえば、> Df <- deriv(~x^2, "x", func=TRUE) > Df(2) [1] 4 attr(,"gradient") x [1,] 4と答えがえられるのですが、カッコ内に打ち込む関数x^2を、最初にfと定義し、それを代入するとうまくいきません。
> f <- expression(x^2) > Df <- deriv(f, "x", func=TRUE) > Df(2) expression(x^2) attr(,"gradient") x [1,] 0ご存じの方がいらしゃれば、教えていただけないでしょうか?お忙しいところ申し訳ありません。よろしくお願いいたします。
> f <- expression(x^2)
> Df <- deriv(f, "x", func=TRUE)
> Df(2)
[1] 4
attr(,"gradient")
x
[1,] 4ringori (2011-10-27 (木) 01:48:51)
お忙しいところ失礼します。plotを使って折れ線グラフを作成しているのですが、x軸の目盛りの内容の一部を文字列で指定したく思っています。少し調べた所test <- c(0.8699, 0.9029, 0.9133, 0.9014, 0.9160) plot(test, type="l", lwd=2, xlab="test", ylab="AUC", xaxt="n") mtext(1:4, 1, 1, at=1:4) mtext("Q", 1, 1, at=5)として近いものは出来たのですが、目盛りの線が無くなってしまうのと、もっとスマートな方法があればご教授いただければと思います。 もし、過去の投稿等見逃していましたら申し訳ありません。環境は
sessionInfo() R version 2.10.1 (2009-12-14) i386-pc-mingw32です。よろしくお願いします。
plot(test, type="l", lwd=2, xlab="test", ylab="AUC", xaxt="n") axis(side=1, at=1:5, label=c(1:4, 'Q'))
Toy (2011-10-26 (水) 14:12:37)
お世話になります。
Windows 7 で R version 2.13.0 を使用しています。
様々な記入形態がある文字列について抽出処理を行おうとしています。
下記のような文字ベクトルについてx <- c("マツホ−ム", "タケハウス", "ウメコウムテン", "カブ)マツホ−ム", "タケハウスカブシキガイシャ", "ウメコウムテンカブ")次のような例では、正しく結果が表示されます。
> grep("_*ウメコウムテン_*", x, value = TRUE) [1] "ウメコウムテン" "ウメコウムテンカブ"ところが、次の例では
> grep("_*タケハウス_*", x, value = TRUE) character(0)と全く結果が得られず、次の例では、
> grep("_*マツホ−ム_*", x, value = TRUE) [1] "マツホ−ム" "ウメコウムテン" "カブ)マツホ−ム" "ウメコウムテンカブ"と余計なものまで出力されてしまいます。
どうしてこのような結果になるのか、お教えいただければ幸いです。
> x <- c("マツホ−ム", "タケハウス", "ウメコウムテン",
+ "カブ)マツホ−ム", "タケハウスカブシキガイシャ", "ウメコウムテンカブ")
> grep("_*ウメコウムテン_*", x, value = TRUE)
[1] "ウメコウムテン" "ウメコウムテンカブ"
> grep("_*タケハウス_*", x, value = TRUE)
[1] "タケハウス" "タケハウスカブシキガイシャ"
> grep("_*マツホ−ム_*", x, value = TRUE)
[1] "マツホ−ム" "カブ)マツホ−ム"nya (2011-10-23 (日) 01:21:25)
read.csv でカンマ区切りのCSVファイルを読み込んだ後、ある特定の文字列から始まる(たとえば"hoge test")行数まで読み飛ばす方法はありませんか?
この行数はファイルによって異なるため、read.csvのskip=xxxでは対応できず、困っております。
ご教授いただければ幸いです。
data <- read.csv("csvfile.csv")
data <- data[which(data[,1] == "hoge test"):nrow(data),]
とか -- 2011-10-23 (日) 07:32:05x <- "csvfile.csv"
table <- read.table(x, sep="\n", header=F)
c <- grep("Well", table$V1) - 1
read.table(x, sep = ",", header = T, skip = c)
どうかよろしくお願いします。 -- nya 2011-10-23 (日) 09:57:06x <- "csvfile.csv"
table <- read.table(x, sep="\n", header=F, blank.lines.skip = F, fill = T)
c <- grep("Well", table$V1) - 1
read.table(x, sep = ",", header = T, skip = c, blank.lines.skip = F, fill = T)
とすることで解決しました。x <- "test.csv"
table <- readLines(x)
c <- grep("Well", table) - 1
read.csv(x, skip = c, blank.lines.skip = FALSE)複数のブロック行列 (2011-10-18 (火) 22:35:10)
A = (3,7,4,6,...,)のような列ベクトルがあり、I[N*N]を単位行列としたとき、B = (I[3*3], I[7*7], I[4*4], I[6*6], ...)のような、Aの要素を次元とするブロック対角行列Bを作成したいのですが、方法が分からず苦慮しております。
ご教授願えませんでしょうか。
A <- c(3, 7, 4, 6) (mlist <- list(diag(1, A[1]), diag(1, A[2]), diag(1, A[3]), diag(1, A[4]))) lapply(A, diag)結果を比較すると同じものができるのが分かるでしょう。
A <- c(3, 7, 4, 6) mlist <- lapply(A, function(N) matrix(1, N, N)) # このようにする library(Matrix) bdiag(mlist)のようにすればよいでしょう。 -- 河童の屁は,河童にあらず,屁である。 2011-10-19 (水) 16:52:55
tau (2011-10-17 (月) 22:09:13)
R 2.13.1を使用しております。
下記のようにt.testを行いました。
> group1 [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11] [,12] [,13] [,14] [,15] [,16] [1,] 15 12 12 14 14 12 16 11 19 18 15 11 10 9 7 15 [2,] 14 16 12 9 14 16 14 11 11 6 7 11 16 12 9 9 [3,] 16 10 11 16 20 20 16 18 17 16 12 8 14 12 17 10 > group2 [,1] [,2] [,3] [,4] [1,] 18 19 12 16 > group3 [,1] [,2] [,3] [,4] [1,] 14 17 17 15 > group4 [,1] [,2] [,3] [,4] [1,] 13 19 11 17 > t.test(group1,group2,var.equal=F) Welch Two Sample t-test data: group1 and group2 t = -1.9197, df = 3.667, p-value = 0.1337 alternative hypothesis: true difference in means is not equal to 0 95 percent confidence interval: -7.810966 1.560966 sample estimates: mean of x mean of y 13.125 16.250 > t.test(group1,group3,var.equal=F) Welch Two Sample t-test data: group1 and group3 t = -2.9049, df = 6.241, p-value = 0.02597 alternative hypothesis: true difference in means is not equal to 0 95 percent confidence interval:~ -4.8156168 -0.4343832 sample estimates: mean of x mean of y 13.125 15.750 > t.test(group1,group4,var.equal=F) Welch Two Sample t-test data: group1 and group4 t = -0.9899, df = 3.474, p-value = 0.3861 alternative hypothesis: true difference in means is not equal to 0 95 percent confidence interval:~ -7.463495 3.713495 sample estimates: mean of x mean of y 13.125 15.000
group1とgroup2,3,4の平均値を比較していますが、group2とgroup4の平均値の中間の値を取るgroup3でのみP値が小さくなるのはなぜでしょうか?
ご教示頂ければ幸いです。
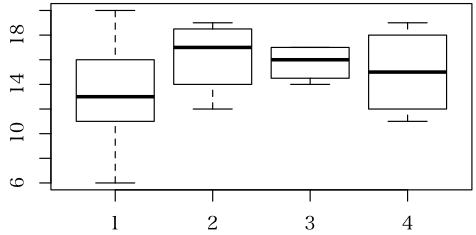
pp (2011-10-13 (木) 15:34:41)
こんにちは。
複数回答のアンケートを作って読み込んだところ、下記のように表示されます。(データフレーム)> data 1 にんじん,たまねぎ 2 にんじん 3 にんじん,たまねぎ,じゃがいもこれをtable(data)とすると上記がそのまま出てしまいますが、
にんじん 3 たまねぎ 2 じゃがいも 1とするような方法はないでしょうか。(文字をカンマで分割したい)
初心者で恐縮ですが、ご回答いただけますと幸いです。
> table(unlist(strsplit(as.character(data[,1]), ",")))
じゃがいも たまねぎ にんじん
1 2 3
内側の as.character は,データを読み込むときに文字列が factor にならないようにしておけば不要。 -- 河童の屁は,河童にあらず,屁である。 2011-10-13 (木) 16:08:04Minako (2011-10-12 (水) 08:31:41)
はじめまして。R初心者(31歳前田敦子似)です。
データフレームのhogeという変数名を入力すると、その変数のサマリーや検定結果などをresult.txtという名前のファイルに保存する関数を作ろうと思ってます。計算結果の前に、どの変数について調べたか記載したいので、
あるデータフレームに年齢が入力されたageという変数名があるときに、ageage(x)という関数ageage(x)<-function(x) { m<-sprintf("title\t%s\n",x) cat(m,file="result.txt",append=T) }を作って、result.txtに、title age と表示させたいです。
ところがこれでageage(age)を実行するとtitle 50 title 54 title 76などとageが年齢を含んだベクトルとして理解されて、全部記載されてしまいます。関数のxに入力された変数名を文字列として取得するためにはどのようにすればよいでしょうか??
> d <- data.frame(age=c(50, 54, 76), sex=c("male", "female", NA))
> deparse(substitute(d$age))
[1] "d$age"
> attach(d)
> deparse(substitute(age))
[1] "age"
> ageage <- function(x) {
+ m <- sprintf("title\t%s\n", x)
+ cat(m, file="result.txt", append=T)
+ }
> ageage(d$age)
> ageage(age)func <- function(data.frame, vector)
{
for (i in vector) {
variable.name <- colnames(data.frame)[i]
values <- data.frame[,i]
cat("variable=", variable.name, " n=", length(values),
" mean=", mean=mean(values, na.rm=TRUE),
" s d=", sd=sd(values, na.rm=TRUE), "\n")
}
}
と定義しておけば,以下のようになるだろうと言うこと(まあ,趣味の問題かも知れないけど)
> func(iris, 1:4) variable= Sepal.Length n= 150 mean= 5.843333 sd= 0.8280661 variable= Sepal.Width n= 150 mean= 3.057333 sd= 0.4358663 variable= Petal.Length n= 150 mean= 3.758 sd= 1.765298 variable= Petal.Width n= 150 mean= 1.199333 sd= 0.7622377
tx (2011-10-11 (火) 11:00:45)
プレートリーダーが出力する以下のようなフォーマットのエクセルファイルから、Rへのデータの読み込みを試みておりますが、Plate Repeat Well Type Time Luciferase (CPS) Time Luciferase (CPS) 1 1 A01 M 00:00:06.80 53530 00:02:38.05 47900 1 1 A02 M 00:00:07.07 107230 00:02:38.31 94150 1 1 A03 M 00:00:07.34 103510 00:02:38.58 95070
取り込むと、一秒以下の部分が削られてしまいました。
読み込みにはこちらのサイトに書かれていた方法を用いました。
> library(RODBC) > sheet <- odbcConnect("Excel Files") > tab <- sqlQuery(sheet, "select * from [List ; Plates 1 - 1$]") > t <- as.numeric(difftime(tab[,7], tab[,5], units="secs")) > t[1] [1] 152
コンマ秒の部分を扱う方法を教えていただけると嬉しいのですが、
どうぞよろしくお願い致します。
> (x <- difftime("2011-10-11 00:02:38.05 JST", "2011-10-11 00:00:06.80 JST", units="secs"))
Time difference of 151.25 secs
> as.numeric(x)
[1] 151.25tx (2011-10-11 (火) 07:18:37)
POSIXct形式の時刻の差を使って乗除計算をしたいのですが、defftimeの値を使っての割り算をRは認めてくれません。"difftime"オブジェクトを数値の配列に変換する方法、または他の方法での時間差の計算法がありましたら御教授いただけませんでしょうか。どうぞよろしくお願い致します。
tetora (2011-10-10 (月) 00:22:25)
既出でしたらすみません。Rの出力をGIF形式で保存したいのですが、デフォルトでGIF出力する関数が見当たりませんでした。こちらのページを参照すると、パッケージでもGIF形式での保存に対応したものはないようなのですが、どうしてもGIF形式で保存したい場合は、一度別の形式(jpeg,pngなど)で保存した後、外部のプログラムで形式変換するしか方法はないのでしょうか?
まつだ (2011-10-07 (金) 15:32:59)
image関数を使って、1または0のバイナリデータの表示を試みているのですが、データ量が多い時にうまく表示できずに困っています。
例えば、以下のようなデータを作ります。x <- c(rep(0, 30000), 1, rep(0, 19999))これをimage関数を使って表記するために以下を実行します。
m <- matrix(x, nrow=50000, ncol=2) par(mfrow=c(2,1)) # plot関数との表示比較のため image(m, col=c("white", "black")) plot(1:nrow(m), m[,1], type="l")plot関数では、50000データの中に1つだけある1のデータを表記できているようですが、image関数ではそれが消えてしまいます。
このようなときに、皆さんはどのように対処なされていますでしょうか。
ご教授いただければ幸いです。
ちなみに、以下くらいのデータであれば問題はないようです。x <- c(rep(0, 300), 1, rep(0, 199)) m <- matrix(x, nrow=500, ncol=2) par(mfrow=c(2, 1)) image(m, col=c("white", "black")) plot(1:nrow(m), m[,1], type="l")どうぞよろしくお願いします
plot(1:nrow(m), m[,1], type="l") # あなたのプログラムだとスパイク状に描画される plot(1:nrow(m), m[,1], type="n") segments(1:nrow(m), 0, 1:nrow(m), m[,1]) # 垂直線を描く
> あなたの環境で,ピクセル数を指定せずに plot または image を使って表示されるグラフの実際のピクセル数は幾つですか?
大変申し訳ありませんが、自分のPCの環境をちゃんと把握しておらず、即答できません。
何とか調べてお答えしたいと思いますが、当面ピクセル数の問題として、plotで対応できるように試みたいと思います。
> plot ではピークみたいに書かれるのがいやだということなら,該当箇所に垂直線を書くようにすればよいでしょう。
ご提案ありがとうございます。plotでの表示を試みているのですが、実際のデータは17280行×183列のデータで、各行で異なる値の数列が含まれています。imageで表示する際のy軸は時系列として表示しています。データ量は違いますが、最近投稿された質問のケースとよく似ており、以下の投稿と質問を参考に作図しておりました。
image における時系列軸の反転
http://www.okada.jp.org/RWiki/?%A3%D1%A1%F5%A3%C1%20%28%BD%E9%B5%E9%BC%D4%A5%B3%A1%BC%A5%B9%29%2F13#m560db05
ごちゃごちゃしていて、大変申し訳ありませんが、大体以下のようなコードで描いております。
st <- c("2008/12/19", "2009/3/24")
st <- strptime(st, "%Y/%m/%d", tz="")
y.days <- seq(st[1], st[2], by="1 day")
x.hours <- seq(0, 48, length=3600*48/10)
m <- matrix(sample(c(0, 1), (3600*48/10)*length(y.days), rep=T, prob=c(10, 1)),
17280, length(y.days))
par(oma=c(2, 1.5, 1, 1))
y.lim <- c("2008/12/17", "2009/3/26")
y.lim <- strptime(y.lim, "%Y/%m/%d", tz="")
image(x.hours, y.days, m, col=c("white", "black"), yaxt="n", xaxp=c(0, 48, 4),
xlab="hours", ylab=NA,
xlim=c(-0.1, 48), ylim=c(as.numeric(y.lim[2]), as.numeric(y.lim[1])))
y.gr <- c("2008/12/18 12", "2009/3/24 12")
y.gr <- strptime(y.gr, "%Y/%m/%d %H", tz="")
y.gr <- seq(y.gr[1], y.gr[2], by="1 days")
abline(h=y.gr, col="gray")
axis(2, at=as.numeric(y.days[(1:4)*20]), labels=as.character(y.days[(1:4)*20],
format="%Y/%m/%d"), las=2)
これを、plotを使って再現するにはどのようにすればよいのか、いいアイデアが浮かびません。 作図は、アイデア次第で色々な図が描けると承知しているのですが、 まだまだRの関数の知識も圧倒的に少く、どのようにして描いていいものか困っております。 自分の勉強不足は十分承知しておりますが、もし、教えても構わないという方がいらっしゃいましたら、 ご教授いただけると幸いです。 -- まつだ 2011-10-07 (金) 18:39:08png(, length(x1), length(y1))
par(mar = c(0, 0, 0, 0))
image(x.hours, y.days, m, col = c("white", "black"))
dev.off()
プロットを使いたいならこんな感じでどうでしょう。
x <- x.hours[matrix(1:length(x.hours), length(x.hours), length(y.days))[m == 1]] y <- y.days[matrix(1:length(y.days), length(x.hours), length(y.days), byrow = TRUE)[m == 1]] plot(x, y, pch = 20, cex = 1) # 真っ黒になるときはcex = 1の値を小さくしてみる。ちなみに、wikiで">"(半角)を行頭に書きたいときはチルダを入れて"~>"としてください。
for(i in 1:ncol(m)){
segments(x.hours[m[,i]==1], y.days[i], x.hours[m[,i]==1], y.days[i+1])
}
ループを作図に使うという発想はこれまでありませんでしたが、試行錯誤しているうちに思いつきました。まだまだ勉強することがたくさんありますが、今回の件はすごくためになりました。ありがとうございました。-- まつだ 2011-10-09 (日) 00:42:13st <- c("2008/12/19", "2009/3/24")
st <- strptime(st, "%Y/%m/%d", tz="")
y.days <- seq(st[1], st[2], by="1 day")
x.hours <- seq(0, 48, length=3600*48/10)
m <- matrix(sample(c(0, 1), (3600*48/10)*length(y.days), rep=T, prob=c(10, 1)),
17280, length(y.days))
par(oma=c(2, 1.5, 1, 1))
y.lim <- c("2008/12/17", "2009/3/26")
y.lim <- strptime(y.lim, "%Y/%m/%d", tz="")
plot(c(-0.1, 48), c(as.numeric(y.lim[2]), as.numeric(y.lim[1])), type="n",
yaxt="n", ylab="")
y.days2 <- rep(y.days, each=17280)[m==1]
x.hours2 <- rep(x.hours, 96)[m==1]
mk <- diff(y.days)[1]
segments(x.hours2, y.days2, x.hours2, y.days2+mk)
y.gr <- c("2008/12/18 12", "2009/3/24 12")
y.gr <- strptime(y.gr, "%Y/%m/%d %H", tz="")
y.gr <- seq(y.gr[1], y.gr[2], by="1 days")
abline(h=y.gr+mk/2, col="gray")
axis(2, at=as.numeric(y.days[(1:4)*20]), labels=as.character(y.days[(1:4)*20],
format="%Y/%m/%d"), las=2)shannon (2011-10-05 (水) 18:38:21)
質問のタイトルが不適当かもしれませんが、よろしくお願いします。a <- c(0, 0, 1, 1, 0, 0, 0, 1, 0, 0)上のような"0"と"1"が並んでいるベクトル(a)に対して、
a2 <- c(0, 1, 1, 0, 1, 0)"0"が2つ以上ならんでいる部分を削除して、"1"の前後にそれぞれ"0"が1つだけ入るように(a2)したいのですが、forループを使って
a2 <- 0 for (i in 1:9) { if (a[i]==0 && a[i+1]==0) next a2 <- c(a2, a[i+1]) }のようにすると、ベクトルの要素数が膨大になった場合にかなり時間がかかってしまいます。
そこで0が2つ並んでいる要素番号(上記のaでいえば、要素番号1,5,6,9)に、FALSEを当てはめたような、論理ベクトル(a3)を作成して、下のように計算したいのですが、方法が分からず悩んでいます。forループを使わずに、a3のようなベクトルを作るにはどうしたらよいのでしょうか。もしくは別の方法でa2を作りだす良い方法はないでしょうか。分かりにくい質問で申し訳ないですが、ご教授お願いします。> a3 <- c(FALSE,TRUE,TRUE,TRUE,FALSE,FALSE,TRUE,TRUE,FALSE,TRUE) > a2 <- a[a3] > a2 [1] 0 1 1 0 1 0
b <- rle(a) b$lengths[which(b$values==0)] = 1 a2 <- inverse.rle(b)
afromonkey (2011-10-05 (水) 16:26:51)
統計ソフトRで、横軸をy軸、縦軸をx軸にして線グラフやヒストグラムを描くには、どうすればよいのでしょうか。
barplot関数の場合は引数に horiz = T を与えればよいということが分かったのですが、 plot や hist などの他の関数についてはどのように設定すればよいでしょうか。
よろしくお願いします。
set.seed(10); x <- rnorm(100)
hi <- hist(x, plot=F)
plot(0, 0, xlim=range(hi$counts), ylim=range(hi$breaks), type='n',
xlab='', ylab='', las=1, bty='L')
for(i in seq(length(hi$counts))) {
rect(0, hi$breaks[i], hi$counts[i], hi$breaks[i+1], col=grey(0.8),
border=grey(0.7))
}
box(bty='L')set.seed(10); x <- rnorm(100) h <- hist(x, plot = FALSE) barplot(h$counts, space = 0, horiz = TRUE) plot.window(xlim = range(h$counts), ylim = range(h$breaks)) axis(2)ところで、線グラフについては里見さんがおっしゃってるようにplot(y, x)で十分だと思うのですが、それでは駄目なんですか?
365 (2011-10-04 (火) 22:28:06)
Rの2.13.1以降でRコマンダーで2バイト文字のファイル名などを使用すると不正なマルチバイト文字があるというエラーが出ます。
Rconsoleでは当該ファイル名は認識され、データもロードできますので、Rコマンダーの方の問題かと思うのですが原因がよくわかりません。過去に同様な投稿も見つけられませんでした。
また、R2.13.0以前では同様なエラーは出ませんでした。
OSはwindowsXP sp3、RcmdrはそれぞれのRバージョンで更新しています。
バージョンアップが出来ずに困っています。どなたかお心当たりのある方、ご教授下さい。
西田 (2011-10-01 (土) 15:48:10)
あるパッケージに含まれる動作を拡張して使いたいと思っています。
今まではlibrary()で読み込んだ後、関数等を書き換えていたのですが、今回拡張したい部分がどうやらC言語で書かれているようで(.Call)書き換え方がわかりません。
パッケージの中身を直接いじることができれば一度で済みますし、余計な不具合の原因にもならないと思い質問させて頂きました。どうかご教授ください。
tak (2011-09-30 (金) 16:41:40)
下のような、縦方向にケース番号、横方向にデータ番号を配置した欠損値ありの行列(1)があるとします。(実際はケース数、データ数の種類がもっと多いものを使います。)> data data1 data2 data3 data4 data5 case1 NA 10 100 1000 10000 case2 2 NA 200 NA 20000 case3 3 30 300 3000 30000 case4 NA 40 NA 4000 NA case5 5 NA 500 5000 50000 case6 6 60 600 NA NA case7 7 70 700 7000 70000 case8 NA 80 800 8000 NA case9 9 90 NA 9000 90000 case10 10 NA 1000 10000 100000上の行列に対して、ケース毎の類似度を計算した行列(2)が下のようになります。(例えば1行2列は、case1のcase2との類似度です。『case1の、case2との類似度』と『case2の、case1との類似度』は同様に計算しているので対称行列になっています。)
> similarity [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [1,] 0.00 0.16 0.46 0.50 0.77 0.84 1.38 1.41 1.61 1.73 [2,] 0.16 0.00 0.20 0.00 0.60 0.67 1.00 0.67 1.17 1.61 [3,] 0.46 0.20 0.00 0.17 0.46 0.63 1.05 1.00 1.42 1.61 [4,] 0.50 0.00 0.17 0.00 0.11 0.25 0.50 0.67 0.84 0.67 [5,] 0.77 0.60 0.46 0.11 0.00 0.17 0.46 0.47 0.80 1.15 [6,] 0.84 0.67 0.63 0.25 0.17 0.00 0.21 0.33 0.53 0.67 [7,] 1.38 1.00 1.05 0.50 0.46 0.21 0.00 0.20 0.47 0.69 [8,] 1.41 0.67 1.00 0.67 0.47 0.33 0.20 0.00 0.17 0.31 [9,] 1.61 1.17 1.42 0.84 0.80 0.53 0.47 0.17 0.00 0.20 [10,] 1.73 1.61 1.61 0.67 1.15 0.67 0.69 0.31 0.20 0.00そこで、行列(1)に存在する欠損値を類似度によって補完する処理を作成したいのですが、どのようにすればよいのか思いつかず困っています。
処理の内容は、欠損値補完対象のケース番号と類似度の高い上位k個のケース(但し、kは任意で、補完対象のデータと同じ番号のデータが欠損値であるケースは除く)の、同じ番号のデータの平均値を入れるようにします。
例えば(k=3の場合)、case1のdata1が欠損値になっていますが、case1と類似度の高い上位3ケースは、case10, case9, case8です。しかし、case8のdata1は欠損値なので、次に類似度の高いcase7を使って、case10, case9, case7のそれぞれのdata1である10,9,7の平均をとって、8.66を補完値とします。この処理を全ての欠損値に対して行います。
説明がわかりづらくて申し訳ありませんが、宜しくお願いします。
n <- nrow(data) # 列数
m <- ncol(data) # 行数
suf <- integer(3) # 類似度が高いもの3つの添え字を入れる
for (i in 1:n) { # 各 case について
for (j in 1:m) { # 各 data について
if (is.na(data[i, j])) { # もし,欠損値なら,類似度によりランク付け
# ties.method は average 以外ならどれでもよい?
hi <- rank(similarity[1,], ties.method="first")
# 類似度が高く,かつ,データが欠損値でない case の添え字を
# suf に格納(3つまで)
l <- 0
for (k in n:1) {
if (!is.na(data[hi[k], j])) { # 欠損値でないなら
l <- l+1 # カウンタを増やす
suf[l] <- k # 添え字を格納
if (l == 3) break # 3つ見つかったら脱出
}
}
if (l == 3) { # 3つないときには NA のままにする?
# (平均値でよいと思うけど)
data[i, j] <- mean(data[hi[suf], j])
}
}
}
}
実行結果
data1 data2 data3 data4 data5 case1 8.666667 10 100.0000 1000 10000.00 case2 2.000000 80 200.0000 9000 20000.00 case3 3.000000 30 300.0000 3000 30000.00 case4 8.666667 40 833.3333 4000 86666.67 case5 5.000000 80 500.0000 5000 50000.00 case6 6.000000 60 600.0000 9000 86666.67 case7 7.000000 70 700.0000 7000 70000.00 case8 8.666667 80 800.0000 8000 86666.67 case9 9.000000 90 833.3333 9000 90000.00 case10 10.000000 80 1000.0000 10000 100000.00
hi <- rank(similarity[1,], ties.method="first") # 変更前 hi <- rank(similarity[i,], ties.method="first") # 変更後
hi <- rank(similarity[i,], ties.method="first")
hi2 <- hi
for (a in 1:max(hi)) {
for (b in 1:max(hi)) {
if (hi[b] == max(hi) + 1 - a) {
hi2[a] <- b
break
}
}
}ifelse(is.na(data), t(apply(similarity, 2,
function(i) apply(data[order(i, decreasing = TRUE), ], 2,
function(x) mean(x[head(which(!is.na(x)), 3)])))), data)
data1 data2 data3 data4 data5
case1 8.666667 10.00000 100.0000 1000.000 10000.00
case2 2.000000 73.33333 200.0000 8666.667 20000.00
case3 3.000000 30.00000 300.0000 3000.000 30000.00
case4 8.666667 40.00000 633.3333 4000.000 66666.67
case5 5.000000 60.00000 500.0000 5000.000 50000.00
case6 6.000000 60.00000 600.0000 4666.667 43333.33
case7 7.000000 70.00000 700.0000 7000.000 70000.00
case8 3.333333 80.00000 800.0000 8000.000 20000.00
case9 9.000000 90.00000 200.0000 9000.000 90000.00
case10 10.000000 36.66667 1000.0000 10000.000 100000.00ohno (2011-09-30 (金) 15:37:38)
C言語などで使うgoto文のように、指定行へジャンプしたい時に使う関数はあるのでしょうか?
下の行へジャンプする場合は、以下のようにジャンプ先の命令文の前までをwhileなどの条件式で囲って途中でbreakすれば、一応はジャンプできますが、
この方法では上の行へジャンプする時が複雑になるので、なにか良い方法がないかと考えています。while (1) { ... if (1) break ... } ... #ここへジャンプ良い方法がありましたらご教授お願いいたします。
do-san (2011-09-29 (木) 15:30:24)
例えば、文字列"abcde"があったとして、文字パターン"d"にマッチするのが何番目かを調べるにはどうすればよいのでしょうか?(答えが4になる。)
文字列"abcde"を一文字ずつ分解・ベクトル化して、charmatchで調べる、というようなことをすればいいのでしょうか?
> strsplit("abcdedcba","")[[1]]
[1] "a" "b" "c" "d" "e" "d" "c" "b" "a"
> which(strsplit("abcdedcba","")[[1]]=="d")
[1] 4 6BCD (2011-09-27 (火) 18:38:03)
時折、名前のマッチを行うと不具合が生じます。これは何故でしょうか? 今回起こった不具合までの経過は以下のようになります。どなたか解決法をご存知の方がいらっしゃれば教えていただけないでしょうか?
数値と値に対する名前が入っているベクトルA、ベクトルBがあります。> AB <- c(names(A), names(B)) # 名前を取り出して一つのベクトルにします。 > CD <- AB[duplicated(AB)] # 重複している名前を一つにする。 ここで以下のような行列xがあります。 V1には名前が入っていて、ベクトルAとベクトルBの名前は この中に全て含まれています。 > x V1 V2 1 名前1 値 2 名前2 値 3 名前3 値 : > x[x$V1 == CD, ] # CDの要素と行列xの名前とが 一致する行を取り出します。これらを実行しても最後に名前が一致する行が取り出されないのです。この時エラーが出るのですが「長いオブジェクトの長さが短いオブジェクトの長さの倍数になっていません 」と表示されます。
> CD <- c("foo", "bar", "baz")
> (x <- data.frame(V1=c("abc", "foo", "zoo", "boo",
"baz", "yah", "bar"), V2=1:7))
V1 V2
1 abc 1
2 foo 2
3 zoo 3
4 boo 4
5 baz 5
6 yah 6
7 bar 7
> x[x$V1 %in% CD,]
V1 V2
2 foo 2
5 baz 5
7 bar 7BCD (2011-09-27 (火) 17:58:16)
c("水", "金", "地", "火", "木", ......) のようなベクトルがあります。
ここから c("水", "地", ....) を除いたベクトルを作りたいのですが、ifやforを使わずに簡単に行う方法はありますか?ご存知の方がいらっしゃいましたらどうか教えてください。
a <- c("水", "金", "地", "火", "木", "土", "天", "海")
b <- c("水", "地", "土")
a[!a %in% b]宗二 (2011-09-26 (月) 11:25:26)
plot関数を使用する際に、グラフィックスパラメータ"pch"で点の形を選択できますが、用意されている形状以外で、任意に点の形を変えることはできるのでしょうか。
具体的には、一点ごとに角度と長さの異なる矢印をプロットしたいのですが、良い方法がありますでしょうか。
spin <- function(x0, y0, factor=1, rotation=2)
{
r <- seq(0.1, 1.0, length=100)*factor
theta <- seq(0, -rotation*2*pi, length=100)
x <- r*cos(theta)+x0
y <- r*sin(theta)+y0
lines(x, y)
}
plot(0:10, 0:10, type="n", axes=FALSE, xlab="", ylab="")
spin(3, 5, 0.5)
spin(5, 5, 1)
spin(7, 5, 0.2)
spin(3, 7, 0.5, rotation=5)
spin(5, 7, 1, rotation=4)
spin(7, 7, 0.2, rotation=1)
square (2011-09-25 (日) 18:59:28)
マルコフ連鎖とは、乱数発生の方法みたいですが、sample関数による乱数発生とは何が違うのでしょうか?
どんな分布もsampleで発生できると思うのですが。
ランゲル・ハンス (2011-09-21 (水) 08:49:14)
いつも掲示板を参考にしております。
これまでExcelで計算していたのですが、Rでもぜひ計算できるようにしたいと思います。
ベースラインy=cと回帰直線y=ax+bの交点から閾値を求めたいと思います。
次のようなデータ(dataXY)があります。X Y 4 1.1 4.1 0.6 4.2 0 4.3 0.9 4.4 0.9 4.5 0 4.6 0 4.7 0 4.8 0.7 4.9 0.9 5 0 5.1 1 5.2 1.1 5.3 0 5.4 0.4 5.5 1.5 5.6 0.9 5.7 1.5 5.8 2.1 5.9 2.6 6 3.2 6.1 4.3 6.2 6.1 6.3 8.3 6.4 10 6.5 11.9 6.6 15.3 data <- read.table("dataXY.txt", header=T)(1)データの1行目から16行目までのYの値の平均cを求める。
data1 <- data[1:16,] c <- mean(data1$Y) c(2)データの20行目から27行目までのX,Yの値を一次回帰してy=ax+bのaとbを求める。
data2 <- data[20:27,] L <- lm(Y~X, data= data2) L(3)y=cとy=ax+bの交点から閾値(X座標)を求める。
(4)最後に作図する。plot(data) abline(h=c, col=2) abline(L, col=4)(3)をご教示いただけないでしょうか?
また、グラフ中に交点の座標を表示したいと思います。
どうぞよろしくお願いいたします。
(x <- (c-coefficients(L)[1]) / coefficients(L)[2])
points(x, c, pch=19)
text(x, c, sprintf("(%.3f, %.3f)", x, c), pos=4, xpd=TRUE)
(簡単なものは自分で計算する。なんでもかんでも「パッケージにあるはず」とないものねだりはしない方がよいでしょう。投稿も整形しました。どんな風に整形されたか見ておくとよいでしょう。) -- 河童の屁は,河童にあらず,屁である。 2011-09-21 (水) 10:13:16宗二 (2011-09-20 (火) 15:57:37)
maptoolsパッケージの"readShapeLines"関数を使って、GISデータをプロットしようとしているのですが、plot()関数内の引数xlim,ylimを使って範囲指定しても、実際のプロット範囲(緯度経度)がずれてしまいます。
練習用にshapefileライブラリにあるtokyok.zipを使って、以下のプログラムでプロットしたのですが、library(maptools) map <- readShapeLines("tokyok.shp") lon1 <- 138 ### 経度138E-141E lon2 <- 141 lat1 <- 35 ### 緯度35N-35.5N lat2 <- 35.5 par(usr=c(lon1, lon2, lat1, lat2)) plot(map, xlim=c(lon1, lon2), ylim=c(lat1, lat2))作図領域を確認すると
> par("usr") [1] 138.00000 141.00000 33.90625 36.59375というように、(緯度の)指定範囲がずれてしまいます。
これは何が原因なのでしょうか?また、修正方法はあるのでしょうか?
> sessionInfo()
R version 2.13.1 Patched (2011-08-31 r56868)
Platform: x86_64-apple-darwin9.8.0/x86_64 (64-bit)
locale:
[1] ja_JP.UTF-8/ja_JP.UTF-8/C/C/ja_JP.UTF-8/ja_JP.UTF-8
attached base packages:
[1] stats graphics grDevices utils datasets
methods base
other attached packages:
[1] maptools_0.8-9 lattice_0.19-33 sp_0.9-83
foreign_0.8-45
loaded via a namespace (and not attached):
[1] grid_2.13.1 tools_2.13.1
> par("usr")
[1] 137.88000 141.12000 32.23003 38.26997library("maptools")
map <- readShapeLines("tokyok.shp")
lon1 <- 138 ### 経度138E-141E
lon2 <- 141
lat1 <- 35 ### 緯度35N-35.5N
lat2 <- 36 # 35.5だと何もプロットされないので変更してます。
plot(map, xlim = c(lon1, lon2), ylim = c(lat1, lat2), setParUsrBB = TRUE)まあくん (2011-09-20 (火) 11:35:53)
pdfを作成するときに、複数ページのpdfの作成はできないでしょうか?。複数のグラフを1度に作成していますが、現在は各ページごとにpdfファイルを作成し、手動で1つのpdfファイルにしています。
pdf("test.pdf")
plot(1:10)
hist(1:10)
dev.off()tau (2011-09-19 (月) 22:56:26)
いつも、色々と教えていただきましてありがとうございます。
暴露Yが疾患Xの発症に及ぼす影響を調べたいと考えています。
調査の対象はA県、B県の県民です。両県の県民共に暴露を受けており、暴露前後での疾患の発症数が判明しています。
A県では暴露前1ヶ月で100例、暴露後1ヶ月で110例の疾患Xの発症があり、B県ではそれぞれ、80例、120例の発症がありました。A県よりB県で暴露による疾患の発症率が増加したことを証明したいと考えています。
1.A県とB県の人口が分かれば単位人口当たり(/1000人など)の発症率を計算してそれを比較と言うことになるかと思われますが、この場合はどのような検定が用いられるのでしょうか?
2.また、A県とB県の人口が十分に大きいと言うことは分かるものの、詳細な人口が分からない場合、上記の仮説を証明する方法はありますでしょうか?(暴露前後での発症数の比を取ってその比を検定する方法はありますでしょうか)
ご教示いただければ幸いです。
西田 (2011-09-15 (木) 00:08:11)
複数の入力値と制約条件から一つの出力が最適になるように分散共分散行列利用の進化的戦略cmaesというパッケージを使ったプログラムを作ろうと頑張っています。
制約条件である説明変数の下限上限がlower(0,0,0),upper(10,10,10)のように定数であればなんとか動いてくれるようになったのですが、下限上限が変動する、具体的にはx1+x2+x3=10のような条件が追加されたときにきちんと動かず困っています。
現在はcmaes内部で、新しく子集合を作る際にx <- xmean + σ # 親集合の重心周辺に点を取っているのだと思う(σは変数) x <- 10 * ( x / sum(x) ) #xの合計が5なら5で割ってその後10倍 ...##色々計算 よくなっていたら更新 ...##くり返しとして無理やりx1+x2+x3=10を実現しています。が、これできちんと最適化してくれているのか不安です。できればcmaes関数を変更することなく、つまりその外部でx1+x2+x3=10という条件を満たしたいと思っています。 何かいい方法はないでしょうか。(cmaes関数自体にコダワリはないです)
Saito (2011-09-14 (水) 16:58:49)
いつもお世話になっております。ネットや過去ログで調べましたが、見つからなかったので質問させてください。mgcvパッケージの中に、一般化加法モデルが扱えるgamという関数があります。これで回帰式の推定を行うとそれらしい曲線が引けるのですが、その曲線の関数形が知りたいのです。以下にサンプルを示します。> n <- 30 > x <- seq(-n, n) > y <- 0.04*x^3+0.03*x^2+0.02*x+rnorm(2*n+1, 0, 1) # 真の関係 > plot(x, y) > > res <- lm(y ~ x) # 直線回帰 > func <- function(x){ + coef(res)[1] + coef(res)[2]*x # これが直線回帰の回帰式 + } > points(x, func(x=x)) > > res2 <- gam(y~s(x, k=3)) # 加法モデル( # デフォルトは薄板スプライン)による回帰 > points(x, predict(res2), type="l", col=2, lwd=3) # predictによる予測で何故か直線回帰と一致 > coef(res2) #回帰係数が何か出力されてはいるがヘルプを見ても分からない (Intercept) s(x).1 s(x).2 9.309025e+00 -1.199402e-09 3.932092e+02 > > res2 <- gam(y~s(x, k=4)) #節点の数を増やすと曲線となる > points(x, predict(res2), type="l", col=2, lwd=3) > coef(res2) (Intercept) s(x).1 s(x).2 s(x).3 9.309025 -843.683785 -129.386077 1162.199292パッケージを扱うのに問題はありませんが、結局、どのような回帰式になったのかを知りたいのです。ヘルプを見れば書いてあるはずなのですが、私には見つけられませんでした。どこかの本に、回帰式の求め方が書いてある、という情報だけでも構いません。
どなたか、ご教授頂けると幸いです。どうぞよろしくお願い致します。
くま (2011-09-14 (水) 15:50:47)
lmで得られた回帰直線式をグラフに描くのに、ablineを使っていますが、これを使うと、直線は横軸の全領域にわたって引れてしまいます。指定した横軸の範囲にのみその直線を描くにはどうすればよいでしょうか。たとえば、グラフの横軸の範囲のxlim=c(0,100)のときに、回帰直線式を描く範囲をc(20,80)にすることは可能でしょうか。
> set.seed(1)
> x <- seq(0, 100)
> y <- 0.02*x+rnorm(101, 0, 1)
> plot(x, y)
>
> res <- lm(y ~ x)
> func <- function(x){
+ coef(res)[1] + coef(res)[2]*x
+ }
> points(x, func(x=x), type="l", lwd=3, col=2)
> x2 <- seq(0, 80)
> points(x2, func(x=x2), type="l", lwd=3, col=3)
lines(x.range, coefficients(a)[1]+x.range*coefficients(a)[2], col=2)でよいでしょう。二点を結ぶ直線を描くだけです。全然うまい方法ではないですが,間違いなく結果の出る方法です。 -- 河童の屁は,河童にあらず,屁である。 2011-09-14 (水) 18:07:41
(2011-09-08 (木) 13:46:05)
行列の任意の(全ての)列に、欠損値NAがどれだけあるか、割合を算出したいのですが、どうすればよいでしょうか。全ての列に対して、それぞれNAの割合を求めたいです。
例えば、100行10列の行列において、3列目にNAが40個あれば、NAの割合は40% ← この40(または0.4)を求めたい
宜しくお願いします。
> a <- matrix(sample(c(1:9,NA),1000,replace=TRUE),100)3列のNAの割合は次のようにします。
> sum(is.na(a[,3]))/nrow(a) [1] 0.12全ての列に対して同様にしたい場合は、sapply(),apply(),for(),などで一括処理を行います。
> b <- list(a1=a,a2=a) > is.na(b) a1 a2 FALSE FALSE
manabu (2011-09-06 (火) 12:03:05)
本で勉強していて、以下のような問題がありました。
サンプルサイズをn=1、3、5、10と変化させた時に、標本分布の形状がどのように変わるか調べてみましょう。
※母集団分布は標準正規分布N(0,1^2)
答えは以下のように書いてありました。curve(dnorm(x, sd=sqrt(1/10)), -3, 3) curve(dnorm(x, sd=sqrt(1/5)), -3, 3, add=TRUE) curve(dnorm(x, sd=sqrt(1/3)), -3, 3, add=TRUE) curve(dnorm(x, sd=sqrt(1/1)), -3, 3, add=TRUE)母集団の標準偏差や分散が1である時に標本の偏差の二乗の合計が1と言えるのは何故でしょうか?
じゃんぽけ (2011-09-05 (月) 10:41:06)
Rを利用してアンケート分析を実施したいのですが、
SPSSのWEIGHT(重み付け)の様な機能はどうすれば再現できるでしょうか?
> d <- table(x)
b
a 1 2 3
female 13 2 6
male 6 14 9
data.frame 関数を使って
> data.frame(d)
a b Freq
1 female 1 13
2 male 1 6
3 female 2 2
4 male 2 14
5 female 3 6
6 male 3 9
のようなデータフレームにできる。このデータフレームの Freq を重みとして,元のデータを再構成することはできる。そのデータを使えば何でもできる。SPSS の weight も内部ではこんな風にして,重み分のデータの複製を作っているのじゃないかな。てるよ (2011-09-01 (木) 11:59:52)
投稿の前にテキスト整形のルールを読んでも、ほとんど意味がわかりませんでした。お見苦しい投稿になっていましたら、どうぞお許しください。
image() における時系列軸の反転について教えていただけたら幸いです。
"2008/12/19"から"2009/3/24"の96日間に、等間隔でデータを記録したデータがあります。等間隔とは、1時間に1点で1日に24点を記録したデータで、要素数が94日×24点の2304です。このデータは1または0のバイナリデータです。
このデータで1がどのような時間帯に現れるのかを調べるために、縦軸(y軸)に日付、横軸(x軸)に時刻(0時から24時)をとってプロットした図を書くために以下のコマンドを実行しました。st <- c("2008/12/19", "2009/3/24") st2 <- strptime(st, "%Y/%m/%d", tz="") y.days <- seq(st2[1],st2[2], by="1 day") x.hours <- seq(0, 24, by=1) m <- matrix(sample(c(0, 1), 24*length(y.days), rep=T), nrow=24, ncol=length(y.days)) image(x.hours, y.days, m, col=c("white", "black"), yaxt="n", xaxp=c(0,24,4), xlab="hours", ylab=NA) r <- as.POSIXct(round(range(y.days), "days")) axis.POSIXct(2, at=seq(r[1],r[2], by="20 day"), format="%Y/%m/%d", las=2)これでとりあえずグラフは書けたのですが、y軸の時間軸を反転させて、日付の進行を上から下に向かって進むように変更したいと思ったのですが、これがなかなかうまくいきません。
四苦八苦しているうちに、image(..., ylim=c(as.numeric(y.days[96]), as.numeric(y.days[1])))を書くことでプロット自体の反転はできたのですが、今度は
r <- as.POSIXct(round(range(y.days), "days")) axis.POSIXct(2, at=seq(r[1],r[2], by="20 day"), format="%Y/%m/%d", las=2)を実行してもy軸に日付が表示されなくなってしまいました。
y軸の時系列軸を反転させた後も、日付を表示するうまい方法がありましたら教えていただけると幸いです。
image(x.hours, y.days, m, col=c("white", "black"),
yaxt="n", xaxp=c(0,24,4),xlab="hours", ylab=NA,
ylim=c(as.numeric(y.days[96]), as.numeric(y.days[1])))
axis(2,at=as.numeric(y.days[(1:4)*20]),
labels=as.character(y.days[(1:4)*20]), las=2)
labelsのフォーマットにもう少し手を入れる必要がありますが、台風12号のため学生も教職員もすぐに帰宅しろと学内放送がかかっているので、あとはご自分で。BCD (2011-08-29 (月) 15:17:00)
初歩的なことかもしれないので恐縮なのですが、教えてください。
Linuxのスパコンを使ってRのheatmap.2関数で作図をしているのですが 'サイズ 1.4 Gb のベクトルを割り当てることができません'というエラーがでました。調べてみたところ、メモリが足りないということでそれまでに使ってきた変数を削除したりgc()をしてメモリの節約もしたりしました。windowsではメモリの制限を解除するようなコマンドがあるのは分かりましたが、Linuxではどのようにすれば良いのか分かりませんでした。この問題を解決する方法を教えてください
- それって32bitのRではないでしょうか? 本当に64bitのRですか? -- 2011-09-08 (木) 18:23:10
- 確かに64bitのRだと思います -- BCD 2011-09-17 (土) 20:47:22
BCD (2011-08-28 (日) 12:16:08)
>x 1 2 3 4 . . A 20 50 60 29 . . B 49 74 90 38 . . C 57 49 20 39 . . >Y [1] "C" "A" .....のようなデータフレーム'x'(行、列ともに20000行ある)とxの全行名の一部がある順番に従って格納されているベクトル'Y'(C, A,......7000個。要素の順序は重要)が入っているベクトルがあります。このベクトル'Y'を参照して行名から'x’を抽出&並べ替える方法を探しています。
現在、私は以下のスクリプトでこの作業を実行しています。しかし、データが膨大で時間がかかってしまいます。この作業が早くなる方法をご存知の方がいらっしゃればどうか御教示願います。result <- NULL for (i in 1:length(Y)) { result <- rbind(result, x[rownames(x) == Y[i], ]) }
t(sapply(Y, function(a) x[a,]))というようにすることもできますね。x が2000行×2000列, Y の長さが 1000 のテストデータについて速度を比較したところ,この方法は,x が matrix の場合にはあなたの for を使うやりかたより三倍速いけど,同じ内容の data.frame に対しては,あなたのより四倍遅いので要注意。
result <- matrix(0, length(Y), 2000)
for (i in 1:length(Y)) {
result[i,] <- x[rownames(x) == Y[i], ]
}
サイズの小さいデータでいろいろ試して,最速のやり方を見つけてください。-- 河童の屁は,河童にあらず,屁である。? 2011-08-28 (日) 14:49:11#簡単な例
> set.seed(1234)
> n <- 6; m <- 4
> x <- matrix(rnorm(n^2),nrow=n,ncol=n)
> colnames(x) <- seq(n)
> rownames(x) <- sample(LETTERS,n,rep=TRUE)
> B <- rownames(x); uB <- unique(B)
> A <- sample(uB,m,rep=TRUE)
> z <- factor(B,levels=uB)
> zx <- cbind(z,x) # 行ラベルを数値(因子)化し行列の先頭列に加える
> Ax <- zx[factor(A,levels=uB),][,seq(2,n+1)]
> x
1 2 3 4 5 6
A -1.2070657 -0.5747400 -0.77625389 -0.8371717 -0.6937202 1.1022975
U 0.2774292 -0.5466319 0.06445882 2.4158352 -1.4482049 -0.4755931
C 1.0844412 -0.5644520 0.95949406 0.1340882 0.5747557 -0.7094400
N -2.3456977 -0.8900378 -0.11028549 -0.4906859 -1.0236557 -0.5012581
J 0.4291247 -0.4771927 -0.51100951 -0.4405479 -0.0151383 -1.6290935
B 0.5060559 -0.9983864 -0.91119542 0.4595894 -0.9359486 -1.1676193
> B
[1] "A" "U" "C" "N" "J" "B"
> A
[1] "U" "J" "B" "C"
> z
[1] A U C N J B
Levels: A U C N J B
> zx
z 1 2 3 4 5 6
A 1 -1.2070657 -0.5747400 -0.77625389 -0.8371717 -0.6937202 1.1022975
U 2 0.2774292 -0.5466319 0.06445882 2.4158352 -1.4482049 -0.4755931
C 3 1.0844412 -0.5644520 0.95949406 0.1340882 0.5747557 -0.7094400
N 4 -2.3456977 -0.8900378 -0.11028549 -0.4906859 -1.0236557 -0.5012581
J 5 0.4291247 -0.4771927 -0.51100951 -0.4405479 -0.0151383 -1.6290935
B 6 0.5060559 -0.9983864 -0.91119542 0.4595894 -0.9359486 -1.1676193
> Ax
1 2 3 4 5 6
U 0.2774292 -0.5466319 0.06445882 2.4158352 -1.4482049 -0.4755931
J 0.4291247 -0.4771927 -0.51100951 -0.4405479 -0.0151383 -1.6290935
B 0.5060559 -0.9983864 -0.91119542 0.4595894 -0.9359486 -1.1676193
C 1.0844412 -0.5644520 0.95949406 0.1340882 0.5747557 -0.7094400
# n=10000, m=3500の場合の実行速度
> system.time(Ax <- zx[factor(A,levels=uB),][,seq(2,n+1)])
ユーザ システム 経過
1.804 0.156 1.965> x <- matrix(round(runif(12)*100),ncol=4)
> colnames(x) <- as.character(1:4)
> rownames(x) <- LETTERS[1:3]
> x
1 2 3 4
A 93 68 20 5
B 26 20 17 74
C 86 35 59 64
> Y <- sample(LETTERS[1:3],10000,replace=TRUE)
> head(Y)
[1] "C" "C" "A" "C" "C" "B"
> system.time(x[Y,])
ユーザ システム 経過
0.000 0.000 0.001
> system.time({result <- matrix(0, length(Y), 4);
+ for (i in 1:length(Y)) {
+ result[i,] <- x[rownames(x) == Y[i], ]
+ }})
ユーザ システム 経過
0.080 0.000 0.087> system.time(Ax <- x[A,])
ユーザ システム 経過
1.048 0.080 1.132tau (2011-08-27 (土) 10:46:47)
一様性の検定に関して質問致します。
発症が希な疾患を有する患者が一年間に何人入院したかを2008-2011年の4年間で比較したいと考えています。2008年から2011年までそれぞれ1,2,1,7例ずつ入院があり、2011年のみ入院が多い事を証明しようと思います。
カイ二乗検定を用いて下記のように検定を行いましたが、度数が少ないためか「不正確かもしれません」のコメントがでます。> chisq.test(c(1,2,1,7)) Chi-squared test for given probabilities data: c(1, 2, 1, 7) X-squared = 9, df = 3, p-value = 0.02929 警告メッセージ: In chisq.test(c(1, 2, 1, 7)) : カイ自乗近似は不正確かもしれません分割表でカイ二乗検定を行うとき同様、度数が少ない場合は、この検定法を用いない方が良いのでしょうか?
不適当な場合、Fisherの正確検定を用いるのかと考え、調べてみましたが、同方法による一様性の検定の方法がわかりませんでした。
可能であればFisherの正確検定による一様性の検定の方法を、不適当であれば他の検定方法をご教示頂ければ幸いです。
Wombat (2011-08-25 (木) 09:16:29)
私の書き方が不明瞭だったようなのでもう一度投稿させて下さい。以下のデータは5が各質問に対して「強くそう思う」で1が「まったくそう思わない」になる質問紙尺度と呼ばれるものになります。ID class Q1 Q2 Q3 Q4 Q5 Q6 Q7 Q11 Q15 1 1 a 5 4 4 4 4 2 4 4 5 2 2 a 4 4 3 3 4 4 3 4 2 3 3 a 4 4 3 5 4 4 3 4 5 4 4 a 4 4 1 3 3 4 5 3 5 5 5 a 5 4 1 5 5 3 2 4 4 6 6 a 4 4 4 4 4 2 1 2 1 -- 省略-- 94 94 d 4 4 3 4 4 2 3 4 4 95 95 d 4 3 4 3 3 4 4 4 4 96 96 d 4 4 1 4 3 3 2 4 3 97 97 d 4 5 2 5 5 5 4 4 4 98 98 d 5 4 4 4 3 4 5 4 4 99 99 d 4 4 2 4 4 3 3 4 4 100 100 d 4 4 2 3 4 4 5 4 4 101 101 d 5 4 2 4 4 5 4 4 1 102 102 d 5 4 4 4 3 5 2 4 2これを各クラスごとに項目別にsapply(d01[,3:11],function(x) table(d01$class,x))でクロス集計を行なう(河童さんのおかげです。)と以下のようになります。
$Q1 x 3 4 5 a 2 15 8 b 0 21 6 c 2 19 4 d 0 14 11 $Q2 x 2 3 4 5 a 1 5 19 0 b 1 6 20 0 c 0 8 14 3 d 0 4 17 4 -- 以下省略--これにより、Q1はclass aは強くそう思うと答えた回答者が8人いることがわかりますが、クラスの数が同じではないので、8人ではなくて、32%というふうにパーセントで報告するのが普通になると思うのですが、いちいち手で計算するのは効率が悪いので、この8という頻度を表す数字をパーセントになおす方法をご存知でしたら教えてください。よろしくお願いします。
sapply(d01[,3:11],function(x) { tbl <- table(d01$class,x); print(round(tbl/rowSums(tbl)*100,1)) }))
正しい答えを出せればよいのであって,うまく書く必要はないのです。for でも何でも使って,プログラムを書くのもまっとうな方法です。Wombat (2011-08-24 (水) 19:26:14)
河童さんのおかげでクラスと項目別のクロス集計ができるようになりましたが、論文に書くときにはクラス間で人数の差があるので、パーセントに変換して書きます。1つ1つ計算してもいいのですが、もし、その値を簡単にパーセント値に変換する方法がありましたら教えてください。よろしくお願いします。OSはWindows XP, RのVersionは2.13.1です。
$Q1x 3 4 5 a 2 15 8 b 0 19 3$Q2
x 2 3 4 a 1 5 19 b 1 5 16
sapply(split(d, d[,2]),
function(x) colSums(x[3:9])/nrow(x)*100)
sapply(split(d, d[,2]), function(x) colMeans(x[3:9])*100)
sapply(d[3:9], function(x) tapply(x, d$class, mean)*100)
by(d[3:9], d[,2], colMeans)
一番目のが一番直感的に分かりやすいかな。Wombat (2011-08-24 (水) 01:52:47)
次のようなデータがあります。これはある英語教授法を4つのクラスでおこなって、英語力のどの部分が伸びたかを学生に尋ねたアンケートの結果を表したものです。複数回答項目ですので、各項目ごとに変数を準備して、選択したら1、しなければ0で処理しています。このデータの集計でクラスごとに各項目の和を一度に求めるにはどのようにしたら良いでしょうか。このクラスごとにどの項目が多いかを調べらばいいので、クロス集計でなく、単に和を求めるだけで良いと思います。sapply(dat01[1:25,3:9],sum)でclass aにおける各項目の集計は求めることができますが、何度も[ ]の中の行の値を変えるのはあまり効率の良い方法とは言えないと思います(4回くらいやれよと言われそうですが)ので、よろしくお願いします。OSはWindo coef(res2)(Intercept) s(x).1 s(x).2 s(x).3 9.309025 -843.683785 -129.386077 1162.199292パッケージを扱うのに問題はありませんが、結局、どのような回帰式になったのかを知りたいのです。ヘルプを見れば書いてあるはずなのですが、私には見つけられませんでした。どこかの本に、回帰式の求め方が書いてある、という情報だけでも構いません。
どなたか、ご教授頂けると幸いです。どうぞよろしくお願い致します。
ID class R Wr L Spk Vocab Gr Spd
1 a 0 0 0 0 0 0 0
2 a 0 0 0 0 0 0 0
3 a 0 0 0 0 0 0 1
4 a 0 0 0 0 0 0 0
5 a 1 0 0 0 0 1 1
: 省略
100 d 1 0 0 0 1 0 0
101 d 1 0 0 0 1 0 0
102 d 0 0 0 0 0 0 0
103 d 1 0 0 0 1 0 0sapply(split(d, d[,2]), function(x) colSums(x[3:9]))または,
by(d[3:9], d[,2], colSums)または
sapply(d[3:9], function(x) tapply(x, d$class, sum))お好きな方を。 -- 河童の屁は,河童にあらず,屁である。 2011-08-24 (水) 08:21:32
BCD (2011-08-22 (月) 19:09:35)
plot関数で折れ線グラフを作る時にxlimでグラフの中のx軸の範囲を決めることができますが、その中のある領域だけそのグラフ内で広げるようにすることはできますか?
例えば xlim = C(0, 10000) のグラフの場合、グラフの左端が0で右端が10000になり中心が5000になります。これを操作してグラフのx軸の左端から1/4までが0~4500の範囲、1/4から3/4までが4500~5500の範囲、3/4から右端までが5500~10000の範囲と部分的にx軸を広げたいのです。
set.seed(123456789)
n <- 500
y <- rnorm(n)
x <- sample(10000, n, replace=TRUE)
for (i in 1:n) {
xi <- x[i]
if (xi < 4500) x[i] <- xi/4500
else if (xi < 5500) x[i] <- (xi-4500)/500+1
else x[i] <- (xi-5500)/4500+3
}
x <- x*2500
plot(x, y, xaxt="n")
axis(1, at=0:4*2500,
labels=c(0, 4500, 5000, 5500, 10000))
BCD (2011-08-22 (月) 13:13:44)
read.tableでファイルを読みこむと列ラベルの先頭が数字の場合、その前にXが付加されてしまいます。Xが付加されないようにするにはどうしたらよいですか?(以前、Rjpwikiに投稿があったと思いますが、見つけ出すことができませんでした)ex) 0002jh 0043sss 1 2 4のようなtxtデータをread.tableで読み込むと
X0002jh X0043sss 1 2 4となります。これを解消したいです。
square (2011-08-21 (日) 17:47:20)
下のような複素数の積分はできませんか?
f=function(x) exp(i*x)
integrate(f,0,1)
integrateでは実数限定のようですが。
> f <- function(x) exp(1i*x)
> integrate(function(x) Re(f(x)),0,1)$value+
(1i)*integrate(function(x) Im(f(x)),0,1)$value
[1] 0.841471+0.4596977iアレックス (2011-08-17 (水) 15:58:26)
R 2.13.1を利用しています。要素1,要素2,要素3 りんご,バナナ,みかん バナナ,なし, パイン,もも,みかんという中身のファイル(data.txt)を以下のコマンドで読み込み
data1 <- read.table("C:/lab/kankou.txt",sep=",", header=TRUE) 要素1 要素2 要素3 1 りんご バナナ みかん 2 バナナ なし 3 パイン もも みかんというdata.frame形式のデータを読み込むことはできたのですが、これをtransactionに変換する際に
data.tran <- as(data1, "transactions") data.frame <- as(data.tran, "data.frame")としてdata.frameを開いてみると
1{要素1=りんご,要素2=バナナ,要素3=みかん} 2{要素1=バナナ,要素2=なし,要素3=} 3{要素1=パイン,要素2=もも,要素3=みかん}となってしまいます。これを
1{りんご,バナナ,みかん} 2{バナナ,なし,} 3{パイン,もも,みかん}となるようなtransactionを生成する方法はないでしょうか。
data.frame[,2] <- gsub("要素[1-3]=", "", data.frame[,2])
などとすればよいのでは? -- 河童の屁は,河童にあらず,屁である。 2011-08-17 (水) 16:36:261{りんご,バナナ,みかん}
2{バナナ,なし,}
3{パイン,もも,みかん}
という内容のテキストファイルが欲しいんですか?(たぶん違うのだろうけどね) -- 河童の屁は,河童にあらず,屁である。 2011-08-19 (金) 20:28:34z (2011-08-12 (金) 18:52:21)
Plm Pacakgeを用い、下記の分析を行いたいのですが、うまくインストールできません。Rは2.13.1を利用しており、スペックは満たしているようです。
このぱっページをインストールする方法か、類似の分析方法をご教示頂けましたら、幸いです。自身でも、plmを使わずに分析を行うコードを色々と試行錯誤しているのですが、なかなか解決できていません。宜しくお願い致します。
the Fixed effects model (within),
• the pooling model (pooling),
• the first-difference model (fd),
• the between model (between),
• the error components model (random).
> library(plm)
以下にエラー library(plm) :
'plm' という名前のパッケージはありません
> install.packages(plm)
以下にエラー install.packages(plm) :
オブジェクト 'plm' がありません
スペックと記載しましたのは、下段の内容についてです。
私のPCはWindows XP(32bit)でR2.13.1を使用しています。> utils:::menuInstallLocal()
パッケージ 'plm' は無事に開封され、
MD5 サムもチェックされました
> library(plm)
要求されたパッケージ bdsmatrix をロード中です
エラー: パッケージ 'bdsmatrix' を
ロードできませんでした
追加情報: 警告メッセージ:
1: パッケージ 'plm' はバージョン 2.13.1 の R の下で
造られました
2: In library(pkg, character.only = TRUE,
logical.return = TRUE, lib.loc = lib.loc) :
'bdsmatrix' という名前のパッケージはありません
次に、'bdsmatrix'を同様にWebsiteからダウンロードし、
参照) http://cran.r-project.org/web/packages/bdsmatrix/index.htmlutils:::menuInstallLocal()
以下にエラー gzfile(file, "r") :
コネクションを開くことができません
追加情報: 警告メッセージ:
In gzfile(file, "r") :
圧縮されたファイル 'bdsmatrix_1.0[1]/DE木SCRIPTION'
を開くことができません,
理由は 'No such file or directory' です
> utils:::menuInstallLocal()
以下にエラー gzfile(file, "r") :
コネクションを開くことができません
追加情報: 警告メッセージ:
In gzfile(file, "r") :
圧縮されたファイル 'bdsmatrix_1.0[1]/DESCRIPTION' を
開くことができません,
理由は 'No such file or directory' です
尚、他の方法、とは、「解説書等を読み漁ってFEやREのプログラミング方法を検討しているが、それらも最終的にはplmパッケージのコードが必要になってなかなかうまくいきません。」という意味です。 -- z 2011-08-15 (月) 11:56:51
-- z 2011-08-15 (月) 11:56:51酔鯨 (2011-08-12 (金) 17:05:34)
使用しているOS=Windows XP Rのバージョン=2.10.0
独立変数が、定量データと定性データが存在する混合モデルを重回帰分析をしました。定性データは(0,1)の2値データです。この場合は、重回帰データとして同じ扱いが出来ると市販本に書かれていたので、AIC最小モデルを変数減増法と群馬大青木先生の総当り法で求めました。結果は同じになりました。定性データは、最小モデルには残りませんでした。
単回帰の結果では、定性データでモデルを別々にしなければならないという結果が出たのですが、重回帰の方法に誤りは無いでしょうか?
ryuji (2011-08-11 (木) 23:37:05)
現在、ある昆虫の体の一部から体長を推定するために、これまで採集した標本をもとにRMA回帰を求めました。そこまではRを使ってできたのですが、実はこの昆虫の死骸が多く見つかるため、一体これらの死骸の体長がどのくらい大きさであったかを推定したいと思っております。そこでRMA回帰に代入して体長を求める前に、予め予測区間内に入るかどうか、グラフで表示しておきたいのですが、その方法が分かりません。超初心者で申し訳ないのですが、ご教授願えないでしょうか?よろしくお願いいたします。
tavia (2011-08-11 (木) 20:46:50)
お世話になります。
Rを使って、クラスター分析をしようとしています。
データの読み込みまではできるのですが、その後
d <- as.dist(data)
を実行すると、以下にエラー as.vector(x, mode) : cannot coerce type 'closure' to vector of type 'any'というメッセージが出ます。
このようなメッセージが出たら、どういうところをチェックすればよいでしょうか。
教えていただければ幸いです。
Rのバージョンは2.10.1
OSはWindows Vista Business S.P.2
です。
よろしくお願いいたします。
nob (2011-08-11 (木) 11:31:15)
Rそのものは超初心者ですが、結構前から使っています。
Plamo Linux 4.7.3でのつまずきです。
いつもパッケージをCRANからダウンロードしてconfigure、make、make install でシステムをインストールしています。が、plamo Linuxのバージョンを4.7.3にしたら、
checking for xmkmf... /usr/X11R7/bin/xmkmf
configure: WARNING: I could not determine FPICFLAGS.
configure: error: See the file doc/html/R-admin.html for more information.
で止まってしまいます。2.13ばかりでなく古いバージョンも同様です。こちらのHPを見たらconfigureで失敗した場合不足しているものをconfig.logで確認して再度configureせよとのことだったのですが見ても何が不足なのかよくわかりません。
どなたか同じような件で解決された方がおりましたら、どのように解決されたか教えていただけないでしょうか?
よろしくお願い致します。
Wombat (2011-08-10 (水) 12:18:35)
R超初心者です。御回答よろしくお願いします。以下のようなデータがあります。ID class Q1 Q2 Q3 Q4 Q5 Q6 Q7 Q11 Q15 1 1 a 5 4 4 4 4 2 4 4 5 2 2 a 4 4 3 3 4 4 3 4 2 3 3 a 4 4 3 5 4 4 3 4 5 4 4 a 4 4 1 3 3 4 5 3 5 5 5 a 5 4 1 5 5 3 2 4 4 6 6 a 4 4 4 4 4 2 1 2 1 7 7 a 5 4 2 2 4 3 4 4 2 8 8 a 4 4 2 5 4 3 3 4 3 9 9 a 4 4 2 3 3 4 3 5 5 10 10 a 5 4 4 4 5 3 3 4 4 11 11 a 4 3 2 3 2 3 1 3 3 12 12 a 3 2 3 4 3 5 4 3 2 13 13 a 5 4 2 4 4 4 5 4 4 14 14 a 3 3 4 3 4 3 1 3 2 15 15 a 4 4 3 4 4 4 4 4 3 16 16 a 5 3 2 4 4 3 3 5 5 17 17 a 4 4 2 4 3 3 4 3 3 18 18 a 4 4 2 2 4 3 2 3 4 19 19 a 5 4 4 4 4 2 5 4 5 20 20 a 4 4 3 4 4 5 4 4 5 21 21 a 4 4 1 4 3 4 4 4 2 22 22 a 4 3 2 3 3 5 3 3 3 23 23 a 4 3 3 3 4 3 3 4 3 24 24 a 5 4 1 4 4 4 4 4 2 25 25 a 4 4 3 4 4 5 4 3 3 26 26 b 4 4 1 4 4 5 3 5 5 27 27 b 5 4 3 4 3 3 3 4 3 28 28 b 4 3 2 4 3 5 4 3 2 29 29 b 4 4 2 3 3 4 4 4 2 30 30 b 4 4 2 3 4 4 3 4 3 31 31 b 4 4 4 3 4 4 2 4 4 32 32 b 4 4 4 4 3 4 3 4 3 33 33 b 4 4 4 3 3 4 4 3 3 34 34 b 4 4 5 3 3 4 3 3 3 35 35 b 5 4 3 4 4 3 3 4 4 36 36 b 4 3 1 3 3 4 5 3 1 37 37 b 4 4 5 4 3 4 4 4 2 38 38 b 4 4 5 2 2 4 2 3 3 39 39 b 4 4 4 4 3 4 2 4 3 40 40 b 4 4 3 4 4 3 3 4 4 41 41 b 4 3 2 3 3 4 4 4 2 42 42 b 5 3 3 4 3 4 5 3 2 43 43 b 4 3 2 3 4 5 5 4 5 44 44 b 4 2 3 3 3 4 3 3 3 45 45 b 4 4 3 5 3 3 5 4 1 46 46 b 4 4 3 3 5 4 5 4 4 47 47 b 4 4 3 4 3 4 4 4 2Q1からQ11の数値はリッカートスケール(5が強く思う〜1全くそう思わない)です。この表ではグループ(Class)はaとbしかありませんが、実際のデータではdまであります。例えば、aクラスにおけるQ1における数値の頻度は> table(dat01[1:25,3])で求めることができことは「Rによるやさしい統計書」インターネットでわかりました。しかし、この方法では時間がかかってしまうので、これを一度にグループことの各質問項目におけるリッカートスケールの数値の頻度を求めるにはどのような関数を使えば良いのか教えてください。
> dat01 <- data.frame(ID=1:47,class=c(rep('a',25),rep('b',22)),
Q1=sample.int(5,size=47,replace=TRUE),
Q2=sample.int(5,size=47,replace=TRUE))
> apply(dat01[,3:4],2,function(x){tapply(x,dat01$class,table)})
$Q1
$Q1$a
1 2 3 4 5
3 10 1 5 6
$Q1$b
1 2 3 4 5
4 7 3 4 4
$Q2
$Q2$a
1 2 3 4 5
6 4 2 7 6
$Q2$b
1 2 3 4 5
2 5 4 4 7
Q1とQ2しかサンプルを作っていませんが、実際に使うときには適宜読み替えてください。> d <- read.table("data01.dat", header=TRUE)
> sapply(d[3:11], function (x) table(d$class, x))
$Q1
x
3 4 5
a 2 15 8
b 0 19 3
$Q2
x
2 3 4
a 1 5 19
b 1 5 16
: 以下略ひろ (2011-08-09 (火) 18:14:41)
grepコマンドの実行で,検索元(パラメータ1)の単語の中に「ー」が含まれていると下記通りエラーメッセージが表示されて失敗だと失敗します.他の「日」「月」「年」といった言葉であれば問題なく検索できました.
検索元の単語に「ー」が含まれていても正しく検索できる方法はございますでしょうか?grep("リー","あ","い","う","え")
以下にエラー grep("リー", "あ", "い", "う", "え") : "リー" は不正な正則表現です, 理由は 'Missing ']''
> grep("リー", c("スリー","い","リード","え"))
[1] 1 3nappa (2011-08-04 (木) 12:50:45)
下記のように日本語があると「不正なマルチバイト文字があります」と表示され、うまく動作しません。コメントなのですがダメです。以前は大丈夫だったのですが、エンコードの仕様が変わったためでしょうか?
対策がありましたら、教えてください。#サンプル x <- 2 x R Version: 2.13.1 RコマンダーVersion: 1.6-4
しげ (2011-08-03 (水) 16:04:22)
パッケージ"adapt"をインストースしようとするとIn getDependencies(pkgs, dependencies, available, lib) : package ‘adapt’ is not available (for R version 2.13.1と警告メッセージが出てインストールできないのですが、どのようにしたらインストールできるようになりますか?使用環境は
> sessionInfo() R version 2.13.1 (2011-07-08) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base other attached packages: [1] rgl_0.92.798 loaded via a namespace (and not attached): [1] tools_2.13.1です。かなり初歩的なことだと思いますが、教えてもらえると嬉しいです。よろしくお願いします!
ひろ (2011-07-29 (金) 16:01:26)
小数点(ex 4.564e-05, 4.234e-02 etc.)のデータセットはrankでソートできないのでしょうか?これをソートすると正しく順序づけられなく困っております.並び替える方法はございますでしょうか?お知恵をお貸いただけますでしょうか?
■ コマンド
res.sorted <- res[rank(res$D),]
データセットresの中のD(eviance)の列は小数点データで,これをrankコマンドで昇順に並べた添え字リストを得て,resを並び替えた結果をres.sortedに入れようとしています.
どぜう (2011-07-26 (火) 10:41:16)
お世話になっております。過去ログにも無いようですので、質問させてください。
とあるスペクトルデータがあり、ピーク面積を求めたいのです。前処理でバックグランドデータを引いたデータを用意するまではよかったのですが、ピークのあるチャンネル番号や、そのピークが囲む面積(ピクセル数?)の求め方が分かりません。
よろしくお願い申し上げます。
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください」と書いてあることに注意を払うべきですね。 -- 河童の屁は,河童にあらず,屁である。 2011-07-27 (水) 16:46:42
を読んでください」と書いてあることに注意を払うべきですね。 -- 河童の屁は,河童にあらず,屁である。 2011-07-27 (水) 16:46:42
x <- 0:4*0.25 # 等間隔であるとする
y <- c(0, 4, 10, 7, 2)
plot(x, y, type="l", yaxs="i")
polygon(c(x, rev(x)), c(y, rep(0, 5)),
col="#00FF0040")
abline(h=y, lty=3)
segments(x, y, x, rep(0, 5))
両端が三角形でも,三角形は上底(下底_が0の台形なので,面積は
(y[1]+y[2])*0.25/2 + (y[2]+y[3])*0.25/2 + (y[3]+y[4])*0.25/2 + (y[4]+y[5])*0.25/2これをまとめると,y 座標値の和の 2 倍から両端の y 座標値を引いて台形の高さ(x座標値の間隔)を掛けて 2 で割るのと同じ
(2*sum(y)-y[1]-y[5])*0.25/2答えは 5.5 になる。 -- 河童の屁は,河童にあらず,屁である。 2011-07-27 (水) 19:04:43
> delta.x <- x[2]-x[1] # x 座標の間隔
> sum(y)*delta.x
[1] 3.803799
> system.time(for (i in 1:10000) sum(y)*delta.x)
ユーザ システム 経過
0.068 0.001 0.069
> polygon <- cbind(x, y) # 開曲線
> areapl(polygon)
[1] 3.803799
> system.time(for (i in 1:10000) areapl(polygon))
ユーザ システム 経過
4.943 0.211 4.643
> polygon2 <- cbind(c(x, 0), c(y, 0)) # 閉曲線
> areapl(polygon2)
[1] 3.803799
> system.time(for (i in 1:10000) areapl(polygon2))
ユーザ システム 経過
2.586 0.123 2.337Tau (2011-07-21 (木) 22:41:07)
統計手法の選択についてご教示ください。
ある疾患が、1週目に4例、2週目に5例、3週目に6例、4週目に17例生じたとします。1週目から3週目までの疾患発症数の平均と4週目の疾患発症数の差を検定する場合は、t.testあるいはノンパラメトリックな手法(U.testなど)を用いて良いものでしょうか?(つまり、比較したい2群があって一方は複数のcaseがあり平均値が算出されるのに、他方は一つのcaseしかない場合です)
ご教示いただければ幸いです。
> chisq.test(c(4, 5, 6, 17)) Chi-squared test for given probabilities data: c(4, 5, 6, 17) X-squared = 13.75, df = 3, p-value = 0.003266
sui? (2011-07-14 (木) 15:45:22)
propensity scoreを用いたマッチングの手法について質問させて頂きます。
Rはver.2.13.1、Matching 4.7-14を用いています。
datasetはMaching package内の「lalonde」を用いています。
以下の通り、propensity scoreを算出します。> library(Matching) > data(lalonde) > Y <- lalonde$re78 > Tr <- lalonde$treat > glm1 <- glm(Tr~age+educ+black+hisp+married+nodegr+ re74+re75, family=binomial, data=lalonde)glm1$fittedがpropensity scoreです。 これを用いてMatchingを行います。
> rr1 <- Match(Y = Y, Tr = Tr, X = glm1$fitted) > summary(rr1) Estimate... 2624.3 AI SE...... 802.19 T-stat..... 3.2714 p.val...... 0.0010702 Original number of observations.............. 445 Original number of treated obs............... 185 Matched number of observations............... 185 Matched number of observations (unweighted). 344サマリーを見ると185例でMatchingが行われたようですが、Matchig後のデータセットを表示したいと考えております。お分かりの方がいらっしゃいましたら教示頂けませんでしょうか?
よろしくお願い申し上げます。
内藤 (2011-07-14 (木) 10:28:10)
以下の行列データがあります(本物はもっとデータが大きい)[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [1,] 0 0 0 0 0 0 0 0 0 0 [2,] 0 0 0 0 0 0 0 0 0 0 [3,] 0 0 0 1 0 0 0 0 0 0 [4,] 0 0 0 999 0 0 0 0 0 0 [5,] 0 0 0 999 0 0 0 0 0 0 [6,] 0 1 0 999 0 0 0 0 0 0 [7,] 0 1 0 999 0 0 0 0 0 0 [8,] 0 1 0 999 0 0 0 0 0 0 [9,] 0 1 0 999 0 0 0 0 0 0 [10,] 0 999 0 999 0 0 0 0 0 0 [11,] 0 999 0 999 0 0 0 0 0 0 [12,] 1 999 0 999 0 0 0 0 0 0 [13,] 1 999 0 999 0 0 0 0 0 0999以降の列データを2を8個,3を10個というようにプログラムで変更したいのですが,上手くいきません。
初歩的な質問で申し訳ありませんが,上手いやり方を教えてください。
x[4:13, 4] <- rep(2:4, c(3, 5, 2)) # x[4:13, 4] <- c(rep(2, 3), rep(3, 5), rep(4, 2)) と同じのようにしますけど...
# 乱数発生数
k <- 100
# 解析年数
n <- 50
###### データ入力(塩化物イオン拡散予測) ########
C0 <- rweibull (k, 2.600, 9.631)
D <- rlnorm (k, -17.454, 0.995)
d <- 6.85
###### データ入力(腐食環境) ####################
a <- 1500
c <- 0.005
alf <- 220
fai <- 1.3
Fe <- 7850
################################################
# 誤差関数の設定
erf <- function(x) 2 * pnorm(x * sqrt(2)) - 1
# 塩化物配列の設定
C <- matrix(0, n, k)
###### 塩化物拡散予測 ##########################
for (i in 1:n) {
for (j in 1:k) {
C[i,j] <- C0[j]*(1-erf(d/(2*sqrt(D[j]*
i*31536000))))
}
}
###### 鉄筋腐食予測(年数予測) #################
t1 <- round((a/alf)*log(0.01/((4*a*c)/(fai*Fe))))
t2 <- round((a/alf)*log(0.05/((4*a*c)/(fai*Fe))))
t3 <- round((a/alf)*log(0.20/((4*a*c)/(fai*Fe))))
# 劣化度の配列設定
CORR<- matrix(0, n, k)
###### 劣化度設定 ##############################
for (j in 1:k) {
for (i in 1:n) {
if (C[i, j] < 1.2) CORR[i, j] <- 0
else if(C[i, j] < 2.0) CORR[i, j] <- 1
else CORR[i, j] <- 999
}
}
################################################set.seed(123456789) # ★(テスト用)
# 乱数発生数
k <- 100
# 解析年数
n <- 50
###### データ入力(塩化物イオン拡散予測) ########
C0 <- rweibull (k, 2.600, 9.631)
D <- rlnorm (k, -17.454, 0.995)
d <- 6.85
###### データ入力(腐食環境) ####################
a <- 1500
c <- 0.005
alf <- 220
fai <- 1.3
Fe <- 7850
################################################
# 誤差関数の設定
erf <- function(x) 2 * pnorm(x * sqrt(2)) - 1
# 塩化物配列の設定
C <- matrix(0, n, k)
###### 塩化物拡散予測 ##########################
for (i in 1:n) {
for (j in 1:k) {
C[i,j] <- C0[j]*(1-erf(d/(2*sqrt(D[j]*i*
31536000))))
}
}
###### 鉄筋腐食予測(年数予測) #################
t1 <- round((a/alf)*log(0.01/((4*a*c)/(fai*Fe))))
t2 <- round((a/alf)*log(0.05/((4*a*c)/(fai*Fe))))
t3 <- round((a/alf)*log(0.20/((4*a*c)/(fai*Fe))))
t12 <- t1+t2 # ★
t123 <- t12+t3 # ★
# 劣化度の配列設定
CORR<- matrix(0, n, k)
###### 劣化度設定 ##############################
for (j in 1:k) {
nn <- 0 # ★
for (i in 1:n) {
if (C[i, j] < 1.2) CORR[i, j] <- 0
else if(C[i, j] < 2.0) CORR[i, j] <- 1
else { # ★ ここから
nn <- nn+1
CORR[i, j] <- ifelse(nn <= t1, 2,
ifelse(nn <= t12, 3,
ifelse(nn <= t123, 4, 5)))
} # ★ ここまで
}
}
################################################C <- d/(2*sqrt(D*t(row(C)*31536000))) C <- t(C0*(1-erf(C)))
result <- t(apply(CORR, 2, function(x) {
a <- table(x)
b <- numeric(6)
b[as.integer(names(a))+1] <- a
return(b)
}))result2 <- t(apply(result, 1, cumsum)) # 面グラフを描くために数値を積み上げ
result2 <- cbind(rep(0, nrow(result2)), result2) # 基準線(0)を追加
plot(c(1, nrow(result2)), range(result2), type="n") # plot 枠
for (i in 2:ncol(result2)) { 隣り合う2列が構成する多角形を塗りつぶす
polygon(c(1:nrow(result2), nrow(result2):1), c(result2[, i-1], rev(result2[, i])), col=i)
}
result2 <- t(apply(result, 1, cumsum))
result2 <- cbind(rep(0, nrow(result2)), result2)
color <- gray(seq(1.0, 0.5, length=7))
plot(c(1, nrow(result2)), range(result2),
xlab="経過年数(年)",
ylab="各劣化状態の面積割合(%)", type="n")
for (i in 2:ncol(result2)) {
polygon(c(1:nrow(result2), nrow(result2):1),
c(result2[, i-1],
rev(result2[, i])), col=color[i])
}
legend(2, 40, c("潜伏期", "進展期", "加速期",
"劣化期", "耐力比1.0以下"),
col=1, fill=color, cex=0.7)
順序を耐力比1.0以上,劣化期,加速期,進展期,潜伏期という順に書く場合はどうすればいいでしょうか?Ryohei (2011-07-12 (火) 22:12:18)
以下のxxというデータセットをID別にTIME2対VAR1とTIME2対VAR2の折れ線グラフを重ね描きしたいと考えております。#1に示した図をアウトプットのイメージとして、#2の2種類のグラフを1枚で重ね描きをして変数VAR1とVAR2に対する時間(TIME2)の推移を同時に表現したいと考えております。xyplotではどのようにプログラムを書けばよいのでしょうか?
今回は便宜上2行2列のグラフでしたが、実際には4行4列、5行5列といったグラフを描こうと考えております。
xyplotの特長である、軸ラベルが両端・最上下段のみに表示されすっきりする、IDごとのグラフエリアが大きくなって見やすくなるといった点を活かしたいと思っております。
どうぞよろしくお願いいたします。#1 N <- 4 ; TIME <- seq(6) TIME2 <- rep(TIME, N) ID <- rep(seq(N), each=length(TIME)) VAR1 <- rep(seq(3), length(TIME)/3*N) VAR2 <- round(runif(N*length(TIME), min=1, max=9)) (xx <- data.frame(ID, TIME2, VAR1, VAR2)) layout(matrix(1:4, byrow=TRUE, ncol=2)) old <- par(mar=c(2, 2.7, 1.3, 1), mgp=c(1.6, 0.7, 0)) for (i in 1:N) { plot(xx[xx[,"ID"]==i, "TIME2"], xx[xx[, "ID"]==i, "VAR1"], xlim=c(0, 7), ylim=c(0, 10), xlab="", ylab="", main=sprintf("ID%02d", i), col="red", type="b") points(xx[xx[, "ID"]==i, "TIME2"], xx[xx[, "ID"]==i, "VAR2"], xlim=c(0, 7), ylim=c(0, 10), col="blue", type="b") } par(old) layout(1) #2 #library(lattice) #xyplot(VAR1 ~ TIME2 | ID, data=xx, ylim=c(0, 10), type="b", col="red") #xyplot(VAR2 ~ TIME2 | ID, data=xx, ylim=c(0, 10), type="b", col="blue")
sakura (2011-07-09 (土) 07:57:43)
PCのシステムは、
Microsoft Windows XP
Professional
Version 2002
Service Pack 3
です。
R version 2.130(2011-04-013)で、package:rattleをロードしてdeta mining を学ぼうとしているのですが、パスが通らず?次のエラーが出てしまいます。
どう対処したら良いのか、分かりません。ご教示頂ければ、幸いです。お願いします。Learn more about GTK+ at http://www.gtk.org If the package still does not load, please ensure that GTK+ is installed and that it is on your PATH environment variable IN ANY CASE, RESTART R BEFORE TRYING TO LOAD THE PACKAGE AGAIN 以下にエラー as.GType(type) : Cannot convert RGtkBuilder to GType 追加情報: 警告メッセージ: In unzip(path, exdir = .windows_gtk_path) : zip ファイルから抽出中に書き込みエラーが生じました
Ackin (2011-07-01 (金) 14:02:41)
Office2007、WindowsXPの職場PCにRAndFriendsSetup2130V3.1-15-1.exeをインストールしてRexcelを学び始めました。
インストール時は管理者権限で行いましたが、使用時に権限はありません。C:\Program Files\RExcelにインストール出来たようです。
職場PCはマイドキュメントをサーバにあるP:としてあり、getwd()すると"P:/"となります。
【症状】
ExcelメニューからRexcel>RthroughExcelWorksheetsとしたいのですが見当たりません。
書籍「ExcelでR自由自在」P.34図3.1によるとDemo WorksheetsとAbout Rexcelの間にあるようです。
C: P: を検索しましたがBookFilesTOC.xlsmは見当たりません。
どこからかBookFilesTOC.xlsmを探してきて、パスを通さなければ使えないでしょうか?
library(RthroughExcelWorkbooksInstaller) installRthroughExcel()を実行します.この場合もCドライブへのアクセス権が必要になりますけど. -- ishida 2011-07-02 (土) 17:33:44
ランゲルハンス (2011-06-22 (水) 14:29:19)
いつも掲示板を参考にしております。
三次元散布図を描きたいと思います。
下記のプログラムでは、x、y、z軸の目盛は違っても長さが同じで立方体の中に各点が散布されます。
それぞれの軸を実際の長さの比(10:20:5 =2:4:1)にした直方体の中に点を散布する方法をご教示いただけないでしょうか?
見た目の錯覚を防ぎたいと思います。
よろしくお願いいたします。library(scatterplot3d) x <- runif(100, 0, 10) y <- runif(100, 0, 20) z <- runif(100, 0, 5) scatterplot3d(x, y, z, pch=20)
buta (2011-06-21 (火) 13:07:20)
ヒストグラムの横軸に度数、縦軸に階級を取るにはどのようにすればよいでしょうか。よろしくお願いします。
old <- par(mar=c(3, 7, 1, 2), las=3, xpd=TRUE, crt=90)
a <- hist(rnorm(1000), xlab="", main="")
mtext("Rotated Histogram", side=2, line=5, cex=1.5)
text(max(a$breaks)+15*strheight("H", units="figure"),
-strwidth("H", units="figure")*10, "Value", pos=1)
par(old)
hist2 <- function(x, breaks = "Sturges",
xlab="Counts", ylab="Value",
main="Horizontal Histogram")
{
a <- hist(x, breaks=breaks, plot=FALSE)
class <- length(a$counts)
plot(range(a$count), range(a$breaks),
type="n", xlab=xlab,
ylab=ylab, main=main)
for (i in 1:class) {
rect(0, a$breaks[i], a$counts[i],
a$breaks[i+1])
}
}poro (2011-06-18 (土) 04:00:33)
str <- "" for (i in c("a", "b", "c")) { str <- paste(str, i, sep="") }としていますが、ベクトルをpasteコマンドにそのまま渡して文字を連結してくれないかと期待するのですができないものでしょうか?
石ころ太郎 (2011-06-15 (水) 19:19:09)
お世話になります。hist(c(0.1*1:10), probability=TRUE)を実行したときにdensityが0.1となるべきところが、1.0となってしまいます。いろいろ確認したところ、breaksの閾値が小数となるときにあらわれる現象と見受けられます。
解決法はございますでしょうか?
> x # データ
[1] 0.1 0.2 0.2 0.3 0.3 0.3 0.4 0.4 0.4 0.4
0.5 0.5 0.5 0.5 0.5 0.6 0.6
[18] 0.6 0.6 0.6 0.7 0.7 0.7 0.7 0.8 0.8 0.8
0.9 0.9 1.0
> a <- hist(x,probability=TRUE) # 元のデータのヒストグラム
> a$breaks # 階級幅は 0.2
[1] 0.0 0.2 0.4 0.6 0.8 1.0
> a$density # 各階級の density
[1] 0.500000 1.166667 1.666667 1.166667 0.500000
> sum(diff(a$breaks)[1]*a$density) # 確率の合計
[1] 1
> b <- hist(x*10,probability=TRUE) # 元のデータを 10 倍すると
> b$breaks # 階級幅は 2(元のデータを使った場合の 10倍)
[1] 0 2 4 6 8 10
> b$density # 各階級の density(元のデータを使った場合の 1/10)
[1] 0.0500000 0.1166667 0.1666667 0.1166667 0.0500000
> sum(diff(b$breaks)[1]*b$density)
[1] 1 # 確率の合計はやはり 1
三十路 (2011-06-14 (火) 21:23:44)
c(1,2,3,4,5,100,1001,1002,1003,1004,1005)
をsummaryで見ると、Min. 1st Qu. Median Mean 3rd Qu. Max. 1.0 3.5 100.0 466.4 1002.0 1005.0という結果が表示されるのですが、この3rd Qu.は間違ってますよね。
c(1,2,3,4,5,10,101,102,103,104,105)
ならば正しく、Min. 1st Qu. Median Mean 3rd Qu. Max. 1.00 3.50 10.00 49.09 102.50 105.00が返されるのですが。
HS (2011-06-14 (火) 18:15:53)
R (2.13.0, x86_64-pc-mingw32/x64) 上で RColorBrewer (1.0-2) を使いたいのですが,> library(RColorBrewer) エラー: パッケージ 'RColorBrewer' は R 2.10.0 以前に造らました。新しくインストールして下さいとなってしまいます.64bit環境のためなのでしょうか?
解決方法等ご存知でしたらご教示ください.
TK (2011-06-14 (火) 12:45:30)
線形混合モデルを使った時に、複数の固定効果の相対的な重要性を検討するために、分散分析表を出力しようとしています。分散分析表は、自由度、平方和(Sum of Square)、平均平方(Mean Square)、F値、P値からなっていることが多いので、このような標準的な分散分析表を執筆中の論文に含めたいと考えています。標準的な表を載せる方が読者も理解しやすく、査読者からも受け入れられやすいと考えるからです。
しかし、lme()を使ってもlmer()を使っても、このような出力ができません。Rで呼び出せるOrthodontのデータを例にすると、以下のような作業です。
使用環境は、Windows XP、R2.13.0、nlme_3.1-100、lme4_0.999375-39 です。library(nlme) res.lme <- lme(distance ~ age + Sex, random= ~ 1|Subject, data=Orthodont) anova(res.lme) library(lme4) res.lmer <- lmer(distance ~ age + Sex + (1|Subject), data=Orthodont) anova(res.lmer)lme()の場合、分子自由度、分母自由度、F値、P値が出力されますが、平方和と平均平方が出力されません。
lmer()の場合、分子自由度、平方和、平方平均、F値が出力されますが、P値が出力されません。lmer()がなぜP値を出力しないかは、https://stat.ethz.ch/pipermail/r-help/2006-May/094765.htmlにDouglas Batesさんの解説があるのを見つけました。混合モデルにおける分母自由度とP値の計算は複雑な問題なので、lmer()では出力しないということのようです。ということはlme()が出力するP値も何らかの問題があるものなのでしょうが、lme()のモデルの平方和が分かれば、分散分析表を完成させられるので、今回の論文ではlme()に基づこうと考えました。
lmer()が返す平方和を用いようかと考えたのですが、lme()とlmer()とでは変数によってF値が微妙に異なっていますので、平方和も異なっていそうです。どうしたらいいのでしょうか。
Orthodont$X1<-as.factor(rep(letters[1:3],36))
res.lme2 <- lme(distance ~ age + X1,
random= ~ 1|Subject, data=Orthodont)
summary(res.lme2)
anova(res.lme2)
summary(res.lme2)を見るとX1の係数は2つあり、それぞれの有意性が示されているのに対し、anova(res.lme2)ではX1について一つの有意性が示されているので、分散分析表のようにその変数による分散を示しているのではないでしょうか。 また、anova.lme()の中身を書き換えてsum(c0i^2) を出力することをためしてみました。そうするとsum(c0i^2)の値は、anova(res.lmer)の平方和の値と比べると、ageでは0.4879倍、SEXでは0.4882倍となっていました。一方でF値はほとんど同じですので、sum(c0i^2)は平方和ではないのではないでしょうか。ご指摘のようにFval[i] <- sum(c0i^2)/nDF[i]というようにF値が計算されていますが、F値は(その変数の平方平均)/(残差平方平均)と計算するとすると、この式が意味が理解できません。 -- KT 2011-06-14 (火) 14:56:42タカ (2011-06-13 (月) 17:27:24)
いつもお世話になっております。
GPA(一般プロクラステス分析)について質問です。Acid Strange Hard Acid1 Strange2 Hard2 C1 2.0 2 2.0 2 1 2.0 C2 4.0 2 1.0 4 3 4.0 C3 7.0 1 2.5 11 1 7.0 C4 4.5 2 1.5 1 2 2.8 C5 5.0 3 4.0 5 6 4.0以上のデータ(test)に対して、
library(FactoMineR) res.GPA <- GPA(test, group=c(3, 3))としたのですが、「以下にエラー if (a >= 0) prob <- pgamma(rvstd - (-2/a), shape = (4/a^2), scale = (a/2), : TRUE/FALSE が必要なところが欠損値です」とエラーが表示されてしまいます。 御存じの方いらっしゃいましたら、ご教授の程よろしくお願いいたします。
使用環境は、WindowsXP、R 2.13.0です。
> traceback() 2: coeffRV(Xi, Xj) 1: GPA(test, group = c(3, 3))coeffRV という関数で最終的にエラーが起きたことがわかります。
Acid Strange Hard Acid1 Strange2 Hard2 C1 2.0 2 2.0 2 1 2.0 C2 4.0 2 1.0 4 3 4.0 C3 7.0 1 2.5 11 1 7.0 C4 4.5 2 1.5 1 2 2.8 C5 5.0 4 6.0 4 5 4.0データの中身自体に何か原因があるのでしょうか?繰り返しの質問になってしまいますが、よろしくお願いいたします。-- タカ 2011-06-14 (火) 09:47:44
> a <- NaN
> if (a >= 0) 1
以下にエラー if (a >= 0) 1 :
TRUE/FALSE が必要なところが欠損値です
> a <- -Inf
> if (a >= 0) 1
> if (a < 0) 2
[1] 2
しかし,pgamma が返す値は NaN になっている。エラーメッセージ出ないときにも結果が間違っている(不適切な)ことはある。> test
Acid Strange Hard Acid1 Strange2 Hard2
C1 2.0 2 2.0 2 1 2.0
C2 4.0 2 1.0 4 3 4.0
C3 7.0 1 2.5 11 1 7.0
C4 4.5 2 1.5 1 2 2.8
C5 5.0 4 6.0 4 5 4.0
> res.GPA <- GPA(test, group=c(3, 3))
以下にエラー
if (a >= 0) prob <- pgamma(rvstd - (-2/a),
shape = (4/a^2), scale = (a/2), :
TRUE/FALSE が必要なところが欠損値です
なんで動いたんでしょ。変ですね。 -- 河童の屁は,河童にあらず,屁である。 2011-06-14 (火) 11:18:40> test
Acid Strange Hard Acid1 Strange2 Hard2
C1 2.0 2 2.0 2 1 2.0
C2 4.0 2 1.0 4 3 4.0
C3 7.0 1 2.5 11 1 7.0
C4 4.5 2 1.5 1 2 2.8
C5 5.0 4 6.0 4 5 4.0
> (res.GPA <- GPA(test, group=c(3, 3)))
**Results of the Generalized Procrustes Analysis (GPA)**
There are individuals, characterized by variables
*Results are available in the following objects :
name description
1 "$RV" "RV Coefficients between partial
configurations"
2 "$RVs" "standardized RV Coefficients
between partial configurations"
3 "$simi" "procrustes similarity indexes
between partial configurations"
4 "$scaling" "isotropic scaling factors"
5 "$dep" "PCA of initial configuration "
6 "$consensus" "coordinates of the consensus
configuration"
7 "$Xfin" "coordinates of partial
configurations"
8 "$PANOVA" "list of Procrustes Analysis of
Variance tables"
9 "$correlation" "Correlations by sets"
何が起きているのかさっぱりですね。バグなんですかね。 -- タカ 2011-06-14 (火) 11:42:54M_Saito (2011-06-11 (土) 16:12:29)
初めて投稿します。この投稿欄にあるhiro?氏と同じような状況ですが、libraryからneuralのパッケージが削除されているようです。そこでhiro?氏の記事を参考にインストールを試みましたが駄目です。当方はOS WindowsXp, R2.12.2です。ダウンロードしたneuralアーカイブは.nueral1.4.2.1.tar.gzですので解凍後ファイルの中身を確かめ再度そのファイルをzipに変換してインストールを試みました。しかし次のようなメッセージが出てしまい、完了しません。> utils:::menuInstallLocal() 以下にエラー gzfile(file, "r") : コネクションを開くことができません 追加情報: 警告メッセージ: In gzfile(file, "r") : 圧縮されたファイル 'neural/DESCRIPTION' を開くことができません, 理由は 'No such file or directory' です > install.packages("C/neural.zip",repose=NULL) ファイル名から 'repos = NULL' を推測 以下にエラー zip.unpack(pkg, tmpDir) : zip ファイル 'C/neural.zip' が見付かりません > install.packages("C/neural.zip",contriburl=NULL) 以下にエラー zip.unpack(pkg, tmpDir) : zip ファイル 'C/neural.zip' が見付かりませんファイルは作業デレクトリに置いてあります。どこが間違っているのか、又その解決策をご教授願いたいのです。下らない質問で済みません、宜しく御願い致します。
うー (2011-06-02 (木) 17:03:52)
ロジスティック回帰をしています。
量xと反応の割合pについて、glm関数でロジスティック回帰式を出しました。
xとpについてプロットを行い、この図に回帰曲線を加えたいと思います。
単回帰であればabline関数とlm関数を用いてコマンド一つで回帰直線を描くことができますが、ロジスティック回帰についてはそれが見当たりません。
いくつか本やウェブページを調べたのですが、独立変数を非常に細かくとってpredictとpointsで作図するとか、glmで求めた予測値についてlines関数で補間するなど、ちょっと回りくどい感じがします。
curve関数で書いてしまおうかとも思ったのですが、高水準作図関数なので実データとの作図の順番が逆になってしまいます。
何かいい方法はないでしょうか? 自分で関数を書く以外ないでしょうか?
plot.glm <- function(a)
{
x <- a$data$x
y <- a$data$y
plot(x, y)
points(x, a$fitted.values, col="red")
d <- data.frame(x=seq(x[1], x[length(x)],
length=500))
lines(d$x, predict(a, d, type="response"),
col="red")
}
# 使用例
d <- data.frame(x=1:10, y=c(0,0,0,1,0,1,1,1,1,1))
a <- glm(y~x, data=d, family=binomial)
plot(a)
# 当然ながら,logit 以外にも対応
plot(glm(y~x, data=d,
family=binomial(link="probit")))
plot(glm(y~x, data=d, family=gaussian))ぺーぺー (2011-06-01 (水) 21:35:13)
相関行列をテキストファイルに出力したいのですが、どうしても列名(1行目のデータ)が1列左へズレてしまいます。
例として、以下のコードで再現可能です。
a<-iris[,1:4] b<-cor(a) write.table(b,"C:/b.txt",append=F, quote=F,col.names=T,row.names=T)
コンソール上でbを出力すると、1行1列目は空白としてきちんと表現されますが、テキストファイルに出力すると、1行1列目に、本来1行2列目の"Sepal.Length"がズレこんできます。
現状、テキストファイルの頭にスペースを追加して読み込んで対応しているのですが、気持ちが悪いのでコード上の問題があるならば解決したいです。
宜しくお願い致します。
はと (2011-05-29 (日) 16:21:03)
R2.13.0を利用しております。
pairs()で対数軸を利用する方法について質問があります。
plot()では、plot(data-frame,log="xy")で対数軸に出来るので、pairs(data-frame,log="xy")としたところ、一応対数軸になったものの、「"log"はグラフィックスパラメータではありません」とメッセージが出るとともに、図内のラベル(データフレームのheader)が表示されなくなってしまいます。
dataのlogをとることも考えましたが、相関係数は元のdataで計算したいのです。
よろしくお願い致します。
たろ (2011-05-23 (月) 17:46:02)
初心者です。データフレームの中の変数 variable (数字と文字列の混ざった値です)が 20, 29, 2A, 2B の値のデータを選び出したいのですが、すべてのケースを列挙するのではなく、subset(data, data["variable"] == "2*")のような書き方で一遍に取り出すことは可能でしょうか。
ようやくRが楽しくなってきたとこ (2011-05-19 (木) 18:49:52)
http://software.ssri.co.jp/statweb2/column/column0708.html に示されている、トレーニング前と後の違いを見るような、対応のあるデータを一覧できるグラフを作成したいのですが、Rではどうやればよいか教えていただければ幸いです。
set.seed(123456)
x <- data.frame(before=rnorm(10),
after=rnorm(10, mean=0.5))
plot(c(1, 2), range(x), type="n", xlab="",
xaxt="n", ylab="value",
xlim=c(0.8, 2.2))
junc <- sapply(1:nrow(x),
function(i) segments(1, x[i, 1], 2, x[i, 2], col=i))
axis(1, at=1:2, labels=colnames(x))
text(2, x$after, 1:nrow(x), col=1:nrow(x),
pos=4, xpd=TRUE)
R始めて1ヶ月 (2011-05-07 (土) 12:28:53)
ニューラルネットワークのパッケージnnetについて質問です。
すでにデータAを学習したネットワークに対して、さらに別のデータBを学習させるにはどうすれば良いのでしょうか?先にAとBを結合して読ませるには時間がかかるために困っております。
別のパッケージならできるという情報もお待ちしております。
質問者 (2011-05-06 (金) 11:07:52)
お世話になります。
fv1からfv25という変数にそれぞれread.csv()で数値データ(30KB~90KBなどまちまち)を代入しました。
これらのデータを順番に同じ処理をしたく以下のようなコードを書きました。for (i in 1:25) { eval(parse(text = paste("fv <- fv", i))) # 変数fvにi番目のfvを代入 #処理 }コードを実行すると
以下にエラー parse(text = paste("fv <- fv", i)) : <text>:1:10: 予想外の 数値定数 です 1: fv <- fv 1 ^というエラーが出てきてしまいます。
この原因が分かる方がいましたら、どうかご教授願います。環境: R version 2.12.1 (2010-12-16) Platform: x86_64-pc-mingw32/x64 (64-bit)
# データが個々のファイルに用意されており,ファイル名が連番を含む規則的なものであるとき
# 入力すべきファイル名を生成する
file.name <- sprintf("file%03d.dat", 1:200)
# file001.dat 〜 file200.dat まで
for (fn in file.name) {
cat(sprintf("file: %s\n"))
# どのファイルを処理した結果かを明記しておく
data <- read.csv(fn)
# いつも同じデータフレームに読み込む
# data というデータフレームについて処理
}
eval(parse( を使わなくては書けないというのはそんなに多くない。 -- 河童の屁は,河童にあらず,屁である。 2011-05-06 (金) 16:46:43初心者KT (2011-05-04 (水) 09:19:54)
三次元の座標を持つ点AからEが存在します
下記の様な三角形が三つあります。
各三角形は連続して隣接しています。
下記の三角形の表と裏を別の色で塗ることは可能でしょうか。
またZの値によって、gradationがかかる様な塗り方も可能でしょうか。
実際には、100個以上の三角形の組み合わせによる構造物を扱う予定です。
三角形1
Point X (mm) Y (mm) Z (mm)
A -14.54 8.39 60.82
B -12.36 10.37 61.72
C -9.4 7.88 61.2
三角形2
A -14.54 8.39 60.82
D -15.77 5.74 60.3
E -10.51 6.07 61.2
三角形3
A -14.54 8.39 60.82
E -10.51 6.07 61.2
C -9.4 7.88 61.2
z <- volcano
x <- 10*(1:nrow(z))
y <- 10*(1:ncol(z))
brks <- quantile(z, seq(0,1, len = 21))
zi <- z[-nrow(z),-ncol(z)]
cols <- terrain.colors(20)[cut(zi,brks,
include.lowest = TRUE)]
persp(x, y, 2*z, theta = 120, phi = 15,
col = cols, scale = FALSE,
ltheta = -120, border = NA, box = FALSE)
ポリゴンやグリッドの中をグラデーションで塗ることはできませんが、このように1つ1つのグリッドを単色で彩色することによって、擬似的なグラデーションを実現することができます。もっと細かいグリッドを用意すればさらに見栄えはよくなるでしょう。質問者 (2011-05-03 (火) 21:29:33)
パッケージを更新しようとすると以下のような警告出ます。警告: パッケージ 'urca' の前のインストールを取り除くことが出来ませんでした パッケージ 'zoo' は無事に開封され、MD5 サムもチェックされました ダウンロードされたパッケージは、以下にあります C:\Users\***\AppData\Local\Temp\RtmpVFYyw4\downloaded_packages install.packages(update[instlib == l, "Package"], l, contriburl = contriburl, 中で警告がありました: 'lib = "C:/PROGRA~1/R/R-212~1.1/library"' は 書き込み可能ではありません 以下にエラー install.packages(update[instlib == l, "Package"], l, contriburl = contriburl, : パッケージをインストール出来ませんでした
解決策はありますでしょうか。ちなみに作業状態ですが、Rを終了する時にワークスペースは保存しないを選んで終了し、Rを再起動後すぐにパッケージの更新を実行しました。
質問者 (2011-05-03 (火) 13:43:25)
あるパッケージをインストールした後、そのバージョンの確認が必要になったのですが、確認方法はありますでしょうか。
str(ip <- installed.packages(priority = "high")) ip[, c(1,3:5)] plic <- installed.packages(priority = "high", fields="License") ## what licenses are there: table( plic[,"License"] )
> library(help=zoo) により表示されるウインドウの一部 Package: zoo Version: 1.6-5 > library(help=MASS) により表示されるウインドウの一部 Package: MASS Priority: recommended Version: 7.3-13
Toy (2011-05-02 (月) 15:14:07)
お世話になります。
初歩的な質問で恐縮です。
ESSで日本語を表示する方法について教えてください。
まず.emacsに、次のように記述しました。;;essのロード (load "c:/Meadow/site-lisp/ess/Lisp/ess-site.el")そしてess-site.elの299行目を、次のように書き換えました。
(setq-default inferior-R-program-name "c:/r/bin/i386/Rterm")その上でMeadowを起動し、Alt+x rと入力するとRは起動します。
しかし、起動した後の初期画面には日本語は表示されず、作成されたプロットでも日本語は文字化けしてしまいます。
どのように設定すれば、日本語が正しく表示されるのか、御教示いただければ幸いです。
当方の環境は、以下のとおりです。Editor:Meadow 3.01-dev OS:Windows XP R:Version 2-13-0
(set-language-environment "Japanese")の次に、以下のコードを付け加えることで、グラフ表示の際の日本語の文字化けが解消しました。
(set-default-coding-systems 'shift_jis)
(set-terminal-coding-system 'shift_jis)
(set-keyboard-coding-system 'shift_jis)
(set-buffer-file-coding-system 'shift_jis)
(require 'ess-site)
(setq ess-pre-run-hook
'((lambda () (setq S-directory default-directory)
(setq default-process-coding-system
'(shift_jis . shift_jis))
)))
なお、既に記述してあった、次のコードは削除しました。
;;essのロード (load "c:/Meadow/site-lisp/ess/Lisp/ess-site.el")ただし、依然として初期画面は日本語表示ではありませんし、エラーメッセージも日本語ではありません。 これはESSの仕様なのでしょうか? -- Toy 2011-05-06 (金) 17:11:27
R初心者 (2011-05-02 (月) 11:14:32)
R、統計ともに超のつく初心者です。
RおよびRコマンダーを用いて、クラスター分析を行い、樹形図を描きたいと思っております。
Rコマンダーのインストールはできたのですが、クラスター分析を選択した後、次の選択欄が全てグレイ表示になってしまい選択できません。
Rコマンダー入門によりますと、「現在の状況において利用できないものはグレイで表示され,選択できないようになっている.」との記載があり、現在、それらの項目が使用できない状況にあることはわかるのですが、他に何が足りないのかがわかりません。
クラスター解析をする際にインストールが必要なパッケージ等があるのでしょうか。
使用OSはWindowsXP, Rのバージョンは2.13.0,データはテキスト形式で読み込みは可能でした。
どうかご教授お願いいたします。
cyako (2011-05-01 (日) 18:44:20)
R初心者ですが、心理学論文を書くのにコレスポンデンス分析、もしくは数量化?�爐鰺僂い燭い塙佑┐討い泙后? そこで、解説サイトや本を参考にしつつ、下記�のスクリプトで実行しました。
(windows7、Rのバージョンは2.13.0、データはexcelで作成したカテゴリーデータです。)�1つ目に使用したスクリプト > df <- read.table("clipboard", header=TRUE) > df > library(MASS) > corresp(df, nf=2)このスクリプトで1つ目のデータはなんとか結果?を出すことができました。
しかし、clipboardの内容(別のカテゴリーデータ)を変えて、同じスクリプトで実行してみても下記�のようなエラーが出て結果が出ません。�2つ目のエラー > df <- read.table("clipboard", header=TRUE) > df > library(MASS) > corresp(df, nf=2) 以下にエラー corresp.matrix(as.matrix(x), ...) : empty row or column in table←これが表示がされますこのようなエラーに対してどのように対処すればいいのでしょうか?
もしくは、そもそもスクリプトが間違っているのでしょうか?
初歩的な質問で大変申し訳ないのですが、ご指導いただきたく存じます。
> library(MASS)
> (x <- matrix(c(1,0,0, 0,0,0, 0,0,1, 1,0,1), 4))
# 第2列が全部 0 です
[,1] [,2] [,3]
[1,] 1 0 1
[2,] 0 0 1
[3,] 0 0 0
[4,] 0 0 1
> corresp(x)
以下にエラー corresp.matrix(x) :
empty row or column in table
> (x <- matrix(c(1,0,0, 0,1,0, 0,0,0, 1,0,1), 4))
# 第3行が全部 0 です
[,1] [,2] [,3]
[1,] 1 1 0
[2,] 0 0 1
[3,] 0 0 0
[4,] 0 0 1
> corresp(x)
以下にエラー corresp.matrix(x) :
empty row or column in table
このほか,分かりにくいですけど見た目は全部 0 であるような行や列が内にもかかわらず同じようなエラーが起きることもありますが,原因は結局はダミー変数に展開したときに同じ状況が生じるというのが原因です。まあ,サンプルサイズが小さいときには起こりがちということもあるでしょう。十分なデータを蓄積後に分析をすることをお勧めします。hiro (2011-05-01 (日) 11:46:01)
Mac OS X 上で R 2.13.0 を使用しています。以前は標準エディタ上で ctrl+H で直前の1文字を消去できていたように思うのですが、このバージョンではヘルプが表示されてしまいます。ctrl+H の機能をもとへ戻す方法があれば教えてください。
* Added menu item 'Show Help for current
Function' CTRL+H
to context menu of RConsole and each Script
Editor. It
parses backwards from the current cursor's
position to
to find the current function and opens the
help page.
If the user did select a text chunk CTRL+H
will try
to come up with help page of the selected
text.
If nothing was found it sets the focus to
the Help
Search Field.
この機能ですね。 右クリックしたときに出てくるコンテキストメニューの項目で、カーソル位置にある関数を読み取り、そのヘルプを表示してくれる新機能です。このコンテキストメニューのショートカットキーをInterface Builderあたりで簡単に変更できるとよさそうですが... -- 岡田 2011-05-20 (金) 14:43:20
R初心者 (2011-04-30 (土) 18:30:19)
仮にRでDBからデータフレームに対して数量化I類を行って、stepを噛ましてAIC最小のモデルを選ぶとします。また、summaryで各カテゴリ変数に対しての有意な結果が分かっているとします。
この時点で、「有意じゃない」カテゴリ内の変数に関して除去しつつ、lmで再計算を行うにはどのようにすれば良いのでしょうか。オリジナルのデータ、あるいはデータフレームの内容を変更するしか手立てが無いのでしょうか。
質問者 (2011-04-30 (土) 09:54:22)
Aployに次のような三次元配列を定義します。Apoly <- array(c(1,-0.5,0.3, 0,0.2,0.1, 0,-0.2,0.7, 1,0.5,-0.3), c(3,2,2))Aployを1次元の添え字ごとに2次元配列として表示するために次のようなコマンドを打ち込みました。
Apoly[1,,] Apoly[2,,] Apoly[3,,]結果は次の通りです。
> Apoly[1,,] [,1] [,2] [1,] 1 0 [2,] 0 1 > Apoly[2,,] [,1] [,2] [1,] -0.5 -0.2 [2,] 0.2 0.5 > Apoly[3,,] [,1] [,2] [1,] 0.3 0.7 [2,] 0.1 -0.3これらを一つのコマンドで実行する方法はないでしょうか。例えばApoly[1:3,,]を試しましたが、Apolyの表示方法と同じの3次元の添え字順に2次元配列として表示され、うまくいきません。
> Apoly[1:3,,] , , 1 [,1] [,2] [1,] 1.0 0.0 [2,] -0.5 0.2 [3,] 0.3 0.1 , , 2 [,1] [,2] [1,] 0.0 1.0 [2,] -0.2 0.5 [3,] 0.7 -0.3
> lapply(1:3, function(x) Apoly[x,,])
[[1]]
[,1] [,2]
[1,] 1 0
[2,] 0 1
[[2]]
[,1] [,2]
[1,] -0.5 -0.2
[2,] 0.2 0.5
[[3]]
[,1] [,2]
[1,] 0.3 0.7
[2,] 0.1 -0.3> junk <- apply(Apoly, 1, print)
[,1] [,2]
[1,] 1 0
[2,] 0 1
[,1] [,2]
[1,] -0.5 -0.2
[2,] 0.2 0.5
[,1] [,2]
[1,] 0.3 0.7
[2,] 0.1 -0.3> aperm(Apoly,c(2,3,1))
, , 1
[,1] [,2]
[1,] 1 0
[2,] 0 1
, , 2
[,1] [,2]
[1,] -0.5 -0.2
[2,] 0.2 0.5
, , 3
[,1] [,2]
[1,] 0.3 0.7
[2,] 0.1 -0.3R初心者 (2011-04-30 (土) 05:53:05)
DBにRに接続しながらlm関数で数量化I類を試しています。
Rで数量化I類を行うと、キチンと回帰結果を返してくれるのですが、一方、基準となるカテゴリカルデータをこちらから指定したいのですが、その方法が分からないのです。
Interceptや、基準となるカテゴリー内のデータ名はlm関数の引数に与える事が可能なのでしょうか。
> x <- c("foo", "bar", "baz", "bar")
> (a1 <- factor(x))
[1] foo bar baz bar
Levels: bar baz foo
> as.integer(a1)
[1] 3 1 2 1
> (a2 <- factor(x,
levels=c("bar", "baz", "foo")))
[1] foo bar baz bar
Levels: bar baz foo
> as.integer(a2)
[1] 3 1 2 1
> (a3 <- factor(x,
levels=c("foo", "baz", "bar")))
[1] foo bar baz bar
Levels: foo baz bar
> as.integer(a3)
[1] 1 3 2 3miyucka (2011-04-30 (土) 00:25:29)
こんばんわ.いつもお世話になっています
環境はMac OSX 10.4.11です.
Rcmdrを使いたくて
library(Rcmdr)
と入力するとError in structure(.External("dotTclObjv", objv, PACKAGE = "tcltk"), class = "tclObj") : [tcl] invalid command name "font". Error : .onAttach は 'attachNamespace' で 失敗しました エラー: 'Rcmdr' に対するパッケージ もしくは名前空間のロードが失敗しましたとなってしまいます.
パッケージのアップデートをCRANのバイナリもソースも両方やってみました
(違いは理解してませんがとりあえず...)
BioConductorもやってみましたが変化無しです.
おかしなことをしたのかと思ってもとからもっていたRを消して
もう一度インストールして
library(Rcmdr)と入力しても要求されたパッケージ tcltk をロード中です Tcl/Tkインターフェースのロード中 終了済 要求されたパッケージ car をロード中です 要求されたパッケージ MASS をロード中です 要求されたパッケージ nnet をロード中です 要求されたパッケージ survival をロード中です 要求されたパッケージ splines をロード中ですという記述はでるものの,結果は変わりません
他のQ&Aのページでは勝手に治ったという結果でバグだったのか?
という記述で終ってました.
これはどこをどうしたらいいのでしょうか???
ご指導頂ければ嬉しいです.よろしくお願い致します.
初心者 (2011-04-27 (水) 13:21:40)
立体的なグラフの表現方法について
初心者KT? (2011-01-31 (月) 22:24:19)
R初心者です。
自分は数学系、物理系、工学系の専門教育は受けたことがなく、医学系の仕事をしています。しかし今回幾つかの解析や、グラフによる表記を行う必要性に迫られています。
30個の点が、xyzを与えられています。
1. 30個の点が、“最も平均的に通る平面”(平面Aとする)を設定しました
(以前のこのコーナーで質問に答えていただき、平面Aは、ax+by+cz+d=0 と表現した時、optim関数でa = 0.09792275, b = 0.37648968 c = -0.32677544, d = 1となりました)。
2. 平面Aの上に81個の点の様な構造物がドーム状にかぶさっています(曲面Bとします)。
� 30個の点と平面A
� 30個の点と平面A,および平面B
� �平面Aと平面B
のそれぞれの3種類の組み合わせを、3次元的にグラフで綺麗に表すには、どの様な方法があるでしょうか。
LatticeからCloundやwireframeなどを使おうとしましたが、なかなかうまくいきません。よろしくお願いします。
理想的には、平面Bは高さによって(Zの値によって)、色が変化するように表せたら最高です。平面Bに関する理想的な色の使い方参考例として、PDFで添付します。
30個の点
X Y Z
56.8458 56.3972 95.2184
58.1262 55.2657 96.9028
61.0080 53.8846 97.6925
63.7117 52.5191 98.1147
65.9630 51.1603 98.0884
68.0598 49.8240 97.4626
70.3595 48.6390 96.2383
73.0480 47.8221 94.6007
76.0860 47.6060 92.8562
79.2539 48.1658 91.3380
82.2386 49.5663 90.3180
84.7249 51.7420 89.9494
86.4678 54.5090 90.2453
87.3370 57.6021 91.0912
87.3282 60.7262 92.2840
86.5478 63.6101 93.5838
85.1780 66.0516 94.7681
83.4311 67.9458 95.6757
81.5042 69.2908 96.2331
79.5429 70.1705 96.4590
77.6191 70.7182 96.4472
75.7289 71.0677 96.3317
73.8088 71.3037 96.2427
71.7678 71.4224 96.2643
69.5302 71.3165 96.4051
67.0781 70.7936 96.5927
64.4841 69.6327 96.6992
61.9234 67.6774 96.5962
59.6531 64.9476 96.2303
57.9525 61.7413 95.6913
曲面Bの構成要素
X Y Z
66.5214 52.3693 100.1890
69.0172 55.2805 98.6580
71.1465 57.6134 98.0792
73.4542 60.2416 96.9707
74.2816 61.4516 95.2830
75.8534 63.4528 93.5104
78.1356 65.5883 94.6485
79.3441 66.4813 96.3968
81.8057 68.7355 97.8613
69.4359 50.9051 99.5420
71.1025 54.5844 99.2688
71.9337 56.6633 98.3382
72.8589 58.7782 97.9511
73.5783 60.6862 96.7916
74.3058 62.7413 95.2100
74.8671 64.3807 93.8145
76.4086 67.2760 95.2155
78.2037 70.3165 97.9255
72.8598 48.6686 97.5556
73.0553 53.8495 97.0382
73.1433 56.6261 96.5826
73.3167 59.6391 96.9136
73.3231 61.7117 95.9461
73.3418 63.9996 95.0022
73.3420 65.9646 94.0229
73.5266 67.9228 94.9986
73.9282 71.3346 97.5466
76.6417 48.3653 95.4308
75.5219 52.4555 96.4391
74.9038 54.7740 97.3086
74.0257 57.8910 97.6356
73.1674 60.8503 97.5045
72.5119 63.0188 96.9356
71.6926 65.7293 96.2245
71.1617 67.6709 96.7142
70.0804 71.6452 97.8153
81.0857 49.4645 93.7371
77.7238 54.0901 94.3492
75.9176 56.5834 95.7828
74.4364 58.6303 97.2458
73.4941 59.9329 98.2465
72.1696 61.7559 98.5740
71.0152 63.3431 98.6402
69.0276 66.0708 98.0351
66.2486 69.8901 97.9546
85.2473 52.2150 93.0255
81.2928 54.9562 92.2683
79.3713 56.2273 93.0238
76.9566 57.8602 93.3170
75.3852 58.7892 95.9743
72.8658 60.3924 98.1361
70.5020 62.0146 97.9852
67.0597 64.3694 97.9051
62.4689 67.4921 98.1250
88.0380 56.8771 94.4088
84.4938 57.8743 92.7572
81.0642 58.7234 92.3931
78.9147 59.1500 93.2910
75.6953 59.7277 95.2876
72.8867 60.1105 98.3210
68.8648 61.0533 98.4587
65.0531 61.9484 98.5723
59.3881 63.4470 96.9480
87.7385 61.3387 95.9346
84.2589 61.3849 93.4009
80.7455 61.1783 92.9229
78.1388 60.8384 94.1021
74.5410 60.2960 96.3323
70.9528 59.7412 98.6698
67.4871 59.4730 98.7284
63.3083 59.2362 98.0871
58.8063 58.9720 97.4714
85.0653 65.4926 97.8556
83.9668 65.5278 94.3416
80.8889 64.4879 91.7567
77.4482 62.5331 93.9201
75.0270 61.1638 95.4027
73.0548 59.9064 97.5156
70.2444 58.5210 97.9352
66.1804 56.5065 98.6132
62.4675 54.5575 99.9249
 -- 私も医学系 2011-04-27 (水) 15:18:18
-- 私も医学系 2011-04-27 (水) 15:18:18library(misc3d)
## ptsは30点のデータフレームです
## range(pts[,1])
## range(pts[,2])
a <- -0.001709210
b <- 0.001085967
c <- -0.009890907
d <- 1
v <- surfaceTriangles(seq(55,90,len=30),
seq(45,75,len=30),
function(x, y){-(a/c)*x-(b/c)*y-(d/c)},
color2 = "green")
p <- pointsTetrahedra(pts[,1],pts[,2],
pts[,3],size = 0.1)
## drawScene.rgl(list(v, p))
drawScene(list(v, p))
ついでにBの方の参考コードも示します。
library(geometry)
library(rgl)
tetramesh(delaunayn(as.matrix(B)),B)
# Bは曲面Bのデータフレーム
NT (2011-04-23 (土) 16:50:39)
R2.12.2をアンインストールしようとしたら「このプログラムは64-bit版Windows上のみでアンインストール可能です。」という表示がでてきます。
使用OSはWindowsXP Proffesional Ver.2002 Servic Pack3です。
私としてはこれまでRがバージョンアップされる度に、古いバージョンと入れ替えてきており、問題は生じておりませんでした。
tadashi (2011-04-21 (木) 13:24:10)
PHP には、10進法を2進法に変換するdecbin とい関数があります。Rでは同様の関数はあるでしょうか? つくるのは簡単で、方法もわかり、つくってもいますので、作り方を知りたいわけではないです。このライブララリを読むと、こんな関数があるよというのがごく当たり前にあるのでしたら、ぜひ教えてください。
まつよ (2011-04-20 (水) 12:36:01)
下のようなデータフレーム"data"がを用いて、glm関数による回帰分析を行った後に、predict関数のse.fit=Tを指定したときに出力される$se.fitについて教えてください。> data x y 1 1 2 2 2 3 3 3 5 4 4 9 5 5 4 6 6 4 7 7 10 8 8 12 9 9 15 10 10 18 11 11 19 12 12 19 13 13 20 14 14 22 15 15 25 16 16 29 17 17 30 18 18 35 19 19 36 20 20 35 > m=glm(y~x, data, family=gaussian(link=identity)) > predict(m, newdata=data.frame(x=x), se.fit=T) $fit 1 2 3 4 5 6 -0.3428571 1.5458647 3.4345865 5.3233083 7.2120301 9.1007519 7 8 9 10 11 12 10.9894737 12.8781955 14.7669173 16.6556391 18.5443609 20.4330827 13 14 15 16 17 18 22.3218045 24.2105263 26.0992481 27.9879699 29.8766917 31.7654135 19 20 33.6541353 35.5428571 $se.fit 1 2 3 4 5 6 0.9877626 0.9129465 0.8408734 0.7723116 0.7082816 0.6501237 7 8 9 10 11 12 0.5995491 0.5586215 0.5295821 0.5144480 0.5144480 0.5295821 13 14 15 16 17 18 0.5586215 0.5995491 0.6501237 0.7082816 0.7723116 0.8408734 19 20 0.9129465 0.9877626 $residual.scale [1] 2.292081おそら$se.fitの値は標準誤差(SE)なのだと思いますが、この値から標準誤差(SD)の値を求めることは可能なのでしょうか?例えば、rnorm()などの関数を使ってデータを再現し、ブートストラップ法でデータの信頼区間を求めるにはSEよりもSDがわかった方が便利な気がするのですが。
Googleで検索して少し調べてみるとSE=SD/sqrt(N)の関係があるそうですが、ここのときのNの値には何を使えばいいのでしょうか?N=nrow(data)でいいのでしょうか?
ちなみにsessionInfo()で出力される使用環境は以下の通りです。> sessionInfo() R version 2.12.1 (2010-12-16) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods [7] base loaded via a namespace (and not attached): [1] MASS_7.3-9
michi (2011-04-18 (月) 19:03:14)
R 2.12.2を使用しています(mac).
&ref(sorafenib SHARP trial NEJM.pdf)のfigure 3のような,Cox proportional hazard modelの結果をforest plotにする方法(パッケージ)をご存知でしょうか?
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください
を読んでください![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください
を読んでください![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください -- 河童の屁は,河童にあらず,屁である。 2011-04-18 (月) 19:35:14
を読んでください -- 河童の屁は,河童にあらず,屁である。 2011-04-18 (月) 19:35:14kazusata (2011-04-18 (月) 18:39:14)
ご回答ありがとうございます。
close.screen( ) をこまめに入れてみます。
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください
を読んでくださいkazusata (2011-04-17 (日) 21:52:27)
windows7で、R ver.2.12.0を便利に使わせて頂いています。
以下の使用で、思いどおりに行かず、ヘルプをお願い致します。
jpegデバイスに複数のグラフを書く中で、screen(n, new=FALSE)で、自由にscreenを移動したいのですが、以下の手順で、前に描いたグラフパーツが消えてしまい、new=FALSEが効かないのですが、何か他に設定が必要なのでしょうか?jpeg(file.name, quality = 50) split.screen(c(8,4) ) screen(1) plot(x,y,type="n") screen(2) ...... screen(1, new = FALSE) liens(x,y) ........ dev.off()これで、jpegを見ると、座標軸が消えている。
pdf("test.pdf", width=10, height=5)
split.screen(c(1, 2))
x <- 1:10
y <- x^2
screen(1)
plot(x,y,type="n")
screen(2)
plot(x,y,type="n")
screen(1, new=FALSE)
lines(x,y)
close.screen(1) # これを入れると期待どおり
screen(2, new=FALSE)
points(x, y)
close.screen(all=TRUE)
dev.off()
hana (2011-04-13 (水) 17:21:10)
Mac OS X 10.6.7でR ver. 2.12.2を使用しています初心者です.
Kaplan-Meierの生存曲線はこちらの既出の方法で描けたのですが,たとえば再発率の解析などでy軸の0から曲線を描くにはどうすれば良いでしょうか(生存曲線の上下反転のような)?
またnumber at riskをグラフに盛り込むにはどうすればよいでしょうか?
よろしくご教示ください.
require(km.ci)
data(rectum.dat)
require(survival)
fit <- survfit(Surv(time, status) ~ 1,
data=rectum.dat)
plot(fit$time, 1-fit$surv, type="s")
text(fit$time, 1-fit$surv, fit$n.risk,
pos=3, xpd=TRUE)sizu (2011-04-08 (金) 14:16:51)
よろしくお願いします。
R.12.2.2のインストール後、install.packages( )で、下記に貼り付けたエラーメッセージが出て失敗するようになり困っています。utils:::menuInstallPkgs()でも同じで、「以下にエラーメッセージを・・・」の後と同じメッセージがでます。
OSはWindowsXP (SP3) 、インストール方法は、full installを選択しました。
ミラーサイトは、Tsukuba, hyogo どちらも試しました。
また、私のPCでは、下記メッセージの「圧縮されたファイル・・・」以下に記載されている、「Rimp70CFbVb]の下には「downloaded_packages」というフォルダが入っており中身は空で、メッセージに書かれてあるよう名前のフォルダは見あたりません。
R2.12.1までは、普通に動いていました。バージョンアップしてからも、前に作成したワークスペース(.rdata)から起動した場合に限って、普通に動いていたこともありました。
現在では、R2.12.1、R2.12.2 いずれをインストールしなおしても、できませんし、以前保存したワークスペースから起動しても同じです。
インストール方法等については、Google などでいろいろ検索し、試行錯誤をしましたが、解決できず困っております。よろしくお願いします。
以下にエラーメッセージを貼り付けます。> install.packages("Rcmdr") パッケージを ‘C:\Documents and Settings\ Administrator\Local Settings\Application Data\ R-core/R/win-library/2.12 中にインストールします (‘lib’ が指定されていないので) 以下にエラー gzfile(file, mode) : コネクションを開くことができません 追加情報: 警告メッセージ: In gzfile(file, mode) : 圧縮されたファイル 'C:\DOCUME~1\ADMINI~1\LOCALS~1\ Temp\RtmpNubfVC/libloc_C %3a%5cDocuments%20and%20Settings%5cAdministrator %5cLocal%20Settings%5cApplication%20Data%5cR-core%2fR%2fwin- library%2f2.12Version,Priority,Depends,Imports, LinkingTo,Suggests,Enhances,OS_type,License,Archs,Built.rds' を開くことができません, 理由は 'No such file or directory' です
miyuka (2011-04-07 (木) 22:14:08)
R初心者です.おねがいします.
環境はMac OS X 10.4.11.R version 2.10.1 です.
クリップボードからデータを読み込みたいのですがx <- read.delim("clipboard") 以下にエラー file(file, "rt") : コネクションを開くことができません : 追加情報: 警告メッセージ: In file(file, "rt") : クリップボードを開くことができないか,中身がありませんとなります.
ファイルのアクセス権の問題かと思いアクセス権をユーティリティから修復したのですがうまくいきません.
ディレクトリはファイルのあるところ,デスクトップ,ホームで試しましたが全て同じ結果です.
何がおかしいか教えて頂ければ幸いです.
K (2011-04-01 (金) 03:58:55)
こちらの論文(PDF)の図4のようなグラフはなんと呼ぶのでしょうか? また、このグラフをRで作成するパッケージはありますでしょうか?
名前だけでも分かれば検索できるようになると思うので、どうぞよろしくお願いします。
初心者KT (2011-03-31 (木) 12:02:24)
初心者的質問です。
CSV fileからデータをimportすると、時々データ行列の一番下に XXX levels: X, X, X, X, と表示が出ます(Xは数字)。このXXX levels・・・の表示を伴ったデータ行列になってしまった場合は、足し算や引き算などの行列の単純な計算を行う時に”因子に対しては無意味です“との表示が出て、計算が出来なくなってしまいます。
またこの様な時は通常のplotも不可能となります。
また行列の値を[ ] を用いて取り出しても、やはり値の下に、XXXlevels・・・の表示があります。
CSV fileから、一部分のみコピーして、他のExcel fileにペーストしてからCSV形式に保存し、それをRにimportすると、XXX levelsの表示が消えて、行列計算が可能となる場合もあれば、そのままLevels・・・・表示が出て、単純な行列計算が不可能な場合もあります。
このXXX levelsの表示は何でしょうか?
また消す方法はあるのでしょうか?
宜しくお願いします。
下記にその表示をコピーします。> AllL[2,3] [1] 87.5242 47 Levels: 84.7952 86.1777 86.4596 86.4788 86.5069 86.6655 ... Z > AllL[3,3] [1] 88.8609 47 Levels: 84.7952 86.1777 86.4596 86.4788 86.5069 86.6655 ... Z > AllL[2,3]- AllL[3,3] [1] NA 警告メッセージ: In Ops.factor(AllL[2, 3], AllL[3, 3]) : - 因子に対しては無意味です
Toy (2011-03-29 (火) 16:08:34)
お世話になります。
latticeのマルチプロットで、gridにより各パネルごとに注釈を付けたいと考えております。
しかし、注釈をベクトルで値を変えながら付けていきたいのですが、上手くいきません。
どなたか、解決策を御存知の方、御教授いただければ幸いです。library(lattice) library(grid) levels <- levels(as.factor(ethanol$C)) xyplot(NOx ~ E|C, data=ethanol, panel = function(x, y) { panel.xyplot(x, y) panel.loess(x, y) # 各パネルごとにCの水準を示したい。 grid.text(label=paste("C=", levels, sep=""), x=unit(0.05, "npc"), y=unit(0.95, "npc"), just="left")})
library(lattice)
library(grid)
levels <- levels(as.factor(ethanol$C))
p <- xyplot(NOx ~ E|C, data=ethanol,
panel = function(x, y) {
panel.xyplot(x, y)
panel.loess(x, y)})
print(p)
update(p,
index.cond = list(rep(1, length(levels))))
panel.locs <- trellis.currentLayout()
i <- 1
for (row in 1:nrow(panel.locs))
for (column in 1:ncol(panel.locs))
if (panel.locs[row, column] > 0)
{
trellis.focus("panel", row = row,
column = column,
highlight = FALSE)
grid.text(label=paste("C=",
levels[i], sep=""),
x=unit(0.05, "npc"),
y=unit(0.95, "npc"),
just="left")
trellis.unfocus()
i <- i + 1
}
お騒がせしました。-- Toy 2011-03-30 (水) 15:08:47hevel (2011-03-28 (月) 16:35:42)
gamを使って一般化加法モデル解析をしています。
gamの中で平滑化スプライン関数sを使って、“s(x, df=4, spar=0)”のように等価自由度を変更設定できると言うことなのですが、設定できません。
つまり、“df=某”という式を入れると、「ExtractVars 中のモデル式が不正です 」と言うメッセージが出ます。
バグでしょうか?それとも何か別のパッケージを入れておかなければならないのでしょうか?
> library(mgcv)
> data(airquality)
> airq.gam <- gam(Ozone~s(Solar.R)+s(Wind)+s(Temp),
data=airquality)
として計算するとエラーは出ないのですが、
> airq.gam2 <- gam(Ozone~s(Solar.R, df=10)+
s(Wind, df=10)+s(Temp, df=10), data=airquality)
とするとエラーが出ます。等価自由度を変更して解析できないか試そうとしたのですが、こういうやり方はそもそも間違っているのでしょうか? -- hevel 2011-03-28 (月) 22:31:28> library(gam)
> data(airquality)
> airq.gam2 <- gam(Ozone~s(Solar.R, df=10)+
s(Wind, df=10)+s(Temp, df=10), data=airquality)> library(mgcv) > data(airquality) > airq.gam <- gam(Ozone~s(Solar.R)+s(Wind)+s(Temp), sp=c(1,3,7),data=airquality) > print(airq.gam)を実行すると
Estimated degrees of freedom: 1.5182 1.4512 1.1420 total = 5.111303などが出てきます。これが、sp=c(1,3,7)に対応する等価自由度です。 spはsmoothing parameter(平滑化パラメータ)です。 竹澤 2011-03-29 (火) 14:16:07
ken (2011-03-18 (金) 21:41:05)
はじめまして。
お世話になります。
私はpackage(cluster)内の関数pamを用い、クラスター分析を行っています。
たとえば、> plot(pam(data, 3), ask=TRUE)で、Silhouette plotやClusplotを作成した場合、グラフ上に個々のデータラベル(個体番号)を表示させるにはどうしたらよいのでしょうか。
上の例で、Silhouette plotやClusplot上で3クラスターに分けることができても、どの個体がどのクラスターに属しているのかが分からず困っています。
大変初歩的な質問で恐縮ですが,御教示いただければ幸いです.
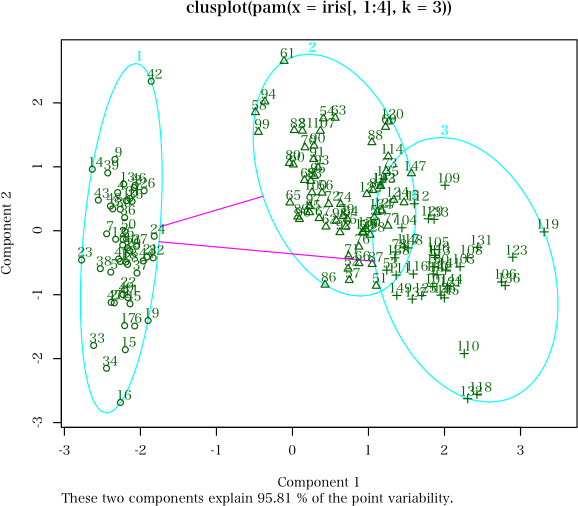
> a <- pam(iris[,1:4],3)
> cbind(iris[,5], a$clustering)
[,1] [,2]
[1,] 1 1
[2,] 1 1
[3,] 1 1
:
[149,] 3 3
[150,] 3 2
> table(iris[,5], a$clustering)
1 2 3
setosa 50 0 0
versicolor 0 48 2
virginica 0 14 36taipapa (2011-03-14 (月) 17:40:04)
お世話になります.
長文になりますが,お許しください.
ある疾患104人の治療効果の判定をロジスティック回帰モデルで行っています.
応答変数はOutcome は良と悪の2値で,説明変数は,9つのカテゴリー変数(a to i),6つの連続変数(J to O)からなり,eventであるOutcomeが悪は25例でした.
まず,下記の用にフルモデルを作成しました.> FullModel <- glm(Outcome ~ a+b+c+d+e+f+g+h+i+J+K+L+M+N+O, family=binomial, data=mydata)モデル選択はstepで行ったのですが,得られたモデルは,以下の通りです.
summary(stepModel) Call: glm(formula = Outcome ~ a+b+c+d+ J + K, family = binomial, data = mydata) Deviance Residuals: Min 1Q Median 3Q Max -1.53608 -0.65721 -0.36439 -0.09402 2.83204 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -6.7502 1.8614 -3.626 0.000287 *** a 0.8752 0.6204 1.411 0.158350 b -1.2636 0.5959 -2.120 0.033974 * c 1.4079 0.8331 1.690 0.091028 . d 0.8834 0.6045 1.461 0.143914 T1 3.6828 0.9318 3.952 7.74e-05 *** T2 -0.8963 0.4643 -1.930 0.053555 . --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 114.717 on 103 degrees of freedom Residual deviance: 81.622 on 97 degrees of freedom AIC: 95.622しかし,イベントが25しかないのに,説明変数が6つもあり,良いモデルとは言えず,含まれる変数も臨床的に意味のなさそうなものが入っており,困りました.
そこで,遺伝的アルゴリズムを用いてAIC/BICがもっとも小さいモデルを探してくれるglmulti (http://cran.r-project.org/web/packages/glmulti/index.html)と言うパッケージを用いてみました.
やり方やオプションは,付属のマニュアルにしたがいました.基準はデフォルトのAICcです.GeneticModel <- glmulti(Outcome ~ a+b+c+d+e+f+g+h+i+J+K+L+M+N+O, family=binomial, data=mydata, method="g", report = FALSE, marginality = TRUE, deltaB = 0, deltaM = 0.01, conseq = 6, sexrate = 0.15, imm = 0.2, level = 1)これで得られたモデルは4つの説明変数を有し,かつ,臨床的にも納得のいくものでした.
さらに,このモデルは,anovaを用いた尤度比検定で上記のstepで得られたモデルより有意に優れていました.
glmのsummaryは以下のとおりです.> summary(GeneticModel.glm) Call: glm(formula = Outcome ~ a+b+J+K, family = binomial, data = mydata) Deviance Residuals: Min 1Q Median 3Q Max -1.6986 -0.5952 -0.4402 -0.1794 2.3542 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -5.3365 1.5652 -3.410 0.000651 *** a -1.1344 0.5692 -1.993 0.046268 * b 1.1870 0.7798 1.522 0.127945 J 3.4808 0.8931 3.897 9.72e-05 *** K -0.8159 0.4454 -1.832 0.066993 . --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 114.717 on 103 degrees of freedom Residual deviance: 85.879 on 99 degrees of freedom AIC: 95.879 Number of Fisher Scoring iterations: 5さらに,このモデルは,anovaを用いた尤度比検定で上記のstepで得られたモデルより有意に優れていました.
これで決まりかと思ったのですが,説明- pam(iris[,1:4],3)変数bのp値が0.05をかなり超えてます(0.128).~またオッズ比を見ると,
exp(cbind(OR=coef(GeneticModel.glm), confint(GeneticModel.glm))) Waiting for profiling to be done... OR 2.5 % 97.5 % (Intercept) 0.00481246 0.0001591826 0.08237885 a 0.32160075 0.1006644837 0.96164214 b 3.27732742 0.8119375358 18.53407469 J 32.48671705 6.5308248197 224.16425104 K 0.44222572 0.1700117573 0.98476474やはり,説明変数bのオッズ比の95%信頼区間は0.81 to 18.5で1を跨いでいます.大部分は1より大ですが.
お聞きしたいのは,このようにinformation criteriaが低いという基準でモデルを選択したところ,一部の変数が有意でない場合どうしたら良いかということです.
基準がAICcなので,有意差検定は気にしないで,このモデルで解析を進めてよいものでしょうか?
primitiveな質問で恐縮ですが,ご教示いただければ幸いです.
みゅ (2011-03-07 (月) 16:11:34)
突然このような現象が起こりました。
エラーが表示されるのですがその意味も対処方法も分かりません。
ご存じの方がおられましたら教えてください。よろしくお願い致します。
(環境は、MacOSX10.6.6、R version 2.9.2です。)
まずtest.Rを作りました。
x <- c(1,2,3,4,5) y <- c(10,20,30,40,50) plot(x,y)
つぎにこれを読み込みました。
>load("./test.R") エラー: ファイルのマジック・ナンバーが不正です (ファイルが壊れているかもしれません)データはロードされませんでした 追加情報: Warning message: ファイル 'test3.R' はマジックナンバー 'x <- ' を持ちます 二度を越えるセーブバージョンの使用は廃止予定です
のように表示されます。「マジックナンバー'○○○○○'」の○○○○○の部分には、読み込むファイル(test.R)の一行目の文字が入っているようです。
これまで、このような方法で、このようなエラーが出た事が無いのですが、
どのように対処すればうまく動くようになるでしょうか?
miya (2011-03-07 (月) 14:47:25)
いつもお世話になっております。
Rのコマンドで、データマトリックスに対して「二重中心化」という処理を行ってくれるものはありますか?
論文に出てきたのですが、どのような処理なのかよくわかりません。
ご存じの方いらっしゃいましたら、ご教示いただけると幸いです。
お願いいたします。
set.seed(12345)
# 元の行列
(a <- matrix(sample(12), 3))
# 二重中心化行列
(b <- t(t(a-rowMeans(a))-colMeans(a))+mean(a))
# これも可
# b <- a-outer(rowMeans(a), colMeans(a), "+")+mean(a)
実行結果
> set.seed(12345)
> (a <- matrix(sample(12), 3))
[,1] [,2] [,3] [,4]
[1,] 9 11 7 12
[2,] 10 4 3 1
[3,] 8 2 5 6
> rowMeans(a)
[1] 9.75 4.50 5.25
> colMeans(a)
[1] 9.000000 5.666667 5.000000 6.333333
> mean(a)
[1] 6.5
# 二重中心化やりま〜〜〜す
> (b <- t(t(a-rowMeans(a))-colMeans(a))+mean(a))
[,1] [,2] [,3] [,4]
[1,] -3.25 2.0833333 -1.25 2.4166667
[2,] 3.00 0.3333333 0.00 -3.3333333
[3,] 0.25 -2.4166667 1.25 0.9166667
# 二重中心化の効果
> rowMeans(b) # 行平均
[1] 0.000000e+00 2.220446e-16 -2.220446e-16
> colMeans(b) # 列平均
[1] 0.00000e+00 -5.92119e-16 0.00000e+00 5.92119e-16
> mean(b) # 全体の平均
[1] 0使用2日目 (2011-02-28 (月) 22:12:33)
はじめまして。
Tipsに紹介されているRepeated measured ANOVAの例題を解こうとrep.aovを使ってみたのですが,> 以下にエラー anova(ret.lm, X = anvX[[j]], M = anvM[[j]], idata = lvI, test = "Spherical") : 使われていない引数 (X = ~1, M = ~b, idata = list(b = 1:4), test = "Spherical")と表示され,解析できません。解決策をご教示いただければ幸いです。
OS:Windows7 R version 2.8.1 (2008-12-22) i386-pc-mingw32 locale: LC_COLLATE=Japanese_Japan.932; LC_CTYPE=Japanese_Japan.932; LC_MONETARY=Japanese_Japan.932; LC_NUMERIC=C; LC_TIME=Japanese_Japan.932 attached base packages: [1] datasets graphics stats utils grDevices tcltk methods base other attached packages: [1] Rcmdr_1.4-8 car_1.2-12
ハローあべちゃん (2011-02-26 (土) 16:22:44)
R version 2.12.0 (2010-10-15) Platform: i386-pc-mingw32/i386 (32-bit)です。
パッケージgplotsのplotmeans()について疑問です。
ヘルプによると、"Plot Group Means and Confidence Intervals"となっていて、平均と信頼区間をプロットするという関数のように書いてありますが、http://cse.naro.affrc.go.jp/takezawa/r-tips/r/68.htmlによると、plotmeans()は平均値±標準偏差をプロットすると説明がされています。実際に自分で試してみたとところ、標準偏差がプロットされているとも、信頼区間がプロットされているともいえないものでした。
plotmeansはいったい何をプロットしているのでしょうか?単に自分の使い方やためしたものが誤っているだけなのでしょうか?
ためしたものデータフレームDF group data 1 1 77.4 2 1 78.2 3 1 78.1 4 1 77.8 5 1 77.9 6 2 78.3 7 2 78.2 8 2 78.4 9 2 77.3 10 2 79.1 11 3 79.2 12 3 79.3 13 3 79.1 14 3 78.2 15 3 79.3 16 4 78.9 17 4 78.8 18 4 78.1 19 4 78.1 20 4 78.9 DF$group <- factor(DF$group) plotmeans(DF$data ~ DF$group) result <- aov(DF$data ~ DF$group) result.pre <- predict(result, interval="confidence") points(1:4, unique(result.pre[,"lwr"]), pch=2, col=2) points(1:4, unique(result.pre[,"upr"]), pch=2, col=2) m <- sapply(1:4, function(x) mean(DF$data[DF$group == x]) ) s <- sapply(1:4, function(x) sd(DF$data[DF$group == x]) ) points(1:4, m+s, pch=3, col=3) points(1:4, m-s, pch=3, col=3)
カワウソ (2011-02-26 (土) 09:54:29)
種ごとに各変数の分散を求めることは以下のように簡単にできますが,種ごとに2変数の共分散を求めるにはどうすればよいのでしょうか?> tapply(iris$Sepal.Length,iris$Species,var) setosa versicolor virginica 0.1242490 0.2664327 0.4043429たとえば,Sepal.LengthとPetal.Lengthの共分散を,
setosa versicolor virginica 0.01635510 0.1828980 0.3032898と出力したいのですが,2変数の場合applyは使えませんよね?byとかaggregateも試してみたのですが,上手くいきません.
> sapply(levels(iris$Species),
function(x) cov(as.matrix(iris[iris[5]==x,
c("Sepal.Length","Petal.Length")]))[1,2])
setosa versicolor virginica
0.01635510 0.18289796 0.30328980> sapply(split(iris, iris$Species),
function(x)
cov(x$Sepal.Length, x$Petal.Length))
setosa versicolor virginica
0.01635510 0.18289796 0.30328980 河童の屁は,河童にあらず,屁である。 (2011-02-25 (金) 22:16:32)
以下の二つが,違うものだと解釈されるのは,それもそうかなあと思うものの,ほかの関数では(変数の指定において)同じものと解釈されることもあるようで,その区別がどこにあるのかなあとか,どちらかに統一(広い解釈の方に)した方がよいのではないかなあと。まあ,どちらで指定するか自明でしょう。という意見はともかくとして,一般ユーザとは異なる,「コア」なメンバーはかたくなな解釈を続けるんでしょうか。# 変数名で指定した場合 > head(iris[Petal.Length]) 以下にエラー `[.data.frame`(iris, Petal.Length) : オブジェクト 'Petal.Length' がありません # 変数名の文字列で指定した場合 > head(iris["Petal.Length"]) Petal.Length 1 1.4 2 1.4 3 1.3 4 1.5 5 1.4 6 1.7
> X <- data.frame(x=1:3, y=10:12) > X["x"] x 1 1 2 2 3 3 > x <- 1 > X[x] x 1 1 2 2 3 3 > x <- 2 > X[x] y 1 10 2 11 3 12 > x <- 3 > X[x] 以下にエラー `[.data.frame`(X, x) : undefined columns selected
hevel (2011-02-25 (金) 17:21:54)
平滑化スプラインを使った回帰計算の後の結果を利用して偏残差プロットを書こうとして行き詰まりました。
初めはsmooting.spline関数を使っていたのですが、回帰にはsregを使うべきであることを発見しました。
http://www.image.ucar.edu/GSP/Software/Fields/Help/sreg.html
しかし、この計算結果を使って偏残差プロットを描けなくて困ってます。
日時と気温のデータ(下記のようなデータ)を使った場合で試しています。day,1,2,3,4,5,6,7,8,9,10,11,12,13,14, 15,16,17,18,19,20 temp,25,20,18,22,23,24,28,26,21,23,24, 27,26,22,25,26,24,20,23,25※ 投稿の都合上横並べですが、実際には縦に並んだtextファイルです。
このデータから下記のような計算を行いました。y <- read.table("temp.txt", header=T) day<-y[, 1] temp<-y[, 2] library(fields) library(spam) y.sr<-sreg(day, temp, df=5, method = "GCV")これでエラー表示なく計算が完了したのですが、この計算結果がうまく利用できません。
残差プロットや偏残差プロットを描きたいのですが、どうすれば計算結果を使って描けるでしょうか?
ぐし (2011-02-24 (木) 16:08:20)
参考書を読んでいて、以下のような文がありました。
データフレームx から、列がID とweight のラベルの列のみ抽出するには次のように実行する
subset(x ,, c(“ID”, “weight”) )
ここで、,,は何を示しているのかhelp(subset)をつかって調べたところ、## S3 method for class 'data.frame'
subset(x, subset, select, drop = FALSE, ...)
の部分から、,,はsubset, select,のsubsetとselectを省略したのではないかと思いました。しかし、c(“ID”, “weight”)の部分はselectでないなら一体何でしょうか?
GARCH (2011-02-23 (水) 11:30:34)
garch関数を使用してパラメータの推計を行いたいのですが、返ってくる結果に***** FALSE CONVERGENCE *****とあり収束できていないようですが、以下のようにパラメータは返ってきます。このパラメータは使用してもいいものなのでしょうか。また、FALSE CONVERGENCEを解決する方法はございますでしょうか。何卒よろしくお願いいたします。
Coefficient(s): Estimate Std. Error t value Pr(>|t|) a0 3.569e-06 5.868e-07 6.082 1.19e-09 *** a1 8.174e-02 6.977e-03 11.716 < 2e-16 *** b1 8.949e-01 9.361e-03 95.603 < 2e-16 ***
ぐし (2011-02-22 (火) 16:29:28)
reshapeの意味がよくわかりません。
一応help(reshape)で調べました。
まずDescriptionに、
This function reshapes a data frame between ‘wide’ format with repeated measurements in separate columns of the same record and ‘long’ format with the repeated measurements in separate records.
とありますが、このなかのwideとlongの意味がわかりません。
次に、timevarとidvarの違いがよくわかりません。timevarが変数そのものであり、idvarは変数の名前だと書いてあるように思うのですが、引数の取り方も違いがないように見えます。以下、説明文です。
timevar the variable in long format that differentiates multiple records from the same group or individual.
idvar Names of one or more variables in long format that identify multiple records from the same group/individual. These variables may also be present in wide format.
よろしくお願いします。
name month x 1 A 1 43.0 2 A 2 41.9 3 A 3 43.6 4 B 1 67.4 5 B 2 27.4 6 B 3 33.9 7 C 1 56.5 8 C 2 59.0 9 C 3 57.5 10 D 1 47.7 11 D 2 43.0 12 D 3 55.1被験者(name)が A〜D の 4 人で,それぞれの人が 1, 2, 3 月(month)に測定され,そのデータ(x)が記録されています。
w <- reshape(data, idvar="name", timevar="month",
v.names="x", direction="wide")
とすれば,"wide" フォーマットで表現したデータフレームになります。
> w name x.1 x.2 x.3 1 A 43.0 41.9 43.6 4 B 67.4 27.4 33.9 7 C 56.5 59.0 57.5 10 D 47.7 43.0 55.1"wide" と "long" は相互に変換できるので,もし,w を "long" にするには,
l <- reshape(w, direction="long")とすれば,以下のように(元の)"long" フォーマットで表したデータフレームになります(順番が変わりますけど,内容は同じです)。
> l
name month x
A.1 A 1 43.0
B.1 B 1 67.4
C.1 C 1 56.5
D.1 D 1 47.7
A.2 A 2 41.9
B.2 B 2 27.4
C.2 C 2 59.0
D.2 D 2 43.0
A.3 A 3 43.6
B.3 B 3 33.9
C.3 C 3 57.5
D.3 D 3 55.1
解析プログラムによって,いずれかのデータ形式しかサポートしないと言うことがあるために,このようなデータフレーム形式の変換を行う必要があるわけですね。 -- 河童の屁は,河童にあらず,屁である。 2011-02-22 (火) 17:17:25tyai (2011-02-21 (月) 16:50:07)
文献等で記載されているfSeriesパッケージのgarchFit関数を使用しようとしましたところ、エラー: 関数 "garchFit" を見つけることができませんでしたというエラーになってしまいます。
> help(package="fSeries")でfSeriesを見たところ、確かにgarchFit関数がないのですが、実際どのパッケージにあるのかご教示いただけますでしょうか?
miura (2011-02-20 (日) 21:18:31)
はじめまして。
表題の件で、Googleなど検索してみたのですが、解決策を見いだせませんでしたので、質問させていただきます。
e1071パッケージでsvm関数を利用して、データを解析しておりましたが、predict関数でprobability = TRUEを指定した場合と、指定しない場合で、異なる結果が得られてしまいました。原因を特定できずに、困っております。
環境は、R version 2.12.1 (2010-12-16) Platform: x86_64-apple-darwin9.8.0/x86_64 (64-bit)です。(Windows7のR-64bitでも同様の結果でした。)
kernlabパッケージのspamデータで、同様の現象が再現できましたので、そのときの手順をお示しいたします。> library(kernlab) - spamデータセットのみ利用しました > library(e1071) > data(spam) > m1 <- svm(type~., data=spam, probability = TRUE) > p1 <- predict(m1, spam) > p2 <- predict(m1, spam, probability = TRUE) > table(p1, p2) p2 p1 nonspam spam nonspam 2834 14 spam 0 1753上記のように、同じモデルに対して、同じデータを与えたにもかかわらず、probability = TRUEを指定すると、結果が異なっています。その理由を特定できずに、悩んでおります。
よろしくお願いいたします。
質問者 (2011-02-20 (日) 03:11:35)
ネットで R の問題集みたいなのをみつけて勉強しています。その中で問題の意味と答えの意味がわかりません。使用verは2.12.1です。
問題(空欄補充)
データフレーム measurement の1列目以外のデータに対して1 列目を基準として分割したの要約統計量を求めるには、コンソールから(?)を実行する。
解答by(measurement[-1], measurement[1], summary)まず、問題の「1 列目を基準として分割したの要約統計量」とはつまり「1列目と2列目以降を分割した要約統計量」と理解したのでいいのでしょうか?
それと、解答がおかしいような気がするのですが、私の勘違いでしょうか?
実際にRでやってみると解答通りのコマンドだとエラーになりますし、そもそも列を扱うなら[,-1]のようにカンマが必要な気がします。
実際にRでやって見る際のデータフレームはmeasurement <- matrix(1:110, nrow=10, ncol=11, byrow=T)と作りました。
よろしくお願いします。
yamada (2011-02-18 (金) 10:55:14)
http://127.0.0.1:26704/library/stats/html/arima.sim.html
内の用語に関する質問です。
�変数nの説明文中の、before un-differencing の意味
�変数n.statの説明文中の、burn-in period の意味
についてご存じの方がいればご教示ください。一般的な意味ではなく、ARIMAモデルに即した具体的な意味です。よろしくお願いします。
ランゲル・ハンス (2011-02-15 (火) 08:07:36)
いつも掲示板を拝見しております。
下記のデータがあるとします。
因子ごとの平均値(mean)の棒グラフを描き、棒の先端に最小値(lower)と最大値(upper)の範囲を示すエラーバーをつけたいと思います。
いろいろ検索してみたのですが、このタイプの棒グラフ+エラーバー(最小〜最大)は見つけられませんでした。
どうぞよろしくお願いいたします。data factor mean lower upper A 50 40 55 B 30 19 45 C 55 25 60
muchlonger (2011-02-13 (日) 16:17:27)
勝ち負け引き分けの時系列データがあります。
勝率(遡n個、勝ち数/n)が安定状態から外れてきたかどうかを最新の10データくらいで判定したいです。
ヒント、検索キーワードをいただけないでしょうか。
yamada (2011-02-12 (土) 23:04:12)
パッケージvarsをダウンロードした後、読み込むと以下のようなエラーメッセージがでました。どうすれば解決するでしょうか。> local({pkg <- + select.list(sort(.packages(all.available = TRUE)), + graphics=TRUE) + if(nchar(pkg)) library(pkg, character.only=TRUE)}) 要求されたパッケージ MASS をロード中です 要求されたパッケージ strucchange をロード中です 要求されたパッケージ zoo をロード中です 要求されたパッケージ sandwich をロード中です Error in gzfile(file, "rb") : コネクションを開くことができません 追加情報: 警告メッセージ: In gzfile(file, "rb") : 圧縮されたファイル 'C:/Users/fujimot/Documents/R/win-library/ 2.12/sandwich/data/Rdata.rdx' を 開くことができません, 理由は 'No such file or directory' です エラー: パッケージ 'sandwich' をロードできませんでしたなお、使用環境は以下の通りです。
> sessionInfo() R version 2.12.1 (2010-12-16) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base
taka (2011-02-10 (木) 13:46:31)
エクセルで書いたグラフのように、グラフの上にポインターをもっていくとそこの場所や値が表示したいのですが、どうのようにすればよいのでしょうか?
istya (2011-02-08 (火) 20:43:35)
グーグルアースで作成したkmlファイルをArcGISで表示させることが目的です。
グーグルアースについては本HPでも取り上げられており、それらしいことは書いてあるのですが、まだ理解できませんので、質問させて頂きます。
■内容
グーグルアースにて、いくつかプロットした地点のまとまりをkml形式で保存したファイルを対象に、Rを使ったshpファイルへの変換はどうすればよいのでしょうか?
以下を参考にしてみましたが、readOGRの指定すら分かりません。library(rgdal) x<-readOGR("c:/GISdata/gunma.kml","gunma") writeOGR(x2,"c:/GISdata/gunmak","gunmak", "ESRI Shapefile")以上、よろしくお願い致します。
R Version 2.12.1(2010-12-16)
$ ogr2ogr -f "ESRI Shapefile" "hoge" KML_Samples.kml $ ls hoge Absolute and Relative.dbf Google Campus.dbf Paths.dbf Absolute and Relative.prj Google Campus.prj Paths.prj Absolute and Relative.shp Google Campus.shp Paths.shp Absolute and Relative.shx Google Campus.shx Paths.shx Extruded Polygon.dbf Highlighted Icon.dbf Placemarks.dbf Extruded Polygon.prj Highlighted Icon.prj Placemarks.prj Extruded Polygon.shp Highlighted Icon.shp Placemarks.shp Extruded Polygon.shx Highlighted Icon.shx Placemarks.shxogr2ogrはgdalに含まれます。rgdalパッケージが動くなら、gdalは入っているでしょうから、すでにインストールされていると思います。ogr2ogrを無理矢理Rの中で使うなら、
> system("ogr2ogr -f "ESRI Shapefile" "hoge
" KML_Samples.kml")
となるでしょう。次に、readOGR()を使う場合ですが、
> M <- readOGR("KML_Samples.kml",
"Absolute and Relative")
> class(M)
[1] "SpatialPolygonsDataFrame"
attr(,"package")
[1] "sp"
とすれば、SpatialPolygonsDataFrameクラスが作成されるので、writeSpatialShape()などでshapefile形式に保存できると思います。ただし、この場合は、どうやって"Absolute and Relative"というレイヤー名を調べるのかという問題が残ります。これはkmlの中を見るか、system()からogrinfoを利用するしかないと思います(もしかしたら、私が知らないだけでよい方法があるかも)。
br;おかしいとは思ったが,数式の書き方にも,(数学的)常識が必要。- ' を持ちます
二度を越えるセーブバージョンの使用は廃止予定です
のようなエラーが出た事が無いのですが、
どのように対処すればうまく動くようになるでしょうか?
>kmlを直接shapefile形式に変換した方が早いと思いますがいかがでしょうか。
それが目的なので、非常に有益な情報で感謝致します。
変換できるようになりました。
ただ、日本語が文字化けしてしまいます。
何か対策はあるんでしょうか? -- istya 2011-02-10 (木) 12:03:38
しまだ (2011-02-07 (月) 23:08:42)
インターバルカメラで取得した風景画像(群)からコントラスト比の時間変化を求めたいのです。もちろんそのような関数があればありがたいのですが、探したところbiOpsやrimageでも見あたらなかったので、お伺いする次第です。
以下のような手順を考えております。
(1)画像を読み込み
(2)複数のピクセルをランダムサンプリングし
(3)そこから最大輝度と最小輝度を得て
(4)コントラスト比=最大輝度/最小輝度
Rでこのようなことは可能でしょうか?R version 2.11.1 (2010-05-31) i386-pc-mingw32
> library(biOps)
> data(logo) # 画像例
> str(logo)
int [1:275, 1:362, 1:3] 255 255 255 255 255
255 255 255 255 255 ...
- attr(*, "type")= chr "rgb"
- attr(*, "class")= chr [1:2]
"imagedata" "array"
> # RGB値を輝度値に変換(RGB値がこの順に並んでいるとして)
> lum <- 0.298912 * logo[,,1] + 0.586611 *
logo[,,2] + 0.114478 * logo[,,3]
> str(lum)
num [1:275, 1:362] 255 255 255 255 255 ...
> fivenum(lum) # 5数要約
[1] 0.0000 124.1494 165.8013 255.0003 255.0003
> hist(lum) # ヒストグラム
> plot.ecdf(lum) # 経験分布関数Cu (2011-02-05 (土) 22:09:49)
お世話になります。使用環境はCentOS 5.1 、 R 2.10.1 です。
以下のようなC言語で記述されたスクリプトがあります。
1.カレントディレクトリにあるデータファイルを読み込む
2.計算結果をテキストファイルで保存する
3.gnuplotで画面上にグラフを表示するとともに、そのグラフをpng形式で保存する
コマンドラインでこのスクリプトを実行すると正常に動作しますが、Rからsystem()で実行すると処理が途中で止まってしまい、プロンプトも帰ってきません。「ctl+Z」でRに戻ってくることはできました。
具体的には2の計算結果のテキストファイルは保存されるのですが、3の画面上に表示されたグラフが不完全な状態で停止するので、gnuplotへうまく命令が伝わっていないように感じています。
「Rから他言語利用」を見てみましたが、コマンドラインで実行できるのにRで実行できない理由がわかりませんでした。
提供できる情報が抽象的ですが、何か対策をご存知でしたら教えて頂ければ幸いです。
絵馬 (2011-02-05 (土) 13:30:02)
こんにちは。
以下のようなdataの各要素に、prefixを行方向に連結したいと考えています。data <- matrix(1:9, 3, 3) prefix <- c("1:", "2:", "3:") ・・・ 処理結果 1:1 2:4 3:7 1:2 2:5 3:8 1:3 2:6 3:9paste()やapply()を組み合わせていろいろやってみたのですがうまくできません。
また、実際にはdataの列数はfor文の中で1~100列程度に変化します。
なるべくif文を使わずにスマートに記述したいので、何か良いアイデアがあれば。
宜しくお願いします。
> (Data <- matrix(as.character(data), 3, 3))
[,1] [,2] [,3]
[1,] "1" "4" "7"
[2,] "2" "5" "8"
[3,] "3" "6" "9"
> (Prefix <- matrix(prefix, 3, 3, byrow=TRUE))
[,1] [,2] [,3]
[1,] "1:" "2:" "3:"
[2,] "1:" "2:" "3:"
[3,] "1:" "2:" "3:"
> matrix(paste(Prefix, Data, sep=""), 3, 3)
[,1] [,2] [,3]
[1,] "1:1" "2:4" "3:7"
[2,] "1:2" "2:5" "3:8"
[3,] "1:3" "2:6" "3:9"> t(matrix(paste(prefix, t(data), sep=""),
ncol(data)))
[,1] [,2] [,3]
[1,] "1:1" "2:4" "3:7"
[2,] "1:2" "2:5" "3:8"
[3,] "1:3" "2:6" "3:9"
> t(apply(data, 1, function(x) paste(prefix, x, sep=""))) # なども> matrix(paste(prefix, t(data), sep=""),
ncol=ncol(data), byrow=TRUE)
[,1] [,2] [,3]
[1,] "1:1" "2:4" "3:7"
[2,] "1:2" "2:5" "3:8"
[3,] "1:3" "2:6" "3:9"akira (2011-02-01 (火) 12:47:03)
いつもありがとうございます。
e1071パッケージのpredict.svmで超平面からの距離はdecision valuesで正しいのでしょうか?
ここを参考に思い込んでいたのですが。> data <- subset(iris,subset=Species!="versicolor") > data <- data.frame(class=factor(data$Species), data[,1:4]) > model <- svm(class~.,data=data[-1,],lernel="linear") > predict(model,data[1,]) 1 setosa Levels: setosa virginica > predict(model,data[1,],decision.values=TRUE) 1 setosa attr(,"decision.values") setosa/virginica 1 1.080803 Levels: setosa virginicaこの1.080803が超平面からの距離ではないのでしょうか?
初心者KT (2011-01-31 (月) 22:24:19)
R初心者です。
この質問の背景から説明します。
自分は数学系、物理系、工学系の専門教育は受けたことがなく、医学系の仕事をしています。しかし今回幾つかの解析をする必要性に迫られています。
その内容は、下記のようなものです。
30個の点が、xyzを与えられています。
1. その30個の点の中心の座標を得る。
2. その30個で囲まれた平面の面積を得る。しかし30個の点は同一平面上にはない。
3. 30個の点が、“最も平均的に通る平面”(平面Aとする)を設定する
4. 平面Aの上に81個の点の様な構造物がドーム状にかぶさっている(曲面Bとする)。
5. しかし曲面Bの部分によっては、平面Aより下に存在する。
6. この曲面Bの表面積を測定する
7. 曲面Bと平面Aで囲まれ、かつ平面Aより上にある部分の体積を測定する
8. 曲面Bと平面Aで囲まれ、かつ平面Aより下にある部分の体積を測定する
この様なことが、Rで可能でしょうか。
よろしくお願いします。
30個の点X Y Z 56.8458 56.3972 95.2184 58.1262 55.2657 96.9028 61.0080 53.8846 97.6925 63.7117 52.5191 98.1147 65.9630 51.1603 98.0884 68.0598 49.8240 97.4626 70.3595 48.6390 96.2383 73.0480 47.8221 94.6007 76.0860 47.6060 92.8562 79.2539 48.1658 91.3380 82.2386 49.5663 90.3180 84.7249 51.7420 89.9494 86.4678 54.5090 90.2453 87.3370 57.6021 91.0912 87.3282 60.7262 92.2840 86.5478 63.6101 93.5838 85.1780 66.0516 94.7681 83.4311 67.9458 95.6757 81.5042 69.2908 96.2331 79.5429 70.1705 96.4590 77.6191 70.7182 96.4472 75.7289 71.0677 96.3317 73.8088 71.3037 96.2427 71.7678 71.4224 96.2643 69.5302 71.3165 96.4051 67.0781 70.7936 96.5927 64.4841 69.6327 96.6992 61.9234 67.6774 96.5962 59.6531 64.9476 96.2303 57.9525 61.7413 95.6913曲面Bの構成要素
X Y Z 66.5214 52.3693 100.1890 69.0172 55.2805 98.6580 71.1465 57.6134 98.0792 73.4542 60.2416 96.9707 74.2816 61.4516 95.2830 75.8534 63.4528 93.5104 78.1356 65.5883 94.6485 79.3441 66.4813 96.3968 81.8057 68.7355 97.8613 69.4359 50.9051 99.5420 71.1025 54.5844 99.2688 71.9337 56.6633 98.3382 72.8589 58.7782 97.9511 73.5783 60.6862 96.7916 74.3058 62.7413 95.2100 74.8671 64.3807 93.8145 76.4086 67.2760 95.2155 78.2037 70.3165 97.9255 72.8598 48.6686 97.5556 73.0553 53.8495 97.0382 73.1433 56.6261 96.5826 73.3167 59.6391 96.9136 73.3231 61.7117 95.9461 73.3418 63.9996 95.0022 73.3420 65.9646 94.0229 73.5266 67.9228 94.9986 73.9282 71.3346 97.5466 76.6417 48.3653 95.4308 75.5219 52.4555 96.4391 74.9038 54.7740 97.3086 74.0257 57.8910 97.6356 73.1674 60.8503 97.5045 72.5119 63.0188 96.9356 71.6926 65.7293 96.2245 71.1617 67.6709 96.7142 70.0804 71.6452 97.8153 81.0857 49.4645 93.7371 77.7238 54.0901 94.3492 75.9176 56.5834 95.7828 74.4364 58.6303 97.2458 73.4941 59.9329 98.2465 72.1696 61.7559 98.5740 71.0152 63.3431 98.6402 69.0276 66.0708 98.0351 66.2486 69.8901 97.9546 85.2473 52.2150 93.0255 81.2928 54.9562 92.2683 79.3713 56.2273 93.0238 76.9566 57.8602 93.3170 75.3852 58.7892 95.9743 72.8658 60.3924 98.1361 70.5020 62.0146 97.9852 67.0597 64.3694 97.9051 62.4689 67.4921 98.1250 88.0380 56.8771 94.4088 84.4938 57.8743 92.7572 81.0642 58.7234 92.3931 78.9147 59.1500 93.2910 75.6953 59.7277 95.2876 72.8867 60.1105 98.3210 68.8648 61.0533 98.4587 65.0531 61.9484 98.5723 59.3881 63.4470 96.9480 87.7385 61.3387 95.9346 84.2589 61.3849 93.4009 80.7455 61.1783 92.9229 78.1388 60.8384 94.1021 74.5410 60.2960 96.3323 70.9528 59.7412 98.6698 67.4871 59.4730 98.7284 63.3083 59.2362 98.0871 58.8063 58.9720 97.4714 85.0653 65.4926 97.8556 83.9668 65.5278 94.3416 80.8889 64.4879 91.7567 77.4482 62.5331 93.9201 75.0270 61.1638 95.4027 73.0548 59.9064 97.5156 70.2444 58.5210 97.9352 66.1804 56.5065 98.6132 62.4675 54.5575 99.9249
p <- persp(range(A$x),range(A$y),
matrix(rep(0,4),ncol=2),
zlim=range(A$z),theta=10,
phi=50,r=10)
xyz <- trans3d(A$x,A$y,A$z,p)
polygon(xyz$x,xyz$y,col="red")
> S <- function(p1,p2,p3) {
w1 <- p1-p3; w2 <- p2-p3
area <- as.vector(0.5*sqrt((w1%*%w1)*
(w2%*%w2)-(w1%*%w2)^2))
return(area)
}
> S(c(1,2,3),c(1,1,1),c(5,0,2))
[1] 4.716991
> D <- c(56.8458,56.3972,95.2184,58.1262,...)
> D <- matrix(D, ncol=3, nrow=30, byrow=TRUE)
> F <- function(x) sum((x[1]*D[,1]+x[2]*D[,2]+
x[3]*D[,3]+1)^2)/sum(x^2)
> (x <- optim(c(0.1,0.1,0.1),F)) # 適当な初期値で最適化
$par
[1] 0.09792275 0.37648968 -0.32677544
$value
[1] 1366.468
$counts
function gradient
116 NA
$convergence
[1] 0
$message
NULL
> (x <- optim(x$par,F)) # 上で求めた解を初期値として繰り返す
$par
[1] -0.001709210 0.001085967 -0.009890907 # これが解 a,b,c ?
$value
[1] 64.76301
$counts
function gradient
158 NA
$convergence
[1] 0
$message
NULL
↓アニメーションにならないならブラウザを変えましょう
> library(geometry)
> eps <- 10^{-14} #ほとんどゼロの幅
# xy平面の単位正方形にepsの厚みを加えた三次元立方体の8個の頂点の座標を並べた行列
> E <- rbind(c(0,0,0),c(1,0,0),c(0,1,0),c(1,1,0),
c(0,0,eps),c(1,0,eps),c(0,1,eps),c(1,1,eps))
> x <- convhulln(E, options = "FA") # オプションFAで実行
> x
$hull # Eの8個の点の凸包の表面を形成する12組の三角形分割
[,1] [,2] [,3]
[1,] 4 3 1 # 3点 E[4,],E[3,],E[1,] からなる
[2,] 4 2 1 # 三角形という意味
[3,] 6 2 1
[4,] 6 5 1
[5,] 6 4 2
[6,] 6 4 8
[7,] 7 3 1
[8,] 7 5 1
[9,] 7 4 3
[10,] 7 4 8
[11,] 7 6 5
[12,] 7 6 8
$area # 凸包の表面積
#(eps を極小に取ったため、上下の正方形の面積)
[1] 2
$vol # 凸包の体積 1x1xeps
[1] 1e-14
> D # 質問者の例の点群
[,1] [,2] [,3]
[1,] 56.8458 56.3972 95.2184
[2,] 58.1262 55.2657 96.9028
[3,] 61.0080 53.8846 97.6925
(途中省略)
[29,] 59.6531 64.9476 96.2303
[30,] 57.9525 61.7413 95.6913
> x <- convhulln(D, options = "FA")
> x # その凸包は56組の三角形からなる
$hull
[,1] [,2] [,3]
[1,] 11 10 1
[2,] 13 24 14
[3,] 26 13 30
(途中省略)
[55,] 8 9 1
[56,] 8 7 1
$area # 凸包の表面積
[1] 1194.405
$vol # 体積
[1] 1264.764
> length(unique(as.vector(x$hull)))
[1] 30 # この場合凸包は30個の点すべてを通る
# 81個の点群の場合
> x <- convhulln(DD, options = "FA")
> x$area
[1] 1116.008
> x$vol
[1] 1644.317
> length(unique(as.vector(x$hull)))
[1] 24 # 凸包は24個の点だけで構成されている!
> # 超平面 ax+by+cz+d=0 に
# 点 p=c(X,Y,Z) から下ろした垂線の足の座標
> # v=c(a,b,c) と置く。
# sum(v*ft(v,d,p))+d=0 となる
> ft <- function(v,d,p)
p - (sum(v*p)+d)/(sum(v*v))*v
> # p=(X,Y,Z) に対する aX+bY+cZ+d の値
> # v=c(a,b,c)。c > 0 の時
# fft(v,d,p) > 0 (< 0)
# ならば点 p は超平面の上(下)側
> fft <- function(v,d,p) sum(v*ft(v,d,p))+d
> # 先に計算した超平面Aに対する a,b,c,dの値。
# cの値が正になるように調整
> v <- c(0.001709210,-0.001085967,0.009890907)
> d <- -1
# v,d を固定した関数 fft
> FFT <- function(p) fft(v,d,p)
# 81個の点に対する ax+by+cz+d の値
> x <- apply(DD,1,FFT)
# 超平面Aの上及び上側にある点のインデックス
> xupper <- which(x >= 0)
# 超平面Aの下側にある点のインデックス
> xlower <- which(x < 0)
# v,d を固定した関数 ft
> FT <- function(p) ft(v,d,p)
# 超平面Aの上側にある点の平面Aへの射影 の座標行列
> xyzupper <- t(apply(DD[xupper,],1,FT))
# 超平面Aの下側にある点の平面Aへの射影 の座標行列
> xyzlower <- t(apply(DD[xlower,],1,FT))
>
> # 例:平面Aの上側にある4つの点から定められる面
#(二つの三角形からなる)
> # と平面Aで囲まれる図形の表面積と体積を求める
> # 二つの三角形ごとに処理、共通する面の面積
# (の二倍)を最後に引く
> D0 <- rbind(c(60,60,100),c(70,60,120),
c(60,70,110),c(70,70,100))
> tri1 <- c(1,2,4) # その三角形分割
> D1 <- D0[tri1,] # 対応座標だけを取り出す
> # D1 と D1 の点から平面Aに下ろした点の
# 凸包(三角柱)の表面積と体積を求める
> x1 <- convhulln(rbind(D1,t(apply(D1,1,FT))),"FA")
> c(x1$area,x1$vol)
[1] 419.3777 211.3755
> tri2 <- c(1,3,4) # もうひとつの三角形分割
> D2 <- D0[tri2,] # 対応座標だけを取り出す
> x2 <- convhulln(rbind(D2,t(apply(D2,1,FT))),"FA")
> c(x2$area,x2$vol)
[1] 353.1260 360.3836
> eps <- 10^{-10} # 極小の幅
# 二つの三角形分割に共通する二点 c(1,4)
> tri3 <- intersect(tri1,tri2)
> # 共通辺をxy方向に少し平行移動してできる
# 平行四辺形の4頂点の座標行列
> D3 <- rbind(D0[tri3,],D0[tri3,]+c(eps,eps,0))
> x3 <- convhulln(rbind(D3,t(apply(D3,1,FT))),"FA")
> c(x3$area,x3$vol) # 体積はほとんどゼロ
[1] 8.284987e+01 9.942196e-10
# D0と平面Aで囲まれる図形の体積
> x1$vol+x2$vol
[1] 571.7591
# D0と平面Aで囲まれる図形の面積(共通側面面積の2倍を引く)
> x1$area+x2$area-x3$area
[1] 689.6538
> # Dは空間点群のx,y,z座標を並べた3列行列
> # TはDの点をある方法で三角形分割したものの
# 点番号を並べた3列の整数行列
> # (任意の二つの三角形は辺を除いて重ならないとする)
> # ax+by+cz+d=0が参照超平面Aを定義(c=0でないとする)
> # Dの点はすべてAの上側か下側にあるとする
> Poly.av <- function(D,T,a,b,c,d) {
+ require(geometry) # 必要なパッケージを読み込み
+ nD <- nrow(D) # 点の総数
+ nT <- nrow(T) # 三角形分割の総数
+ eps <- 10^{-10}*c(1,1,0) # 僅かな平行移動用ベクトル
+ Area <- 0; Volume <- 0 # 面積、体積初期値
+ # 超平面 ax+by+cz+d=0 に
+ # 点 p=c(X,Y,Z) から下ろした垂線の足の座標
+ FT <- function(p)
+ p - (sum(c(a,b,c)*p)+d)/(a^2+b^2+c^2)*c(a,b,c)
+ for (i in seq(nT)) { # 各三角形について繰返し
+ cat("番号 ",i," 対応三角形 (",T[i,],")",fill=TRUE) # デバッグ用
+ D0 <- D[T[i,],] # 対応座標行列
+ D1 <- t(apply(D0,1,FT)) # D0の各点から超平面に下ろした足の座標行列
+ x <- convhulln(rbind(D0,D1),"FA")
+ Area <- Area + x$area # 三角柱の共通側面の面積の2倍分余計に加算
+ Volume <- Volume + x$vol
+ }
+ for (i in 1:(nT-1))
# すべての三角形の組 i,j について繰返し
+ for (j in (i+1):nT) {
+ itsc <- intersect(T[i,],T[j,]) # 共有頂点番号
+ if (length(itsc)==2) { # 二つの三角形が一辺を共有すれば
+ cat("共通辺 (", itsc, ")",fill=TRUE) # デバッグ用
# その辺をxy方向で少し平行移動した平行四辺形
+ D0 <- rbind(D[itsc,],D[itsc,]+eps)
+ x <- convhulln(rbind(D0,t(apply(D0,1,FT))),"FA")
+ Area <- Area - x$area # 共有側面の面積の(約)2倍を引く
+ }}
+ return(list(area=Area,volume=Volume))
+ }
>
> # 簡単な例:6点からなる点群
> D <- rbind(c(60,60,120), #P1
+ c(70,60,110), #P2
+ c(80,60,160), #P3
+ c(70,70,100), #P4
+ c(80,70,140), #P5
+ c(80,80,100)) #P6
> T <- rbind(c(1,2,4),c(2,3,4),
c(3,4,5),c(4,5,6)) #その三角形分割(の例)
> v <- c(0.001709210,-0.001085967,
0.009890907,-1) # 参照超平面の係数
> (x <- Poly.av(D,T,v[1],v[2],v[3],v[4]))
番号 1 対応三角形 ( 1 2 4 )
番号 2 対応三角形 ( 2 3 4 )
番号 3 対応三角形 ( 3 4 5 )
番号 4 対応三角形 ( 4 5 6 )
共通辺 ( 2 4 )
共通辺 ( 3 4 )
共通辺 ( 4 5 )
$area
[1] 2285.054
$volume
[1] 994.3007
> D <- c(56.8458,56.3972,95.2184,58.1262,...)
> D <- matrix(D, nrow=30,ncol=3,byrow=TRUE)
> (ctr <- apply(D,2,mean))# 30個の点群の重心座標
[1] 73.21028 60.43731 95.08736
> DD <- c(66.5214, 52.3693, 100.1890,69.0172, ...)
> DD <- matrix(DD,nrow=length(DD)/3,ncol=3, byrow=TRUE)
> v <- c(0.001709210,-0.001085967,
0.009890907,-1) # 参照超平面の係数
> vv <- v[-4]
vv <- vv/sqrt(sum(vv^2)) # (a,b,c)を長さ1に
> # 座標行列 D を点 cg を原点、
# 方向ベクトル vv が (0,0,1) となるように変換
> Rot <- function(D,vv,cg) {
+ D0 <- sweep(D,2,cg) # Dの各座標からcgを引く
# オイラー角 x[1],x[2],x[3]に対する回転行列
+ tmp1 <- function(x) {
+ a <- x[1]; b <- x[2]; c <- x[3]
+ R1 <- rbind(c(1,0 ,0 ),
+ c(0,cos(a),-sin(a)),
+ c(0,sin(a),cos(a) ))
+ R2 <- rbind(c(cos(b) ,0,sin(b)),
+ c(0 ,1,0 ),
+ c(-sin(b),0,cos(b)))
+ R3 <- rbind(c(cos(c),-sin(c),0),
+ c(sin(c),cos(c) ,0),
+ c(0 ,0 ,1))
+ R1%*%R2%*%R3 }
# vvをtmp1(x)で回転したベクトルとc(0,0,1)とのズレ
+ tmp2 <- function(x) {
+ W <- tmp1(x)%*%vv-c(0,0,1)
+ sum(W*W) }
+ # 方向ベクトル vv を (0,0,1) に移す
# 回転行列のオイラー角を求める
+ # 初期値 (0,0,0) から始め念の為に
# 最適化を三回繰り返す
+ angle <- optim(optim(optim(c(0,0,0),
tmp2)$par,tmp2)$par,tmp2)$par
+ t(tmp1(angle)%*%t(D0)) # 変換した座標行列を返す
+ }
> DX <- Rot(DD,vv,apply(D,2,mean))
> DX[1:2,]
[,1] [,2] [,3]
[1,] -5.098094 -9.352273 4.733394
[2,] -3.240725 -5.936903 3.342893
# tripackパッケージを読みこみ
> library(tripack)
# z座標が非負の点だけを取り出す
> DXupper <- DX[which(DX[,3]>=0),1:2]
# そのドロネー三角分割を計算
> tri.upper <- tri.mesh(DXupper[,1],DXupper[,2])
# 三角分割のプロット
> plot(tri.upper)
# 三角分割要約
> summary(tri.upper)
triangulation:
Call: tri.mesh(x = DXupper[, 1],
y = DXupper[, 2])
number of nodes: 25
number of arcs: 65
number of boundary nodes: 7
number of triangles: 41
number of constraints: 0
# 三角分割(各列が三角形のノード 番号)
> triangles(tri.upper)[,1:3]
node1 node2 node3
[1,] 1 21 9
[2,] 1 9 8
[3,] 1 8 17
(途中省略)
[40,] 22 24 23
[41,] 23 24 25
>
# z座標が非負の点だけを取り出す
> DXlower <- DX[which(DX[,3]<=0),1:2]
# そのドロネー三角分割を計算
> tri.lower <- tri.mesh(DXlower[,1],DXlower[,2])
# 三角分割のプロット
> plot(tri.lower)
# 三角分割要約
> summary(tri.lower)
triangulation:
Call: tri.mesh(x = DXlower[, 1], y = DXlower[, 2])
number of nodes: 56
number of arcs: 156
number of boundary nodes: 9
number of triangles: 101
number of constraints: 0
# 三角分割(各列が三角形のノード 番号)
> triangles(tri.lower)[,1:3]
node1 node2 node3
[1,] 1 12 51
[2,] 1 51 50
[3,] 1 50 48
(途中省略)
[100,] 49 51 52
[101,] 52 53 54
まめ (2011-01-28 (金) 09:29:53)
R初心者です。
Rコマンダーで箱ひげ図を描画する時に、箱ひげ図の層別プロットができません。
何か考えられることはありますでしょうか?
ぐし (2011-01-28 (金) 03:04:17)
いつもRを起動するとGUIプリファレンスのフォントの設定を変更しなければ日本語が表示されません.
起動と同時にMSgothicで表示させる方法はないのでしょうか?
使用OSはwin7で、Rは2.4.1です。
??? (2011-01-27 (木) 15:54:11)
setwd("D:\\") d1<-read.table("data3.csv", header=TRUE, sep=",") head(d1) n.d1 <- nrow(d1) mean.d1 <- apply(d1, 2, mean) sd.d1 <- sd(d1) cor.d1 <- cor(d1) data.frame(n.d1, mean.d1, sd.d1, cor.d1) library(sem) model.1 <- specify.model() x1 <- f1, b11, NA x2 <- f1, b21, NA x3 <- f1, b31, NA x4 <- f1, b41, NA x5 <- f1, b51, NA x6 <- f1, b61, NA x7 <- f1, b71, NA x8 <- f1, b81, NA x9 <- f1, b91, NA x10 <- f1, b101, NA x11 <- f1, b111, NA x12 <- f1, b121, NA x13 <- f1, b131, NA x14 <- f2, b142, NA x15 <- f2, b152, NA x16 <- f2, b162, NA x17 <- f2, b172, NA x18 <- f2, b182, NA f1 <-> f1, NA, 1 f2 <-> f2, NA, 1 f1 <-> f2, c12, NA sem.model.1 <- sem(model.1, cor.d1, N=nrow(d1)) summary(sem.model.1)とうつと,
以下にエラー scan(file, what, nmax, sep, dec, quote, skip, nlines, na.strings, : scan 関数は 'a real' を期待したのに、得られたのは 'N=nrow(d1))'と出ます。
ちなみに,スクリプトの「N=nrow(d1)」を「N=n.d1」に変えても結果は一緒でした。
scan関数なんて使っていないのになぜでしょう??
白雪姫 (2011-01-26 (水) 03:04:34)
こんにちは。
R version 2.11.1 を mac で使っております。
例えばv <- rep( c(1, 2, 3, 5, 6), c(4, 3, 4, 2, 1) )というベクトルがあります。
require(gtools) combinations( length(v), 3, v, set=FALSE, repeats=FALSE )とすると、364 個の答が返ってきます。(364*3 の matrix です。)
この中で unique な raw だけに興味があります。つまり、c(1,2,3) というベクトルが48回繰り返されていますが、求める答は 1,2,3 が1つしか含まれないものです。
もう少し長い v で combination の長さも長い応用を考えているので、効率の良い答を探しています。
よろしくお願いします。
効率の悪いのは、こんな感じです。unique( combinations( length(v), 3, v, set=FALSE, repeats=FALSE ) )combinations() を全て羅列してから条件に合うものだけを取り出す、というのが何となく効率が悪いような気がします。
getComb <- function(v, n)
{
search <- function(x, y)
{
if (y == 1) {
a <<- rbind(a, rep(v1, um[1,]))
}
else if (x == 1) {
if ((t <- um[1, 1]-um[y, 1]) >= 0) {
um[1, 1] <<- t
search(nc, y-1)
um[1, 1] <<- um[1, 1]+um[y, 1]
}
}
else {
search(x-1, y)
while (um[y, 1] && um[1, x]) {
um[y, x] <<- um[y, x]+1
um[y, 1] <<- um[y, 1]-1
um[1, x] <<- um[1, x]-1
search(x-1, y)
}
um[y, 1] <<- um[y, 1]+um[y, x]
um[1, x] <<- um[1, x]+um[y, x]
um[y, x] <<- 0
}
}
f1 <- table(v)
v1 <- as.numeric(names(f1))
um <- matrix(0, 2, length(v1))
um[,1] <- c(n, sum(f1)-n)
um[1,] <- f1
a <- NULL
search(nc, 2)
return(a)
}
v <- rep( c(1, 2, 3, 5, 6), c(4, 3, 4, 2, 1) )
n <- 7
system.time(b <- unique(combinations(
length(v), n, v, set=FALSE, repeats=FALSE )))
system.time(a <- getComb(v, n))
実行結果は以下の通り
unique(combinations(... による b とは,出力順が異なる
> head(a)
[,1] [,2] [,3] [,4] [,5] [,6] [,7]
[1,] 3 3 3 3 5 5 6
[2,] 2 3 3 3 5 5 6
[3,] 1 3 3 3 5 5 6
[4,] 2 2 3 3 5 5 6
[5,] 1 2 3 3 5 5 6
[6,] 1 1 3 3 5 5 6
> tail(a)
[,1] [,2] [,3] [,4] [,5] [,6] [,7]
[87,] 1 1 2 2 2 3 3
[88,] 1 1 1 2 2 3 3
[89,] 1 1 1 1 2 3 3
[90,] 1 1 1 2 2 2 3
[91,] 1 1 1 1 2 2 3
[92,] 1 1 1 1 2 2 2あびこ (2011-01-22 (土) 15:23:13)
現在、自宅でRのパッケージ(sem)をインストールしようとしています。
OSはwin7、Rは2.4.1です。管理者権限でログインしています。Rをインストールする際、設定はすべてデフォルトにしています。初心者なので特にRにインストールしたりはしていません。ネットには繋がっています。
ファイルを探してもsemのファイルはありませんでした。
[パッケージ]→[パッケージのインストール]でインストールしようとしました。しかし以下のようにエラーがでます。英語の意味はわかるのですが、IT関連の知識は余り無いので、どうすれば解決するのかがわかりません。パッケージをインストールするにはどうすればいいのでしょうか?
よろしくお願いします。
以下、エラー文です--- Please select a CRAN mirror for use in this session ---~ 警告:unable to access index for repository http://www.stats.ox.ac.uk/pub/RWin/bin/ windows/contrib/2.4 URL 'http://essrc.hyogo-u.ac.jp/cran/bin/windows/ contrib/2.4/sem_0.9-8.zip' を試しています Content type 'application/zip' length 245050 bytes 開かれた URL downloaded 239Kb 以下にエラーzip.unpack(pkg, tmpDir) : ファイル 'C:/Program Files/R/R-2.4.1/library/fileef56f57/ sem/chtml/sem.chm' を開くことができません > library(sem) 以下にエラーlibrary(sem) : 'sem' という名前のパッケージはありません
yamada (2011-01-21 (金) 10:57:37)
パッケージ"urca"をダウンロードして登録しましたが、ヘルプファイル"C:\Program Files\R\R-2.12.1\library\urca\html"内の
リンク先をクリックしても当該ファイルが存在しません。
例えば最初の.spcvをクリックしても"file:///C:/Program%20Files/R/R-2.12.1/library/urca/html/urca-internal.html"が存在しません。
解決方法はありますでしょうか。
R version 2.12.1 (2010-12-16)
Platform: i386-pc-mingw32/i386 (32-bit)
locale:
[1] LC_COLLATE=Japanese_Japan.932
LC_CTYPE=Japanese_Japan.932
[3] LC_MONETARY=Japanese_Japan.932
LC_NUMERIC=C
[5] LC_
[2,] TIME=Japanese_Japan.932
attached base packages:
[1] stats graphics grDevices utils
datasets methods base
other attached packages:
[1] urca_1.2-4
loaded via a namespace (and not attached):
[1] grid_2.12.1 lattice_0.19-13 nlme_3.1-97 tools_2.12.1 みゅ (2011-01-20 (木) 16:17:19)
ABCDEをEDCBAのように逆順に並べたいのですが、これに対応するfunctionは有りますか?
数字なら例えばrev()で良いと思うのですが、> A <- 1:5 > A [1] 1 2 3 4 5 > rev(A) [1] 5 4 3 2 1一般に文字列の場合はどのようにするのでしょうか。
1文字ずつに分割した文字列について、order()などで順序番号の逆順を生成してから再度並び替えるとやるのでしょうか???
もしfunctionが有るようなら教えて下さい。調べましたが解りません。
> A <- "ABCDE"
> system(paste("echo",A,"|rev"),intern=TRUE)
[1] "EDCBA"> a <- "abcdefghij" > paste(rev(unlist(strsplit(a, ""))), collapse="") [1] "jihgfedcba" > Napoleon <- "Able was I ere I saw Elba" > paste(rev(unlist(strsplit(Napoleon, ""))), collapse="") [1] "ablE was I ere I saw elbA"
せこがに (2011-01-19 (水) 07:32:58)
べき分布x^-uに従う乱数を発生させたいと思っています。xを確率変数として、uとxの最小値(a)だけを与えて乱数が発生させれたらいいなと思っています。下に示した関数を定義してx^-uに従う乱数を発生させようとしているのですが、これでうまくいっているのでしょうか?それとxの最大値を無限大に拡張したいのですが、どなたかアイディアをお持ちの方はいらっしゃいませんか?pdf <- function (n, u=1, a=1) { x <- runif(n, min=a, max=1000) p <- x^-u y <- s vv ample(x, n, replace=T, prob=p) return(y) }
rpower <- function(n, m=1, M=100, b=0.1)
{
((M^-b-m^-b)*runif(n)+m^-b)^(1/-b)
}
> xの最大値を無限大に拡張したいcentury (2011-01-19 (水) 00:27:39)
初歩的なことなのかもしれませんが、optim関数について教えてください。
以下のようなプログラムがあります。flb <- function(x) { p <- length(x); sum(c(1, rep(4, p-1)) * (x - c(1, x[-p])^2)^2) } ## 25-dimensional box constrained optim(rep(3, 25), flb, NULL, method = "L-BFGS-B", lower=rep(2, 25), upper=rep(4, 25)) # par[24] is *not* at boundaryの中の
sum(c(1, rep(4, p-1)) * (x - c(1, x[-p])^2)^2)はどういった計算でしょうか。
c(1, rep(4, p-1))と
(x - c(1, x[-p])^2)^2は計算できるのですが、sumが良く分かりません。
自分でsum(c(1,2,3)*(1,2,3))と書いてもエラーで上手く行きません。
なんで元々のプログラムではうまくいくのでしょうか?
実行したら普通にできるのですが、何をしているのか理解したく、お教え頂けますと幸いです。
また、lower=rep(2, 25), upper=rep(4, 25)というのはxに初期値として入れたrep(3,25)が25個のデータなので、その全てに2以上4以下という制約があるということでしょうか。
> (x <- c(3, 5, 2)) # 例えば,こんな例を [1] 3 5 2 > (p <- length(x)) # x の長さ(要素数) [1] 3 # 1 と 4 を p-1 個繰り返した要素を持つベクトル > c(1, rep(4, p-1)) [1] 1 4 4 # 1 と x の p 番目の要素を除いたものを要素とするベクトル > c(1, x[-p]) [1] 1 3 5 > c(1, x[-p])^2 # その二乗 [1] 1 9 25 > x - c(1, x[-p])^2 # x から引き算 [1] 2 -4 -23 > (x - c(1, x[-p])^2)^2 # さらにその二乗 [1] 4 16 529 > c(1, rep(4, p-1)) * (x - c(1, x[-p])^2)^2 # ベクトルの積 [1] 4 64 2116 > sum(c(1, rep(4, p-1)) * (x - c(1, x[-p])^2)^2) # その要素の和 [1] 2184> xに初期値として入れたrep(3,25)が25個のデータなので、その全てに2以上4以下という制約があるということでしょうか
河童の屁は,河童にあらず,屁である。 (2011-01-18 (火) 22:33:38)
お作法というのもいろいろ流儀があって,一通りでは済まないとは思うものの,せめて
{ } では,インデントしましょう
<- の前後には半角空白を置きましょう
, の後には半角空白を置きましょう
for, if, else if の後には半角空白を置きましょう
{ の左側には半角空白を置きましょう
少なくとも ==, !=, >, <, <=, >=, &, &&, |, ||, ! の前後には半角空白を置きましょうa <- function(b, c) { d <- e+f if (g > 0) { h <- i+8 } else { h <- i+90 } return(h*900) }
century (2011-01-17 (月) 14:44:50)
最尤推定で多くのパラメータが含まれた尤度関数を扱っているのですが、optim関数ではあてはまりが悪く、nlm関数で再度やってみたいと思っています。そこで、プログラムの最後のoptim関数の部分だけnlm関数に変えれば実行できると思ったのですがうまくいきませんでした。
具体的には、optim(par=c(0.927, 0.982, 0.091, 0.058, 0.604, 1.129, 0.368, 0.326), fn=Likelihood(ε, R), control=list(fnscale=-1, trace=TRUE))を
nlm(Likelihood, c(0.927, 0.982, 0.091, 0.058, 0.604, 1.129, 0.368, 0.326), hessian=TRUE)にしたのですが、それ以外にどこを直すと回るのでしょうか。
※c()は初期パラメータです。。。
色々nlm関数のページも見ましたが、分からず、御教授頂けますと幸いです。
あと、一部のパラメータだけ違う制約を置くことが可能でしたらお教え頂けますでしょうか。
> f <- function(x) sum((x-1:length(x))^2)
> nlm(f, c(10,10))
$minimum
[1] 4.303458e-26
$estimate
[1] 1 2
$gradient
[1] 2.757794e-13 -3.099743e-13
$code
[1] 1
$iterations
[1] 2
> optim(c(10, 10), f,
control=list(reltol=1e-14))
$par
[1] 0.9999998 1.9999994
$value
[1] 4.218356e-13
$counts
function gradient
103 NA
$convergence
[1] 0
$message
NULLε<-x[,10]
R<-x[,5]
y<-c(1:305)
Likelihood<-function(ε,R){
return(function(par){
p<-par[1]
q<-par[2]
ω_0<-par[3]
α_0<-par[4]
β_0<-par[5]
μ_0<-0.1
ω_1<-par[6]
α_1<-par[7]
β_1<-par[8]
μ_1<--0.2
LogL<-0
E<-(1-p)/(2-p-q) # t=0の定常確率(スタート)
F<-(1-q)/(2-p-q)
ε_0<-ε[1] # t=1の確率密度関数計算
R_1<-R[2]
A<- q*E+(1-p)*F
B<- (1-q)*E+p*F
sigma_0_1<-sqrt(ω_0+α_0*ε_0^2+β_0*ε_0^2)
sigma_1_1<-sqrt(ω_1+α_1*ε_0^2+β_1*ε_0^2)
f1<-1/sqrt(2*pi*sigma_1_1^2)*exp(-(R_1-μ_1)^2/
(2*sigma_1_1^2))
f0<-1/sqrt(2*pi*sigma_0_1^2)*exp(-(R_1-μ_0)^2/
(2*sigma_0_1^2))
f<- f0*A+f1*B
LogL<-LogL+log(f)
E<-f0*A/(f0*A+f1*B)
F<-f1*B/(f0*A+f1*B)
G<-E
H<-F
C<-A
D<-B
ε_1<-ε[2] #t=2
R_2<-R[3]
A<-q*E+(1-p)*F
B<-(1-q)*E+p*F
sigma_0_2<-sqrt(ω_0+α_0*ε_1^2+β_0*
(sigma_0_1^2*C+sigma_1_1^2*D))
sigma_1_2<-sqrt(ω_1+α_1*ε_1^2+β_1*
(sigma_0_1^2*C+sigma_1_1^2*D))
f1<-1/sqrt(2*pi*sigma_1_2^2)*
exp(-(R_2-μ_1)^2/(2*(sigma_1_2)^2))
f0<-1/sqrt(2*pi*sigma_0_2^2)*
exp(-(R_2-μ_0)^2/(2*(sigma_0_2)^2))
f<-f0*A+f1*B
LogL<-LogL+log(f)
E<-f0*A/(f0*A+f1*B)
F<-f1*B/(f0*A+f1*B)
A=q*E+(1-p)*F
B=(1-q)*E+p*F
C=q*G+(1-p)*H
D=(1-q)*G+p*H
sigma0t<-sigma_0_2
sigma1t<-sigma_1_2
z<-2 #t=3以降
ε_t<-0
R_t<-0
for(i in y){
z<-z+1
ε_t<-ε[c(z)]
R_t<-R[c(z+1)]
A<-q*E+(1-p)*F
B<-(1-q)*E+p*F
sigma0t<-sqrt(ω_0+α_0*ε_t^2+
β_0*((sigma0t)^2*C+(sigma1t)^2*D))
sigma1t<-sqrt(ω_1+α_1*ε_t^2+
β_1*((sigma0t)^2*C+(sigma1t)^2*D))
f1<-1/sqrt(2*pi*(sigma1t)^2)*
exp(-(R_t-μ_1)^2/(2*(sigma1t)^2))
f0<-1/sqrt(2*pi*(sigma0t)^2)*
exp(-(R_t-μ_0)^2/(2*(sigma0t)^2))
f<-f0*A+f1*B
LogL<-LogL+log(f)
E<-f0*A/(f0*A+f1*B)
F<-f1*B/(f0*A+f1*B)
C<-A
D<-B
}
return(LogL)
})
}
optim(par=c(0.927,0.982,0.091,0.058,
0.604,0.9,0.368,0.326),
fn=Likelihood(ε,R),
control=list(fnscale=-1, trace=TRUE))
このプログラムで計算されたパラメータに書き換えて、最初の
Likelihood<-function(ε,R){
return(function(par){
と最後の
return(LogL) }) }を外したら最終的なLogLの値が計算され、それらしい値になったのですが、この確認ではうまく計算できているか確かめたことにはならないでしょうか・・・
> ε<-x[,10]
エラー: オブジェクト 'x' がありません
> R<-x[,5]
エラー: オブジェクト 'x' がありません
:
エラー: オブジェクト 'z' がありません
> return(LogL)
エラー: オブジェクト 'LogL' がありません
> })
エラー: 予想外の '}' です ( "}" の)
> }
エラー: 予想外の '}' です ( "}" の)
> optim(par=c(0.927,0.982,0.091,0.058,
0.604,0.9,0.368,0.326),fn=Likelihood(ε,R),
control=list(fnscale=-1, trace=TRUE))
Nelder-Mead direct search function minimizer
以下にエラー function (par) :
関数 "Likelihood" を見つけることができませんでした
こんな単純なエラーが出ないように提示することが要求されるのです。つうといえばかーというようにはなかなか行かないなあ。 -- 河童の屁は,河童にあらず,屁である。 2011-01-17 (月) 20:36:11> x <- matrix(runif(305*2), 305) > ε<-x[,10] エラー: 添え字が許される範囲外です > R<-x[,5] エラー: 添え字が許される範囲外ですあなたのいいたかったのは「2行×305列の行列」?何でそうなのか(2×305の行列?)もよく分からないし,たとえ,そこをパスしても,その後のエラーは起きます。あなたが,プログラムをちゃんとコピーペーストしたなら,あなたのコンソールでも同じエラーが起きているはずなのです。そうでないというなら,あなたがプログラムのコピーペーストを誤ったということなんです。
set.seed(12345) ε<-runif(308) R<-runif(309)としてやってみると。以下のようなエラーが50個以上(関数の参照ごとにほぼいつも起きるのだろう)
44: In log(f) : 計算結果が NaN になりました 45: In sqrt(ω_0 + α_0 * ε_t^2 + β_0 * ((sigma0t)^2 * C + ... : 計算結果が NaN になりましたまあ,それはほうっておいて,結果を見てみると,収束が非常に遅くて,maxit のデフォルトの500回では収束しない。10000にしてやってみたら6000回ほどでどうにか収束する。
NewFunctionName <- function(par) { 関数名は何でも良い
p<-par[1]
以下同じ(この中で εや R を参照すると,大域変数が参照されるので,引数で渡す必要がない
return(LogL)
}
このように定義した関数を使って
nlm(NewFunctionName, c(0.927,0.982,0.091, 0.058,0.604,0.9,0.368,0.326), hessian=TRUE)で動く。
y <- c(1:7)
ε <- c(0.104794415, 0.042511256, 0.125477647,
0.314732511, -0.411718232,
0.670690239, -0.006636651, -1.099916039,
-0.380745473, 1.415399685)
R <- c(0.043765052, 0.019836787, 0.103638308,
0.289968381, -0.442985542,
0.665004049, - 0.050993233, -1.11928331,
-0.362827922, 1.406915882)
Likelihood <- function(ε, R) {
return(function(par) {
p <- par[1]
q <- par[2]
ω_0 <- par[3]
α_0 <- par[4]
β_0 <- par[5]
ω_1 <- par[6]
α_1 <- par[7]
β_1 <- par[8]
μ_0 <- 0.1
μ_1 <- -0.2
LogL <- 0
E <- (1-p)/(2-p-q) # t=0 の定常確率(スタート)
F <- (1-q)/(2-p-q)
ε_0 <- ε[1] # t=1 の確率密度関数計算
R_1 <- R[2]
A <- q*E+(1-p)*F
B <- (1-q)*E+p*F
sigma_0_1 <- sqrt(ω_0+α_0*
ε_0^2+β_0*ε_0^2)
sigma_1_1 <- sqrt(ω_1+α_1*
ε_0^2+β_1*ε_0^2)
f1 <- 1/sqrt(2*pi*sigma_1_1^2)*
exp(-(R_1-μ_1)^2/(2*sigma_1_1^2))
f0 <- 1/sqrt(2*pi*sigma_0_1^2)*
exp(-(R_1-μ_0)^2/(2*sigma_0_1^2))
f <- f0*A+f1*B
LogL <- LogL+log(f)
E <- f0*A/(f0*A+f1*B)
F <- f1*B/(f0*A+f1*B)
G <- E
H <- F
C <- A
D <- B
ε_1 <- ε[2] # t=2
R_2 <- R[3]
A <- q*E+(1-p)*F
B <- (1-q)*E+p*F
sigma_0_2 <- sqrt(ω_0+α_0*ε_1^2+
β_0*(sigma_0_1^2*C+sigma_1_1^2*D))
sigma_1_2 <- sqrt(ω_1+α_1*ε_1^2+
β_1*(sigma_0_1^2*C+sigma_1_1^2*D))
f1 <- 1/sqrt(2*pi*sigma_1_2^2)*
exp(-(R_2-μ_1)^2/(2*(sigma_1_2)^2))
f0 <- 1/sqrt(2*pi*sigma_0_2^2)*
exp(-(R_2-μ_0)^2/(2*(sigma_0_2)^2))
f <- f0*A+f1*B
LogL <- LogL+log(f)
E <- f0*A/(f0*A+f1*B)
F <- f1*B/(f0*A+f1*B)
A <- q*E+(1-p)*F
B <- (1-q)*E+p*F
C <- q*G+(1-p)*H
D <- (1-q)*G+p*H
sigma0t <- sigma_0_2
sigma1t <- sigma_1_2
z <- 2
ε_t <- 0
R_t <- 0
for (i in y) { # t=3 以降
z <- z+1
ε_t <- ε[c(z)]
R_t <- R[c(z+1)]
A <- q*E+(1-p)*F
B <- (1-q)*E+p*F
sigma0t <- sqrt(ω_0+α_0*ε_t^2+
β_0*((sigma0t)^2*C+(sigma1t)^2*D))
sigma1t <- sqrt(ω_1+α_1*ε_t^2+
β_1*((sigma0t)^2*C+(sigma1t)^2*D))
f1 <- 1/sqrt(2*pi*(sigma1t)^2)*
exp(-(R_t-μ_1)^2/(2*(sigma1t)^2))
f0 <- 1/sqrt(2*pi*(sigma0t)^2)*
exp(-(R_t-μ_0)^2/(2*(sigma0t)^2))
f <- f0*A+f1*B
LogL <- LogL+log(f)
E <- f0*A/(f0*A+f1*B)
F <- f1*B/(f0*A+f1*B)
C <- A
D <- B
}
return(LogL)
})
}
optim(par=c(0.9, 0.9, 0.3, 0.3, 0.4,
0.9, 0.2, 0.5), fn=Likelihood(ε, R),
control=list(fnscale=-1, trace=TRUE))
教えて頂いた通りにnlm関数をやってみたら回りました。本当にありがとうございます。
このエラーなのですが、sqrt関数を使っているので、その中の関数値が負になるとNaNになってしまうようです。そこで推定する際の各パラメータ条件を付けたいのですが、L-BFGS-B法だと全てのパラメータに共通の制約ですよね。各パラメータ非負という条件と、par[1]<1、par[2]<1、par[4]+par[5]<1、par[7]+par[8]<1を本来であれば加えたいのですが、可能でしょうか。
εとRの参照がおかしいというのはzとz+1がずれているということでしょうか?
参照するデータが元々のデータと違うのでひとつずれています。紛らわしくて申し訳ありません。
maxit のデフォルトの500回を変更する方法も教えて頂けますでしょうか・・・optim(rep(3, 25), flb, NULL, method = "L-BFGS-B",
lower=rep(2, 25), upper=rep(4, 25))
# par[24] is *not* at boundaryhf (2011-01-15 (土) 23:17:40)
「Rの基礎とプログラミング技法」(U.リゲス)p.176,8.2節に,latticeパッケージでできることについて,「例えば入力データのカテゴリー変数について,その水準ごとにグラフィックスを作成することができる。あるいは変数が多い場合,各変数ごとのグラフィックスを並べて描画できる」と書いてあります。この記述の前半部分の説明はその後に出てくるのですが,後半部分(「あるいは」以降)の方法についてその後の記述から読み取ることができませんでした。「○○を見れば書いてある」というようなことをご存知の方はお教え願えないでしょうか。よろしくお願いします。
splom (~ USArrests[c(3, 1, 2, 4)] | state.region, pscales = 0, types = c("g", "p", "smooth"))barchart (VADeaths, groups = F, layout = c(2,2))
dat.long <- stack (data.wide)
histogram( ~ values | ind, data = data.long,
layout = c(2,2))
latticeではないですけど
library( ggplot2)
p <- ggplot(diamonds, aes(carat)) +
geom_histogram(binwidth = 1)
p + facet_grid(cut ~ .)library(lattice)
x <- as.data.frame(matrix(round(rlnorm(100*10,
log(25), 0.4)), 100))
# xの中身を確認しておく
names(x) <- sprintf("Q%02d", 1:10)
histogram( ~ values | ind, data = stack(x),
layout = c(5, 2))
set.seed(123)
x <- as.data.frame(matrix(round(rlnorm(
100*10, log(25), 0.4)), 100))
# xの中身を確認しておく
names(x) <- sprintf("Q%02d", 1:10)
layout(matrix(1:10, byrow=TRUE, ncol=5))
old <- par(mar=c(2, 2.7, 1.3, 1),
mgp=c(1.6, 0.7, 0))
for (i in 1:ncol(x)) {
hist(x[, i], breaks=seq(-5, 100, by=5),
xlim=c(0, 80), ylim=c(0, 25),
xlab="", main=sprintf("Q%02d", i),
col="darkorchid1")
}
par(old)
layout(1)
Jun (2011-01-15 (土) 23:11:54)
お世話になります。
組み込み関数の定義を調べたい場合、
1.関数名を()なしで入力する ex. order
2.methods()とgetS3metod()を組み合わせる ex.mean
という方法がありますが、なぜこのような違いが生じるのでしょうか?
関数の定義を隠す意味がよく分からないのですが、何か目的があってのことなのですか?
何かご存知でしたらご教授願います。
century (2011-01-14 (金) 01:19:36)
こんばんは。
Rを使って複数のパラメータからなる尤度関数を最大化し、最尤推定でパラメータを求めたいのですが、一部のパラメータに非負制約が必要です。
optim関数の最後に「trace=TRUE」と付けたのですが、どうにも負の値を返してきます。
どなたかお分かりになりますでしょうか?宜しくお願い致します!
cat (2011-01-13 (木) 03:25:26)
こんばんは。
R version 2.9.2 を使用している者です。a <- c("a.1", "a.2", "b.1", "b.2", "c.1", "c.2") b <- array(a, dim=6)上記のようなデータにsplitを使用し "a.1", "a.2" と "b.1", "b.2" と "c.1", "c.2" のように頭から2つずつ取り出したいので
y <- seq(1, 6, 2) split(b, y)としましたが(seqの基準点で分けたい)
$`1` [1] "a.1" "b.2" $`3` [1] "a.2" "c.1" $`5` [1] "b.1" "c.2"のような出力になります。
本掲示板を拝見しましたが解答を見つけられなかったので、どなたかご教示願えないでしょうか。
宜しくお願い致します。
> a <- c("a.1", "a.2", "b.1", "b.2",
"c.1", "c.2")
> split(a, rep(1:3, each=2))
$`1`
[1] "a.1" "a.2"
$`2`
[1] "b.1" "b.2"
$`3`
[1] "c.1" "c.2"yamada (2011-01-12 (水) 23:08:56)
RGuiのファイル→「履歴の読み込み」は、「履歴の保存」で保存したコマンドを使えるようにしますが、どういうときに有用なのでしょうか。作業スペースを保存するとコマンドを保存するので、次回開いた時は自動的に前回までのコマンド履歴がロードされますし、使用中の回のコマンドも使えます。「履歴の保存と読み込み」の存在意義がわかりません。
「ファイルの保存」は何をしたかというログを保存するだけで、開いてもコマンドが使えないので、過去を記録する以外には使い道はないですよね。
[Workspace restored from /Users/foo/.RData] [History restored from /Users/foo/.Rhistory]って表示されますよ。 -- 河童の屁は,河童にあらず,屁である。 2011-01-16 (日) 14:32:13
ミヤ (2011-01-12 (水) 11:34:36)
現在、PLS回帰分析の勉強をしているのですが、library(pls) data(yarn) yarn.plsr <- plsr(density ~ NIR, 6, data = yarn, validation = "CV")とした後に、
plot(yarn.plsr, "prediction", which = "train", 6, line = T)によって、実測値と予測値の対散布が見れると思うのですが、このとき決定係数(R2)を知ることはできないでしょうか?
よろしくお願いいたします。
ss.t <- sum((yarn$density- mean(yarn$density))^2) ss.e <- sum(yarn.plsr$residuals[,, yarn.plsr$ncomp]^2) 1-ss.e/ss.t
(2011-01-11 (火) 11:44:55)
計算結果で、左に出る[1]って表示を消すことはできないのでしょうか?
なつ (2011-01-10 (月) 11:03:58)
いつもお世話になっております。Rを使って,特定の値の組み合わせが出現する回数を計算したいと考えています。
例えば,特定の組み合わせ="0,1"としまして(順序もこのまま),a <- c(0, 1, 1, 1, 0, 0, 1, 1) b <- c(1, 1, 1, 1, 1, 0, 1, 0)という2つのオブジェクトがあるとします。
この場合,aからは2,bからは1という値を計算したいということです。
何か良い方法がありましたら,教えて頂ければ幸いです。
よろしくお願いいたします。
> sum(unlist(strsplit(gsub("01", "@",
paste(a, collapse="")), "")) == "@")
[1] 2
> sum(unlist(strsplit(gsub("01", "@",
paste(b, collapse="")), "")) == "@")
[1] 1> a <- c(1, 1, 1, 0, 0, 1) > aa <- a[-length(a)] > sum( aa[which(aa==0)+1] == 1 ) [1] NA > a <- c(1, 1, 1, 0, 0, 0) > aa <- a[-length(a)] > sum( aa[which(aa==0)+1] == 1 ) [1] NA
> sum(a[-length(a)]*10^8+a[-1]==0*10^8+1) [1] 2
とし (2011-01-09 (日) 17:37:25)
path worst cost pathpattern throughput stddev Numpulse Nummeasure AveF 1 □ 0 1 0 217.7 0.682583 90441 140519 0.985497 2 ○ 1 1.83 1 118.447 0.255297 163628 254691 0.984438 3 □□ 0 2 0 94.7204 0.29984 392181 609967 0.994922 4 △ 2 2.93 2 74.3604 0.188577 258852 404117 0.985847 5 □□□ 0 3 0 98.3713 0.170828 476040 739740 0.980546 6 □×○□ 0 4 0 97.9405 0.171691 753243 1172179 0.98989このようなデータフレームを用いて、x軸にpathの記号列(〇や、□×○□など)を、y軸に他のパラメータをプロットしようとしていたのですが、記号列同士が被ってしまうので、以下のようにして強引に縦向きで出力させました
df <- read.table("ex_data.txt", header=T) sortlist <- order(df$throughput) df <- df[sortlist,] cs <- as.character(df$path) asdf <- barplot(df$throughput, 0.1, ylim=c(0, 250), xlab="", ylab="", axes=F, ann=F) axis(1, at=asdf, labels=FALSE) axis(2, at=seq(0, 250, 10)) library(gridBase) vps <- baseViewports() pushViewport(vps$inner, vps$figure, vps$plot) grid.text(cs, x=unit(asdf, "native"), y=unit(-1, "lines"), just="right", rot=90) popViewport(3)しかし、
警告メッセージ: 1: In grid.Call.graphics("L_texta.1", as.graphicsAnnot(x$label), ... : 'mbcsToSbcs' 中の 'テ療療療冷味笆。笆。笆。 ' で変換に失敗: <e2>をドットで置き換えましたとなってしまい、.png出力だと問題ないのですが、.eps出力だと×以外の文字(□、○、△b.1)が3個の.に置き換えられてしまいます。
画像出力部のコードは以下のとおりです。dev.copy2eps(file="throughput+cost.eps") dev.copy(png, "throughput+cost.png") dev.off().epsに全角文字を出力するにはどのようにすればいいのでしょうか?
よろしくお願い致します。
のの (2011-01-09 (日) 07:17:48)
データフレーム内の変数名を簡単に変更(rename)する方法はあるのでしょうか?
一応冗長ですが以下の方法で実現はできています。# dataオリジナル設定 > data <- data.frame(x=1:10, y=1:10, z=1:10) > names(data) # 変数名の確認 [1] "x" "y" "z" > data$newx=data$x # 新しい変数名を定義 # データフレームから変数xを削除 > data <- data[setdiff(colnames(data), "x")] > names(data) # 変数名の確認 [1] "y" "z" "newx"変数の削除はデータフレームTips大全を参考にしました。
他にも> library(gdata) > data <- rename.vars(data, c("x", "y", "z"), c("first", "second", "third"))という方法があるようですが、変数名を全部列挙しないといけないようで使いにくいです。
良い方法があればお教えください。
> data <- data.frame(x=1:10,
y=1:10, z=1:10)
> names(data)
[1] "x" "y" "z"
> names(data)[2] <- "Y"
> names(data)
[1] "x" "Y" "z"
> names(data)[names(data)=="x"] <- "X"
> names(data)
[1] "X" "Y" "z"
> names(data)[2:3] <- c("A","B")
> names(data)
[1] "X" "A" "B"
> names(data)[names(data) %in% c("X", "B")] <-
c("foo", "bar")
> names(data)
[1] "foo" "A" "bar"> data <- data.frame(x=1:10, y=1:10, z=1:10)
# x, z のみを foo, bar に変更(付値を忘れないように)
> data <- rename.vars(data, c("x", "z"),
c("foo", "bar"))
Changing in data
From: x z
To: foo bar
> names(data)
[1] "foo" "y" "bar"
> data <- rename.vars(data, "y", "ほげほげ")
Changing in data
From: y
To: ほげほげ
> names(data)
[1] "foo" "ほげほげ" "bar"data <-を忘れて使っていました。ご指摘ありがとうございます。 -- のの 2011-01-09 (日) 19:32:08
てるてるぼうず (2011-01-09 (日) 02:01:34)
以前も同じ質問をさせていただいたのですが、どなたか情報をお持ちでないでしょうか?
関数のヘルプファイルを表示したいと思い、以下を実行するのですが、
?…またはhelp(…)で毎回以下のようなエラーが出ます> ?glm 警告メッセージ: In file.show(temp, title = gettextf("R Help on '%s'", topic), delete.file = TRUE) : file.show():ファイル 'C:\DOCUME~1\蜿、蟾晏忽蠢予 LOCALS~1\Temp\RtmpIDu7yb\Rtxt678418be' は存在しません > help("glm") 警告メッセージ: In file.show(temp, title = gettextf("R Help on '%s'", topic), delete.file = TRUE) : file.show():ファイル 'C:\DOCUME~1\蜿、蟾晏忽蠢予 LOCALS~1\Temp\RtmpIDu7yb\Rtxt3d6c4ae1' は存在しません検索エンジン等で調べてはみましたが、未だ解決していません。 環境は以下の通りです。
Microsoft Windows XP Professional Version 2002 Service Pack 3 Intel(R) Core(TM)2 Duo CPU E8400 @ 3.00GHz 2.99 GHz、976 MB RAMRのバージョン
R version 2.12.1 (2010-12-16)
R初心者 (2011-01-08 (土) 21:01:26)
はじめまして。大学生でゼミでRを扱っているものです。大学のパソコンではRでパッケージのダウンロードができるのですが、家でやったら> utils:::menuInstallPkgs() --- このセッションで使うために、CRANのミラーサイトを選んでください --- install.packages(NULL, .libPaths()[1L], dependencies = NA, type = type) 中で警告がありました: 'lib = "C:/PROGRA~1/R/R-212~1.1/library"' は書き込み可能ではありません 以下にエラー sprintf(msg, userdir) : <83>シ<e3>Ν縺吶k縺溘a縺ォ蛟倶ココ逧<84>縺ェ繝ゥ繧、繝悶Λ繝ェ '%s' 繧剃ス懊j縺溘>縺ョ縺ァ縺吶°<ef>シ<9f> に 不正なマルチバイト文字がありますという表示がでてしまいダウンロードできませんでした。zipでも駄目でした。どうすればいいかご教示ください。
> F <- function(x) sum((x[1]*DD[,1]+
x[2]*DD[,2]+x[3]*DD[,3]+1)^2)/sum(x^2)
# 適当なパラメータ初期値で始める
> (x <- optim(c(0.1,0.1,0.1),F))
$par
[1] 0.09792275 0.37648968 -0.32677544
$value
[1] 1366.468
$counts
function gradient
116 NA
$convergence
[1] 0
$message
NULL
# 上で求めた解を初期値として繰り返す
> (x <- optim(x$par,F))
$par
# これが求めたい a,b,c の値?
[1] -0.001709210 0.001085967 -0.009890907
$value
[1] 64.76301
$counts
function gradient
158 NA
$convergence
[1] 0
$message
NULL
y