![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
新規投稿はできません。
初心者のための R および RjpWiki に関する質問コーナー
過去の記事のアーカイブ。
きむ (2011-01-07 (金) 18:12:19)
いつも参考にさせていただいています。
現在、緯度経度で座標が指定されている地域標準メッシュ(3次メッシュ)の座標をある地点を基準としたx,y座標に換算しようとしています。
そこで、RjpWikiの、「RでGPS」を参考にlibrary(sp)のspDistsN1という関数を用いて距離を出そうとしているですがうまくいきません。library(sp) ll <- matrix(c(34, 34, 138, 139), ncol=2) km <- spDistsN1(ll, ll[1,], longlat=T) sum(zapsmall(km))とすると110.96kmとなりますが正確には92.38kmが正?また、
library(sp) ll <- matrix(c(34, 35, 139, 139), ncol=2) km <- spDistsN1(ll, ll[1,], longlat=T) sum(zapsmall(km))とすると結果が0になってしまいます。使用方法に誤りがあるのだと思いますがhelp等でも良くわかりません。すみませんが使用方法をご指導ください。
また、今のところ上記関数を用いて基準点からの距離で各メッシュの座標を設定しようとしていますが、直接緯度経度をx,y座標(平面直角座標系)に変換できる関数等がありましたら教えていただけると助かります。基本的な質問で恐縮ですがよろしくお願いいたします。
> library(sp)
> ll <- matrix(c(34, 35, 139, 139), ncol=2)
> km <- spDistsN1(ll, ll[1,], longlat=T)
> sum(zapsmall(km))
[1] 84.13467
> ll
[,1] [,2]
[1,] 34 139
[2,] 35 139
> km
[1] 0.00000 84.13467
> zapsmall(km)
[1] 0.00000 84.13467
> sum(zapsmall(km))
[1] 84.13467
どうなっているんでしょうね。 -- 河童の屁は,河童にあらず,屁である。 2011-01-07 (金) 19:04:12> library(sp) > ll <- matrix(c(139, 139, 34, 35), ncol=2) > km <- spDistsN1(ll, ll[1,], longlat=T) > sum(zapsmall(km)) [1] 110.8055なおRのバージョンは2.10.1であり、これを緯度-経度で入力すると結果が0になります。(ver2.9.2では河童様の結果のように84.13km)
> library(sp) > ll <- matrix(c(34, 35, 139, 139), ncol=2) > km <- spDistsN1(ll, ll[1,], longlat=T) > sum(zapsmall(km)) [1] 0次に東経138-139度、北緯34度の距離を出す場合(当初質問の1番目のケース)、測量計算で92.38kmとなるのに対して、経度-緯度で入力すると値が0となります。(逆に緯度-経度で入力すると110.96km)。
> library(sp) > ll <- matrix(c(138, 139, 34, 34), ncol=2) > km <- spDistsN1(ll, ll[1,], longlat=T) > sum(zapsmall(km)) [1] 0
yamada (2011-01-07 (金) 13:46:05)
表のような結果をsink(),print()でテキストファイルに出力しています。実際にクリップボードに格納したいのは、一列目の行ラベルを除いた、jp,hk,usの列だけですが、テキストファイルで範囲選択をすると、行ラベルを含んでしまいます。現在は、エクセルでスペース区切りを指定して開いていますが、スペース区切り等開くまでの操作が手間です。そこで、行ラベルを出力しないか、あるいは、行ラベル付きのデータで行ラベル除いて出力する方法はないでしょうか。jp uk us 1 -3.030863e-02 -2.523841e-02 -4.387615e-03 2 -1.594305e-02 -1.853893e-02 -1.499784e-02 3 9.720749e-03 8.167201e-03 1.648989e-02 4 -1.138466e-02 5.133008e-03 1.188763e-03
tadashi (2011-01-05 (水) 15:08:35)
やりたいことは、1列しかないCSVファイルを2つ読み込んで、共通部分を出力したいのです。できるだけ簡単に行う方法はあるでしょうか?
read.cvs で読み込むと下記のようにうまくいきません。
CSVから、データの作り方がうまくないのだと思いますが、どのコマンドを使えばいいのかがわかりません。> a <- read.csv('temp.1', header=FALSE) > b <- read.csv('temp.2', header=FALSE) > a V1 1 1 2 2 3 3 > b V1 1 3 2 4 3 5 > intersect(a, b) data frame with 0 columns and 0 rows
松代 (2010-12-31 (金) 14:08:35)
グレンジャー因果性の検定量をrestrictで制約した残差と制約無し残差を使って計算すると、教科書の計算結果(Eview)やエクセルの計算結果と大幅な違いが出ました。原因は制約付残差の違いでした。restrictの計算精度は悪いのでしょうか。
hashi (2010-12-30 (木) 17:22:31)
いつもお世話になっております。
デンドログラムの回転方法ですが、青木先生著「Rによる統計解析」P230、図6.26を「初級Q&A アーカイブ(6)クラスター図を横向きで描画したい」の方法、horiz = T を前項のスクリプトのどの場所に挿入すれば回転したデンドログラムが表現できるのでしょうか。以下前項のスクリプトです。set.seed(123) x <- round(matrix(rnorm(100), ncol=5), 3) d <- dist(x) ans <- hclust(d^2, method="ward") pdf("cluster.pdf", height=375/72, width=500/72) plot(ans, hang=-1) dev.off()hang=-1の後ろに書き加え計算すると、
"horiz"はグラフィックスパラメータではありませんとメッセージが出ます。
> hc <- hclust(dist(USArrests)^2, "cen") > par(mar = c(4,1,1,7)) > plot(as.dendrogram(hc), horiz = T)は,ちゃんと動くのだから,
set.seed(123)
x <- round(matrix(rnorm(100), ncol=5), 3)
d <- dist(x)
ans <- hclust(d^2, method="ward")
pdf("cluster.pdf", height=375/72, width=500/72)
plot(as.dendrogram(ans), horiz=TRUE) # @@@ ここをこのように変更する
dev.off()
ということでしょう。 -- 河童の屁は,河童にあらず,屁である。 2010-12-30 (木) 17:35:14akira (2010-12-30 (木) 14:20:04)
いつもありがとうございます.
mvpartパッケージのrpart関数について質問です.
ヘルプを見ると、分岐指標に"gini"と"information"を選べるような記載がありますが、引数"parms"を変更しても結果が変わらないように思います.
同志社大の金先生のHPには、引数"split"とありますが、rpart関数、rpart.control関数は引数"split"を持たないようです.
一方、rpartのコード(40〜63行目ぐらいと思っていますが…)ではparmsが規定しているように見えます.
ご存知の方、いらっしゃいませんか?
> library(rpart)
# @@@ information を使用
> fit <- rpart(Kyphosis ~ Age + Number + Start, data=kyphosis,
+ parms=list(split='information'))
> fit2 <- rpart(Kyphosis ~ Age + Number + Start, data=kyphosis,
+ parms=list(split='gini'))
>
> library(rpart)
> fit <- rpart(Kyphosis ~ Age + Number + Start, data=kyphosis,
+ parms=list(split='information'))
> fit
n= 81
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 81 17 absent (0.79012346 0.20987654)
2) Start>=12.5 46 2 absent (0.95652174 0.04347826) *
3) Start< 12.5 35 15 absent (0.57142857 0.42857143)
6) Age< 34.5 10 1 absent (0.90000000 0.10000000) *
7) Age>=34.5 25 11 present (0.44000000 0.56000000)
14) Number< 4.5 12 5 absent (0.58333333 0.41666667) *
15) Number>=4.5 13 4 present (0.30769231 0.69230769) *
# @@@ gini を使用(デフォルト)
> fit2 <- rpart(Kyphosis ~ Age + Number + Start, data=kyphosis,
+ parms=list(split='gini'))
> fit2
n= 81
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 81 17 absent (0.7901235 0.2098765)
2) Start>=8.5 62 6 absent (0.9032258 0.0967742)
4) Start>=14.5 29 0 absent (1.0000000 0.0000000) *
5) Start< 14.5 33 6 absent (0.8181818 0.1818182)
10) Age< 55 12 0 absent (1.0000000 0.0000000) *
11) Age>=55 21 6 absent (0.7142857 0.2857143)
22) Age>=111 14 2 absent (0.8571429 0.1428571) *
23) Age< 111 7 3 present (0.4285714 0.5714286) *
3) Start< 8.5 19 8 present (0.4210526 0.5789474) *松代 (2010-12-28 (火) 15:54:34)
パッケージVARSを使うために解説書どおりに以下のコマンドを打ち込みましたが、最後コマンドの後に“以下にエラー Canada$e : $ operator is invalid for atomic vectors”という警告が出ました。対応策をご存じの方教えてください。> library(vars) > data(Canada) > layout(matrix(1:4, nrow = 2, ncol = 2)) > plot.ts(Canada$e, main = "Employment", ylab = "", xlab = "")
これ以上の情報はディレクトリ '/Library/Frameworks/R.framework/Versions/2.12/Resources/library/vars/doc' にある以下のビニエット中にあります: vars: VAR, SVAR and SVEC models (source, pdf)いずれにせよ「現行の文書中にはない」。私は,新しい文書を見ているので,古い文書に間違った(当時は正しかったのだろう)情報が書いてあるかもしれないという認識はなかった。
松代 (2010-12-25 (土) 00:07:03)
Windows7+R64 2.12.1でパッケージadaptをインストールできません。解決方法をご教示ください。> install.packages("adapt")~ パッケージを ‘C:\Users\fujimot\Documents/R/win-library/2.12’ 中にインストールします (‘lib’ が指定されていないので) --- このセッションで使うために、CRANのミラーサイトを選んでください --- 警告メッセージ: In getDependencies(pkgs, dependencies, available, lib) : package ‘adapt’ is not available
install.packages("cubature",dep=T) # インストール
library(cubature) # パッケージの呼び出し
?adaptIntegrate # 関数のヘルプ
f <- function(x) cos(x) # 1変数関数の場合
adaptIntegrate(f, 0, pi/2)
g <- function(x) { # 2変数関数の場合
exp(-(x[1]^2+x[2]^2)/2)/(2*pi)
}
adaptIntegrate(g, c(-3, -3), c(3, 3))ランゲル・ハンス (2010-12-21 (火) 09:00:38)
いつも掲示板を参考にさせていただいております。
以前も同じような質問をさせていただいたので、大変恐縮です。
NとMを10区間に区切って、その区間内に入るdの合計と平均を求めたいと思います。
下記の例(results1,results2)ではいくつかNAが出ます。NAを0として計算する方法を教えていただけないでしょうか?
最終的には区間にすべて色の入るlevelplotを作図したいと考えています。N <- c(0, 1, 3, 7, 12, 20, 30, 45, 50, 81) M <- c(10, 20, 40, 60, 65, 75, 90, 95, 98, 100) d <- runif(10, 0, 10) data <- data.frame(cbind(N, M, d)) by1 <- cut(data$N, seq(0, 100, 10), right=TRUE, include.lowest=TRUE) by2 <- cut(data$M, seq(0, 100, 10), right=TRUE, include.lowest=TRUE) results1 <- cbind(tapply(data$d, list(by1, by2), sum)) results2 <- cbind(tapply(data$d, list(by1, by2), mean)) results1 results2 library(lattice) levelplot(results1) levelplot(results2)どうぞよろしくお願いいたします。
なつ (2010-12-20 (月) 22:13:28)
Rを使って,特定の値が連続して出現する回数の最大値を計算する方法を探しています。
例えば,特定の値=1としまして,a <- c(0, 1, 1, 1, 0, 0, 1, 1) b <- c(1, 1, 1, 1, 1, 0, 1, 0)という2つのオブジェクトがあるとします。
この場合,aからは3,bからは5という値を計算したいということです。
何か良い方法がありましたら,教えて頂ければ幸いです。
初歩的な質問で申し訳ありませんが,よろしくお願いいたします。
max(diff(c(0, which(b != 1), length(b)+1))-1)
yos (2010-12-19 (日) 20:39:01)
よろしくおねがいします.
1分おきで1日(0~1440分)に計測された,ある値があります.
1列目が,時間(0,1,2,...,1440)
2列目が,その時間における値(10, 40, ..., 50)
3列目が,グループ(A, B, C)
これを,
X軸を,例えば1時間おきの24分割
Y軸を,A, B, Cの3分割
で,値の平均値をプロットしたいのですが,色々調べてもどうしても方法が分かりません.
ご教授いただけないでしょうか
set.seed(123) d <- data.frame(time=0:1440, value=rnorm(1441), group=sample(factor(LETTERS[1:3]), 1441, replace=TRUE)) d$time2 <- d$time%/%60 d$time2[d$time2 == 24] <- 23 d$time2 <- factor(d$time2) matplot(tapply(d$value, list(d$time2, d$group), mean), type="l")
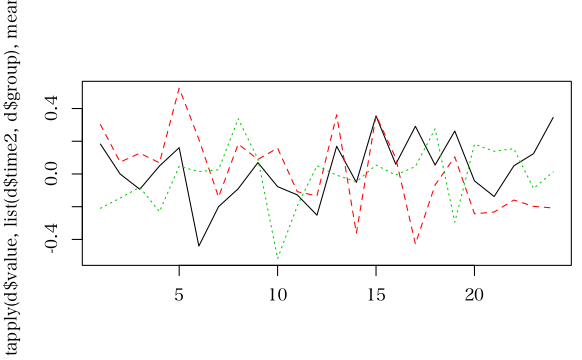
image(x=0:23, y=1:3, tapply(d$value, list(d$time2, d$group), mean), yaxt="n", ylab="", xlab="") axis(2, at=1:3, labels=LETTERS[1:3], pos=-0.5)
Kai (2010-12-12 (日) 15:31:30)
こんにちは。
lm()でデータAの一次回帰式を求め、predict()でデータBを回帰します。A_res <- lm(formula=Volume ~ Girth, data=trees) # データAの回帰結果 B <- trees+rnorm(dim(trees)[1]*dim(trees)[2]) # データBの作成 B_res <- predict(A_res, B$Girth) # データBの回帰B_resの回帰結果から自由度調整済み決定相関係数を求めたいのですが、どのようにすればよいのでしょうか?
ヘルプファイルを読む限りではse.fitという引数が関係ありそうな気がしたのですが、そこから先がよくわかりませんでした。
Rは2.11.1, OSはMac 10.5.8です。宜しくお願いします。
> trees <- data.frame(Volume=c(56, 54, 33, 58, 49), Girth=c(62, 44, 37, 57, 50)) > A_res <- lm(formula=Volume ~ Girth, data=trees) > B <- trees+rnorm(dim(trees)[1]*dim(trees)[2]) > B_res <- predict(A_res, newdata=B) # 予測値 > Se <- sum((B$Volume-B_res)^2) # 残差平方和 > n <- nrow(B) # データ数 > St <- var(B$Volume)*(n-1) # 全平方和 > MSe <- Se/(n-2) # 残差の平均平方 > MSt <- St/(n-1) # 全体の平均平方(不偏分散) > (CorrectedR2 <- 1-MSe/MSt) # 自由度調整済み決定係数という名前には当てはまらない,訳のわからない統計量 [1] 0.3946343
kouka (2010-12-11 (土) 22:07:24)
R全くの初心者です。igraphのパッケージは下記のようにインストールされるのですが、> utils:::menuInstallLocal() パッケージ 'igraph' は無事に開封され、MD5 サムもチェックされましたパッケージを読み込む際に、
> library(igraph) エラー: パッケージ 'igraph' は 'arch=i386' に対してインストールされていません。と表示されます。原因を教えていただけませんか?
YK (2010-12-11 (土) 19:53:30)
はじめまして。
被験者間因子と被験者内因子を含む分散分析に関する質問です。
先日、中級Q&Aに投稿した内容ですが、初級Q&Aが適切ではないかというご指摘をいただき、こちらに再投稿します。
Rのaov、nlmeパッケージのlme、carパッケージのAnovaと岡田先生のrep.aovで同じ結果が得られるのですが、SPSSや桐木先生のWEBの結果と一致しません(SPSSと桐木先生の結果は一致します)。
下記にサンプルを示します。
P値はfactor1, factor2, timeの順に0.044, 0.441, 0.017となりますが、SPSSや桐木先生のWEBでは順に0.042, 0.397, 0.575となります。
ダミーの結果を加えて被験者内因子の水準を3にしてみても結果は一致しません。
他のサンプルデータでも結果が一致するときとしないときがあるようです。
SPSSはType IIIで計算していますが、R側でtype IIIを指定しても一致しません。
原因についてどなたかご教示いただけましたら幸いです。ID <- factor(rep(c(1:32),2)) factor1 <- factor(c(0, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1, 0, 1, 0, 0, 1, 0, 0, 0, 0, 1, 0, 0, 1, 0, 0, 1, 0, 1, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1, 0, 1, 0, 0, 1, 0, 0, 0, 0, 1, 0, 0, 1, 0, 0, 1, 0, 1, 0)) factor2 <- factor(c(0, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 0, 1, 0, 1, 0, 0, 0, 0, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 0, 1, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 0, 1, 0, 1, 0, 0, 0, 0, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 0, 1, 0)) time <- factor(c(rep(1,32), rep(2,32))) result <- c(102.56, 101.80, 123.72, 74.16, 34.12, 61.52, 54.08, 115.08, 58.96, 60.24, 111.68, 122.20, 45.04, 104.84, 121.48, 125.20, 45.48, 217.00, 39.40, 157.48, 119.96, 65.80, 4.44, 39.40, 65.80, 90.20, 45.48, 117.72, 83.04, 156.40, 42.72, 110.92, 135.52, 22.60, 129.08, 127.76, 149.12, 69.92, 73.24, 97.12, 114.92, 54.56, 170.08, 71.60, 179.96, 57.08, 199.88, 126.48, 59.56, 120.40, 53.56, 127.12, 66.48, 75.24, 268.64, 90.88, 102.48, 147.84, 116.84, 84.40, 147.84, 138.76, 122.96, 120.04) df <- data.frame(ID, factor1, factor2, time, result) rep.aov(result ~ I(time) + B(factor1, factor2) + S(ID), df)
net (2010-12-10 (金) 19:24:00)
R初心者です.
ネットワーク分析をして,ネットワーク図を描くときに,辺の太さを辺ごとに調整できるとされています.太さを与えるデータはベクトルでも隣接行列でもよいという解説がありました.
たとえば,重み付きグラフの隣接行列(たとえばwg)でグラフを描き,その重みに応じて各辺の太さを変えたい場合,辺の太さを指定するedge.lwdをつかって具体的にどのように表現したらいいでしょうか.
ちなみに,wgは作業ディレクトリにあるcsvファイルで,
wg<-as.matrix(read.csv("XXXXX(ファイル名)".csv))
で既に定義してあるとします.
私は,
gplot(wg,
edge.lwd = wg)
として,グラフを描こうとしたら,エラーで「使われていない引数があります」として,edge.lwd = wgがでてしまいました.
具体的なスクリプトを教えていただけたら幸いです.
library(sna)
wg <-as.matrix(read.csv("test.csv",header=TRUE))
gplot(wg,
edge.lwd = wg)
test.csvのなかみ
A,B,C,D,E,F
0,8,0,0,0,0
0,0,0,1,0,1
0,6,0,0,0,0
0,0,0,0,0,0
0,0,0,0,0,2
1,0,0,0,2,0しょう (2010-12-10 (金) 02:48:02)
スペース区切りのテキストファイル(数百万行*9列)をRに読み込もうとしています。
ただ、元データの8列目が「備考」欄のため、scan(file="xxx.txt", sep="")を使うと備考のない部分は区切りのスペースと同じ扱いを受けてしまい、matrixを使って行列化するときにずれてしまいます。
read.tableを使うと「'289' 行目には,8 個の要素がありません」とエラーがでてしまい、読み込んでくれません。(288行目までは備考がなく、289行目で初登場のため)
read.delim("xxx.txt")を使うと、空白の備考欄に「NA」を入れてくれ、一番理想に近いのですが、1行目に備考がないため8列のフレームが作られてしまいます。
read.delimで列数を設定できればよいのですが、可能でしょうか?googleやRの書籍などで探したのですが見当たりませんでした。解決方法があればお教え願えると幸いです。よろしくお願いいたします。
R version=2.9.2, OS=Vista
ファイル
1 2 3 4 5 6 7 8
11 12 13 14 15 16 17 18
21 22 23 24 25 26 27 28 comment
31 32 33 34 35 36 37 38
実行結果
> read.delim("test.txt", header=FALSE, sep=" ")
V1 V2 V3 V4 V5 V6 V7 V8 V9
1 1 2 3 4 5 6 7 8
2 11 12 13 14 15 16 17 18
3 21 22 23 24 25 26 27 28 comment
4 31 32 33 34 35 36 37 38 #ファイル 1 2 3 4 5 6 7 8 11 12 13 14 15 16 17 18 21 22 23 24 25 26 27 28 31 32 33 34 35 36 37 38 41 42 43 44 45 46 47 48 51 52 53 54 55 56 57 comment 58 61 62 63 64 65 66 67 comment 68 71 72 73 74 75 76 77 78 #実行結果 read.delim(file="test.txt",sep="",header=F) V1 V2 V3 V4 V5 V6 V7 V8 1 1 2 3 4 5 6 7 8 2 11 12 13 14 15 16 17 18 3 21 22 23 24 25 26 27 28 4 31 32 33 34 35 36 37 38 5 41 42 43 44 45 46 47 48 6 51 52 53 54 55 56 57 comment 7 58 NA NA NA NA NA NA 8 61 62 63 64 65 66 67 comment 9 68 NA NA NA NA NA NA 10 71 72 73 74 75 76 77 78
4 31 32 33 34 35 36 37 38 NA 5 41 42 43 44 45 46 47 48 NA 6 51 52 53 54 55 56 57 comment 58 7 61 62 63 64 65 66 67 comment 68 8 71 72 73 74 75 76 77 78 NAみたいになるだけでしょう。その後で8列と9列をちゃんと解釈して代入し直せばよいですけど(面倒)。
txt <- readLines("test.txt")
n <- length(txt)
dat <- matrix(0, n, 9)
for (i in 1:n) {
str <- unlist(strsplit(txt[i], " "))
if (length(str) == 8) {
str[8:9] <- c("", str[8])
}
dat[i,] <- str
}
dat
実行結果
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
[1,] "1" "2" "3" "4" "5" "6" "7" "" "8"
[2,] "11" "12" "13" "14" "15" "16" "17" "" "18"
[3,] "21" "22" "23" "24" "25" "26" "27" "" "28"
[4,] "31" "32" "33" "34" "35" "36" "37" "" "38"
[5,] "41" "42" "43" "44" "45" "46" "47" "" "48"
[6,] "51" "52" "53" "54" "55" "56" "57" "comment" "58"
[7,] "61" "62" "63" "64" "65" "66" "67" "comment" "68"
[8,] "71" "72" "73" "74" "75" "76" "77" "" "78"まさ (2010-12-08 (水) 17:45:28)
R commanderを動かそうとしている初心者です。MacOSX10.6、Rのバージョンは2.12.0です。install.packages("Rcmdr", dependencies=TRUE)
の後に、library(Rcmdr)
と入力すると以下のメッセージが来ました。
要求されたパッケージ tcltk をロード中です
Tcl/Tkインターフェースのロード中
となり、それから特に何も起こりません。 この後少し打ち込もうとすると、それ以上入力できず、終了せざるをえない状況です。良い解決策を教えていただけないでしょうか?またX11は立ち上がった状況でやってはいますが、解決できていません。よろしくご教授下さい。
also installing the dependencies ‘aplpack’, ‘relimp’ URL 'http://cran.md.tsukuba.ac.jp/bin/macosx/leopard/contrib/2.12/aplpack_1.2.3.tgz' を試しています Content type 'application/x-gzip' length 2077295 bytes (2.0 Mb) 開かれた URL ================================================== downloaded 2.0 Mb URL 'http://cran.md.tsukuba.ac.jp/bin/macosx/leopard/contrib/2.12/relimp_1.0-2.tgz' を試しています Content type 'application/x-gzip' length 37864 bytes (36 Kb) 開かれた URL ================================================== downloaded 36 Kb URL 'http://cran.md.tsukuba.ac.jp/bin/macosx/leopard/contrib/2.12/Rcmdr_1.6-2.tgz' を試しています Content type 'application/x-gzip' length 2647612 bytes (2.5 Mb) 開かれた URL ================================================== downloaded 2.5 Mb ダウンロードされたパッケージは、以下にあります /var/folders/mL/..../downloaded_packagesのようなログがコンソールに出ましたか?出ていないなら,インストールはうまくいっていないでしょう。
要求されたパッケージ tcltk をロード中です
Tcl/Tkインターフェースのロード中 終了済
要求されたパッケージ car をロード中です
要求されたパッケージ MASS をロード中です
要求されたパッケージ nnet をロード中です
要求されたパッケージ survival をロード中です
要求されたパッケージ splines をロード中です
Rcmdrのバージョン 1.6-2
次のパッケージを付け加えます: 'Rcmdr'
The following object(s) are masked _by_ '.GlobalEnv':
partial.cor
The following object(s) are masked from 'package:tcltk':
tclvalue
となるはずなので,やはりどこかおかしいのでしょう。なお,【X11については,ユーザは何にもしなくてもかまいません】。どこらあたりから脇道にそれているのかわかりますか?質問君 (2010-11-27 (土) 18:29:37)
plotの使いかたで質問です。x <- c(10, 20, 30, 40, 50, 60, 70, 80, 90, 100) y <- c(5, 6, 7, 1, 3, 5, 6, 7, 1, 3)これらのデータで、x[1]の時、y[1]をプロットし、x軸にそのままxの値を10〜100まで表示したいのですが、どうやればよいのでしょうか?
よろしくお願いします。
plot(x, y, xaxt="n") axis(1, at=x)とでもするかな?グラフの横幅はある程度大きくしないと総ての目盛り数字は書かれない(逆に言えば,総ての目盛り数字が書かれるまで横幅を大きくしてね)。
ランゲル・ハンス (2010-11-26 (金) 15:14:51)
aggregate関数について質問させていただきます。
以下のようなデータフレーム(data)があったとき、cut 関数で N を 10 区間に区切って、その区間における d の合計を求めたいと思います。ある区間において NA がある場合、NA を 0 として合計する方法を教えていただけないでしょうか?下記の例では (50, 60], (60, 70], (70, 80] の区間の合計を 0 としたいと思います。
どうぞよろしくお願いいたします。N <- c(0, 1, 3, 7, 12, 20, 30, 35, 50, 91) d <- runif(10, 0, 10) data <- data.frame(cbind(N, d)) by1 <- cut(data$N, seq(0, 100, 10), right=TRUE, include.lowest=TRUE) agg <- aggregate(data$d, by=list(by1), FUN="sum") agg
> cbind(sapply(levels(by1), function(x) sum(d[by1 == x])))
[,1]
[0,10] 21.574116
(10,20] 8.055110
(20,30] 3.415935
(30,40] 2.436897
(40,50] 9.260387
(50,60] 0.000000
(60,70] 0.000000
(70,80] 0.000000
(80,90] 0.000000
(90,100] 5.495600
> cbind(sapply(split(d, by1), function(y) sum(y))) # なども> table(by1)
by1
[0,10] (10,20] (20,30] (30,40] (40,50] (50,60] (60,70] (70,80] (80,90] (90,100]
4 2 1 1 1 0 0 0 0 1
> sapply(split(d, by1), function(x) length(x)) # これでもよいですけどね。大げさです。
[0,10] (10,20] (20,30] (30,40] (40,50] (50,60] (60,70] (70,80] (80,90] (90,100]
4 2 1 1 1 0 0 0 0 1超初心者 (2010-11-20 (土) 15:53:12)
空行の実行方法とプロンプト上で改行を行なう方法を教えてください!!
マニュアル等いろいろ探したんですが、全くわかりません。
初歩的すぎて申し訳ございませんが、よろしくお願いします。
> library(sem)次に,read.moments 関数,入力しますね。
> cor.1 <- read.moments(diag=FALSE, names=c("年齢", "個人年収",
+ "教育年数", "職業威信"))
そうすると,コンソールには「1:」というプロンプトが出ます。プロンプトに応じて,
1: 0.12107847 2: -0.28075478 0.2329769 4: -0.04963399 0.3954726 0.2921118を入力しますね。この入力が終わるとプロンプトは
7:になります。このあと,何も入力せずに,リターンキーを押すのです(つまり,空行を入力したことになるんだけど)。
Read 6 itemsと表示され,入力が完了した事になります。
cor.1を入力すれば,cor.1 の内容が表示されるでしょう。 -- 河童の屁は,河童にあらず,屁である。 2010-11-21 (日) 19:21:16
困り果ててます (2010-11-19 (金) 18:48:40)
日本語を含む固定長ファイルを読み込みたいのですが,うまくいきません。123あ 456 321いう789のような内容の test.txt というファイルがあり,1件目のデータをV1="123", V2="あ ", V3="456" と読みたいのですが,
read.fwf("test.txt", width=c(3, 4, 3))だと意図したように読めません。
readChar("test.txt", c(3, 4, 3), useByte=TRUE)としてみたのですが,1行目しか読めません。
解決法をご存知の方がいらっしゃいましたら是非 ご教示ください。
よろしくお願いします。
> read.fwf("test.txt", width=c(3, 2, 3))
V1 V2 V3
1 123 あ 456
2 321 いう 789
Macintosh 版の R では,全角でも半角でも,「一文字は一文字」ということですね。lc <- Sys.getlocale("LC_CTYPE")
Sys.setLocale("LC_CTYPE", "C")
read.fwf("test.txt", width=c(3, 4, 3))
Sys.setLocale("LC_CTYPE", lc)
とすればよいのかな?前後に余分な面倒な指定が必要なのか?Windows では,読み取り幅を,バイト単位で指定するということか。 -- 河童の屁は,河童にあらず,屁である。 2010-11-19 (金) 20:38:40> sessionInfo() R version 2.10.1 (2009-12-14) i386-pc-mingw32 locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods baseです。もとのデータを編集するのは極力避けたいので,半角の空白2文字を全角空白1文字にするといった対処法は避けたいのと,例では全角文字しか出しませんでしたが,2行目の「いう」に相当するところに「ABCD」などの半角文字が入るケースもあり,半角2文字を全角1文字に変換するだけでは意図した読み込みができません。 他の環境のことはわかりませんが,Windowsでは読取幅を指定すると全角半角を問わず1文字単位で数えるようなので,読み取り幅をバイト単位で指定する方法がわからず,質問しました。-- 困り果ててます 2010-11-22 (月) 14:59:38
> lc <- Sys.getlocale("LC_CTYPE")
> Sys.setlocale("LC_CTYPE", "C")
[1] "C"
> a <- read.fwf("test.txt", width=c(3, 4, 3))
> Sys.setlocale("LC_CTYPE", lc)
[1] "Japanese_Japan.932"
> a
V1 V2 V3
1 123 あ 456
2 321 いう 789
のように、ちゃんとよめたけどなあ。 -- 河童の屁は,河童にあらず,屁である。 2010-11-22 (月) 15:27:44> lc <- Sys.getlocale("LC_CTYPE")
> Sys.setlocale("LC_CTYPE", "C")
[1] "C"
> test <- read.fwf("test.txt", width=c(3, 4, 3))
> Sys.setlocale("LC_CTYPE", lc)
[1] "Japanese_Japan.932"
> test
V1 V2 V3
1 123 あ 4 56
2 321 いう78 9
やはり文字単位でしか読み込めませんでした... -- 困り果ててます 2010-11-22 (月) 21:26:55con <- file(description="test.txt", open="r", encoding="shift-jis")
lc <- Sys.getlocale("LC_CTYPE")
Sys.setlocale("LC_CTYPE", "C")
test <- read.fwf(con, width=c(3, 4, 3))
Sys.setlocale("LC_CTYPE", lc)
test
close(con)
> test
V1 V2 V3
1 123 あ 4 56
2 321 いう78 9
のように,文字コードを指定して読み込もうとしてみたのですが,やはり結果は同じでした。 -- 困り果ててます。 2010-11-22 (月) 22:47:03> lc <- Sys.getlocale("LC_CTYPE")
> Sys.setlocale("LC_CTYPE", "E")
[1] "English_United States.1252"
> test <- read.fwf("test.txt", widths=c(3, 4, 3))
> Sys.setlocale("LC_CTYPE", lc)
[1] "Japanese_Japan.932"
> test
V1 V2 V3
1 123 あ 456
2 321 いう 789
ありがとうございました。 -- 困り果ててます 2010-11-23 (火) 14:11:15質問君 (2010-11-18 (木) 22:04:57)
エクセルでMM法を乱数で実行してるのですが、Rの乱数の方が良いと聞いたので、Rでも試したいのですが、エクセルとの擬似乱数比較について、Rが優れているなど分かりやすく、紹介されてるサイトや記事はないのでしょうか?たしかにエクセルよりも、乱数関数は多いです。性能的にはどうなのでしょうか?
探しても見当たりませんでした。もし、ご存知の方がいらっしゃれば、お願いします。
NT (2010-11-17 (水) 20:14:57)
3 段階評価 (0, 1, 2) の尺度の項目母数を grm 関数によって求めてみました。
出力結果の二行のみ示すと以下の通りとなります。Extrmt1 Extrmt2 Dscrmn Item1 -1.719 -0.126 1.553ここで Extrmt1 とは評価点 0 と 1 の項目曲線が交わる地点のθの値、Extrmt2 とは評価点 1 と 2 の項目曲線の交わる地点のθの値と解釈してよいのでしょうか。
ご教示願います。
Saito (2010-11-17 (水) 01:37:43)
いつもお世話になっております。
この手の話は既出かと思い検索しましたが、見つけることが出来なかったので質問させてください。
ある座標セットaを持っています。この座標セットには、座標とそれに付随する価が入っています。もう一つ別の座標セットbを持っています。こっちには、座標しか入っていません。今、座標bに最も近い座標aの持つ付随値を補間したいと考えています。下記にサンプルプログラムを示します。> ###座標とそれに付属する価### > a <- data.frame( + expand.grid(Long=seq(11, 13), + Lat=seq(20, 23)), + value=seq(1, 12) + ) > ###マッチさせる数### > num <- 1000 > > ###座標しか分かっていない### > b <- data.frame(long=runif(num, 11, 13), + lat=runif(num, 20, 23) + ) > head(b) long lat 1 12.00220 21.48183 2 11.14583 21.13535 3 12.31510 20.28334 4 11.54138 21.18872 5 11.66167 20.76173 6 12.52289 21.27551 > ###全ての組み合わせから最近接距離を持つ座標を特定### > dis <- NULL > for(i in 1 : nrow(b)) { + dis <- sqrt((b[i, 1] - a[, 1])^2 + (b[i, 2] - a[, 2])^2) + b[i, 3] <- a[which(dis==min(dis))[1], 3] #i番目のbがどのaに最も近いか探し出 してマッチング + } > head(b) long lat V3 1 12.00220 21.48183 5 2 11.14583 21.13535 4 3 12.31510 20.28334 2 4 11.54138 21.18872 5 5 11.66167 20.76173 5 6 12.52289 21.27551 6
しかし、実際にはaもbも超巨大で、この方法だとものすごく時間がかかることがあります。上記プログラムをもっと高速化するにはどうすればよいでしょうか。どなたかわかる方がいらっしゃいましたら、ご助言頂けると幸いです。
> a <- data.frame(
+ expand.grid(Long=seq(slong, elong),
+ Lat=seq(slat, elat)),
+ value=seq(1, elong*elat)
+ )
> b <- data.frame(long=runif(num, slong, elong),
+ lat=runif(num, slat, elat)
+ )
>
> dis <- NULL
> pp_a <- ppp(x=a[, 1], y=a[, 2], c(slong, elong), c(slat, elat))
> pp_b <- ppp(x=b[, 1], y=b[, 2], c(slong, elong), c(slat, elat))
>
> system.time(
+ for(i in 1 : nrow(b)) {
+ dis <- sqrt((b[i, 1] - a[, 1])^2 + (b[i, 2] - a[, 2])^2)
+ b[i, 3] <- a[which(dis==min(dis))[1], 3] #i番目のbがどのaに最も近いか探し 出 してマッチング
+ }
+ )
user system elapsed
17.19 0.00 17.20
>
> system.time(
+ b[, 4] <- a[nncross(pp_b, pp_a)$which, 3]
+ )
user system elapsed
0.01 0.00 0.02
>
> head(b)
long lat V3 V4
1 54.32715 76.365028 7554 7554
2 90.84222 7.004393 691 691
3 88.88978 93.650574 9389 9389
4 61.72731 8.535613 862 862
5 73.77037 6.405291 574 574
6 95.42776 92.221599 9195 9195カワウソ (2010-11-15 (月) 19:11:55)
はじめまして。
n個の従属変数(y1,y2,…,yn)と一つの独立変数xに関して,if構文を使って以下のような作業を一度に行うにはどのようにプログラムを書いたらよいのでしょうか?lm(y1 ~ x + g) lm(y2 ~ x + g) ・ ・ ・ lm(yn ~ x + g)
> iris.ancova <- for (i in 2:4) lm(iris[,i]~iris[,1] + iris[,5]) > summary(iris.ancova) Length Class Mode 0 NULL NULL > iris.ancova <- for (i in 2:4) summary(lm(iris[,i]~iris[,1] + iris[,5])) > iris.ancova NULL
for (i in 2:4) print(summary(lm(iris[,i]~iris[,1] + iris[,5])))
ダルビ (2010-11-10 (水) 21:24:20)
初めまして。
Rコマンダーで特定のデータセットを削除するにはどうすればよいのでしょうか?
いろいろなデータセットができてしまって困っています。
お手数ですが、よろしくお願います。
Jam (2010-11-08 (月) 17:07:02)
初めまして。
clusterライブラリで利用できるk-meansとpamの挙動について質問です。
2次元のxy座標で示せるデータをk-meansとpamでクラスタリングをしていて気になることがありました。それはpamの実行結果が毎回同じであるということです。
私の解釈として、どちらの手法も初期のランダムサンプルに結果が依存するため、クラスタリング結果は同じデータ・設定で実行したとしても毎回変わると思っています。確かにk-meansでは実行の度に結果は変わることは確かに確認できるのですが、pamではクラスタリング結果が毎回同じです。
クラスタリング結果が同じというのは、plotした図、result$centers (k-meansの場合)、result$medoids (pamの場合)を見て判断しています。データ数は1000弱です。
これはどういうことなのでしょうか?pamのランダムサンプルはクラスタリング結果に依存する、つまり結果は毎回多少でも異なってくると思うのです。k-menansの$cnetersは実行のたびに異なるが、pamの$medoidsは毎回同じ値になることは以下のプログラムで確認しました。
検証プログラム。library(cluster) # (x, y)形式のデータを読み込む data <- read.table(var.in_file, header=F, sep="\t") ################ # pam clustering # ################ data.clust <- pam(data, 10) data.clust$medoids ################ # k-means clustering # ################ data.clust <- kmeans(data, 10) data.clust$centers
MKI (2010-11-06 (土) 05:26:32)
散布図にLOESS()で平滑化曲線を加えたあと、その信頼区間を合わせてグラフに描画したいのですがそれらしい関数が見あたらず、もしどなたかご存じでしたら教えて下さい。できれば信頼区間を95%以外にも自分で変更できれば助かります。
nan (2010-10-31 (日) 18:35:42)
初めまして。
リストの成分にアクセスしたいのですが、例えば以下のようなデータについて> test <- function(x) {y=log(x); z=sin(x); return(list(value=x, log=y, sin=z))} > test(1:3) $value [1] 1 2 3 $log [1] 0.0000000 0.6931472 1.0986123 $sin [1] 0.8414710 0.9092974 0.1411200$logの[0.0000000]や$sinの[0.9092974]にアクセスするにはどのようにすれば良いのでしょうか?
宜しくお願いします。
> foo <- test(1:3) > class(foo) [1] "list" > class(foo$log) [1] "numeric" > foo$log[1] [1] 0 > foo$sin[2] [1] 0.9092974
のの (2010-10-31 (日) 04:07:34)
データフレームxの中の列名(変数名)aについて> attach(x) > x <- ifelse(a=="", NA, a)とすると、空の値を欠損値NAと指定できます。
しかし、aがfactorだった場合には、factorの性質が失われてしまうようです。
何かもっとエレガントな方法はあるのでしょうか?> x <- subset(x, a != "") > table(a, useNA="always")これも違うようです。
MKI (2010-10-29 (金) 22:29:49)
多変量解析を行い、意味のありそうな説明変数を取り出す作業をした後、それをブートストラップ法によってreliabilityを確認するよう求められました。Rでやってみたいと思いますが、「entry criteriaをp<=0.10にしてretention criteriaをp<0.05で確認する」ように指示されましたがstepAICでできるのでしょうか。方法としては
1.スプレッドシートからランダムにサンプリング(重複あり)
2.一般化線形モデルに当てはめ
3.stepAIC???で残った説明変数を抽出
4.1〜3を1000回繰り返す
といった感じになるかと思うのですが3.の部分が方法として正しいのかどうか分かりません。あるいは同じようなこと(多変量解析+ブートストラップ法)ができるパッケージはありますか。もしご存じでしたら教えて下さい。
hashi (2010-10-28 (木) 05:34:07)
Q&A (初級者コース)/11であった「任意の X 軸と曲線との交点(Y 値)の値の算出」の逆のことをしたいのです。
以下がデータです。x <- c(2:10) y <- c(0.084, 3.642, 12.472, 27.262, 47.035, 67.439, 83.974, 92.767, 100) plot(x, y, xlim = c(2, 10), ylim = c(0, 100)) lines(spline(x, y, n = 20), col = 2)このとき、abline(h = 5) を作図したときスプライン曲線との交点を読みたいのですが、どのようにしたらよいのでしょうか。
どうかよろしくご教授お願いいたします。
solve.spline <- function(x, y, y.val, x.init)
{
fun <- splinefun(x, y)
x <- x.init
repeat {
x2 <- (y.val-fun(x))/fun(x, 1)+x
if (abs(x-x2) < 0.0001) return(mean(c(x, x2)))
x <- x2
}
}
使用例
> solve.spline(x, y, 5, 4.5) # y=5 になる
[1] 3.205662 # x の値
> solve.spline(x, y, 30, 6) # y=30 になる
[1] 5.150513 # x の値
> fun <- splinefun(x, y) # これがスプライン曲線の関数
> fun(5.150513) # x=5.150513 のとき
[1] 30.00001 # y=30.00001 になってるMKI (2010-10-27 (水) 13:17:49)
データフレームの各列毎にある条件(たとえば0.1以上など)を満たす要素をカウントするにはどうしたらよろしいでしょうか。apply関数で各列のmeanなどは簡単に得られますが、似たような方法でカウント数を得ることは可能でしょうか。
sh (2010-10-27 (水) 09:17:04)
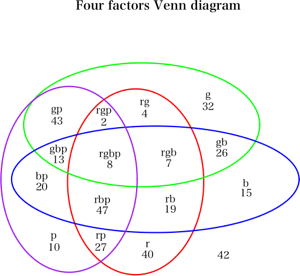
4つの集合のベン図をRで描くにはどうすればよろしいでしょうか? [参考文献] http://ja.wikipedia.org/wiki/ベン図 多数の集合のベン図 http://www.ats.ucla.edu/stat/r/faq/venn.htm R FAQ: How can I generate a Venn diagram in R? vennDiagram Can't plot Venn diagram for more than 3 sets
venn4 <- function(counts, col=c("red", "green", "blue", "purple"), lwd=2, labels=NULL, delta.y=7, ...)
{
elp <- function(x0, y0, a, b, col, lwd)
{
theta <- 0:360/180*pi
x <- x0+a*cos(theta)
y <- y0+b*sin(theta)
lines(x, y, col=col, lwd=lwd)
}
plot(c(0, 254), c(0, 202), type="n", axes=FALSE, xlab="", ylab="", ...)
x0 <- c(112, 116, 125, 68)
y0 <- c( 88, 134, 90, 90)
a <- c( 45, 78, 95, 45)
b <- c( 75, 50, 43, 75)
for (i in 1:4) {
elp(x0[i], y0[i], a[i], b[i], col=col[i], lwd=lwd)
}
# "" r g rg b rb gb rgb p rp gp rgp bp rbp gbp rgbp
loc.x <- c(170, 120, 160, 118, 185, 135, 169, 134, 58, 89, 60, 91, 50, 90, 61, 95)
loc.y <- c( 30, 30, 150, 144, 80, 67, 112, 104, 36, 36, 138, 137, 85, 66, 107, 103)
text(loc.x, loc.y-!is.null(labels)*delta.y, labels=counts)
if (!is.null(labels)) {
text(loc.x, loc.y+delta.y, labels=labels)
}
}
venn4(d$counts, labels=c( "", "r", "g", "rg", "b", "rb", "gb", "rgb", "p", "rp", "gp", "rgp", "bp", "rbp", "gbp", "rgbp"),
main="Four factors Venn diagram")
> d
r g b p counts
1 0 0 0 0 42
2 1 0 0 0 40
3 0 1 0 0 32
4 1 1 0 0 4
5 0 0 1 0 15
6 1 0 1 0 19
7 0 1 1 0 26
8 1 1 1 0 7
9 0 0 0 1 10
10 1 0 0 1 27
11 0 1 0 1 43
12 1 1 0 1 2
13 0 0 1 1 20
14 1 0 1 1 47
15 0 1 1 1 13
16 1 1 1 1 8
venn4(d$counts, labels=c( "", "r", "g", "rg", "b", "rb", "gb", "rgb", "p", "rp", "gp", "rgp", "bp", "rbp", "gbp", "rgbp"),
main="Four factors Venn diagram")
ランゲル・ハンス (2010-10-26 (火) 10:29:21)
いつも掲示板を参考にさせていただいています。
さて、下記の行列mの各行の値を逆順にして、行列m2を作りたいと思います。
行列操作でmからm2を作る方法、あるいはdからm2を作る方法をご教示いただけないでしょうか?d <- c(1, 2, 3, 4, 5, 6, 7, 8, 9) m <- matrix(d, 3, 3, byrow=TRUE) d2 <- c(3, 2, 1, 6, 5, 4, 9, 8, 7) m2 <- matrix(d2, 3, 3, byrow=TRUE)どうぞよろしくお願いいたします。
yorudan (2010-10-25 (月) 17:22:10)
ハフ変換を行うためのパッケージはありますか!?
のの (2010-10-24 (日) 03:42:03)
library(gregmisc)を導入して、excelシートからデータを直接取り込む方法を試しています。
いくつかおかしな挙動をみつけたので書き込みます。
こちらの環境は、MacOS10.6.4, GUI版R version 2.11.1 (2010-05-31) x86_64-apple-darwin9.8.0 です。
他の環境で、同じ状況が再現するのかも教えて欲しいです。
1. エクセルシートの中で、"'’"または"…"を使っていると次のエラーが出る。Wide character in print at /Library/Frameworks/R.framework/Resources/library/gdata/perl/xls2csv.pl line 262.調べた範囲では、たぶん内部処理でperlに文字列を渡すときに文字コードエラーを起こしているのだと思います。ただデータは正しく取り込まれるので実質的な問題はないように思います。
これがサンプルファイルです。
2. サンプル数が増えてしまう
こちらの方が問題が大きいです。Rはエラー表示しませんが、変なレコードが追加されて、データフレームに影響を与えます。
試行錯誤して、""',"この文字列を含むとおかしくなることは再現できました。
これがサンプルファイルです。
まだ、library(gregmisc)開発サイトの方は良く調べていないのでこれから調べて何か分かればこの記事を更新する予定です。
MKI (2010-10-22 (金) 13:29:37)
図(グラフ)の中に平均(変数)+/-標準偏差(変数)を書き込みたいのですがexpressionを使用してもうまくいきません。ひとつひとつ数字を書き込めば
可能ですが、変数などと組み合わせて表示する方法はないでしょうか。
plot(hist(rnorm(10000)))
mean <- c(3.2, 4.5, 6.8)
sd <- c(0.2, 0.1, 0.6)
x <- c(-3, -2, 2)
y <- c(1100, 1300, 1500)
for (i in 1:3) {
str <- sprintf("text(%f, %f, labels=expression(%f%%+-%%%f))", x[i], y[i], mean[i], sd[i])
eval(parse(text=str))
}
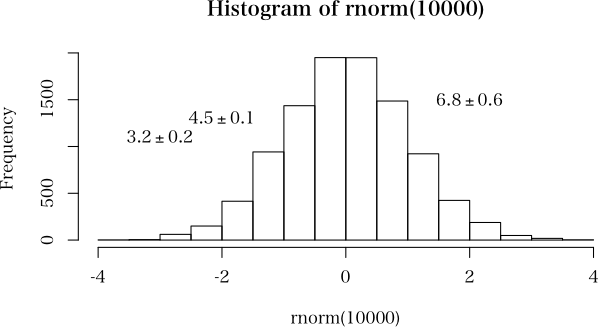
plot(hist(rnorm(10000)))
mean <- c(3.2, 4.5, 6.8)
sd <- c(0.2, 0.1, 0.6)
x <- c(-3, -2, 2)
y <- c(1100, 1300, 1500)
str <- sprintf("text(%f, %f, labels=expression(%f%%+-%%%f))", x, y, mean, sd)
eval(parse(text=str))しげゆき (2010-10-18 (月) 10:39:18)
XPでR2.11.1の初心者です。よろしくお願いします。
scatterplot3dでプロットの色、サイズを変えた図を作成していますが、色はデータどおりいきましたが、サイズがデータと食い違ってうまくいきませんでした。
以下データで左3列がXYZ軸、colorが色4種類、sizeが5段階です。sisu dens length color size 1 -115 1.4 9.8 A 5 2 -62 1.6 7.0 A 3 3 108 1.7 452.0 B 2 4 174 1.8 202.0 B 1 5 184 1.7 141.0 B 4 6 11 1.7 10.2 C 4 7 23 1.7 40.0 C 5 8 -14 1.8 232.0 D 5 9 127 1.7 132.0 D 3 scatterplot3d(x=d$sisu, y=d$dens, z=d$length, color=c(2,3,4,5)[unclass(d$color)], cex.symbols=c(1,2,3,4,5)[unclass(d$size)])cexを書き出しましたが、データどおりでおかしくありませんでした。
> c(1,2,3,4,5)[unclass(d$size)] [1] 5 3 2 1 4 4 5 5 3どこか間違っているのでしょうか。
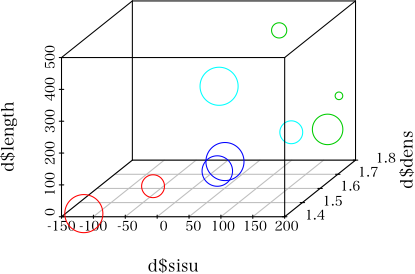
のの (2010-10-14 (木) 11:05:44)
いつもお世話になります。 例えば、次のようなベクトルが二つあるとき、x <- c(1:10) y <- c(20:40) test(x)とtest(y)の結果が1でtest(c(x,y))の結果が2となるような関数testを作りたいのですが、何か良い方法があれば是非教えて下さい。 test <- function(i) { text <- as.character(match.call()[2]) if (grep("^c", text) != 1) {1} else {grep(",", text)} }のようになるのかと考えましたが上手くいきません。
> test <- function(arg)
+ {
+ sum(unlist(strsplit(deparse(substitute(arg)), ""))==",")+1
+ }
> x <- 1:10
> y <- 21:26
> z <- 31:48
> test(x)
[1] 1
> test(c(x, y))
[1] 2
> test(c(x, y, z))
[1] 3Saito (2010-10-13 (水) 13:00:02)
いつもお世話になっています。
過去ログ等検索しましたが、見つからなかったので質問させてください。
座標と座標に付与されたデータからなる3列のデータセットがあります。今、座標が細かすぎるので、もう少し粗い解像度に変換したいのですが、その変換プログラムが上手くいきません。以下に例を示します。> ###仮想データセット### > ###x, yが座標で、zが値### > mat <- data.frame(expand.grid(x=1:6, y=1:4), z=1:24) > mat x y z 1 1 1 1 2 2 1 2 3 3 1 3 4 4 1 4 5 5 1 5 6 6 1 6 7 1 2 7 8 2 2 8 9 3 2 9 10 4 2 10 11 5 2 11 12 6 2 12 13 1 3 13 14 2 3 14 15 3 3 15 16 4 3 16 17 5 3 17 18 6 3 18 19 1 4 19 20 2 4 20 21 3 4 21 22 4 4 22 23 5 4 23 24 6 4 24 > ###y軸方向に足し合わせたときの行列を用意### > mat2 <- data.frame(matrix(0, ncol=ncol(mat), nrow=nrow(mat)/2)) > > ###x軸方向にも足し合わせたときの行列を用意### > mat3 <- data.frame(matrix(0, ncol=ncol(mat), nrow=nrow(mat2)/2)) > > ###y軸の値がx軸方向にいくつ詰まってるのか確認### > a <- as.numeric(summary(as.factor(mat[, 2]))[1]) > > ###y軸方向に何度足せばよいのか確認### > b <- length(levels(as.factor(mat[, 2]))) > > ###等比数列でy軸の値をx軸方向に詰まっている分だけ足し合わせ### > for (i in 1 : (b/2)) { + mat2[((i-1)*a+1):(i*a), ] <- + mat[((2*a)*(i-1)+1):((2*a)*(i-1)+a), ] + + mat[((2*a)*(i-1)+(1+a)):((2*a)*(i-1)+(a*2)), ] + } > mat2 X1 X2 X3 1 2 3 8 2 4 3 10 3 6 3 12 4 8 3 14 5 10 3 16 6 12 3 18 7 2 7 32 8 4 7 34 9 6 7 36 10 8 7 38 11 10 7 40 12 12 7 42 > ###同様にx軸についても### > for(i in 1 : (nrow(mat2)/2)){ + mat3[i, ] <- mat2[(2*(i-1)+1), ] + mat2[(2*i), ] + } > mat3 X1 X2 X3 1 6 6 18 2 14 6 26 3 22 6 34 4 6 14 66 5 14 14 74 6 22 14 82 > ###最終的に得たい座標セット### > ###周囲4つの値の平均となってほしい### > mat3/4 X1 X2 X3 1 1.5 1.5 4.5 2 3.5 1.5 6.5 3 5.5 1.5 8.5 4 1.5 3.5 16.5 5 3.5 3.5 18.5 6 5.5 3.5 20.5 >一応、上記のへたくそなプログラムでもおおよそのやりたいことは出来ているのですが、実は、まだ上記に含めていない条件が二つあります。
一つは、周囲4つの平均でなく周囲9つの平均や16の平均(つまり正方形で扱いたい)と任意に変えたいときに、上記のプログラムでは一から書き直しになります。
そこを明示的に加えたいのですが、上手く書けませんでした(例ではそもそも24行しかないので、9や16は難しいですが…)。
もう一つの条件は、z軸にNAが含まれている座標があるのですが、そのときはNAを抜いた数で平均を返したいのです。
例えば、
mat[1, 3] <- NA等とした時には、そのまま上記プログラムを走らせると最後までNA表記となり、やりたいことができません。
4で割るのではなく、そのときだけ上記例ですと3で割ってほしいのですが、複雑で出来ませんでした。
さらに、このデータ、実際は結構大きな行列(6万×3くらいです)ですので出来るだけベクトルで処理したいと思っています。
高速化した例ですと、大変助かります。
どなたか上記の条件で変換が出来る方がいらっしゃいましたら、ご教授頂けると幸いです。
関数
func <- function(mat, n2)
{
n <- sqrt(n2)
nr <- nrow(mat)
nc <- ncol(mat)
stopifnot(nr%%n == 0 && nc%%n == 0)
ans <- NULL
for (j in 0:(nc%/%n-1)*n) {
ind.j <- (j+1):(j+n) # j+1 から j+n までの列
for (i in 0:(nr%/%n-1)*n) {
ind.i <- (i+1):(i+n) # i+1 から i+n までの行
ans <- c(ans, mean(ind.i), mean(ind.j),
mean(mat[ind.i, ind.j], na.rm=TRUE))
}
}
return(data.frame(matrix(ans, ncol=3, byrow=TRUE)))
}
実行例
mat <- matrix(1:108, 9, 12)
mat[4,5] <- mat[7,3] <- mat[9,6] <- NA # 欠損値を適当にちりばめる
mat
(ans <- func(mat, 9)) # 9×12 行列を,3行×3列単位でまとめる
実行結果
> mat
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11] [,12]
[1,] 1 10 19 28 37 46 55 64 73 82 91 100
[2,] 2 11 20 29 38 47 56 65 74 83 92 101
[3,] 3 12 21 30 39 48 57 66 75 84 93 102
[4,] 4 13 22 31 NA 49 58 67 76 85 94 103
[5,] 5 14 23 32 41 50 59 68 77 86 95 104
[6,] 6 15 24 33 42 51 60 69 78 87 96 105
[7,] 7 16 NA 34 43 52 61 70 79 88 97 106
[8,] 8 17 26 35 44 53 62 71 80 89 98 107
[9,] 9 18 27 36 45 NA 63 72 81 90 99 108
> (ans <- func(mat, 9))
X1 X2 X3
1 2 2 11.000
2 5 2 14.000
3 8 2 16.000
4 2 5 38.000
5 5 5 41.125
6 8 5 42.750
7 2 8 65.000
8 5 8 68.000
9 8 8 71.000
10 2 11 92.000
11 5 11 95.000
12 8 11 98.000
性能評価
> mat <- matrix(1:(500*500), 500) # セル数が25万
> system.time(func(mat, 4)) # これが,一番時間のかかる場合(単位は秒)
ユーザ システム 経過
35.414 49.723 85.492
> system.time(func(mat, 25))
ユーザ システム 経過
1.599 1.213 2.886
> system.time(func(mat, 100))
ユーザ システム 経過
0.277 0.030 0.407
###作成して頂いたfuncへ自分のデータを変換### > mat2 <- data.frame(expand.grid(x=1:6, y=1:4), z=1:(6*4)) > mat3 <- matrix(mat2[, 3], 6, 4) > func(mat3, 4) #確認 X1 X2 X3 1 1.5 1.5 4.5 2 3.5 1.5 6.5 3 5.5 1.5 8.5 4 1.5 3.5 16.5 5 3.5 3.5 18.5 6 5.5 3.5 20.5上記の変換で、作っていただいた関数用にデータ加工する方法はわかりました。本当にありがとうございました。ただ、実際のデータは座標が1から始まらないので変換する必要がありました。座標系を適当に1から与えてサンプルプログラムを提示した私のミスです。申し訳ありません。そこで、1から始まらない座標系にも作っていただいたプログラムが適用できる様に、以下のようなfunc2を作成してみました。
> ###1から始まらない座標系を与える###
> mat4 <- data.frame(expand.grid(x=seq(11, 16),
+ y=seq(3, 6)), z=1:24)
>
> ###番地を付け足す###
> mat5 <- data.frame(mat4, expand.grid(x2=1:length(levels(as.factor(mat4[, 1]))),
+ y2=1:length(levels(as.factor(mat4[, 2])))
+ ))
>
> ###座標を元の座標に戻すように改造###
> func2 <- function(mat, n2, MAT)
+ {
+ n <- sqrt(n2)
+ nr <- nrow(mat)
+ nc <- ncol(mat)
+ stopifnot(nr%%n == 0 && nc%%n == 0)
+ ans <- NULL
+ for (j in 0:(nc%/%n-1)*n) {
+ ind.j <- (j+1):(j+n) # j+1 から j+n までの列
+ for (i in 0:(nr%/%n-1)*n) {
+ ind.i <- (i+1):(i+n) # i+1 から i+n までの行
+
+ ###合致するものをsubsetで探してくる###
+ ans <- c(ans, mean(MAT[ind.i, 1]), mean(
+ subset(MAT[, 2],
+ MAT[, 4]==ind.i&MAT[, 5]==ind.j)),
+ mean(mat[ind.i, ind.j], na.rm=TRUE))
+ }
+ }
+ return(data.frame(matrix(ans, ncol=3, byrow=TRUE)))
+ }
###整合性の確認###
> func(mat3, 4)
X1 X2 X3
1 1.5 1.5 4.5
2 3.5 1.5 6.5
3 5.5 1.5 8.5
4 1.5 3.5 16.5
5 3.5 3.5 18.5
6 5.5 3.5 20.5
###整合性の確認###
> func2(mat3, 4, mat5)
X1 X2 X3
1 11.5 3.5 4.5
2 13.5 3.5 6.5
3 15.5 3.5 8.5
4 11.5 5.5 16.5
5 13.5 5.5 18.5
6 15.5 5.5 20.5
> 性能評価
> mat <- data.frame(expand.grid(x=101:600, y=101:600),z=1:(500*500))
> mat2 <- matrix(mat[, 3], 500, 500)
> mat3 <- data.frame(mat, expand.grid(x2=1:as.numeric(summary(as.factor(mat[, 1]))[1]),
+ y2=1:as.numeric(summary(as.factor(mat[, 2]))[1])
+ ))
>
> system.time(func2(mat2, 4, mat3))
ユーザ システム 経過
539.02 0.12 539.36 #約9分かかります。
> system.time(func2(mat2, 25, mat3))
ユーザ システム 経過
83.35 0.00 83.35
> system.time(func2(mat2, 100, mat3))
ユーザ システム 経過
20.77 0.00 20.77
もう目的は十分達成されていると思うので、お時間があれば、で構いません。
もし、座標を元に戻すやり方をもっと早く処理する方法をご存じの方がいらっしゃいましたらご教授頂ければ幸いです。 -- Saito 2010-10-14 (木) 14:17:19func <- function(mat, rv, cv, n2) # ★★ rv, cv を追加
{
n <- sqrt(n2)
nr <- nrow(mat)
nc <- ncol(mat)
stopifnot(nr%%n == 0 && nc%%n == 0)
ans <- NULL
for (j in 0:(nc%/%n-1)*n) {
ind.j <- (j+1):(j+n) # j+1 から j+n までの列
for (i in 0:(nr%/%n-1)*n) {
ind.i <- (i+1):(i+n) # i+1 から i+n までの行
ans <- c(ans, mean(rv[ind.i]), mean(cv[ind.j]), # ★★ rv, cv の平均を求める
mean(mat[ind.i, ind.j], na.rm=TRUE))
}
}
return(data.frame(matrix(ans, ncol=3, byrow=TRUE)))
}
#実行例
mat <- matrix(1:108, 9, 12)
mat[4,5] <- mat[7,3] <- mat[9,6] <- NA # 欠損値を適当にちりばめる
rv <- c(1,3,6,9,11,20,27,35,42) # ★★ 行方向の座標ベクトル(下の mat の表示の表側を参照)
cv <- c(2,4,6,7,8,11,13,16,19,22,25,27) # ★★ 列方向の座標ベクトル(下の mat の表示の表頭を参照)
rownames(mat) <- rv # これは付けなくても良いけど
colnames(mat) <- cv # 〃
mat
(ans <- func(mat, rv, cv, 9)) # 9×12 行列を,3行×3列単位でまとめる
> mat
2 4 6 7 8 11 13 16 19 22 25 27
1 1 10 19 28 37 46 55 64 73 82 91 100
3 2 11 20 29 38 47 56 65 74 83 92 101
6 3 12 21 30 39 48 57 66 75 84 93 102
9 4 13 22 31 NA 49 58 67 76 85 94 103
11 5 14 23 32 41 50 59 68 77 86 95 104
20 6 15 24 33 42 51 60 69 78 87 96 105
27 7 16 NA 34 43 52 61 70 79 88 97 106
35 8 17 26 35 44 53 62 71 80 89 98 107
42 9 18 27 36 45 NA 63 72 81 90 99 108
> (ans <- func(mat, rv, cv, 9)) # 9×12 行列を,3行×3列単位でまとめる
X1 X2 X3
1 3.333333 4.000000 11.000
2 13.333333 4.000000 14.000
3 34.666667 4.000000 16.000
4 3.333333 8.666667 38.000
5 13.333333 8.666667 41.125
6 34.666667 8.666667 42.750
7 3.333333 16.000000 65.000
8 13.333333 16.000000 68.000
9 34.666667 16.000000 71.000
10 3.333333 24.666667 92.000
11 13.333333 24.666667 95.000
12 34.666667 24.666667 98.000> mat2 <- data.frame(expand.grid(x=11:16, y=3:6), z=1:(6*4))
> mat3 <- matrix(mat2[, 3], 6, 4)
> rv <- c(11:16)
> cv <- c(3:6)
> func(mat3, rv, cv, 4) #確認
X1 X2 X3
1 11.5 3.5 4.5
2 13.5 3.5 6.5
3 15.5 3.5 8.5
4 11.5 5.5 16.5
5 13.5 5.5 18.5
6 15.5 5.5 20.5
自分の実際のデータに適用しても上手く動きました。非常に勉強になりました。重ね重ね、本当にありがとうございました。-- Saito 2010-10-14 (木) 18:45:12JJ (2010-10-12 (火) 20:17:51)
まったくの初級者です。
ポリコリック相関係数を用いた因子分析(最尤法、プロマックス)を3日間ほど悩んでいます。最終的に因子得点も算出したいと思っています。
現在までのところ、
factanal(covmat=cor(データ), factors=3,promax,regression)
という感じまでは来たのですが。。。数字は出ますが、これは一般の因子分析なのかと思っています。
ちなみにpolychor(x,y,)関数の場合、相関係数にはなので、相関係数行列にはなりませんよね。
まことに見当はずれかもしれませんが、誰か力を貸していただければありがたいです。
上記の corのままでは
library(psych) library(polycor) data(bfi) dat <- bfi[1:17] # 17項目のデータ pcr <- polychoric(dat) pcr # polychoric関数はrhoやtauといった複数の値を返す。polychoric関数のヘルプ参照 ans <- factanal(covmat=pcr$rho, factors=2, rotation="promax") ans
sayaka (2010-10-12 (火) 14:05:00)
以前、「T,Fを文字列として出力するには」でお世話になりました。
うまく説明するのが難しいのですが、下記の data のようなベクトルがあるとき、399,400,401・・のように数値が続いている場合に"何個続いているか"を下記の a のように出力したいと考えているのですが、どのようにすれば求めることが出来るでしょうか。
いろいろと試行錯誤したのですが、どうしても思いつきません。
data はあるデータ中のエラーデータの行番号を抽出したもので、エラーデータの開始位置と長さより削除するプログラムがあるために長さが必要です。
開始位置は下記のposiで求めました。
どうぞよろしくお願いします。> data [1] 14 41 73 152 296 297 399 400 401 402 418 419 420 421 422 423 424 425 426 427 428 429 [23] 430 431 451 452 453 454 460 461 466 500 501 502 503 504 505 506 507 508 > posi <- data[diff(c(0,data)) != 1] > posi [1] 14 41 73 152 296 399 418 451 460 466 500 > a [1] 1 1 1 1 2 4 14 4 2 1 9
菊亭 (2010-10-06 (水) 22:12:34)
現在XPでRを使っております。そのうち7にアップグレードしょうかと画策中ですが、VistaとRの相性がかなり悪かった(というかVistaが酷かった)記憶があります。7とRには既知の問題はありますでしょうか?
ランゲル・ハンス (2010-10-02 (土) 09:51:51)
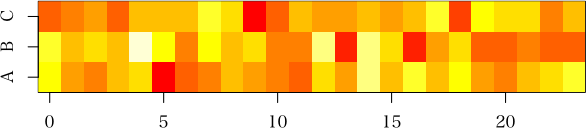
plotrixにあるcolor2D.matplot関数について質問させていただきます。library(plotrix) x <- c(1, 0, 10, 5, 3, 6, 4, 5, 4, 3, 8, 5, 1, 0, 7, 5) data <- matrix(x, ncol=4) color2D.matplot(data, c(1, 0), c(0, 1), c(0, 1), show.values=TRUE)このmatirixの数値をカラー表示のマトリックスに変換して数値も表示したいと思います。
上記の例では変換できるのですが、マイナスがある場合のスケール変換の方法と色表示の方法をご教示いただけないでしょうか?
例えばyの場合についてy <- c(1, 0, 10, 5, -3, 6, -4, 5, 4, 3, 8, 5, 1, 0, 7, 5)よろしくお願いします。
隣は何をする人ぞ (2010-10-01 (金) 21:34:02)
psych パッケージを使って因子分析を行ったとき、因子負荷量の大きい順にソートしたときとしなかったときで、因子負荷量の値が異なったものが表示されます。単に因子単位に因子負荷量の絶対値の大きい順に並べ替えて表示するだけだと思うのですが、なぜ因子負荷量の値が異なるのでしょうか。訳がわかりません。library(psych) data(bfi) fa.parallel(bfi) fpa.out <- factor.pa(bfi, nfactors=3, rotate="promax") print(fpa.out) # 分析に使用した変数順そのまま print(fpa.out, sort=TRUE) # 因子負荷量の大きい順に並べ替えるで分析を行いました。並び順は異なっても、各変数の因子負荷量は同じはずです。
まず、print(fpa.out) の結果です。いくつかの変数だけを選択して掲載します。########## デフォルト(sort=FALSE)のとき > print(fpa.out) Factor Analysis using method = pa Call: factor.pa(r = bfi, nfactors = 3, rotate = "promax") Unstandardized loadings based upon covariance matrix PA1 PA2 PA3 h2 u2 H2 U2 A1 -0.22 0.08 0.01 0.0600 0.94 0.0600 0.94 : E4 0.74 -0.06 -0.15 0.4823 0.52 0.4812 0.52 : gender 0.20 0.15 0.00 0.0490 0.95 0.0490 0.95 education -0.03 -0.04 0.10 0.0099 0.99 0.0099 0.99 age 0.04 -0.09 0.14 0.0398 0.96 0.0398 0.96 PA1 PA2 PA3 SS loadings 3.30 2.66 2.15 Proportion Var 0.12 0.09 0.08 Cumulative Var 0.12 0.21 0.29 Standardized loadings item PA1 PA2 PA3 h2 u2 A1 1 -0.22 0.08 0.01 0.0600 0.94 : E4 14 0.73 -0.06 -0.15 0.4812 0.52 : gender 26 0.20 0.15 0.00 0.0490 0.95 education 27 -0.03 -0.04 0.10 0.0099 0.99 age 28 0.04 -0.09 0.14 0.0398 0.96次に、print(fpa.out, sort=TRUE) の結果です。
########## sort=TRUE を指定したとき > print(fpa.out, sort=TRUE) Factor Analysis using method = pa Call: factor.pa(r = bfi, nfactors = 3, rotate = "promax") Unstandardized loadings based upon covariance matrix PA1 PA2 PA3 h2 u2 H2 U2 E4 0.74 -0.06 -0.15 0.4823 0.94 0.339 0.66 : A1 -0.22 0.08 0.01 0.0600 0.73 0.076 0.92 gender 0.20 0.15 0.00 0.0490 0.58 0.078 0.92 : age 0.04 -0.09 0.14 0.0398 0.99 0.039 0.96 education -0.03 -0.04 0.10 0.0099 0.96 0.010 0.99 PA1 PA2 PA3 SS loadings 3.30 2.66 2.15 Proportion Var 0.12 0.09 0.08 Cumulative Var 0.12 0.21 0.29 Standardized loadings item PA1 PA2 PA3 h2 u2 E4 14 0.62 -0.05 -0.12 0.339 0.66 : A1 1 -0.25 0.09 0.01 0.076 0.92 gender 26 0.25 0.19 0.00 0.078 0.92 : age 28 0.04 -0.09 0.13 0.039 0.96 education 27 -0.03 -0.04 0.10 0.010 0.99Unstandardized loadings based upon covariance matrix については、並べ替えしてもしなくても、各変数の因子負荷量は同じです(これが当たり前だと思います)。
Standardized loadings については、並べ替えしたのとしないとでまるっきり違うものが表示されています。
なぜでしょう。
一応、psych クラスの print メソッド(psych:::print.psych) のソースもたどっては見たのですけど、明らかなバグというのではなく、書かれているプログラムがなぜそのようなパスをたどらなければならないのかがよくわかりませんでした(それ自身がバグと言うことなのかも知れませんが)。
多くの人が使っているパッケージなので、いまだにバグがあるとも思えませんが、不思議に思いましたので質問させて頂きます。
I finally had time to find the bug. It was not in factor.pa as I had hoped, but it was in the print routine which thus affected fa, as well as factor.pa. The sorted loadings are now sorted correctly, as are the communalities and uniquenesses. I have fixed this for version 1.0.93 which should be released sometime soon.だそうです。 -- 隣は何をする人ぞ 2010-11-28 (日) 23:12:19
のの (2010-09-29 (水) 02:16:00)
MacでGUI版Rを使っています。version.string R version 2.11.1 (2010-05-31)
エディタにコマンドを連ねて、メニュー>編集>実行をする場合、途中でエラーがあっても、止まらずに最後まで流れてしまいます。
コマンドリストの任意の場所でRの実行を止める方法を探しています。
readline(),stopifnot()などを試しましたがだめでした。
今のところquit()を入れると、そこで、保存するかどうか聞いてくるので目的は達成できているのですが、何かもっと良い方法はないでしょうか?
> try({ old <- options(warn=2) # これと,
+ a <- sqrt(-9)
+ print(,)
+ print("ok")
+ options(old) }) # これの対で囲む
Error in sqrt(-9) : # 最初のエラーでストップ
(警告から変換されました) 計算結果が NaN になりました
> try({ old <- options(warn=2)
+ a <- sqrt(9) # 修正した
+ print(,)
+ print("ok")
+ options(old) })
Error in .Internal(print.default(x, digits, quote, na.print, print.gap, :
'x'が見つかりません # 二番目のエラーでストップ
> try({ old <- options(warn=2)
+ a <- sqrt(9)
+ print(a) # ここも直した
+ print("ok")
+ options(old) })
[1] 3 # 全部うまくいった
[1] "ok"
その他に,tryCatch, withCallingHandlers, signalCondition, simpleError, simpleWarning, conditionCall, conditionMessage, withRestart, computeRestarts, findRestart, invokeRestart, invokeRestartInteractively, isRestart, restartDescription, restartFomals, .signalSimpleWarning, .handleSimpleError などを調べると良いでしょう。 -- 河童の屁は,河童にあらず,屁である。 2010-09-29 (水) 12:26:45while(1){ #エディタの最初に記載
+ ここから実行するコマンドリスト
+ 次のコマンド・・・
+ break #ここでRの実行を止める
+ 結果として実行されないコマンド
+ break;} #もし途中にbreakがない場合にはここで終了
このような方法も思いつきましたが、コマンドの結果を出力させるためにcat()を使わないといけなかったりと面倒な感じです。もう少し軽やかな方法があればと思っています。 -- のの 2010-09-30 (木) 04:56:16print(1) print() # Error print(2)と書いて[全て実行]すると、
> print(1) [1] 1 > print() # Error 以下にエラー print.default() : 要素 1 は空です; > print(2) [1] 2のように、エラー以降のprint(2)も実行されてしまう、と。 これを解決したいということであれば、単純にスクリプト全体をプロック化して
{
print(1)
print() # Error
print(2)
}
のようにするのはいかがでしょう。括弧が閉じたところでスクリプトが実行され、エラーが起こるとブロック全体が止まります。
> {
+ print(1)
+ print() # Error
+ print(2)
+ }
[1] 1
以下にエラー print.default() :
要素 1 は空です;
> # print(2)は実行されていない
もちろんこんなことせずとも、全体をファイルに保存してからsourceすれば、エラーの部分で止まって以降の処理は行なわれないわけですが。どうしても[全て実行]を使いたいということであれば、この方法が楽かと思います。 -- 2010-09-30 (木) 16:22:26shumei (2010-09-25 (土) 14:33:13)
Mac版R 2.11.1 をインストールした後、パッケージurcaをインストールしました。
urcaをロードし、パッケージの使用はできましたが、Rを起動するたびに未ロード状態にもどってしまいます。
何冊か書籍に当たってみましたが、該当する記述が見つからず、こちらに投稿させて頂きます。
パッケージのロード状態を維持するにはどうすればいいのでしょうか。それとも、これはRの仕様なのですか?
ご教授下さい。どうぞよろしくお願い致します。
/.Rprofileと教えるところを単に.Rprofileと書くと、説明された方は?.Rprofileを参照して、R_HOME/etc/Rprofile.siteのことではないかと思う人もいるだろう -- 2010-10-02 (土) 14:33:58
moyu (2010-09-21 (火) 14:54:22)
ヒストグラムで、ある一部の区間において細かく階級を分割したいのですが、どうしたらいいのか分かりません。ちなみに以下の操作をしました。hangseng = read.csv("ASIA/hangseng/hangsengdaily-1986dec31~2010jun11.csv") hangseng.ts = ts(rev(hangseng$Close),start=c(1986,12,31),frequency=248) ts.plot(hangseng.ts) ts.plot(diff(hangseng.ts)) hist(diff(hangseng.ts),breaks=30,col="magenta")histの中のbreaksをどういじったらいいのでしょうか??
breaks one of: ・ a vector giving the breakpoints between histogram cells, ・ a single number giving the number of cells for the histogram, ・ a character string naming an algorithm to compute the number of cells (see ‘Details’), ・ a function to compute the number of cells. In the last three cases the number is a suggestion only.あなたが指定した breaks=30 は,この中の 2 番目のものですよね。一番目の指定方法をとらないとね。どうやって良いかそれでもわからない?下の方の,Example の項にいくつか例があり,そのうちの breaks=c(12,20,36,80,200,1000,17000) というのがそれですよ。-- 河童の屁は,河童にあらず,屁である。 2010-09-21 (火) 17:10:12
初級者です。 (2010-09-20 (月) 18:11:38)
CentOS 5.5 に Rをインストールし、以下の操作をしました。pdf("test.pdf",family="Japan1") plot(1:10,ylab="test test") dev.off() embedFonts("test.pdf")すると、ylabに指定した"test test"文字列の間にある空白の位置に、「・」が表示されてしまいます。
正確には、"test ・test"という感じで、普通に半角スペースが表示されている上に、若干右側によって「・」が重なっているという感じです。
embedFonts()を呼ぶ前の状態では、「・」は表示されていません。
また、その状態で文章のプロパティから使用されているフォントを見ると、「KozMinPro-Regular-Acro」 「KozMinPro-Regular-Acro.Bold」 「KozMinPro-Regular-Acro.BoldItalic」 「KozMinPro-Regular-Acro.Italic」 「Symbol」 「ZapfDingbats」というフォントが使用されていると出ますが、embedFonts()を呼んだ後で同様にフォントを確認すると、「Sazanami-Gothic(埋め込みサブセット)」というフォントが使用されていると出ます。
何らかフォントの設定が足りないとは思い、いろいろ調べてみてはいるのですが、壁に当たった状態です。
同様の現象を解決された方がいらっしゃれば方法をご教示いただけないかと思い、こちらに投稿しました。
よろしくお願いいたします。
/KozMinPro-Regular-Acro << /FileType /TrueType /Path (/usr/local/share/fonts/ipafont/ipam.ttf) /CSI [(Japan1) 6] >> ;みたいに記述すれば良いと思います. -- なかま 2010-09-22 (水) 00:55:05
てるてるぼうず (2010-09-19 (日) 23:22:48)
関数のヘルプファイルを表示したいと思い、以下を実行するのですが、
?…またはhelp(…)毎回以下のようなエラーが出ます> ?glm 警告メッセージ: In file.show(temp, title = gettextf("R Help on '%s'", topic), delete.file = TRUE) : file.show():ファイル 'C:\DOCUME~1\蜿、蟾晏忽蠢予LOCALS~1\Temp\RtmpIDu7yb\Rtxt678418be' は存在しません > help("glm") 警告メッセージ: In file.show(temp, title = gettextf("R Help on '%s'", topic), delete.file = TRUE) : file.show():ファイル 'C:\DOCUME~1\蜿、蟾晏忽蠢予LOCALS~1\Temp\RtmpIDu7yb\Rtxt3d6c4ae1' は存在しません検索エンジン等で調べてはみましたが、未だ解決していません。
環境は以下の通りです。Microsoft Windows XP Professional Version 2002 Service Pack 3 Intel(R) Core(TM)2 Duo CPU E8400 @ 3.00GHz 2.99 GHz、976 MB RAM Rのバージョン R version 2.11.1 (2010-05-31)
sh (2010-09-16 (木) 11:50:21)
regexpr(pattern, text, perl = T)を実行したところ、以下のエラーが出力されました。patternの文字数(59291文字)を減らせば、正常に動作します。patternの文字数を減らすことなく、正常に動作させることは可能でしょうか?環境は、R version 2.11.1 (2010-05-31); x86_64-apple-darwin9.8.0です。以下にエラー regexpr(pattern, text, perl = T) : 追加情報: 警告メッセージ: In regexpr(pattern, text, perl = T) : PCREパターンのコンパイルエラー 'regular expression is too large' at ''
> text [1] "A11" "A12" "A13" "A14" "A15" "A16" "A17" "A18" "A19" "A20" "A21" "A22" [13] "A23" "A24" "A25" "A26" "A27" "A28" "A29" "A30" "A31" "A32" "A33" "A34" [25] "A35" "A36" "A37" "A38" "A39" "A40" > text1 <- gsub(pattern = "A11|A15|A20|A27",NA,text) # マッチしたものをNAで置き換え > text1 [1] NA "A12" "A13" "A14" NA "A16" "A17" "A18" "A19" NA "A21" "A22" [13] "A23" "A24" "A25" "A26" NA "A28" "A29" "A30" "A31" "A32" "A33" "A34" [25] "A35" "A36" "A37" "A38" "A39" "A40" > text2 <- gsub(pattern = "A29|A31|A35|A36|A39",NA,text1) > text2 [1] NA "A12" "A13" "A14" NA "A16" "A17" "A18" "A19" NA "A21" "A22" [13] "A23" "A24" "A25" "A26" NA "A28" NA "A30" NA "A32" "A33" "A34" [25] "A35" NA "A37" "A38" NA "A40" > is.na(text2) [1] TRUE FALSE FALSE FALSE TRUE FALSE FALSE FALSE FALSE TRUE FALSE FALSE [13] FALSE FALSE FALSE FALSE TRUE FALSE TRUE FALSE TRUE FALSE FALSE FALSE [25] TRUE TRUE FALSE FALSE TRUE FALSE > text[is.na(text2)] # マッチした文字列 [1] "A11" "A15" "A20" "A27" "A29" "A31" "A35" "A36" "A39"
> text <- paste('AAA11111', seq(11, 29), sep='')
> pattern = c('AAA1111111', 'AAA1111112')
> text %in% pattern
[1] TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[12] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSEg (2010-09-14 (火) 11:54:44)
例えば,A,B,C 3121234567890,10,20 3122345678901,20,30 3123456789012,30,40という中身のcsvファイルa.csvをread.csvで読み込ませると
> read.csv("a.csv") A B C 1 3.121235e+12 10 20 2 3.122346e+12 20 30 3 3.123457e+12 30 40となってしまいます.浮動小数点ではなくそのままの形で読み込ませるにはどうしたらいいでしょうか.
> (X <- read.csv("a.csv"))
A B C
1 3.121235e+12 10 20
2 3.122346e+12 20 30
3 3.123457e+12 30 40
> options(digits=22)
> X
A B C
1 3121234567890 10 20
2 3122345678901 20 30
3 3123456789012 30 40> options(digits=22) > 123456789012345 [1] 123456789012345 > 1234567890123456 [1] 1234567890123456 > 12345678901234567 # 近似による誤差が生じ始める [1] 12345678901234568 > 123456789012345678 [1] 123456789012345680 > 1234567890123456789 [1] 1234567890123456768 > 12345678901234567890 [1] 12345678901234567168 > 123456789012345678901 [1] 123456789012345683968 > 1234567890123456789012 # 浮動小数点表示になり始める [1] 1.234567890123457e+21
id (2010-09-12 (日) 21:34:03)
密度関数を直接積分することで、分布関数を求めてプロットしようとしているのですが、うまくいきません。
簡単に書きなおすと以下のようなコードで、実行するとエラーを吐きます。> pdf <- function(x) exp(-x) > cdf <- function(x) integrate(f, 0, x) > plot(cdf) 以下にエラー xy.coords(x, y, xlabel, ylabel, log) : 'x' and 'y' lengths differcdf(Inf) などとすると正常な答えを返してくれるので、関数として機能はしているようですがプロットしてくれません。
プロットするにはどうすればよいでしょうか?
ちなみに実際に考えている分布は beta prime distribution です。
この分布の cdf に出てくる 2F1 関数の扱いがわからず、上のような苦肉の策をしようと思っています。
> pdf <- function(x) exp(-x) > cdf <- function(x) integrate(f, 0, x) > plot(cdf) 以下にエラー match.fun(f) : オブジェクト 'f' がありません
> cdf <- function(x) sapply(x,function(t) integrate(pdf,0,t)$value)
sakura (2010-09-12 (日) 17:26:35)
Rで、pooled adjacent violator algorithm を使ったライブラリーには何があるのでしょうか?
mtanaka (2010-09-07 (火) 20:12:53)
for文で任意の関数に連番で命名したオブジェクトを入力する場合どのように記述すればよいのでしょうか?
for文内でのファイル名の記述様式がわかりません。関数 func() オブジェクト(入力データ): hoge01, hoge02, hoge03 オブジェクト(出力データ): piyo01, piyo02, piyo03 実行例 piyo01 <- func(hoge01) piyo02 <- func(hoge02) piyo03 <- func(hoge03)この上記の実行例をfor文で記述したいです。
どなたか、ご助言いただける方がいらっしゃいましたら、宜しくお願いいたします。
hoge01 <- 1 # 関数に作用させる対象
hoge02 <- 2
hoge03 <- 3
hoge04 <- 4
for (i in 1:4) { # それぞれの対象について func を作用させ,結果を格納することの記述
eval(parse(text=paste(sprintf("piyo%02i <- func(hoge%02i)", i, i))))
}
piyo01 # 結果を参照するときに,一々名前を引用しないといけない
piyo02 # 何の処理をすることなく,単に結果をファイルに書き出すだけであっても同じ
piyo03
piyo04
しかし,普通は以下のようにした方がもっと扱いやすいでしょう。
hoge <- 1:4 # 関数に作用させる対象を配列なりリストなりで表す
piyo <- func(hoge) # 関数を作用させ結果も配列やリストに格納する
# 場合によっては,for ループで処理しないといけない場合もあるだろう
piyo # 結果を引用するときも,配列やリストの要素を参照する
eval(parse(text=foo)) は,今のあなたのやりたいことのために使うものじゃないでしょう。> hoge1 <- 1; hoge2 <- 2; hoge3 <- 3; hoge4 <- 4
> N <- 4
> X <- vector("list",N)
> for (i in 1:N) X[[i]] <- eval(parse(text=paste("hoge",i,sep="")))
> str(X)
List of 4
$ : num 1
$ : num 2
$ : num 3
$ : num 4
> foo <- function(x) x^2
> Y <- lapply(X,foo)
> str(Y)
List of 4
$ : num 1
$ : num 4
$ : num 9
$ : num 16
> Y[[1]];Y[[2]];Y[[3]];Y[[4]]
[1] 1
[1] 4
[1] 9
[1] 16rcddnsj (2010-09-06 (月) 23:54:37)
Windows7でR version 2.11.1を使用しております。
サポートベクターマシンを試してみたくて、パッケージe1071をインストールし、関数svmを実行するところまでは、たどり着きました。
しかし、e1071に含まれているはずの関数predict.svmで予測しようとすると、「関数 "predict.svm" を見つけることができませんでした」とのエラーでpredict.svmを呼び出せませんでした。
操作はRcmdrのスクリプトウィンドウから実行しています。
このように、パッケージに含まれているはずの関数が呼び出せない場合、どのような原因とあるいは、対処法が考えられるのでしょうか?
Rの再インストール、パッケージe1071の再インストールなどはもちろん試しています。
sessionInfo() の実行結果は下記の通りです。R version 2.11.1 (2010-05-31) i386-pc-mingw32 locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] splines tcltk stats graphics grDevices utils datasets methods base other attached packages: [1] SparseM_0.86 e1071_1.5-24 class_7.3-2 Rcmdr_1.6-0 car_2.0-2 survival_2.35-8 nnet_7.3-1 [8] MASS_7.3-7 loaded via a namespace (and not attached): [1] tools_2.11.1どなたか、ご助言いただける方がいらっしゃいましたら、宜しくお願いいたします。
Usage:
## S3 method for class 'svm':
predict(object, newdata, decision.values = FALSE,
probability = FALSE, ..., na.action = na.omit)
分かりますよね? -- 2010-09-07 (火) 00:07:05ito (2010-08-31 (火) 20:12:14)
あるベクトルについて、他のベクトルの中のNAに対応する要素だけをNAに変換したいのです。よい方法はないでしょうか。x <- c(1,3,4,6,8,9) y <- c(1,NA,2,NA,3,4)変換後
> x [1] 1 NA 4 NA 8 9となるようにしたいのです。
ifelse(x[is.na(y)],NA)とか考えたのですが、うまくいきません。
よろしくおねがいします。
mapply(max, x, y)mapplyは1つ目の引数に指定した関数を2つ目以降に指定したベクトルから1つずつ取り出して実行する関数。 -- Spica 2010-08-31 (火) 20:18:23
func <- function(x, y)
{
nx <- length(x)
ny <- length(y)
if (nx <= ny) {
return(ifelse(is.na(y[1:nx]), NA, x))
}
else {
return(c(ifelse(is.na(y), NA, x[1:ny]), x[(ny+1):nx]))
}
}
# 長さが同じときには色々な方法がある
> x <- c(1,3,4,6,8,9)
> y <- c(1,NA,2,NA,3,4)
> func(x, y)
[1] 1 NA 4 NA 8 9
# x の長さが短いとき,x と同じ長さに y を切り詰めて,x, y を同じ長さにして処理
> x <- c(1,3,4,6,8)
> y <- c(1,NA,2,NA,3,NA)
> func(x, y)
[1] 1 NA 4 NA 8
# x の長さが長いとき,y と同じ長さに x を切り詰めて処理した後,残りの x を接続
> x <- c(1,3,4,6,8,9,10,11)
> y <- c(1,NA,2,NA,3,4)
> func(x, y)
[1] 1 NA 4 NA 8 9 10 11> 0*y [1] 0 NA 0 NA 0 0 > x + 0*y [1] 1 NA 4 NA 8 9
初心者 (2010-08-30 (月) 16:56:25)
いつもお世話になっております。
pls回帰分析をしたいのですが,Rですと,ベータは求められても,そのベータが有意かの検定はできないのでしょうか。またできるのなら,どのように行うのでしょうか。pls回帰分析を学ぶに当たり,岩田先生のpls回帰入門やAcreMaker様のホームページを見ましたが,有意かどうか扱っていませんでしたので,質問させて頂きました。
使用環境は、R2.11.1です。OSはWindowsVista です。
よろしくお願いします。
Saito (2010-08-27 (金) 08:52:15)
いつもお世話になっております。
似た様な質問はいくつかあったのですが、意外にも、条件抽出が複数で、かつ固定値ではなく、ベクトルで条件抽出をしている例が見当たらなかったので、質問させてください。
ある参照列(座標と価がセット)があるときに、それを参照して、新しい座標から、それにマッチングする価を参照したいと思っています。以下がサンプルプログラムです。
set.seed(1) a <- seq(1, 10) b <- seq(1, 10) c <- rnorm(100) ###参照列### d <- data.frame(expand.grid(a=a, b=b), c=c) ###当てはめたい座標### e <- data.frame(a=sample(a, 50000, rep=T), b=sample(b, 50000, rep=T)) ###これでは上手く動かない### subset(d$c, d$a==e$a & d$b==e$b) ###これでも### subset(d$c, d$a%in%e$a & d$b%in%e$b)
つまり、eの座標軸が与えられたときに、dの座標軸と対応させて、dの三列目(c列)を引っ張って来たいのです。
for文でやろうと思えばできるのですが、実際はもっとeが大きくて、for文が実行スピード的に使えません。%in%も、&が入っていなければ使えたのですが、この場合上手く動作しないようです。おそらく単純な問題だと思うのですが、思うようにいきません。
どなたか、分かる方がいらっしゃいましたら、ご教示いただけると幸いです。
d2 <- matrix(d$c, 10, 10) e2 <- as.matrix(e) d2[e2]つまり,この問題は表引きで,d$a, d$b が添え字の二次元配列で,その要素が d$c。d$a, d$b の添え字はちょうど R の二次元配列の順序と同じなので,d$c を matrix 関数で行列にするだけで済む。e$a, e$b を添え字と見て,その要素を取り出すのだから,単純な表引き。しかし,この場合,別に d2 や e2 など作る必要はなくて,
d$c[e$a+10*(e$b-1)]だけでよいことがわかってしまう。
> Ord <- data.frame(ord=seq(nrow(e))) # 本来の順序を示す作業用変数 > A <- merge(merge(cbind(e,Ord),d),Ord) # 欲しい結果 > AA <- A[,-1] # 作業用変数列が目障りなら > str(e) 'data.frame': 50000 obs. of 2 variables: $ a: int 3 3 6 3 2 6 6 2 3 8 ... $ b: int 6 8 4 8 7 8 4 2 6 7 ... > str(A) 'data.frame': 50000 obs. of 4 variables: $ ord: int 1 2 3 4 5 6 7 8 9 10 ... $ a : int 3 3 6 3 2 6 6 2 3 8 ... $ b : int 6 8 4 8 7 8 4 2 6 7 ... $ c : num 0.3411 0.6107 -0.415 0.6107 -0.0392 ... > str(AA) 'data.frame': 50000 obs. of 3 variables: $ a: int 3 3 6 3 2 6 6 2 3 8 ... $ b: int 6 8 4 8 7 8 4 2 6 7 ... $ c: num 0.3411 0.6107 -0.415 0.6107 -0.0392 ...
もしくは(こちらの方が直感的でわかり易い?)
> ed <- merge(cbind(e,Ord),d) > str(ed) 'data.frame': 50000 obs. of 4 variables: $ a : int 1 1 1 1 1 1 1 1 1 1 ... $ b : int 1 1 1 1 1 1 1 1 1 1 ... $ ord: int 32810 46779 23088 6441 17984 15437 12878 47025 38836 21870 ... $ c : num -0.626 -0.626 -0.626 -0.626 -0.626 ... > ed[order(ed$ord),][,-3] # 上のAAと同じもの
学生 (2010-08-26 (木) 22:48:35)
RでPLSを行おうとして追います。
PLSパッケージをインストールし,読み込んでも,「関数"PLS"を見つけることができませんでした」と返ってきます。
Rになれるための練習として,インターネット「岩田先生のPLS回帰入門」の「wine」についてPLSで分析しようとしているのですが上手くいきません。
何卒よろしくお願い致します。
> library(pls) # pls パッケージを使うために
次のパッケージを付け加えます: 'pls'
The following object(s) are masked from 'package:stats':
loadings
> plsr(foo, ...) # plsr というのが,あなたが使いたい関数ですよね> wine <- read.table("wine.txt")
> Y <- scale(wine[,1:3])
> X <- scale(wine[,4:7])
> library(pls)
次のパッケージを付け加えます: 'pls'
The following object(s) are masked from 'package:stats':
loadings
> wine.pls <- plsr(X, Y, 1:3, validation="CV")
以下にエラー formula.default(object, env = baseenv()) : invalid formula> library(pls)
> wine <- read.table("wine.txt")
> Y <- scale(wine[,1:3])
> X <- scale(wine[,4:7])
> wine.pls <- plsr(Y~X, 3, data=wine)
> summary(wine.pls)
Data: X dimension: 5 4
Y dimension: 5 3
Fit method: kernelpls
Number of components considered: 3
TRAINING: % variance explained
1 comps 2 comps 3 comps
X 70.45 98.35 100.0
Hedonic 70.53 70.71 100.0
Goes_with_meat 93.74 98.51 100.0
Goes_with_dessert 25.72 86.97 87.5
> coefficients(wine.pls, ncomp=2)
, , 2 comps
Hedonic Goes_with_meat Goes_with_dessert
Price -0.27140847 -0.2525422 0.01880366
Suger 0.06339537 0.3206223 0.78764141
Alcohol 0.28567521 0.3619657 0.27127454
Acidity 0.30722143 0.3730769 0.24272634> R574 <- read.csv("R574.txt")
> Y <- scale(R574[,9:10])
> X <- scale(R574[,5,8])
> R574.pls <- plsr(Y~X, 3, data=R574)
以下にエラー mvr(Y ~ X, 3, data = R574, method = "kernelpls") :
Invalid number of components, ncompま (2010-08-26 (木) 16:50:23)
図を作成した際に,x軸やy軸に添えられる数字が指数表示(1e-01,1e+01など)に自動的になります。これを回避したいのですが,どなたか方法を教えて頂けませんでしょうか?
何卒よろしくお願い申し上げます。
scipen.org <- getOption('scipen')
x <- seq(0, 0.001, 0.0001)
plot(x)
options(scipen=10)
plot(x)
options(scipen=scipen.org)atsuo (2010-08-23 (月) 11:52:39)
optimやconstrOptimで解決できないかと、あれこれやってみたのですが、
以下の線形計画問題を解決したいのです。
� 2*θ−2*λ[1]−2*λ[2]−1*λ[3] >= 0
� 1*θ−1*λ[1]−2*λ[2]−2*λ[3] >= 0
� 1 −1*λ[1]−1*λ[2]−1*λ[3] <= 0
� λ[1]+λ[2]+λ[3] >= 1
� λ[1]>=0
� λ[2]>=0
� λ[3]>=0
� �〜�の条件を満たし、θを最小にする問題です。
欲しいのはθの値とλ[1]、λ[2]、λ[3]の値です。
これはDEA分析なのですが、パッケージのFEARでも準備されていない解θが欲しいのです。
環境は、R2.11.1です。OSはWindows7 です。
> sim <- function(trial=10000, mx=1)
+ {
+ count <- 0
+ min.theta <- 10000
+ while(TRUE) {
+ lambda <- runif(3, min=0, max=mx) # 一様乱数発生 条件(5), (6), (7)
+ if (sum(lambda) >= 1) { # 条件(4)
+ theta <- max(lambda%*%c(1, 1, 0.5), lambda%*%c(1, 2, 2)) # θは(1)と(2)を満たす大きい方
+ if (theta < min.theta) { # それまでに見つかったものより小さければ書き出す
+ cat(theta, lambda, "\n")
+ min.theta <- theta # 見つかった解の候補で更新
+ ans <- c(theta, lambda)
+ count <- 0 # 空振り回数の更新
+ }
+ else {
+ count <- count+1 # 空振りが以下の回数続けば現在の解を最良とする
+ if (count > 1000000) return(ans)
+ }
+ }
+ }
+ }
> (ans <- sim()) # 出力は順に,θ,λ[1],λ[2],λ[3]
2.961947 0.9688506 0.2850662 0.711482
2.460659 0.9278959 0.2115464 0.5548352
1.890355 0.7575402 0.4784459 0.08796157
1.857158 0.2751631 0.7211324 0.06986522
途中省略
1.030237 0.9743367 0.00815206 0.01979813
1.029195 0.9880413 0.004979686 0.01559721
1.023945 0.9964487 0.005063805 0.008684346
[1] 1.023944978 0.996448677 0.005063805 0.008684346 # これが最終解
> # 念のために条件のチェック
> 2*ans[1]-2*ans[2]-2*ans[3]-ans[4] >= 0 # (1)
[1] TRUE
> ans[1]-ans[2]-2*ans[3]-2*ans[4] >= 0 # (2)
[1] TRUE
> sum(ans[2:4]) >= 1 # (3), (4)
[1] TRUE
> all(ans[2:4] >= 0) # (5), (6), (7)
[1] TRUE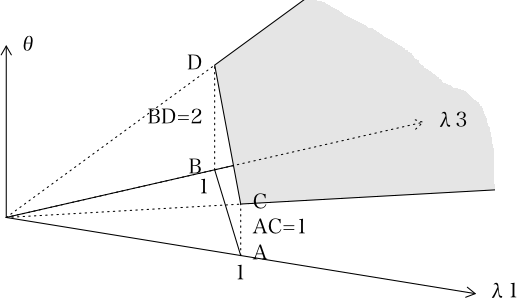
z (2010-08-20 (金) 22:42:44)
Coefficient(s)や、Hessian Matrixに標示される、muやomegaの解釈方法はどこを参照すればよいのでしょうか?ご教示頂ければ幸いです。
例) Coefficient(s):
mu ar1 ma1 omega alpha1
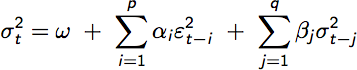
なるほど、貴重なアドバイスを有難うございます。出てきたGarch項を推計式の誤差項に代入する場合には、同時方程式を使う形になるのでしょうか? テキストを見比べて、自分で式を考えてみたのですが、なかなかうまくいきません。 アドバイス頂けると幸いです。宜しくお願い致します。 (fgarchはうまくInstallできなかったため、そちらのマニュアルを確認することは、失念していました。調べ方が甘く、すみませんでした。 ご丁寧にアドバイスをいただいて、有難うございました。)-- z 2010-08-31 (火) 19:05:42
rの初心者 (2010-08-20 (金) 16:28:28)
とあることでRを用いて多変量解析を行う必要が生じまして、ダウンロードしてインストールしました。ところがメッセージ言語は文字化けしているようで日本語で表示されません。ただし「ファイル」「ヘルプ」といった表示は正常です。どうすればよろしいでしょうか? よろしくお願いします。
z (2010-08-20 (金) 14:29:41)
RでTARCH(Threshold GARCH)をLoopで100社*10年分行おうと調べてみたのですが、該当する機能が見つかりませんでした。
RでTARCHするのと同じ結果を得られる機能がございましたらご教示頂けると幸いです。
宜しくお願いします。
青葉ほととぎす (2010-08-20 (金) 14:18:14)
最近、Rでプログラムを始めたものです(Vistaでversion 2.10.1を使用)。
あるプログラムを作っているときに不可解な結果がでて、いろいろ試した結果、forループのところに問題がることが分かってきました。
以下は、問題の部分だけを抜き出したものです。
初め実験1を行っていたのですが問題があり、実験2のように書き変えたところうまくいきました。sim.rate <- seq(0, 1, by=0.01) result.matrix <- matrix(0, nrow=length(sim.rate), ncol=2) # 実験1 for(i in sim.rate) { result.matrix[i*100, 1] <- result.matrix[i*100, 1]+1 } # 実験2 for(i in 1:length(sim.rate)) { result.matrix[i, 2] <- result.matrix[i, 2]+1 }この2つは以下のような異なる結果を出します。
[,1] [,2] [1,] 1 1 中略 [27,] 1 1 [28,] 2 1 [29,] 0 1 [30,] 1 1 中略 [56,] 1 1 [57,] 2 1 [58,] 0 1 [59,] 1 1 中略 [95,] 1 1そこで質問ですが、forループのリストしてベクトルを使ってはいけないのでしょうか?なぜこんな違いが生じるのかも教えていただけると助かります。
> result.matrix[0, 1] numeric(0)もう一つは,i は sim.rate という実数値をとるので,0.05*100 は 5 になるのですが,いつも,期待されるとおりの結果にはならないということに注意が必要です。以下のように,sim.rate の 26 〜 32 番目の要素は 0.25 〜 0.31 ですが,それを使って計算される添え字は as.integer(sim.rate[26:32]*100)つまり,100 倍して,小数部を除いたものなので,29 になってほしいところが 28 になっているのがわかるでしょう(29 はなく,28 が 2 回出ていますよね)。
> sim.rate[26:32] [1] 0.25 0.26 0.27 0.28 0.29 0.30 0.31 > as.integer(sim.rate[26:32]*100) [1] 25 26 27 28 28 30 31ようするに,コンピュータの中での実数は(整数値や,2 進数で循環小数にならずに正確に表現できる実数を除いて),あくまでも「近似値」なのです。近似値を整数倍して小数部を除くと,期待されるのとは違う結果になることがあるということです。
> s <- 0 > for (i in 1:100) s <- s+0.01 # 0.01 を 100 回足し込む > s # 結果を書いて見ると [1] 1 # ちゃんと 1 になっているように見えますが > s == 1 # 1 と等しいかどうか見てみると [1] FALSE # 1 とは等しくないということがわかります
ななしのごんべ (2010-08-12 (木) 19:53:54)
Windows7 Home Premiumで最新のRを入れたら、パッケージがインストールできません。「コンピューターにiconv.dllがないため、プログラムを開始できません。この問題を解決するには、プログラムを再インストールしてみてください。」というエラーウィンドウが出ます。どなたか解決策をご教示いただけるとありがたいです。
しょーじん (2010-08-11 (水) 14:36:14)
積分した値を計算に使おうと思ったところ,「二項演算子の引数が数値ではありません」と出てしまい,計算に使えません.
その値自体を調べると「0.5 with absolute error < 5.6e-15」とでます.
計算に使うためにはどうしたらよいのでしょうか?
> x <- integrate(dnorm, -1.96, 1.96) > x 0.9500042 with absolute error < 1.0e-11 > x + 2 以下にエラー x + 2 : 二項演算子の引数が数値ではありません > str(x) List of 5 $ value : num 0.95 $ abs.error : num 1.05e-11 $ subdivisions: int 1 $ message : chr "OK" $ call : language integrate(f = dnorm, lower = -1.96, upper = 1.96) - attr(*, "class")= chr "integrate" > x$value [1] 0.9500042 > x$value + 2 [1] 2.950004
z (2010-08-07 (土) 23:56:43)
OLSを試みています。
推計式はComp1=α+βcomp0なので、以下の要領で、Syntaxを書いたら、Errorが出ました。--Syntax----- Result1<-lm(comp1~comp0) --error-------- Error in eval(expr, envir, enclos) : object 'comp1' not found利用しているデータは下記の要領で、Comp0-90 まで、Dayは1-150まで。回帰式は、Comp<n>=α+βComp0 を Comp1-90まで順にComp0で回帰する予定です。
day comp0 comp1 comp2 comp3 comp4 1 -0.010873538 -0.017208413 0.009237875 0.000000000 -0.012048193 2 0.007344111 -0.001945525 -0.010297483 0.013313609 0.000000000 3 0.016795163 0.031189084 -0.019653179 0.002919708 0.034146341 4 0.006113627 0.003780718 0.004716981 0.010189229 0.007075472 5 -0.002080986 -0.007532957 0.011737089 0.010086455 0.145199063 6 0.005460344 0.001897533 -0.012761021 -0.021398003 0.022494888Comp2以降はLoopでの処理を試みる予定なのですが、Comp1についての式は間違っていないようなのにエラーが出るので、調べ方にも窮しています。
a <- vector(mode="list", length=90)
for (i in 3:92) {
a[[i-2]] <- lm(d[, 2] ~ d[, i]) # このような使い方のときには,第2引数にデータフレームは指定不要
}
また,投稿の仕方(投稿書式)についても確認してください。読みにくい質問は答えてやろうという気がわかないかも知れませんね。上の質問は書き直しました。あなたが投稿した後に表示されたものと比較してみてください。どのようにすれば上のように表示されるのか調べてください。b <- lapply(paste("comp", 1:90, sep=""), function(x) lm(comp0 ~ eval(parse(text=x)), d))for (i in comp1: comp90){lm(d[,0]~d[,i])}としてみたのですが、以下のようなErrorが出ました。 -- z 2010-08-09 (月) 15:48:16
> for (i in 1:90) {
+ lm(d[, 0] ~ d[, i])
+ }
Error in eval(expr, envir, enclos) : object 'd' not found
> for (i in 3:92) {
+ lm(result1[, 0] ~ result1[, i])
+ }
Error in result1[, 0] : incorrect number of dimensions
> for (i in comp1:comp89) {
+ lm(result1[, 0] ~ result1[, i])
+ }
Error: object 'comp89' not found
> for (i in comp1:comp3) {
+ lm(result1[, 0] ~ result1[, i])
+ }
Error in result1[, 0] : incorrect number of dimensions In addition:
Warning messages:
1: In comp1:comp3 : numerical expression has 150 elements: only the first used
2: In comp1:comp3 : numerical expression has 150 elements: only the first used
> lm(result1[, 0] ~ result1[, i])a <- vector("list", length=4) # lm の結果は list なので,必要なメモリを確保
for (i in 3:6) { # i は順に 3, 4, 5, 6 をとる
a[[i-2]] <- lm(d[, i] ~ d[, i-1]) # i=3 のとき,a[[1]] <- lm(d[,3] ~ d[,2]) が実行される
}
a
# function には順に 1, 2, 3, 4がわたされ,
# lm(eval(parse(text="comp1")) ~ eval(parse(text="comp0")), data=d) が実行される
# 最終的には lm(comp1 ~ comp0, data=d) が実行される "comp1" と comp1 は別物だということに注意
b <- lapply(1:4, function(i) lm(eval(parse(text=sprintf("comp%i", i))) ~
eval(parse(text=sprintf("comp%i", i-1))), data=d))
b
# lapply っていったって,for とそうは変わらないのだ
C <- vector("list", length=4)
for (i in 1:4) {
C[[i]] <- lm(eval(parse(text=sprintf("comp%i", i))) ~
eval(parse(text=sprintf("comp%i", i-1))), data=d)
}
C
それぞれの関数の引用について,なにをやっているのかを確認すること。> a <- vector("list", 10)
> class(a) # vector は第一引数で指定するオブジェクトを
[1] "list"
> length(a) # 第二引数で指定する個数分用意する
[1] 10
> a[[2]] <- c(1, 3, 5) # リストの2番目の要素に付値(代入)する
> a[[2]] # 結果を表示してみる
[1] 1 3 5
> parse(text="comp1")
expression(comp1) # comp1 が作られるが,それは expression である
attr(,"srcfile")
<text>
> comp1 = "test dummy" # 例えば comp1 が "test dummy" という文字列だとすれば
> eval(parse(text=sprintf("comp%i", i)))
# これは,comp1 ということになり,それを print で表示すると "test dummy" という文字列になる
[1] "test dummy"
> print(eval(parse(text=sprintf("comp%i", i)))) # 上と同じ
[1] "test dummy"
これくらい冗長に書けばよいかな? -- 河童の屁は,河童にあらず,屁である。 2010-08-09 (月) 16:19:20gqt<-lapply(1:88, function(i){
gqtest( lapply(1:88, function(i){
lm(eval(parse(text=sprintf("comp%i", i))) ~ eval(parse(text=sprintf("comp%i", 0))), data=dataset)
}), data= dataset,fraction=50, order.by=NULL) })
Error in terms.default(formula) : no terms component
どうアレンジしたら良いのでしょう? 調べ方等、アドバイスを頂けると幸いです。 宜しくお願い致します。 -- z 2010-08-31 (火) 19:00:42
z (2010-08-07 (土) 21:47:40)
二進も三進も行かず、困っています。データを読み込む行はエラーが出ないのですが、二行目から+が出て、どんな構文を打っても、+が続きます。どこが間違っているのでしょうか?
例えば、下記のようになります。dataset<-read.csv("C:/Users/(myname)/Documents/(foldername) /(sheet name).csv”, header=T) + attach (dataset) +
森の熊五郎 (2010-08-05 (木) 16:21:03)
X <- c(0.1, 0.2, 0.3, 0.4) Y <- c(0.3, 0.5, 0.8, 0.5) Z <- c(0.6, 0.5, 0.7, 0.9) sum(X) sum(Y) A <- rbind(X, Y) sum(A) sum(Z) B <- rbind(A, Z) sum(B)この式を繰り返し文 forを使って実行する方法を知りたい。
BASICでは例えば、A(20,10)の各要素を足し算する方法としてS=0 for I=1 to 20 for J=1 to 10 S=S+a(i,j) next nextなる文にて計算が出来ますが、Rの場合、内側のループはsum文で処理をするとして、外側のループをFor文で制御仕様とした場合、どのように文を組めば良いでしょうか?ご教示下さい。上記の例ではsum文とrbind文と使えばよいのですが、rbind文を使わず、for文を使って組む方法を知りたいと思います。
A <- matrix(1:16, nrow=4, ncol=4)
B <- matrix(1:20, nrow=4, ncol=5)
nrA <- nrow(A)
ncA <- ncol(A)
ncB <- ncol(B)
S <- matrix(NA, nrow=nrA, ncol=ncA+ncB)
for (i in 1:nrA) {
for (j in 1:ncA) S[i,j] <- A[i,j]
for (j in 1:ncB) S[i,j+ncA] <- B[i,j]
}
# もし一重ループなら
S <- matrix(NA, nrow=nrA, ncol=ncA+ncB)
for (i in 1:nrA) {
S[i,1:ncA] <- A[i,]
S[i,ncA+1:ncB] <- B[i,]
}a <- rbind(X, Y, Z)
S <- 0
for (i in 1:nrow(a)) {
for (j in 1:ncol(a)) {
S <- S+a[i, j]
}
}
print(S)
# 内側のループはsum文で というへんな要望に答えるなら
a <- rbind(X, Y, Z)
S <- 0
for (i in 1:nrow(a)) {
S <- S+sum(a[i, ])
}
print(S)
# でもそんなことする必要もなくて
a <- rbind(X, Y, Z)
S <- sum(a)
print(S)
# rbind する必要さえなくて
print(sum(c(X, Y, Z)))
# どれも,あなたのプログラムの最後に sum(B) としたのと同じ答えを得ることになりますよ?
# それともまさか,
S <- 0
for (i in c("X", "Y", "Z")) {
S <- S+sum(eval(parse(text=i)))
}
print(S)
# または
S <- 0
for (i in c(X, Y, Z)) {
S <- S+sum(i)
}
print(S)
# みたいなのをお望みで?まさかね〜yoshi (2010-08-02 (月) 14:56:40)
memory.limit()について質問です。
R上でmemory.limit()
[1] 1535memory.limit(T)
[1] 12memory.limit(F)
[1] 10
となります・ここでmemory.limit(4000)
[1] 4000
とした後はmemory.limit()
[1] 4000memory.limit(T)
[1] 12memory.limit(F)
[1] 10
となります。ここで質問なんですが「memory.limit()」「memory.limit(T)」「memory.limit(F)」の違いはなんなのでしょうか?今現在Rのメモリの上限のことで非常に困っています。また、皆さんはこのようなコマンドをどこから学んでいるのでしょうか?もし、良いWEBページ等がありましたら教えてくれると幸いです。
yoshi (2010-08-02 (月) 10:49:35)
パッケージ「R.huge」のパッケージの読み込みが上手くいきません。
私がやったことは
�CRANミラーサイトから「R.huge」のzipファイルをダウンロード
�RのGUIで「ローカルにあるzipファイルからのパッケージのインストール」で「R.huge」のインストール
→RのGUI上では「パッケージ 'R.huge' は無事に開封され、MD5 サムもチェックされました 」と表示
�RのGUIで「パッケージの読み込み」で「R.huge」を選択し実行
→RのGUI上で「要求されたパッケージ R.oo をロード中です
エラー: パッケージ 'R.oo' をロードできませんでした
追加情報: 警告メッセージ:
In library(pkg, character.only = TRUE, logical.return = TRUE, lib.loc = lib.loc) :
'R.oo' という名前のパッケージはありません 」と表示
このように�までは良いと思うのですが、�でエラーが表示されてしまい、パッケージ「R.huge」が読み込まれていないと思います。
これを解決する方法を自分でも探したのですが分らなかったため今回投稿さしていただきました。よろしくお願いします。
aMC (2010-08-01 (日) 06:07:54)
たとえば、2→10なので1桁目、5→101なので2桁目、7→0111なので4桁目、などのように、2進数表記した時、右から数えて最初にゼロが出現する桁数を求める効率のよいやり方はありますでしょうか?ベクトルを与えると、何桁目かをベクトルで返してくれるようなものが欲しいのですが、forループを使うと時間が掛かり過ぎます。
> i2b <- function(x)
+ {
+ res <- integer(32)
+ for (i in 1:32) {
+ res[i] <- x %% 2
+ x <- x %/% 2
+ }
+ return(rev(res))
+ }
> b2i <- function(x)
+ {
+ return(sum(x*2^(31:0)))
+ }
> (a <- i2b(615729635))
[1] 0 0 1 0 0 1 0 0 1 0 1 1 0 0 1 1 0 1 0 0 1 0 0 1 1 1 1 0 0 0 1 1
> (b <- b2i(a))
[1] 615729635
> which(rev(a)==0)[1]
[1] 3
繰り返しておきますが,このプログラムが効率が悪いと言っても,あなたの人生に比べれば取るに足りない時間です。回答を待つ時間が無駄です。 -- 河童の屁は,河童にあらず,屁である。 2010-08-01 (日) 21:48:11cnt <- function(vector) {
x <- abs(vector)
z <- x * 0 + 1
times <- max(floor(log(max(x), 2)), 0)
for(i in 1:times) {
y <- x
x <- x * 0 - 1
x <- x + (y %% 2 != 0) * (y + 1) / 2
z <- z + (x != -1)
}
return(z)
}> foo <- function(x){a <- ceiling(log2(x));
suppressWarnings(min(which((floor(x/2^(0:(a-1)))%%2)==0)))}
> foo(1*2^5+1*2^4+1*2^3+1*2^2+1*2+1)
[1] Inf
> foo(1*2^5+1*2^4+1*2^3+1*2^2+0*2+1)
[1] 2
> foo(1*2^5+1*2^4+1*2^3+1*2^2+0*2+0)
[1] 1
> foo(1*2^5+1*2^4+1*2^3+0*2^2+1*2+1)
[1] 3
> foo(1*2^5+0*2^4+1*2^3+0*2^2+1*2+1)
[1] 3
> foo(1*2^5+0*2^4+1*2^3+1*2^2+1*2+1)
[1] 5qMC (2010-07-31 (土) 22:56:24)
10進数→2進数、あるいは2進数→10進数へと変換する関数はありませんでしょうか?
りりぽん (2010-07-31 (土) 11:56:57)
Rscriptで次のように、test.Rを実行させると、グラフタイトルの日本語文字化けが発生します。Rscriptを使わずに、直接、test.Rを実行すると問題なく日本語が表示されます。どなたか、解決方法をご教授いただけたら幸いです。よろしくお願いいたします。###### test.bat ################### C:\R-2.11.1-x64\bin\Rscript test.R ####### test.R #################### postscript(file="test.eps", horizontal=F, family="Japan1Ryumin") plot(1:10, main="日本語") dev.off() ################################### 使用フォーマット・R環境: ソースコード;UTF-8(BOMなし)エンコード コマンドプロンプト;cp932;MSゴシック R version 2.11.1 (2010-05-31) x86_64-pc-mingw32 locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932
basel_firb (2010-07-29 (木) 15:18:47)
過去に「行列にfor文中の変数を含んだ名前をつける 」という質問があり、内容を読み、構文 eval(parse(text=...)を使用するということらしいのですが、よく分かりませんでした。
データフレームに、変数var1 var2 var3・・・var68というのがあり、for文を使って,h1=var1/var68,h2=var2/var68・・・と分子のvarを1〜67まで繰り返し計算したいのですが、どのようにすればよいのでしょうか?
h <- (d/d[,-68])[,-68]
colnames(h) <- paste("h", 1:67, sep="")
で十分です。 -- 河童の屁は,河童にあらず,屁である。 2010-07-29 (木) 17:09:13sano (2010-07-29 (木) 12:39:07)
こんにちは、hclustで階層的クラスタリングを行おうとすると、サイズ 3.4 Gb のメモリブロックを割り当てることができません。とエラーメッセージが出てしまいます。
OSはwindows7,Rのversionは version 2.11.1(64bit), メモリは48GB積んでいます。しかしwindowsのリソースモニターを見ると、11GB程度でまだ余裕があります。> memory.limit(T)と実行すると、
[1] 7363と表示されるので、Rが物理メモリを7GBしか、OSから割り当てられていないということではないかと思います。
30GB割り当ててもらおうと、> memory.limit(30000)と入力すると、
[1] 49149 警告メッセージ: In memory.size(size) : メモリー限界を減らすことができません。と表示され、うまくいきませんでした。
CPUがデュアルCPUでメモリを片側24GBずつ積んでいるのですが、このことも何か関係があるのでしょうか。
いろいろ調べてみたのですが、どうにもいかず、投稿させて頂きました。
お教えいただければ、幸いです。よろしくお願い致します。
sayaka (2010-07-26 (月) 11:59:13)
いつも参考にさせていただいております。自分でも本やGoogleなどで調べたのですがどうもわからず、質問させていただきました。
以下のようなデータ"test_data.csv"があるとき"T","T" "T","F" "T","T" "F","F" data <- read.csv("test_data.csv",header=F)というようにread.csvを使ってデータを読み込むと、以下のようにTやFが文字列ではなく論理値として出力されてしまいますが、どのようにすれば文字列として認識させることが出来るでしょうか?
> data V1 V2 1 TRUE TRUE 2 TRUE FALSE 3 TRUE TRUE 4 FALSE FALSEどうぞよろしくお願い致します。
> data <- read.csv("test_data.csv",header=F)
> data
V1 V2
1 T T
2 T F
3 T T
4 F F
5 A A
> data[-nrow(data),]
V1 V2
1 T T
2 T F
3 T T
4 F F
> str(data[-nrow(data),]) # 因子水準に "A" が残りますがご愛嬌
'data.frame': 4 obs. of 2 variables:
$ V1: Factor w/ 3 levels "A","F","T": 3 3 3 2
$ V2: Factor w/ 3 levels "A","F","T": 3 2 3 2> data <- read.csv("test_data.csv",header=F)
> is.data.frame(data)
[1] TRUE
> data <- lapply(data, function(x) ifelse(x, "T", "F"))
> is.data.frame(data)
[1] FALSE
> is.list(data)
[1] TRUE
> data <- as.data.frame(data)
> is.data.frame(data)
[1] TRUE
> data
V1 V2
1 T T
2 T F
3 T T
4 F FT,T,A
T,F,A
T,T,A
F,F,A
fixTF <- function(x) {
if (is.logical(x)) {
return(ifelse(x, 'T', 'F'))
}else {
return (x)
}
}
data <- read.csv("test_data.csv", header=FALSE)
data1 <- lapply(data, fixTF)
data1 <- as.data.frame(data1)
data1
V1 V2 V3
1 T T A
2 T F A
3 T T A
4 F F A
以上のようにうまくいきました。もっとよく勉強します。ありがとうございました。-- sayaka 2010-07-27 (火) 11:33:03em (2010-07-21 (水) 01:30:51)
runif()関数を使って一様乱数を生成し、0〜100の範囲に変換してみました。x <- round(runif(100), 2) * 100ここで、7の出ている回数を調べようと
length(x[x==7])とやると、実際にはベクトルxに7が含まれていても、0になってしまします。
また、x[x==7]のような式の結果もnumeric(0)となってしまいます。
どこがまずいのでしょうか?
> x <- 0 > for (i in 1:100) x <- x+0.01 > x # x を表示させると 1 になっているけど [1] 1 > x == 1 # 0.01 を 100 回足しても 1 にはなっていない [1] FALSE > (a <- round(0.0718*100)) [1] 7 > a == 7 # この場合は 7 だけど [1] TRUE > (b <- round(0.0718, 2)*100) [1] 7 > b == 7 # このようにして得られた数値は 7 ではない [1] FALSE > all.equal(b, 7) # testing ‘near equality’ all.equal と identical の違い [1] TRUE > identical(b, 7) # being exactly equal [1] FALSE
hashiro (2010-07-18 (日) 16:50:49)
「Rによる統計解析」を読みながらクラスター分析を行っています。
エクセルで、194の変数、160行のデータです。
R内に読み込んでデンドログラムまでは出来ました。
この時、デンドログラム最下段には1〜160までのNo.が表示されますが、
この数字をエクセルのa列の文字で表示したいのです。
plotの中にlabels= で指定すればいいみたいですが、指定の仕方が悪くエラーが出てしまいます。
指定方法をどうか教えていただけませんか。
t_y (2010-07-16 (金) 10:57:30)
今現在相関ルール抽出のパッケージ「arules」を用いているのですが、分からないことがあります。しかし、言葉で端的に言うのは難しいので例を交えて説明します。
買い物バスケットで以下の例があるとします。TID アイテム集合 1 {パン、牛乳} 2 {ハム、牛乳} 3 {ビール、たばこ}このデータを「data」に代入します。
> data <- list(c("パン", "牛乳"), c("ハム", "牛乳"), c("ビール", "たばこ”))そして transactions 形式のデータを「data.tran」に代入します。
> data.tran <- as(data1, "transactions")次に相関ルール「data.ap」に生成します
> data.ap <- apriori(data.tran)生成された相関ルールは以下の 3 つです。
lhs rhs support 1 {パン} => {牛乳} 0.3333333 2 {ハム} => {牛乳} 0.3333333 3 {ビール} => {たばこ} 0.3333333相関ルールは lhs⇒rhs で「lhs が存在する場合に rhs が存在する」という意味です。
Support は「特定のルールの数÷全てのルール」です。例えば{パン}→{牛乳}の場合、support=1/3 です。
ここでもしアイテム「パン」と「牛乳」が新たなアイテム「食べ物」に属するとします。
属した場合の相関ルールの例を載せますlhs rhs support 1 {食べ物} => {牛乳} 0.6666666 2 {パン} => {牛乳} 0.3333333 3 {ハム} => {牛乳} 0.3333333 4 {ビール} => {たばこ} 0.3333333このように「食べ物」の中に「パン」と「ハム」が含まれているため suppot=2/3 になります。
しかし、data2 に以下のように代入し、> data2 <- list(c(“食べ物”, ”牛乳”), c("パン", "牛乳"), c("ハム", "牛乳"), + c("ビール", "たばこ”))同様に相関ルールを作成すると、
lhs rhs support 1 {パン} => {牛乳} 0.25 2 {ハム} => {牛乳} 0.25 3 {ビール} => {たばこ} 0.25 4 {食べ物} => {牛乳} 0.25のようになり support の値が変化してしまいます。
これを解消する方法を教えて下さい。
Sensory (2010-07-15 (木) 19:17:26)
分散分析を行うと出現するエラーについてです.R,Rcommander,SensomineRを使っています.SensomineRよりpanel performance(分散分析)を起動させた場合,Product35×Panelist5×descriptor9ではエラーは出現しないのですが,Product31×Panelist5×descriptor9では,"置き換えるべき項目数が,置き換える数の倍数ではありませんでした”というエラーが出現します.
何か解決策があれば教えて頂けないでしょうか.よろしくお願い致します.
大学生 (2010-07-12 (月) 20:25:01)
SPSSファイルを、STATAファイルに変換したいのですが。library(foreign) read.spss("datafile",use.value.labels=FALSE)で、SPSSファイルを読み込んだあと、そのまま、STATAファイルに変換して、保存したいのですが。この後のコマンドがわかりません。
Rのバージョンは2.8.1で、MACです。どうかよろしくお願いします。
メジロウ (2010-07-09 (金) 21:24:59)
マトリックスを3次元グラフで描画した時、x、y、zの座標まで、グラフ内に頂点として表示されてしまいます
x、y、zの座標点を取り除き、純粋に数値の座標点のみを頂点として描画するにはどうすれば良いでしょうか?
打ち込んだコマンドラインは以下の通りですjhin <-matrix(c( 1.1479, 0.8587, 0.8719, -1.099, 0.353, 0.972, -1.3226, -0.4005, -0.9295, -0.4804, -0.7637, -1.3671, -0.1707, -1.3382, 1.1815, 0.2106, 0.7653, -1.7421, 0.3761, 1.8047, 0.0608, 0.4473, -1.3014, 0.7972, 1.625, 0.7415, 0.5675, 1.5616, -0.7194, -0.4122), nrow = 10, ncol = 3) jhin rownames (jhin) <- paste("in", 1:10, sep="") colnames (jhin) <- c("X", "Y", "Z") jhin rg <-jhin library(sna) gplot3d(rg, thresh = 1.1, displayisolates = TRUE, suppress.axes = FALSE, displaylabels = TRUE, xlab = "dimension 1", ylab = "dimension 2", zlab = "dimension 3")
library(rgl)
x <- c(0,1,1,0,0,1,1,0)
y <- c(0,0,1,1,0,0,1,1)
z <- c(0,0,0,0,1,1,1,1)
library(scatterplot3d)
plot3d(x, y, z, size=20, col=2)
library(sna)
gplot3d(cbind(x, y, z))
> cbind(x, y, z)
x y z
[1,] 0 0 0
[2,] 1 0 0
[3,] 1 1 0
[4,] 0 1 0
[5,] 0 0 1
[6,] 1 0 1
[7,] 1 1 1
[8,] 0 1 1
> library(sna) > gplot3d((as.matrix(dist(jhin)) <= 1.1)+0)
> (as.matrix(dist(jhin)) <= 1.1)+0
in1 in2 in3 in4 in5 in6 in7 in8 in9 in10
in1 1 1 0 0 0 0 0 0 0 0
in2 1 1 0 0 0 0 0 0 0 0
in3 0 0 1 0 0 0 0 0 0 0
in4 0 0 0 1 0 0 0 0 0 0
in5 0 0 0 0 1 0 0 0 0 0
in6 0 0 0 0 0 1 0 0 0 0
in7 0 0 0 0 0 0 1 0 0 0
in8 0 0 0 0 0 0 0 1 0 0
in9 0 0 0 0 0 0 0 0 1 0
in10 0 0 0 0 0 0 0 0 0 1
ラベルをつけたりなんだらかんだらは自分でやってください。library(rgl) plot3d(jhin, type="s", col=4, radius=0.05) text3d(jhin, text=1:10, adj=2) d <- as.matrix(dist(jhin)) con <- which(d <= 1.5, arr.ind=TRUE) apply(con, 1, function(xy) lines3d(jhin[xy,], lwd=3, col=2))
hello r (2010-07-09 (金) 15:46:59)
ググッたり、ここのDatagram tipsを見たのですが、解決しなかったので質問させていただけると助かります。
CSVファイルから読み込んだファイルに因子が含まれています。t s 1 3755 sd 2 3840 sd 3 3856 si 4 3884 sd 5 4011 si 6 4031 sd 7 4033 sd 8 4115 sd 9 4147 si 10 4157 si
ここから因子ごとにdataframeを分離したいと考えています。
因子でなければv <- dataframe[,"s"] == "sd" dataframeSD <- dataframe[v,]などとして抽出できると思うのですが、最初の因子の比較がDataframeの該当列の因子の取得方法が分からないため、マッチングが行えません。
どのように対処すればよいでしょうか。
もしよろしければご教授願えないでしょうか。
よろしくお願いします。
> v <- dataframe[,"s"] == "sd"
> dataframeSD <- dataframe[v,]
> dataframeSD
t s
1 3755 sd
2 3840 sd
4 3884 sd
6 4031 sd
7 4033 sd
8 4115 sd
split を使う方法を示したのは,その方が汎用性が高いから。 -- 河童の屁は,河童にあらず,屁である。 2010-07-09 (金) 18:06:55> v <- df[,2] == "si" > v [1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
> v <- d[,2] == "si" > v [1] FALSE FALSE TRUE FALSE TRUE FALSE FALSE FALSE TRUE TRUE > class(d[,2]) [1] "factor"
Saito (2010-07-06 (火) 00:54:49)
いつもお世話になっています。
ググっても、過去ログを漁っても、持っている本を読んでも、どうしてもわからなかったので、質問させてください。
都合により、メトロポリス法によるパラメータ推定プログラムを自作しています。が、メトロポリス法によるパラメータ推定がうまくいきません。以下に例を示します。
set.seed(1) iter <- 20000 ###真の値### a <- 10 b <- 2 x <- seq(0.01, 1, length=500) y <- b*x + rnorm(500, a, 1) ###尤度の計算(!!!対数にはしていない!!!)### cost_func2 <- function(a3, b3) { sum(dnorm(y, a3 + b3*x, 1)) } ###乱数と初期値の設定### u <- runif(iter, 0, 1) u2 <- runif(iter, 0, 1) a4 <- b4 <- numeric(iter+1) a4[1] <- b4[1] <- 5 lag <- 4 ###メトロポリス法(?)の実行### for(i in 1 : iter) { a4_2 <- a4[i] + runif(1, -lag, lag) b4_2 <- b4[i] + runif(1, -lag, lag) ###一つ前のサンプルで計算されるcost_funcの値が近すぎると、ほとんど採択される???### a4[i+1] <- ifelse(u[i] < cost_func2(a3=a4_2, b3=b4[i])/ cost_func2(a3=a4[i], b3=b4[i]), a4_2, a4[i]) b4[i+1] <- ifelse(u2[i] < cost_func2(a3=a4[i+1], b3=b4_2)/ cost_func2(a3=a4[i+1], b3=b4[i]), b4_2, b4[i]) } hist(a4[(iter/2):iter]) hist(b4[(iter/2):iter])
疑問点は二つです。メトロポリス法の場合でも、尤度を計算すると思いますが、対数尤度にするとマイナスになる場合があります。メトロポリス法では、棄却するか採択するかのときに、一つ前のパラメータのサンプルと現在のパラメータのサンプルの比をとります。しかし、比を取ってしまうと、マイナス同士が消しあってしまうこともあります。例えば、パラメータ-10が前回のサンプル、-11が今回のサンプルだとします。-10と-11の比は、1.1ですよね。同じく、10と11の比も1.1です。しかし前者は前回のサンプルよりも尤度は小さくなっており、後者は尤度が大きくなっています。しかし、比をとるだけだと、両者とも採択されてしまう気がします。そのため上記のプログラムでは対数にしていません。
もう一つの疑問は、メトロポリス法では、上記のように比を取った後、一定確率(u)で、尤度が小さい方向にサンプルされても、採択する場合があります。しかし、前回のサンプルと今回のサンプルの比が、例えば、先ほどの11、10でサンプルされる順番が逆だったとすれば、10/11=0.91となります。この0.9という数字は、ほとんど1に近く、ずっとこの比でサンプルが続けられるとすると、どんどん間違った方向へサンプルが続いていきます。
このようなことを避けるにはどうすればよいのでしょうか。
上記のプログラムですと、a4とb4の平均値がそれぞれ10と2になればよいのですが、うまくいきません。仮にb4を消して、切片モデルでやるとうまくいくのですが、それでは解決にならないので・・・。
どなたかわかる方がいましたら、ご教授のほどよろしくお願いいたします。
なお、環境はWindows7, R-2.11.1です。
a4[i+1] <- ifelse(cost_func2(a3=a4_2, b3=b4[i]) > cost_func2(a3=a4[i], b3=b4[i]),
a4_2, a4[i])
b4[i+1] <- ifelse(cost_func2(a3=a4[i+1], b3=b4_2) > cost_func2(a3=a4[i+1], b3=b4[i]),
b4_2, b4[i])
:
> mean(a4)
[1] 10.05778
> mean(b4)
[1] 1.890743likelihood <- 1
likelihood2 <- 0
cost_func2 <- function(a3, b3) {
for (i in 1 : length(y)) {
likelihood2 <- likelihood * dnorm(y[i], a + b*x[i], 1)
likelihood <- likelihood2
}
return(likelihood2)
}
などとして、対数を取らずに尤度の積を計算するようにしても、結果はやはり上手く推定できません。-- Saito 2010-07-07 (水) 10:08:08set.seed(1)
iter <- 20000
###真の値###
a <- 10
b <- 2
x <- seq(0.01, 1, length=500)
y <- b*x + rnorm(500, a, 1)
###正規化定数抜きの事後分布の計算###
###桁落ちするので、log(exp(1))を加えて、必ず1以上の数を返すように工夫(テクニックとしてアリ?)###
###マジメにa,bの事前分布を設定。真の値から平均を-1だけずらして、誤差は10として広く取った(注意!!!100だとダメ!)###
cost_func2 <- function(a3, b3) {
sum(log(log(exp(1)) + dnorm(y, a3 + b3*x, 1)*dnorm(a3, a-1, 10)*dnorm(b3, b-1, 10)))
}
###乱数と初期値の設定###
u <- runif(iter, 0, 1)
u2 <- runif(iter, 0, 1)
a4 <- b4 <- numeric(iter+1)
a4[1] <- b4[1] <- 5
lag <- 4
###メトロポリス法の実行###
for(i in 1 : iter) {
a4_2 <- a4[i] + runif(1, -lag, lag)
b4_2 <- b4[i] + runif(1, -lag, lag)
###a, bは一斉に値を更新するように変更###
if (u[i] < cost_func2(a3=a4_2, b3=b4_2)/
cost_func2(a3=a4[i], b3=b4[i])) {
a4[i+1] <- a4_2
b4[i+1] <- b4_2
} else {
a4[i+1] <- a4[i]
b4[i+1] <- b4[i]
}
}
###a, bの事後分布###
hist(a4[(iter/2):iter])
hist(b4[(iter/2):iter])
> mean(a4[(iter/2):iter])
[1] 9.688851
> mean(b4[(iter/2):iter])
[1] 2.424549
しかし、やはり対数の比のところはそのままの問題が残っていますし、事前分布の誤差を大きくとると、上手く推定されないようです。。。引き続き、何かお気づきの点がありましたら、ご教授頂けると幸いです。 -- Saito 2010-07-07 (水) 17:39:32set.seed(1)
iter <- 20000
###真の値###
a <- 10
b <- 2
c <- 1
x <- seq(0.01, 1, length=500)
y <- b*x + rnorm(500, a, c)
###正規化定数抜きの事後分布の計算###
###マジメにa,b.cの事前分布を設定###
cost_func2 <- function(a3, b3, c3) {
sum(log(dnorm(y, a3 + b3*x, c3)*
dnorm(a3, a-1, 100)*dnorm(b3, b-1, 100)*dgamma(c3, 100, 100)))
}
###乱数と初期値の設定###
u <- runif(iter, 0, 1)
a4 <- b4 <- c4 <- numeric(iter+1)
a4[1] <- b4[1] <- c4[1]<- 5
lag <- 0.4
###メトロポリス法の実行###
for(i in 1 : iter) {
a4_2 <- a4[i] + runif(1, -lag, lag)
b4_2 <- b4[i] + runif(1, -lag, lag)
c4_2 <- c4[i] + runif(1, -lag, lag)
###a, b, cは一斉に値を更新するように変更###
if (log(u[i]) < cost_func2(a3=a4_2, b3=b4_2, c3=c4_2)-
cost_func2(a3=a4[i], b3=b4[i], c3=c4[i])) {
a4[i+1] <- a4_2
b4[i+1] <- b4_2
c4[i+1] <- c4_2
} else {
a4[i+1] <- a4[i]
b4[i+1] <- b4[i]
c4[i+1] <- c4[i]
}
}
###a, b, cの事後分布###
hist(a4[(iter/2):iter])
hist(b4[(iter/2):iter])
hist(c4[(iter/2):iter])
> mean(a4[(iter/2):iter])
[1] 10.07981
> mean(b4[(iter/2):iter])
[1] 1.872144
> mean(c4[(iter/2):iter])
[1] 0.9888237
おそらく、これで問題なくコードできたと思います。皆様、本当にありがとうございました。そしてtoto様、分かりやすいコード例をありがとうございました。 -- Saito 2010-07-07 (水) 21:31:30> sum(dnorm(1:10, 0, 1, log = TRUE)) + dnorm(0, 0, 1, log = TRUE) + dnorm(0, 0, 1, log = TRUE) [1] -203.5273 > sum(log(dnorm(1:10, 0, 1)*dnorm(0, 0, 1)*dnorm(0, 0, 1))) [1] -220.0682
set.seed(1)
iter <- 20000
###真の値###
a <- 10
b <- 2
x <- seq(0.01, 1, length=500)
y <- b*x + rnorm(500, a, 1)
###正規化定数抜きの事後分布の計算###
###マジメにa,b.cの事前分布を設定###
cost_func2 <- function(a3, b3) {
sum(log(dnorm(y, a3 + b3*x, 1))) +
log(dnorm(a3, a-1, 100)) + log(dnorm(b3, b-1, 100))
}
###乱数と初期値の設定###
u <- runif(iter, 0, 1)
a4 <- b4 <- numeric(iter+1)
a4[1] <- b4[1] <-5
lag <- 0.4
###メトロポリス法の実行###
for(i in 1 : iter) {
a4_2 <- a4[i] + runif(1, -lag, lag)
b4_2 <- b4[i] + runif(1, -lag, lag)
###a, bは一斉に値を更新するように変更###
if (u[i] < exp(cost_func2(a3=a4_2, b3=b4_2)-
cost_func2(a3=a4[i], b3=b4[i]))) {
a4[i+1] <- a4_2
b4[i+1] <- b4_2
} else {
a4[i+1] <- a4[i]
b4[i+1] <- b4[i]
}
}
###a, b, cの事後分布###
hist(a4[(iter/2):iter])
hist(b4[(iter/2):iter])
> mean(a4[(iter/2):iter])
[1] 10.08433
> mean(b4[(iter/2):iter])
[1] 1.884697
分散パラメータが私の実力不足で上手くコードに含めることができなかったのは残念ですが、とりあえず、メトロポリスが何をやっているかは理解できたので初期の目標は達成できました。皆様、そしてtoto様、本当にありがとうございました。-- Saito 2010-07-08 (木) 07:30:03酔鯨 (2010-07-03 (土) 10:33:29)
spgwrの地理的加重回帰分析のgwrの予測値は、XXX$SDFのテーブル出力を見れば、predの項目(厳密には、ヘッダがずれているので、右に1項目ずらす必要がある。)を見ればよいことは判りました。しかし、この関数を使い予測するためには、被説明変数の値が必要です。つまり、何らかの方法で予測しなければなりません。通常の重回帰で予測をするためには、回帰係数と説明変数の値だけで良いです。地理的重回帰分析では、通常の重回帰の予測と同じように、回帰係数と説明変数だけで予測値を得る関数はないのでしょうか?
tadashi (2010-07-02 (金) 15:05:54)
"Rでアクセスログ" と検索してもでてきません。Rでアクセスログ(にかぎらず、ログデータ)の解析をすることはそれほどないのでしょうか?
もし、awstats 等でやっているようなことをRで代替している事例がありましたら、お教えください。
ちゃーぴー (2010-07-02 (金) 10:04:01)
RjpWiki内の統計解析Tipsにある反復測定分散分析 (Repeated measured ANOVA)(http://www.okada.jp.org/RWiki/index.php?R%A4%CE%C5%FD%B7%D7%B2%F2%C0%CF%B4%D8%BF%F4Tips#content_1_7)を使おうと思い,コードをコピペしようとしました.しかし,print.rep.anovaのコードが,ペーストしている途中でエラーとなります.当方の環境WindowsXP×2,Windows7-64bitでRのバージョンは2.11.0です.なお,64bitマシンでは32bit版,64bit版R両方を使用しています.3台のマシン全てで同様の結果となります.下記のような感じです.どなたか対策をご存知の方いらっしゃいますでしょうか?+ rownames(ttx)[rownames(ttx)=="Residuals"]<-sub("Error: (.*\)","Error(\\1)",names(tx)[i]) エラー: "Error: (.*\)"で始まる文字列の中で '\)' は文字列で認識されないエスケープです
rownames(ttx)[rownames(ttx)=="Residuals"]<-sub("Error: (.*)","Error(\\1)",names(tx)[i])
とすればよいのでしょう。 -- 河童の屁は,河童にあらず,屁である。 2010-07-02 (金) 10:21:28tasosi (2010-07-01 (木) 18:43:27)
Shaffer法の多重比較に関するパッケージを探しています.
色々調べてみたのですが,ANOVA君を活用するという手段はあったのですが,パッケージでは見つかりませんでした.
多重比較で,paired-t-testのP値を補正するのに用いたいと思っていますが,パッケージとしてはないのでしょうか?
R初心者 (2010-07-01 (木) 14:27:54)
はじめまして。
ネットワークの研究をしている大学院生です。データのグラフ化をしたくて一週間ほど前からRの勉強を始めました。
googleとこのwikiでググってみたのですが、分からなかったので質問させていただきました。
環境はMacOSX 10.5.8
Rのバージョンは2.9.0です。
−−−−−−
やろうとしていることの概要
2つのプログラムからの出力をまとめた欠損値を持つCSVを、欠損値を補完したCSVにして、それをlatticeで線グラフ化する。
補完せずとも、線グラフが切れない手法があればそれでもOKです。
−−−−−−
ネットワークのスループットとRTTをTcpdumpを解析して出力するプログラムを書きましたが、都合上RTTとスループットが別に出力されて次のような形式になっています。time, seqnum(シーケンス番号), rtt, throughput 0.0, 3136068389, 0.0469200000006822, 0.0808820000020205, 3136070885, 0.0611399999979767, 0.0999999999985448, , , 24960.0~ 0.142041999999492, 3136074629, 0.0901450000019395, 0.142041999999492, 3136072133, 0.0699139999996987, 0.19999999999709, , , 37440.0 0.21197000000393, 3136077125, 0.0549669999963953, 0.232200999998895, 3136079621, 0.0598790000003646, 0.266950000004726, 3136084613, 0.0701439999975264, .....これをRにCSVとして食わせると、次のようになります。
time seqnum rtt throughput 1 0.000000 3136068389 0.046920 NA 2 0.080882 3136070885 0.061140 NA 3 0.100000 NA NA 24960 4 0.142042 3136074629 0.090145 NA 5 0.142042 3136072133 0.069914 NA 6 0.200000 NA NA 37440 7 0.211970 3136077125 0.054967 NA 8 0.232201 3136079621 0.059879 NAこのNA値を前後の行の値の中間値となるように補完したいです。
なにかそのような関数はありますでしょうか。
補完のやりかたは
「1,2,NA,4,5」-> [1,2,(2+4)/2=3, 4, 5]
のようなもので、連続した値が抜けた際は
「1,2 NA, NA, NA, 6」-> [1,2,3,4,5,6]
となってもらえるのが理想です。
最初は作ろうかと思ったのですが、既にありそうな気がしたので質問させていただきました。
よろしくお願いします。
−−−−−−
自己解決しました。 Vectorを補完する関数を書いたので、行列を「列で分解、適用、合体」することで補完できると思います。complementNaVector <- function(vector) { posVector <- 1:length(vector) naPosVector <- posVector[is.na(vector)] notNaPosVector <- posVector[!is.na(vector)] for (index in naPosVector) { lowerVector <- notNaPosVector[notNaPosVector < index] greaterVector <- notNaPosVector[notNaPosVector > index] if(length(lowerVector) != 0 && length(greaterVector) != 0) { low <- lowerVector[length(lowerVector)] high <- greaterVector[1] distance <- vector[high] - vector[low] stepl2h <- high - low stepl2i <- index - low vector[index] <- vector[low] + (distance * stepl2i / stepl2h) } else { vector[index] <- NA } } return(vector) }行列の補完
complementNaMat <- function(matrix) { iterator <- 2:ncol(matrix) mat <- complementNaVector(matrix[, 1]) for(index in iterator){ vector <- complementNaVector(matrix[, index]) mat <- cbind(mat, vector) } return(mat) } ## 以下でよさそう complementNaMat <- function(matrix) { for (index in 1:ncol(matrix)) { matrix[, index] <- complementNaVector(matrix[, index]) } return(matrix) }
d$time[5] <- d$time[5]+0.000001 plot(d$time, d$rtt) lines(d$time, d$rtt, type="l") points(d$time, complementNA(d$rtt), col=2) # あなたの関数(赤) lines(d$time, complementNA(d$rtt), col=2, lty=3) points(d$time, approx(d$time, d$rtt, xout=d$time)$y, col=4) # approx 関数(青) lines(d$time, approx(d$time, d$rtt, xout=d$time)$y, col=4, lty=4)
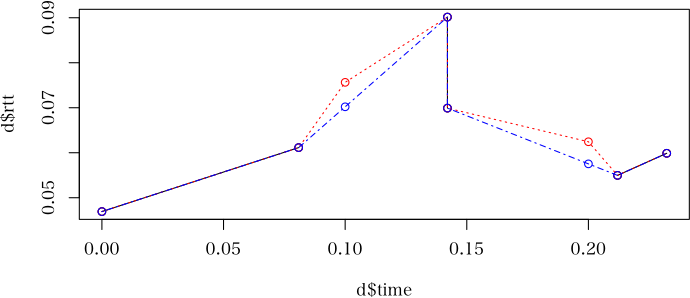
森の熊五郎 (2010-06-27 (日) 01:02:10)
コールセンタに日々大勢の方から電話がかかってきます。1日に何回も、また続けて何日も。そのようなコールセンタにおいて、日々かかってくる電話が何件あって、その電話が何人によってかかっているのか(重複をなくして)。またその結果を、1日単位、1週間単位、1ヶ月単位、一年単位で調べようとしております。本来は電話番号を使って、調べるのですが、個人情報ということもあり、ここでは、仮に下記のような人名が書かれたデータベースがあって、それをもとに調べることとします。
C201004,C201005,C201006
Sato,Sato,Nishihara
Tanaka,Yamada,Sato
Yamada,Kojima,Nishijima
Inoue,Kitano,Inoue
Sato,Shinagawa,Yokosuka
Kitamura,Handa,
Yamamoto,Kitamura,
Ohta,Handa,
Inoue,Nishi,
Kitamura,Yokota,
Kobayashi,Sato,
csvのファイルに上記のようにデータベースがあります。
行方向は日にち、列方向はその日に利用されたお客様のお名前が保存されています。call=read.csv("call2010.csv",header=TRUE) call201004=call$C201004 call201004=call201004[!is.na(call201004)] call201005=call$C201005 call201005=call201005[!is.na(call201005)] call201006=call$C201006 call201006=call201006[!is.na(call201006)] length(call201004) length(unique(call201004)) length(call201005) length(unique(call201005)) length(call201006) length(unique(call201006)) Tcall=c(call201004,call201005) length(Tcall) length(unique(Tcall)) Tcall=c(Tcall,call201006) length(Tcall) length(unique(Tcall))~というプログラムを作りました。
�今回は3日分しかないので、簡単なのですが365日分だとさすがにベタ書きですと大変なので、BasicでいうところのFor next分みたいな文を作り簡単に処理したいのですが可能なのでしょうか?
�文字列のベクトルの結合はc(X,Y)ではうまくいきません。どうすればよいのでしょうか?
�欲を言えばピボットテーブルのように、お客様毎にいつ何件利用されたのかをしりたい。
コールセンタの利用件数は月に何万件もあって膨大なので、excelでは処理できず、Rに挑戦しているのですが、まだまだ素人なので。お手数をおかけしますがご教示下さい。
call <- read.csv("call2010.csv",header=TRUE,as.is=TRUE)
x <- colnames(call)
n <- nrow(call)
N <- 3
call.1 <- call.2 <- rep(NA,N*n)
for (i in seq(1,N)) {
SEQ <- seq(1+(i-1)*n, i*n)
call.1[SEQ] <- call[[i]]
call.2[SEQ] <- rep(x[i],n) }
call.3 <- data.frame(call.2, call.1)
unstack(call.3)
[[1]]
[1] "C201006" "C201006" "C201006" "C201006" "C201006" "C201006" # 欠損値に対応
$Handa
[1] "C201005" "C201005"
$Inoue
[1] "C201004" "C201004" "C201006"
$Kitamura
[1] "C201004" "C201004" "C201005"
$Kitano
[1] "C201005"
$Kobayashi
[1] "C201004"
$Kojima
[1] "C201005"
$Nishi
[1] "C201005"
$Nishihara
[1] "C201006"
$Nishijima
[1] "C201006"
$Ohta
[1] "C201004"
$Sato
[1] "C201004" "C201004" "C201005" "C201005" "C201006"
$Shinagawa
[1] "C201005"
$Tanaka
[1] "C201004"
$Yamada
[1] "C201004" "C201005"
$Yamamoto
[1] "C201004"
$Yokosuka
[1] "C201006"
$Yokota
[1] "C201005"
call.3 # このような一時的データフレームを作っています
call.2 call.1
1 C201004 Sato
2 C201004 Tanaka
3 C201004 Yamada
4 C201004 Inoue
5 C201004 Sato
6 C201004 Kitamura
7 C201004 Yamamoto
8 C201004 Ohta
9 C201004 Inoue
10 C201004 Kitamura
11 C201004 Kobayashi
12 C201005 Sato
13 C201005 Yamada
14 C201005 Kojima
15 C201005 Kitano
16 C201005 Shinagawa
17 C201005 Handa
18 C201005 Kitamura
19 C201005 Handa
20 C201005 Nishi
21 C201005 Yokota
22 C201005 Sato
23 C201006 Nishihara
24 C201006 Sato
25 C201006 Nishijima
26 C201006 Inoue
27 C201006 Yokosuka
28 C201006
29 C201006
30 C201006
31 C201006
32 C201006
33 C201006
call <- read.csv("call2010.csv",header=TRUE,as.is=TRUE)
x <- colnames(call)
n <- nrow(call)
N <- 3
call.1 <- call.2 <- NULL
for (i in seq(1,N)) {
SEQ <- (call[[i]] != "") # 空文字列でないかどうかを判定する論理ベクトル
call.1 <- c(call.1, call[[i]][SEQ])
call.2 <- c(call.2, rep(x[i],sum(SEQ)) }
call.3 <- data.frame(call.2, call.1)
unstack(call.3)
$Handa
[1] "C201005" "C201005"
$Inoue
[1] "C201004" "C201004" "C201006"
(以下略)> # テストデータ作成
> # 作成されるデータファイル Jun0601 〜 Jun0610 の実際の中身をご覧じろ
> set.seed(88541)
> for (i in 1:10) {
+ fn <- sprintf("Jun06%02d", i)
+ n <- sample(100:500, 1)
+ x <- sprintf("%03d-%04d", sample(999, n, replace=TRUE), sample(9999, n, replace=TRUE))
+ write(x, fn)
+ }
> # 以上のデータを使って,色々な集計をしてみる(最終的には,集計範囲を指定する引数を持つ関数にすればよい)
> # 入力制限せず(全部),全部の集計
> x <- NULL
> for (i in 1:10) {
+ fn <- sprintf("Jun06%02d", i)
+ x <- c(x, scan(fn, what=""))
+ }
Read 145 items
Read 109 items
Read 169 items
Read 446 items
Read 412 items
Read 283 items
Read 140 items
Read 140 items
Read 199 items
Read 174 items
> length(x) # 読み込んだ全部のクレーム電話番号の個数
[1] 2217
> table(x) # 集計結果 電話番号のように見えるのは架空のものですからね
x
001-0617 001-5112 002-3684 003-1068 003-3291 004-9049 005-4181 005-4319
1 1 1 1 1 1 1 1
中略
996-7889 996-9818 997-0618 997-8295 998-9462 999-0492 999-2608 999-3352
1 1 1 1 1 1 1 1
999-3655
1
> sum(x=="999-3352") # 999-3352 は 1 件
[1] 1
> sum(x=="123-4567") # 123-4567 は 0 件
[1] 0
> sum(table(x) >= 2) # このデータにおいて,2 件以上のクレーマーはいない
[1] 0
> # などなどなどなどなどなどなどなどなどなどなどなどなどなど
> ######################################
> # 任意の入力 Jun0601, Jun0602 のみ
> x <- NULL
> for (i in 1:2) {
+ fn <- sprintf("Jun06%02d", i)
+ x <- c(x, scan(fn, what=""))
+ }
Read 145 items
Read 109 items
> length(x)
[1] 254
> table(x) # 集計結果 電話番号のように見えるのは架空のものですからね
x
003-1068 006-7349 014-9687 021-9091 023-4157 024-4196 029-7869 035-8444
1 1 1 1 1 1 1 1
中略
979-1463 979-8195 984-7011 990-2678 992-4048 997-0618
1 1 1 1 1 1
意外と,簡単ではないですか?そのうえなにか必要ですか? -- 河童の屁は,河童にあらず,屁である。 2010-06-29 (火) 21:47:47hiro (2010-06-24 (木) 02:54:33)
はじめまして、Ubuntu10.04(64bit)にて、R:2.10.1を使っている者です。
現在、豊田秀樹氏の「データマイニング入門」という書籍を読みながら、Rを学んでおります。
この書籍の第2章の「鉛筆の数え方」のところで、『neuralライブラリ』を用いたサンプルコードが載っているのですが、『neuralライブラリ』をインストールしようにも、install.packages("neural")としても、パッケージを見つけることが出来ないため、インストール出来ずに困っております。
この書籍はそもそもOS:windowsXP、R:2.7.0を前提にして書かれたものなので、neuralライブラリはLinuxには提供されていないライブラリなのでしょうか?
あるいは、neuralライブラリはLinuxにも提供されていたが、古くなったため、今では使われなくなってしまったのでしょうか?
この書籍でRを学びたいので、出来ればneuralライブラリを使いたいのですが、良い解決策をご存知の方がいらっしゃったら、対処方法をご教授下さい。
sudo R CMD INSTALL neural_1.4.tar.gzでOK -- 2010-06-24 (木) 11:51:00
尼河童 (2010-06-23 (水) 00:12:42)
みなさまこんにちは。
ウィンドウズマシーンで R 2.10.1 を使っています。はい、バージョンアップします。
エディタは昔から vim を使っているのですが、簡単に R コードを vim から R に飛ばす方法を模索しています。ウィンドウズというのがネックになりそうです。
素敵な方法をご存知の方はご教授ください。
としろう (2010-06-20 (日) 07:21:25)
plot関数などで作成した図を、png形式で出力する際に、枠やプロットした点以外の背景が透かしのままなのですが、背景を透かしの代わりに白で指定することはできるのでしょうか?
例えば下のプログラムでは、背景が透かしたままで出力されてしまいます。
使用環境はwindows,R-2.11.1です。plot(1:20) pp<-recordPlot() png("test.png",bg="white") replayPlot(pp) dev.off()
png("test.png",bg="white")
plot(1:20)
dev.off()
でできるのは,ちゃんとバックグラウンドが white です。 -- 河童の屁は,河童にあらず,屁である。 2010-06-20 (日) 09:22:19R初心者 (2010-06-19 (土) 21:29:59)
各アルファベットが0/1のコードに(ハフマン)符号化されているデータ「0,1,1,0,0,0,1,1,1,0,0,1,1,0,0,1,1,0,0,0,0,1,1,0,1,1,1,1,0,0,0,1」があるとします.例としまして,A〜Iのデータが以下の0/1のコードに符号化されている状況を考えます.
文字 符号 A 01 B 111 C 110 D 101 E 001 F 000 G 1001 H 10001 I 10000
上記の表でデータを複合化すると「0,1,1,0,0,0,1,1,1,0,0,1,1,0,0,1,1,0,0,0,0,1,1,0,1,1,1,1,0,0,0,1」は「AHCAGICBH」に複合化されます.
さて,データを複合化するための方法の1つが,符号を2分木化し,C言語等のポインタで2分探索法で複合化する方法です.9個の葉を持つ2分木は,9 個(種類)の異なる文字を含む文字列を符号化するために用いることができます.このとき,2分木の各葉を各文字に対応させます.各文字の符号は2分木の根から対応する葉へのパスによって決定します.このとき、左へ向かった場合 0,右へ向かった場合 1 とします.しかし,Rではポインタがなさそうなので,仕方なく以下のような方法で複合化してみました. データをx,上記のリスト(符号,文字)をy,zに格納するプログラムを以下で生成します.
> x <- c(0,1,1,0,0,0,1,1,1,0,0,1,1,0,0,1, # データ + 1,0,0,0,0,1,1,0,1,1,1,1,0,0,0,1) # 結果:AHCAGICBH > y <- c("01","111","110","101","001", + "000","1001","10001","10000") # ハフマン符号 > z <- c("A","B","C","D","E","F","G","H","I") # 対応する文字次に,データを1文字ずつ読み込み,リストyの要素のいずれに合致しているかをチェックし,合致していたらその符号に対応する文字をresultに格納する,というものです.
> result <- c() # 複合結果 > tmp <- "" # バッファ > for (i in 1:length(x)) { + tmp <- paste(tmp, x[i], sep="") # データから1文字読み込み + j <- 1 # 複合化:yのどの値に該当するかチェック + for (j in 1:length(y)) { # + if (y[j] == tmp) { # yのj番目の値に該当した場合 + result <- c(result, z[j]) # zに複合化した結果(j)を格納 + tmp <- "" # バッファを初期化 + break # 繰り返し文から抜ける + } + } + } > result [1] "A" "H" "C" "A" "G" "I" "C" "B" "H"(1) 上記のプログラムでは,文字の種類が増えてくると計算速度が遅くなってしまいます.Rで,木構造を扱うようなことは出来るのでしょうか.
(2) Rで,C言語のポインタのようなことは出来るのでしょうか.
ご教示戴けますと幸いです.
松田紀之 (2010-06-18 (金) 10:04:51)
Mac OS-X (10.5.8)でR2.11.1で文字列処理を試みているうちに,題名にあげた問題が見つかりました.以下,幾つか確認できたことです:
(1) patternの文字が"c"以外のアルファベットなら正常に動く.
(2) 参照されるlistの内容が c("") と単一の場合も正常に動く.
(3) listではなく,grep("c",c("","")) なら正常に動く.
プログラム全体の都合上,list() を対象にしています.何故この問題が起こるのか,またその対処法を教えてください.
nakamura (2010-06-17 (木) 19:18:15)
単純な例として装置からの多数のdataをreadで取り込んで、XDR形式などで保存したいのですが、write.tableとsaveのfile名の指定のところで困ってます。" "で変数( fnとかfile.name[i] )を囲むと、文字列として変数が認識されているようで上手く行きません。ご教授お願いします。単純な例は以下です。”plotでのmain titleの指定”を参考にしました。file.name <- c("map_1.csv", "map_2.csv") # ファイル名のベクトル par(ask=T) for (i in 1:2) { fn <- file.name[i] # ファイル名 dt <- read.csv(fn , header=T, na.strings="NA") # plot(dt, main=fn) # main タイトル付きの描画 write.table(summary(dt), "fn_sum.txt", quote=F) save(dt, file="file.name[i].dat") }
R初心者 (2010-06-17 (木) 12:01:56)
「0100101000010101・・・」のように延々と続くファイルがあり,それを1文字(1ビット)ずつ読み込むことを考えております.> ff <- tempfile() > cat(file=ff,"01000101000100100101\n") > read.fwf(ff, widths=rep(1,20)) V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 V18 V19 V20 0 1 0 0 0 1 0 1 0 0 0 1 0 0 1 0 0 1 0 1(1) ファイルを読み込む際文字数(ビット数)が分からなければ引数widthsを指定するときに困ります….読み込むファイルのサイズや文字数を計算する関数はありませんでしょうか.
(2) 上記では関数read.fwf()で無理やり読み込んでいますが,もっと良い関数があれば教えてもらえませんでしょうか.
どうかよろしくお願いいたします.
> ff <- tempfile()
> cat(file=ff, "01000101000100100101\n")
> read.fwf(ff, widths=rep(1,20))
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 V18 V19 V20
1 0 1 0 0 0 1 0 1 0 0 0 1 0 0 1 0 0 1 0 1
> file.info(ff) # ファイル情報
size isdir mode
C:\\DOCUME~1\\x\\LOCALS~1\\Temp\\RtmpcQhzyb\\file767d7a5a 22 FALSE 666
mtime
C:\\DOCUME~1\\x\\LOCALS~1\\Temp\\RtmpcQhzyb\\file767d7a5a 2010-06-17 15:53:37
.............................................................................
> file.info(ff)[1] # ファイル情報からサイズのみ抽出
size
C:\\DOCUME~1\\x\\LOCALS~1\\Temp\\RtmpcQhzyb\\file767d7a5a 22
>
> size <- as.integer(file.info(ff)[1]-2) # 改行文字の分だけ引き算
> read.fwf(ff, widths=rep(1,size)) # 読み込み
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 V18 V19 V20
1 0 1 0 0 0 1 0 1 0 0 0 1 0 0 1 0 0 1 0 1
> readBin(ff, size=1, what="int", n=size) # 整数値 48/49 のベクトルとして読み込み
[1] 48 49 48 48 48 49 48 49 48 48 48 49 48 48 49 48 48 49 48 49
> readBin(ff, size=1, what="raw", n=size) # raw(16進) 30/31 のベクトルとして読み込み
[1] 30 31 30 30 30 31 30 31 30 30 30 31 30 30 31 30 30 31 30 31
> readBin(ff, size=1, what="int", n=size)-48 # 整数値 として読み込み -> 0,1に変換
[1] 0 1 0 0 0 1 0 1 0 0 0 1 0 0 1 0 0 1 0 1
お忙しいところ本当にありがとうございました.-- R初心者 2010-06-17 (木) 15:54:30AKB (2010-06-15 (火) 23:49:30)
基データ(csv)としてテキスト(301)×単語(1134)のマトリクスがあり,単語間の距離を算出し,デンドログラムの構築までしたいのですが”non-square matrix”と当然返されてしまいます.手順としてはdata <-read.csv("C:/RTest/Real Game.csv", header=T, row.names=1) data # 表示 old.op <- options(max.print=999999) myCosine <- function(x) { ret <- matrix(0, ncol=ncol(x), nrow=ncol(x)) for(i in 1:ncol(x)) { for(j in 1:ncol(x)) { ret[i, j] <- (x[,i] %*% x[,j]) / (sqrt(sum(x[,i]^2)) * sqrt(sum(x[,j]^2))) } } ret } myCosine(data) plot(hclust(as.dist(data)))です.基のデータから単語間の距離に基づいたデンドログラムを構築する方法で何かうまい方法は無いでしょうか?
akira (2010-06-12 (土) 22:53:35)
以下のデータフレームがあったとしてa b c d e f g 1 q p q 4 6 7 6 2 q p p 5 4 5 4 3 p p p 5 4 5 7 4 q q q 3 5 4 6a~cそれぞれの値がpに該当する行を取り出す方法を考えていたのですが、どうしても思いつきませんでした。
[a] d e f g 3 5 4 5 7 [b] d e f g 1 4 6 7 6 2 5 4 5 4 3 5 4 5 7 [c] d e f g 2 5 4 5 4 3 5 4 5 7どなたかお分かりの方、丸投げで申し訳ありませんがご教示頂けないでしょうか?
> subset(x, x[1]=="p")[4:7] d e f g 3 5 4 5 7 > subset(x, x[2]=="p")[4:7] d e f g 1 4 6 7 6 2 5 4 5 4 3 5 4 5 7 > subset(x, x[3]=="p")[4:7] d e f g 2 5 4 5 4 3 5 4 5 7 > x[x[1]=="p",][4:7] # これは余りお勧めしない d e f g 3 5 4 5 7
宗二 (2010-06-11 (金) 02:01:30)
シェープファイルと2次元配列から作った、図の重ね合わせ方で悩んでます。
具体的には、日本の海岸線のシェープファイル(ライン)をplot(シェープファイル, xlim=c(西経度, 東経度), ylim=c(南緯度, 北緯度))というようにプロットした後、2 次元配列として用意した日本周辺の気温データを、同じ緯度経度の範囲で重ねて作図したいのですが、2次元配列をimage、あるいはcontour関数で作図するときに、x軸とy軸の範囲を緯度経度で指定できないものでしょうか?
配列をプロットするときの軸の範囲が、配列の要素番号と対応するのでimage(配列, xlim=c(西経度, 東経度), ylim=c(南緯度, 北緯度))のようにすると、範囲が大きくずれて何も表示されなくなってしまいます。
なにか良い解決方法ありましたらお願いします。
temp0 <- file("...気温のバイナリデータ...", "rb")
width <- 501 ### 気温データの水平格子数
height <- 501 ### 気温データの鉛直格子数
temp <- readBin(temp0,integer(), n=width*height, size=2)
close(temp0)
temp = temp/10.0 ### データ-->気温への変換式です。
temp_arr <- matrix(temp,width,height)
temp_arr = t(apply(temp_arr,1,rev)) ### 地図投影のために配列を回転
image(temp_arr,col=rainbow(300),xlim=c(137,142),ylim=c(36,41),zlim=c(5,30))
### 経度137E-142E, 緯度36N-41N, 温度の範囲5-30℃ で指定したいのですが、
### このままだと図の枠中が白紙のままです。
### 501*501の配列を、0.01度格子で上記の緯度経度に対応させたいのです。
map <- readShapeLines("coastline.shp")
plot(map,xlim=c(137,142),ylim=c(36,41))
### 海岸線のシェープファイルの読み込みとプロット
### こちらは図の枠内にちゃんと海岸線がプロットされます。a <- list(x=seq(137,142,1),y=seq(36,41,1),z=1:6%o%1:6) image(a)
map <- readShapeLines("coastline.shp")
plot(map,xlim=c(137,142),ylim=c(36,41),add=TRUE)
とすれば、重なっていることがわかるはず。coastline.shpがないので検証できませんが。shannon (2010-06-11 (金) 00:54:55)
プログラム実行を途中で中断・再開させる関数、あるいはコマンドはあるのでしょうか?
WindowsのTinn-R(ver:1.19.4.7)から、ショートカットキーでRコンソール(ver:2.11.0)にプログラム全体を送り実行しているのですが、プログラムの途中で、変数の値を確認するために実行を中断し、確認後に実行を再開させる、ということをやりたいのです。
手動で範囲指定してコンソールに送る、以外でいい方法はないでしょうか?
> for (i in 1:10) {
+ print(paste("i =", i))
+ j <- readLines(con=stdin(), n=1)
+ if (j == "stop") break # 早めに実行中断したいならこのようなものを入れておく
+ }
[1] "i = 1"
# リターンキーだけ押した
[1] "i = 2"
ok
[1] "i = 3"
0
[1] "i = 4"
stop # "stop" を入力すると終了
それとも場合に応じていろいろな変数をチェックすることもあるということ?ならば debug 関数を調べてみたら? -- 河童の屁は,河童にあらず,屁である。 2010-06-11 (金) 11:26:181: a <- 1 2: b <- 2 3: #ここで実行を中止したい 4: a <- 10 5: b <- 20示していただいた例文を使って、
1: a <- 1
2: b <- 2
3:
4: for (i in 1:2) {
5: print(paste("i=", i))
6: j <- readLines(con=stdin(), n=1)
7: if (j =="stop") break
8: }
9:
10: a <- 10
11: b <- 20
のようにして実行すると、10行目のa<-10という命令がreadLines関数に渡されて、プログラム実行後のaとbの値は、a=1,b=20になってしまいます。
10行目以降の命令を渡す前に、9行目までで実行を中断したいのですが方法ありますでしょうか。初歩的な勘違いしてたらすみません。 -- shannon 2010-06-16 (水) 00:50:29{ # これと
a <- 1
b <- 2
cat("Hit any key!\n")
if (readLines(con=stdin(), n=1) == "stop") stop("中止しました")
a <- 10
b <- 20
} # これ
こんなところでいかが? -- 河童の屁は,河童にあらず,屁である。 2010-06-17 (木) 10:37:56T (2010-06-10 (木) 16:22:17)
只今卒論のテーマとしてある飲食店の一店舗の様々な日別データをRによって時系列分析しようと考えていまして、日別データを扱った時系列分析に関する本や文献を探しています。
ご存じの方がいらっしゃれば教えていただけると幸いです。
do-san (2010-06-10 (木) 01:08:00)
2次元のバイナリ(もしくは文字列)配列を、90度、もしくは180度回転させたいのですが、そのような関数はあるのでしょうか?
vp (2010-06-06 (日) 21:35:28)
宜しくお願いします。
以下のように filled.contour で描いた図に abline で x=0.5, y=0.5 の線を重ね描きしようと思いましたが、座標が対応しませんでした。data(volcano) filled.contour(volcano, color = terrain.colors, asp = 1) # simple par(new = T) abline(h = 0.5, xlim = c(0, 1), ylim = c(0, 1)) par(new = T) abline(v = 0.5, xlim = c(0, 1), ylim = c(0, 1))何かうまい方法は無いでしょうか?
filled.contour(volcano, color = terrain.colors, asp = 1,
plot.axes={axis(1); axis(2); abline(v=0.5, h=0.5)})data(volcano)
filled.contour(volcano, color = terrain.colors, asp = 1
,plot.axes={axis(1); axis(2);plot(seq(0,1,by=0.1))})
下の図のように、左側にあった作図領域が消去されて、data(volcano)
filled.contour(volcano, color = terrain.colors,xlim=c(0,1),ylim=c(0,1),
plot.axes={axis(1); axis(2);par(new=T);
plot(seq(0,1,by=0.1,xlim=c(0,1),ylim=c(0,1))
})
xlim,ylimで軸の範囲を設定してもずれは直りませんでした。これは何が原因なのでしょうか?-- ですら 2010-06-18 (金) 10:40:58data(volcano)
filled.contour(volcano, color = terrain.colors,
plot.axes = {par(new = T, xaxs = "i", yaxs = "i")
plot(seq(0, 1, by = 0.1), seq(0, 1, by = 0.1),
xlab = "", ylab = "")
axis(1); axis(2)})mm (2010-06-06 (日) 01:59:41)
こんにちは。最近RでGLMに取り組み始めました。
まずは,説明変数が応答変数によって正,負のどちらに影響しているかを調べることが目的です。
この応答変数は,連続変数で正の値を取るため,familyをGammaにしました。
するとEstimateの値が,正規分布を仮定した回帰分析と逆の正,負の関係になってしまい,解釈に困っています。
下の例のように,irisのデータで,familyをgaussianとGammaでGLMを行っても同じ結果です。
指定するfamilyによって,正負の方向が逆転するということはあるのでしょうか?test <- glm(Petal.Length ~ Petal.Width, data=iris) summary(test) Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 1.08356 0.07297 14.85 <2e-16 *** Petal.Width 2.22994 0.05140 43.39 <2e-16 *** #################### testG <- glm(Petal.Length ~ Petal.Width, data=iris, family=Gamma) summary(testG) Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 0.532340 0.017386 30.62 <2e-16 *** Petal.Width -0.172682 0.008915 -19.37 <2e-16 ***
sakura (2010-06-05 (土) 13:13:57)
最近、調べ物をしていたら、Bland Altman plot のことが目にとまりました。信頼性評価に使われるそうですが、馴染みがありません。Rのlibraryに搭載されたものがあるのでしょうか?差分の傾向を知るというのが、ポイントのようですが・・・。ご教示いただければ幸いです。
BlandAltman {ResearchMethods} R Documentation
BlandAltman Plot
Description
Using a graphical user interface (GUI) this function performs a Bland Altman
plot, and allows for manipulation of the variables within the plot.
Usage
BlandAltman(x, y, gui=TRUE, bandsOn=FALSE, biasOn=FALSE, regionOn=FALSE,
smooth=FALSE, sig=2)while (2010-06-03 (木) 03:44:10)
while を使用し、行列 kara 内に while 内処理を length(mg) 回実行したいのですがエラーが出ます。
(df kara 内の candy, drop という変数(行名)それぞれに対応する colMeans を求め、kara 内に格納する)
エラーは以下のように出ます。以下にエラー kara[, i] <- g : 置き換えるべき項目数が,置き換える数の倍数ではありませんでした mg <- c("candy","drop") # データフレーム ame の中の colname を 2 つ選び vector に kara <-matrix(, length(mg)) i <- 0 while(i <- i+1 <= length(mg)) { g <- colMeans(subset(ame, ame[i] == "eat", select = (範囲を指定))) kara[,i] <- g }
> set.seed(123)
> ame <- data.frame(name=sample(c("candy", "drop", "caramel"), 15, replace=TRUE),
+ x1=sample(140, 15, replace=TRUE),
+ x2=sample(140, 15, replace=TRUE),
+ x3=sample(140, 15, replace=TRUE))
> ame
name x1 x2 x3
1 candy 126 135 20
2 caramel 35 127 33
3 drop 6 97 66
4 caramel 46 112 38
5 caramel 134 4 121
6 candy 125 67 7
7 drop 97 107 62
8 caramel 90 31 112
9 drop 140 45 18
10 drop 92 33 79
11 caramel 100 20 29
12 drop 77 59 18
13 caramel 84 58 106
14 drop 41 52 126
15 candy 21 22 53
求めるものも,一行でできますよ。
> sapply(split(ame, ame[,1]), function(d) colMeans(d[2:4]))
candy caramel drop
x1 90.66667 81.50000 75.5
x2 74.66667 58.66667 65.5
x3 26.66667 73.16667 61.5
これでよい?必要に応じ,結果を転置。 -- 河童の屁は,河童にあらず,屁である。 2010-06-04 (金) 15:14:21> aggregate(ame[2:4], ame[1], mean)
name x1 x2 x3
1 candy 90.66667 74.66667 26.66667
2 caramel 81.50000 58.66667 73.16667
3 drop 75.50000 65.50000 61.50000> ame2 <- ame[ ame[,1] %in% c("caramel", "drop"),]
> aggregate(ame2[2:4], ame2[1], mean)
name x1 x2 x3
1 caramel 81.5 58.66667 73.16667
2 drop 75.5 65.50000 61.50000?アロバ (2010-06-02 (水) 19:29:27)
たびたび皆様にご指導いただきたく、よろしくお願いします。source("http://aoki2.si.gunma-u.ac.jp/R/src/km_surv.R", encoding="euc-jp") # 1 は A 群,2 は B 群を表す group <- c(1,2,2,2,2,2,2,2,2,2,2,2,2,2,2,1,2,2,1,1,1,1,1, 1,1,2,2,2,2,2,2,2,2,2,2,2,2,2,1,2,1,1,2,2,1,2) # 1 は死亡,2 は 生存(打ち切り)を表す event <- c(1,0,0,0,0,0,0,0,0,0,0,0,0,1,1,0,0,0,0,0,1,0,1, 1,1,0,0,0,0,0,1,1,0,0,0,0,0,0,0,0,0,0,1,0,0,0) # 生存期間 time <- c(2,84,318,198,198,197,192,306,96,90,88,66,48,264, 24,5,243,216,42,5,20,0,12,144,48,192,192,228, 176,180,84,84,123,117,115,267,98,96,86,63,44,41, 33,20,18,252) a.group <- group == 1 km.surv(time[a.group], event[a.group]) library(survival) # survival ライブラリーを使う dat <- Surv(time[a.group], event[a.group]) # survfit で使うオブジェクトを作る res <- survfit(dat)上記にて入力したのですが、Kaplan-Meier 曲線が A 群 1 本だけのグラフになってしまいます。A, B 両群の曲線を一つのグラフに入れたいのですが。。。 よろしくお願いします。
lo (2010-05-31 (月) 00:05:13)
for (i in 1:10){
の中で
計算結果を行列に格納する工程を書いた後
i番目の結果を格納した行列(仮にkekkaという名前とします)に、新たに、
hogeとi(1から10までの値をとる)をくっつけた、hogeiという名前をつけたいです。
この場合、
hogei<-kekka
とやっても、hoge1とならずに、hogeiとなってしまいます。
paste("hoge",i)<-kekkaとやると、エラーになります。
このような、ループの回数に応じた連続した名称をつけるには、どのようにしたらよいのでしょうか?
vi (2010-05-27 (木) 19:22:41)
質問です。Designパッケージの説明ページ
http://bm2.genes.nig.ac.jp/RGM2/R_current/library/Design/man/val.prob.html
において、Examplesとして、実際に使用するときのコードが載っています。
この中の、後半部分$Survival analysis examples 中に# Survival analysis examples # Generate failure times from an exponential distribution set.seed(123) # so can reproduce results n <- 2000 age <- 50 + 12*rnorm(n) sex <- factor(sample(c('Male','Female'), n, rep=TRUE, prob=c(.6, .4))) cens <- 15*runif(n) h <- .02*exp(.04*(age-50)+.8*(sex=='Female')) t <- -log(runif(n))/h label(t) <- 'Time to Event' ev <- ifelse(t <= cens, 1, 0) t <- pmin(t, cens) S <- Surv(t, ev) # First validate true model used to generate data w <- val.surv(est.surv=exp(-h*t), S=S) plot(w) plot(w, group=sex) # stratify by sex上記のような記載があるのですが、私は、est.survというのが、時間tにおける各ケースの計算上の生存率であると理解しています。その場合、
w <- val.surv(est.surv=exp(-h*t), S=S)の部分の、exp(-h*t)は、比例ハザードを用いて、(時間tにおけるbaseline hazard)^exp(h)とするのが正しいのではないかと疑問に思っています。そうはいっても実際に試すと、きれいなキャリブレーションプロットがかけるのですが。私のest.survの解釈自体が間違っているのでしょうか?どなたか、引数est.survとSについての解釈を教えていただけると幸いです。
jun (2010-05-25 (火) 22:57:47)
クラス "dendrogram" に対するメソッドで葉を並べ替える
reorder(x, wts, ...)
というものがありますが、使い方がよくわかりません。
葉をV1、V2、V3、V4・・・・と昇順に並べたいのですが。
昇順に並べても枝は絶対にクロスしないような距離行列とクラスタリング結果になっています。
wtsとは重みと書いてありましたが何の重みですか?
よろしくお願いします。
使用環境はR version 2.10.0 (2009-10-26)でWindowsVistaです。
1からV1への距離は存在しないので過度に大きな数として99.00を使っています。その他の99.00も同じ意味です。
クラスタリングを単連結で行うため結果に影響は及ぼさないと考えてそのようにしました。> r V1 V2 V3 V4 V5 V6 V7 V8 V9 1 99.000 0.045 99.000 99.000 99.000 99.000 99.000 99.000 99.000 2 0.045 99.000 0.071 99.000 99.000 99.000 99.000 99.000 99.000 3 99.000 0.071 99.000 0.042 99.000 99.000 99.000 99.000 99.000 4 99.000 99.000 0.042 99.000 0.037 99.000 99.000 99.000 99.000 5 99.000 99.000 99.000 0.037 99.000 0.059 99.000 99.000 99.000 6 99.000 99.000 99.000 99.000 0.059 99.000 0.111 99.000 99.000 7 99.000 99.000 99.000 99.000 99.000 0.111 99.000 0.071 99.000 8 99.000 99.000 99.000 99.000 99.000 99.000 0.071 99.000 0.091 9 99.000 99.000 99.000 99.000 99.000 99.000 99.000 0.091 99.000 > plot(hclust(as.dist(r),method="single"))
par(mfrow=1:2) hc <- hclust(as.dist(r),method="single") plot(hc, hang=-1, main="", sub="", xlab="") hc2 <- reorder(as.dendrogram(hc), c(1,2,3,4,5,13,7,8,20)) plot(hc2)
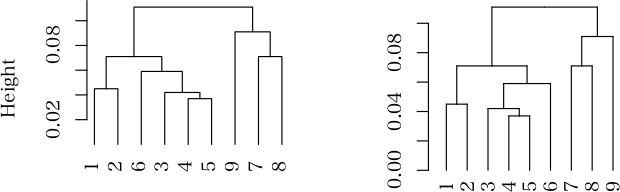
ms (2010-05-25 (火) 20:56:33)
RjpWikiの『Rの関数定義の基本』を参考にさせて頂き、関数定義で変数を既定値にしているのですが、結果を見ると、既定値になっていないようです。
x[2]+x[3]=1 としたいのですが、この書き方ではだめなのでしょうか?
ご意見頂きたくよろしくお願い致します。
使用環境は R2.10.1, XPです。fr <- function(x, y=1) { LL <- 0 pp <- x[4]*(x[2]*(Data[, 19])^x[1] + x[3]*(Data[, 21])^x[1])^(1/x[1]) dp <- x[4]*(x[2]*(Data[, 20])^x[1] + x[3]*(Data[, 22])^x[1])^(1/x[1]) y <- x[2] + x[3] Ppp <- exp(pp) / (exp(pp) + exp(dp)) Pdp <- exp(dp) / (exp(pp) + exp(dp)) Ppp <- (Ppp != 0)*Ppp + (Ppp == 0) Pdp <- (Pdp != 0)*Pdp + (Pdp == 0) Cpp <- Data[, 4] == 1 Cdp <- Data[, 4] == 0 LL <- sum(Cuchi*log(Puchi) + Csoto*log(Psoto)) return(LL) }
- 何を計算しようとしている関数なのか理解できません。何を計算していようがかまわないのだけど,やっていることが何のためにやっているのか分からない。関数が返す LL は return の前にある式で計算され,その計算式で引用される変数はそれ以前のどこにも出てこない(それ以前の計算式は何のために何を計算しているの?)
「x[2]+x[3]=1 としたい」というのも,よくわからない。x の要素は 4 個?で,x[2] と x[3] を足すと 1 になるような関係式がある?そもそも,x はベクトルで引き渡さないといけないようにも見えない。いちいち x[1] みたいに引用しないといけないし。4 つの要素を x1, x2, x3 x4 として引き渡せば,function(x1, x2, x3=1-x2, x4, y) とすれば,少なくとも x3 は 1-x2 という規定値を持つことにはなるでしょう。x[2]+x[3] が 1 になるような組み合わせは無限にあり,規定値になり得ない。(x2+x3=1 になるような解を求めるというような場合もありますが,そのような場合は「規定値」ではなく,「制約条件」という。概念がまるで違う)。
ついでながら,引数で渡される y は規定値 1 を持つが,にもかかわらず他の計算に使われる前に y <- x[2] + x[3] と代入されてしまう。これはあなたが言っている 「x[2]+x[3]=1 としたい」というのとは,まるで違う。引数で渡された x[2], x[3] の値を足して,それを y という変数に代入しているだけ。それによって,x[2] や x[3] の値が別のものになるということではない。-- 河童の屁は,河童にあらず,屁である。 2010-05-25 (火) 21:30:01- 不勉強で申し訳ありません。
LL <- sum(Cpp*log(Ppp)+Cdp*log(Pdp)) の間違いです。計算したかったことは対数尤度を計算したのち、optim(初期値,fn,method,…)で関数の最大化を行いたかったのです。そこで、ヘッセ行列を用いるため、x[1]としています。x[2]+x[3]=1というのは、制約条件として入れたいのですが、このような場合、どのようにすれば、この条件を満たす答えが出るか教えて頂ければ幸いです。お手数掛けます。 -- ms 2010-05-25 (火) 22:15:02- x[2]+x[3]=1 が制約条件なら,x[3] を求めなきゃ良い。計算の途中で x[3] のところを (1-x[2]) とすればよいだけでは?つまり,求めるパラメータは x[1], x[2], x[4] の 3 個だけということ(順序はつめればよいが)。 -- 河童の屁は,河童にあらず,屁である。 2010-05-25 (火) 22:26:14
- もし x[1],x[2] 等にさらに正値等の条件が付くなら、線形不等式制約下での最適化関数 constrOptim がよいかもしれません。RjpWiki 中に解説がありますのでキーワード検索。 -- 2010-05-26 (水) 22:26:14
- ありがとうございます。コメント頂いたとおり、x[2]x[3]は正の条件が付きます。optimで行う方法はありますでしょうか?constrOptimには、hessian はないですよね?質問ばかりですみませんが、よろしくお願いします。 -- ms 2010-05-28 (金) 18:32:13
- ありがとうございます。コメント頂いたとおり、x[2]x[3]は正の条件が付きます。optimで行う方法はありますでしょうか?constrOptimには、hessian はないですよね?質問ばかりですみませんが、よろしくお願いします。 -- ms 2010-05-28 (金) 18:43:07
- constrOptim のソースコードの optim を呼ぶところに hessian=TRUE を加える。 -- 河童の屁は,河童にあらず,屁である。 2010-05-29 (土) 09:24:05
aor (2010-05-21 (金) 11:28:09)
いつも勉強させてもらっています。
nlmeパッケージ、lme関数を使用してマルチレベル分析の勉強をしています。
この出力のうち、 VarCorr関数を使うと分散が出力できますが、その標準誤差を求めたいと思っています。よい方法をご存知の方がいらしたら教えていただけないでしょうか。
小野寺先生らが訳された「基礎から学ぶマルチレベルモデル」 (ナカニシヤ出版) や、石田先生らが訳された「RとS-PLUSによる多変量解析」などで勉強していますが、標準誤差を求める方法はわかりませんでした。
上記の書やインターネットでの検索をすると、「標準誤差はあまり意味がないから信頼区間を使おう」という意見があり、標準誤差の出力方法を記載しているものは見当たりませんでした。
ですが、その意見はその意見として、勉強のため標準誤差の出力方法も知りたいと考えています。
標準誤差を出力するRの方法、あるいは計算式などについて記載されている書籍やサイト (日本語か英語) についてでもお教えいただければ助かります。
どうぞよろしくお願いします。
環境はR2.11.0, Windows Vistaです。# サンプル (lmeのヘルプから) library(nlme) fm2 <- lme(distance ~ age + Sex, data = Orthodont, random = ~ 1) summary(fm2) VarCorr(fm2) intervals(fm2)
yosshiii (2010-05-20 (木) 16:28:42)
連続で質問です。質問ばかりですみません。
行列で01.txt 02.txt 03.txt A 10 3 7 B 1 0 0 C 10 3 7の行列があり、”A”という単語がテキスト01に10回, テキスト02に3回, テキスト03に7回出ているという意味の行列です。
そこで「A」と「C」の各テキスト毎の出現回数が等しいとき, 「A, C」をペアとして抽出するプログラムを作りたいんですけど、効率的なやり方が思い浮かびません。意見お願いします。
#-----------テストデータ作製開始
set.seed(12347)
a <- data.frame(matrix(sample(3, 30, replace=TRUE), 10))
colnames(a) <- sprintf("%02d.txt", 1:3)
rownames(a) <- LETTERS[1:10]
a
#-----------テストデータ作製完了
# 同一行の抽出
dup <- rownames(a[duplicated(a),])
for (i in dup) {
rows <- logical(nrow(a))
for (j in 1:nrow(a)) {
rows[j] = all(a[j,] == a[i,])
}
if (sum(!is.na(rows))) {
cat("-------------\n")
print(a[rows,])
a <- a[!rows,,drop=FALSE]
if (nrow(a) < 2) break
}
}
作られるデータフレーム a は以下の通り
01.txt 02.txt 03.txt A 3 2 3 B 2 1 1 C 3 2 1 D 3 1 1 E 1 1 1 F 3 1 1 G 1 1 1 H 3 2 1 I 2 1 1 J 1 1 1実行結果は以下の通り
------------- 01.txt 02.txt 03.txt D 3 1 1 F 3 1 1 ------------- 01.txt 02.txt 03.txt E 1 1 1 G 1 1 1 J 1 1 1 ------------- 01.txt 02.txt 03.txt C 3 2 1 H 3 2 1 ------------- 01.txt 02.txt 03.txt B 2 1 1 I 2 1 1同一行の抽出部分を以下のようにすれば,
# 同一行の抽出
a <- a[order(a[,1], a[,2], a[,3]),] # ここは,列数分書いてね
n <- nrow(a)
dup <- logical(n)
dup[1] = all(a[1,] == a[2,])
dup[n] = all(a[n,] == a[n-1,])
for (i in 2:(n-1)) {
dup[i] = all(a[i,] == a[i-1,]) || all(a[i,] == a[i+1,])
}
a[dup,]
以下のような結果になります。同じ内容を持つ行がまとめられて配置し直されます。
01.txt 02.txt 03.txt E 1 1 1 G 1 1 1 J 1 1 1 B 2 1 1 I 2 1 1 D 3 1 1 F 3 1 1 C 3 2 1 H 3 2 1
yosshiii (2010-05-19 (水) 12:44:00)
フォルダ内全てのテキストファイルからテキストファイル毎に固有表現の出現頻度をカウントしデータフレーム化を実装したいと思っています。以下にイメージしている実行例を載せます。
フォルダ「test」に01.txt, 02.txt, 03.txtのテキストファイルがありそれぞれ「”アリ” “スズメ”」「”アリ” “リス”」「”アリ” “ハチ”」という固有表現が書かれていたとします。これをデータフレーム化すると01.txt 02.txt 03.txt アリ 1 1 1 スズメ 1 0 0 リス 0 1 0 ハチ 0 0 1のようにしたいと思っています。
そして今現在「count」というリストにcount1=(1 1 0 0), count2=(1 0 1 0), count3=(1 0 0 1) の数値をいれる所まで出来ているのですが、このリストの「count」を上記のようにデータフレーム化したいです。よって、フォルダ内のテキストファイル数が幾つであっても良いように実装したいので、意見お願いします。
l <- list.files(pattern="*.R") # R プログラムを対象に
n <- length(l) # 対象ファイルの個数
res <- matrix(0, 4, n) # 行列にしておく。行数(例では4)はキーワードの種類の数
colnames(res) <- l # 列名をファイル名に
for (i in 1:n) { # 各対象ファイルを処理
x <- readLines(l[i]) # 読み取って
a <- c(any(grepl("function", x)), # キーワードがあれば TRUE,なければ FALSE
any(grepl("plot", x)), # 最終的には 1/0 になる
any(grepl("sqrt", x)),
any(grepl("print", x)))
res[, i] <- a # i 番目のファイルの結果は i 列へ
}
(res <- data.frame(res)) # データフレームに変換
こんな風にもできるということで。 -- 河童の屁は,河童にあらず,屁である。 2010-05-19 (水) 13:30:24だいもん (2010-05-19 (水) 12:05:32)
以下のコマンドをループでまわすことで、ポストスクリプトファイルを自動的に生成しようと試みています。(sheet, ddがループの変数に依存して替わります。)postscript(sheet, horizontal = FALSE, paper = "special", height = 6, width = 6,colormodel="rgb") levelplot(dd[, 4] ~ dd[,1]*dd[,2], , color=TRUE) dev.off()まずこのコマンドをループを遣わずに実行すると、X11()デバイスにはカラーで表示されるものが白黒のポストスクリプトファイルしか作成されません。
さらにループで複数回まわすと、全てのPSファイルが白紙(ファイルサイズ=5kb)になってしまいます。この問題については、recordPlot(), replayPlot()を用いたコードを試してみても同じ結果でした。(ただしこちらではカラーで出る。)
当方の環境はWindows XPで、R2.11.0を利用しています。お知恵を拝借できれば幸いです。
library(lattice) ps.options(onefile=F, paper = "special", height = 4, width = 4) trellis.device(postscript, color=T, file="hoge.eps") mm <- matrix(rnorm(8^2), 8, 8) levelplot(mm) dev.off()なんかかなぁ.
N/A (2010-05-19 (水) 12:02:00)
ラベルで
coefficient β[i]
としたい場合どのようにすればよいでしょうか?ylab="Regression function expression(beta)[i]" ylab=parse( "Regression function",expression(beta),"[i]" )でもできなくて案がつきました・・・
お手数をおかけしますがよろしくお願いします。
nene (2010-05-17 (月) 16:54:23)
Design, Hmisc, survival,cmprskの各パッケージを読み込んだ、R 2.11.0で、とあるコード(R2.3.1で動作確認されているようです)を読み込んで実行すると、関数 "summary.survfit" を見つけることができませんでした、と表示されてしまい、うまく動きません。RSiteSearch("summary.survfit")を行ってみると、summary.survfit関数はきちんと存在するようなのですが、現在は仕様が変わって別の関数になっているのでしょうか?R2.3.1のころの仕様変更歴なども調べてみたのですが、特に記載がありません。お手数をおかけしますがよろしくお願いします。
BURBUR (2010-05-15 (土) 11:55:27)
フォルダ内のファイルを検索したい関数を実装したいのですが> test3 <- function(x) { file<-list.files(path="./x") return(file) } > test3(フォルダ名)と実行するとpath名をクオートのでくくっているため
list.files: './x' は読めないディレクトリです というエラーメッセージが出ます。~ これを防ぐ方法を教えて下さい。~
> test3 <- function(x) {
+ file <- list.files(x)
+ return(file)
+ }
> test3("foo")
[1] "bar" "bar2" "bar3" ...
とすればよいでしょう。test3 に与えるパスの指定方法はご存じのことと思いますので省略。 -- 2010-05-15 (土) 12:54:11ヨッシ (2010-05-14 (金) 16:17:19)
perlではディレクトリ内のファイルを取得する場合
opendir my $dir, "ディレクトリのパス" or die "$!";while(my $name = readdir $dir){ if($name =~ /.*\.sgml/){ #ディレクトリ内のファイルの名前 open F1, "ディレクトリのパス/$name" or die "$!"; my @lines = <F1>; } } } } closedir $dir;
アロバ (2010-05-12 (水) 09:45:56)
par(mfrow=c(1,2)) x<-c(1.12,0,6.82,23.9,17.66,2.67,6.09,0.13,5,0.36,2.07,3.1,10.79,57.29,5.14, 107.91,80.05,0,21.66,45.33,0,57.51,103.7,40.62,110.27,77.03,203.22,85.15) boxplot(x, names=c("x")) #箱ひげ図 y<-c(0,1.74,5.36,0,1.12,7.7,0.85,26.52,4.52,0.33,42.44,37.23,2.55,2.65,61.59) boxplot(y, names=c("y")) #箱ひげ図 par(mfrow=c(1,1))2種類のデータxとyを上記で入力したところ、1つのファイルの中に別々に箱ひげのグラフが出来てしまいます。
質問1;この2つのグラフを共通の縦軸のグラフにするにはどのようにすれば良いか?
質問2;それぞれのグラフの下に"x"、"y"と名前を入れる事は可能でしょうか?
ご多忙のところ大変恐縮です、よろしくお願いします。
使用環境は下記です。R version 2.11.0 (2010-04-22) i386-apple-darwin9.8.0 locale: [1] ja_JP.UTF-8/ja_JP.UTF-8/C/C/ja_JP.UTF-8/ja_JP.UTF-8 attached base packages: [1] stats graphics grDevices utils datasets methods base
boxplot(list(x=x, y=y))ということで良いのでしょうか? -- 2010-05-12 (水) 10:47:56
jb3 (2010-05-11 (火) 21:58:13)
2つ質問があります。
datatableにtime event group 55 0 1 23 1 1 61 1 1 12 1 1 90 0 1以下同様に200行の生存時間timeと、イベントeventに関するデータがあるとします。
これを、読み込んで、<- datasetとして、Rの解説の本に書かれていたのを真似て、> fitteddata <- Surv(dataset$time, dataset$event) > sf <- survfit(fitteddata~group, data = dataset) > plot(sf)と実行すると、kaplan meier 生存曲線を描くことはできたのですが
質問1
もともと1グループしかないので、群分けはいらないと思い、上式の~groupを消すとSurvfit requires a formula or a coxph fit as the first argumentというエラーが出ます。単一群の生存曲線を描くのに、~groupは常に設定が必要でしょうか?
質問2
任意の時間における、kaplan meier法による生存率を計算する関数というのはあるのでしょうか?
Rを覚えたてで、2冊の解説書をみながら悪戦苦闘しています。
よろしくお願いします。
sf <- survfit(fitteddata ~ 1, data = dataset)とすると,sf$timeが経過期間,sf$survが生存率となります.階段関数を返す関数stepfunを使うと,求める関数sは以下のように導くことができます.
s <- stepfun(c(0, sf$time), c(1, 1, sf$surv))群分けをした場合は,群ごとに経過期間と生存率を切り出してから同様にすればいいでしょう.
mtk (2010-05-09 (日) 20:52:56)
MS Windows XP sp3上でRcmdrを使っています。
ver.2.8.0では変数をcharacterに変更するだけで層別化の因子と認識されていました。
ver.2.11.0でRcmdr 1.5-4を起動したところ,これでは認識されませんでした。そこで
データ → アクティブデータセット内の変数の管理 → 数値変数を因子に変換
で指定したところ認識されました。これは仕様の変更なのかトラブルなのか分かりません。
もし仕様の変更なのでしたら層別化の便利な使い方があれば教えてください。
sousou (2010-05-07 (金) 15:55:04)
Rcmdrで適合性検定を行いたいですが、どうやってもうまく行かなかったんですが、どなたが助けていただけませんか?
操作手順は分かりますが(統計量→要約→頻度分布→カイ2乗適合度検定)、例えば、AとBの選択者が同じかとうかを検討することとしますが、Aの選択者は127名で、Bの選択者は71名です。Rcmdrでどのように操作するのかを教えていただければありがたいですが、よろしくお願いします。
X-squared = 15.8384, df = 1, p-value = 6.899e-05となるはず。以下の例をご覧じろ。 -- 河童の屁は,所詮屁である。 2010-05-07 (金) 16:20:28
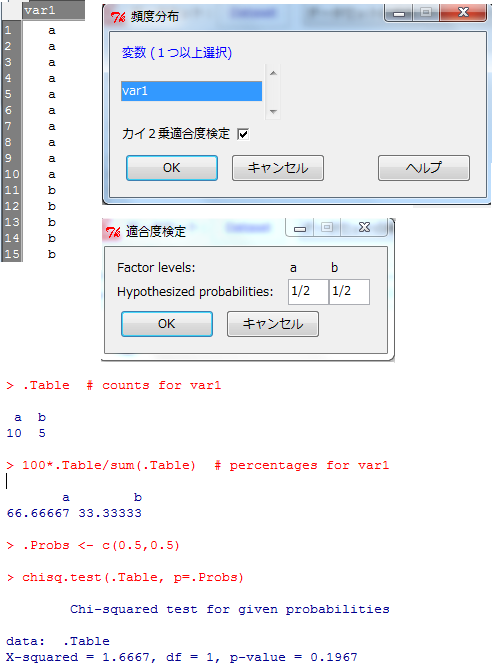
まさお (2010-05-07 (金) 15:51:27)
シェルスクリプト上でRによるさまざまな処理を行うには,どうしたらよいのでしょうか?#!/bin/csh
R
でRは起動するんですけれど,そこで止まってしまいます.R内のコマンドはシェルスクリプト内でどのように書けばよいのでしょうか.
foo [11] > cat script x <- 0 for (i in 1:5) x <- x+sqrt(i) print(x)それを動かす
foo [12] > R --vanilla --silent --fiile=script > x <- 0 > for (i in 1:5) x <- x+sqrt(i) > print(x) [1] 8.382332出力は,ファイルに出したりする方がよいでしょうけど。
#!/bin/bash
R --slave --vanilla << EOF
jpeg("plot.jpg")
plot(sin, xlim=c(0,2*pi))
EOF
詳細は高階さんの『プログラミングR』を買いましょう。 -- 2010-05-08 (土) 07:33:39さっこ (2010-05-07 (金) 12:07:39)
linux(GNU)にRを無事make installできたのですが(suで,/usr/local/r-2.10.1/に),RとタイプしてもRが起動しません(自分のusernameで).cshrcなどに何らかのパスを通す必要があるのでしょうか?
きりんさん (2010-05-04 (火) 11:17:02)
Rで統計解析を行い,その結果をそれぞれの物質名ごとのファイルに格納するちょっとしたツールを作成しています.
これにループ処理を用いて,完成させたいのですがdir.create(“Ala”)などの「“”」に囲まれた部分を配列に置き換えるなどの操作が出来ずに困っています.
他のプログラミング言語を用いずにRのみで行う方法はないでしょうか?
拙い質問ではありますが,ご教授のほどを宜しくお願い致します.データフレーム(ROC_table1.csv) Ala Ethanolamine phosphate Hypotaurine Pro Uridine Glycolate C 551.6935 29.13754 16.681 173.8867 45.10912 50.05477 C 607.481 0 13.91893 168.1533 42.37981 84.32064 C 506.6699 28.35182 16.64904 159.7178 43.60009 299.7703 C 530.9984 20.93959 11.42694 236.6067 42.33055 0 C 466.6074 19.3733 14.7419 176.2133 41.44249 180.9834 C 532.253 18.66473 12.15251 172.6138 40.66632 181.5452 C 449.6792 9.951401 11.69104 130.8715 47.65735 0 C 403.2724 19.33708 0 155.0835 47.37078 230.7595 C 423.5579 19.48771 11.05998 140.1291 38.48613 238.7741 C 463.7251 23.312 0 126.3421 43.01137 248.8566 C 465.0218 28.58722 10.97927 137.6295 37.42445 177.0466 C 485.2845 35.14039 7.268277 143.0229 37.97251 223.5736 D 707.1228 25.1555 11.25501 237.0575 37.70653 278.061 D 658.9739 31.52341 14.1557 197.7347 33.66692 312.7555 D 475.4444 31.67994 11.48172 132.5917 41.70935 545.9441 D 613.6305 23.5282 12.38427 164.597 41.24212 63.77056 D 545.5581 33.57919 21.21189 174.7159 43.30637 419.7285 D 848.2421 32.92102 16.18306 248.8493 30.06797 402.9233 D 507.675 25.12478 17.90848 185.7012 36.81457 315.3726 D 528.8479 20.10386 17.13785 196.1179 40.02536 126.3903 #データの読み込み x <- read.csv("ROC_table1.csv", header=T) data.frame(x) #ROCソースコード source("http://aoki2.si.gunma-u.ac.jp/R/src/ROC.R", encoding="euc-jp") #患者群データを格納 Dise <- (x$X=="D") disease.x <- x[Dise,"Ala"] #健常者群データを格納 Cont <- (x$X=="C") normal.x <- x[Cont,"Ala"] #物質名のフォルダを作成 dir.create("Ala") #新規フォルダへのディレクトリ変更 Direct <- getwd() Direct1 <- paste(Direct,"Ala",sep = "/") setwd(Direct1) #フォルダに計算結果を格納 RR <- ROC0(disease.x, normal.x) write.table(RR, file="Ala.data", sep="\t", row.names=FALSE, quote=FALSE) #フォルダに図を格納 bmp(filename = "Ala.bmp", width = 480, height = 480) ROC0(disease.x, normal.x) dev.off() #ディレクトリを元へ戻す setwd(Direct)
Name <- "Ala"
eval(parse(text=paste("dir.create('", Name, "')", sep="")))さつまいも (2010-05-03 (月) 20:23:20)
初めにヒストグラムを作ってから、X軸の目盛りを変えると最初の目盛りとかぶってしまいます。
どうしたら最初の目盛りを消すことができますか?
ピースケ (2010-04-29 (木) 17:51:37)
英語でテキストマイニングをする方法を探しています。。語の頻度,形態素について分析するパッケージはありますでしょうか。
せーだ (2010-04-29 (木) 12:19:03)
Win7 Pro/WinXP Pro SP2を使っています。
R-2.11.0で両方とも再現しましたので投稿します。
以下のサンプルは、R-2.10.1までは問題なく動作していました。
しかし、2.11.0では、コンボボックス内の日本語だけが文字化けします。require(tcltk) tclRequire("BWidget") tt <- tktoplevel() tkgrid(tklabel(tt,text="好きな果物は?")) fruits <- c("林檎","蜜柑","バナナ","梨") comboBox <- tkwidget(tt,"ComboBox",editable=FALSE,values=fruits) tkgrid(comboBox) OnOK <- function() { fruitChoice <- fruits[as.numeric( tclvalue(tcl(comboBox,"getvalue")))+1] tkdestroy(tt) msg <- paste(fruitChoice,"は美味しいよね",sep="") tkmessageBox(title="Fruit Choice",message=msg) } OK.but <-tkbutton(tt,text=" OK ",command=OnOK) tkgrid(OK.but) tkfocus(tt)2.11.0の新機能説明の項目の中に
Package tcltk now sends strings to Tcl in UTF-8: this means that strings with a marked UTF-8 encoding are supported in non-UTF-8 locales.とあったので、これが影響しているのでしょうか。
また、このような文字化けは、BWidgetやTktable等、Tcl/Tkの拡張ライブラリに由来するウィジェット全般に発生しているのではないかと思います(Tktableでも、2.11.0ではセル中の日本語が化けました)。
もし対処法をご存知の方がいらっしゃれば、ご教示頂きたく存じます。
宜しくお願い致します。
josephine (2010-04-28 (水) 18:34:02)
大量のデータをS-W検定するスクリプトを作っています。
不適当な標本データが原因のS-W検定のエラーを上手く処理して結果をファイルに出力させたいのですが、try()では上手くゆきませんでした。
入力が適切な例> a [1] 0 0 0 1 0 0 0 > try(shapiro.test(a), silent=TRUE) Shapiro-Wilk normality test data: a W = 0.453, p-value = 4.136e-06 > s_shapiro_a <- try(shapiro.test(a), silent=TRUE) > s_shapiro_a Shapiro-Wilk normality test data: a W = 0.453, p-value = 4.136e-06入力が不適切な例
> b [1] 0 0 0 0 0 0 0 > s_shapiro_b <- try(shapiro.test(b), silent=TRUE) > s_shapiro_b [1] "Error in shapiro.test(b) : all 'x' values are identical\n" attr(,"class") [1] "try-error"try-errorの場合にはNAをs_shapiro_bに代入したいのですが、恐れ入りますが、アドバイスを頂けないでしょうか?よろしくお願いいたします。
> Shapiro.Wilk.test(letters) # 数値データでない [1] NA > Shapiro.Wilk.test(c(1, 2)) # 少なすぎるデータ [1] NA > Shapiro.Wilk.test(c(1, 1, 1, 1, 1, 1)) # 全部同じデータ [1] NA > Shapiro.Wilk.test(rnorm(10000)) # 多すぎるデータ [1] NA > Shapiro.Wilk.test(c(1, 4, 3, 6)) # 妥当なデータ Shapiro-Wilk normality test data: c(1, 4, 3, 6) W = 0.9984, p-value = 0.995
orange (2010-04-26 (月) 16:32:03)
グラフィックス参考実例集にあるcontourplot("Cube Root Ozone (cube root ppb)"と同様な等高線図を,色付き(赤←→緑)ではなく,白黒で描きたいのですが,どのようにすればよいでしょうか.どなたかお教え頂けないでしょうか.よろしくお願いします.
require(stats)
attach(environmental)
ozo.m <- loess((ozone^(1/3)) ~ wind * temperature * radiation,
parametric = c("radiation", "wind"), span = 1, degree = 2)
w.marginal <- seq(min(wind), max(wind), length.out = 50)
t.marginal <- seq(min(temperature), max(temperature), length.out = 50)
r.marginal <- seq(min(radiation), max(radiation), length.out = 4)
wtr.marginal <- list(wind = w.marginal, temperature = t.marginal,
radiation = r.marginal)
grid <- expand.grid(wtr.marginal)
grid[, "fit"] <- c(predict(ozo.m, grid))
contourplot(fit ~ wind * temperature | radiation, data = grid,
cuts = 10, region = TRUE,
xlab = "Wind Speed (mph)",
ylab = "Temperature (F)",
main = "Cube Root Ozone (cube root ppb)",
col.regions=gray(seq(0, 1, by=0.01))) # これを加える之事よ gray もオンラインヘルプでね
detach()
前の人の回答は,あなたに対して「オンラインヘルプで contourplot の col.regions 引数について調べたらいかがでしょ?」と言っているわけですよ。それに対して「素人でなもので〜」はないでしょう(^_^;) -- 河童の屁 2010-04-26 (月) 22:43:02uribo (2010-04-25 (日) 22:43:10)
forループで計算を1000回繰り返したのですが、その計算結果の最後の値だけを1000個分抽出する式をつくりたい場合どうすればよいでしょうか?
恐らく意、はじめのforループ計算の値の最後の値のみを出す式を1000回繰り返すとはおもうのですがその最後の値の抽出方がわかりません。もしよろしければどなたかご教授お願いいたします。
> func <- function(n)
+ {
+ for (i in 1:n) {
+ x <- rnorm(1)
+ }
+ return(x)
+ }
> replicate(100, func(sample(10, 1)))
[1] 0.19487424 1.39857201 0.97981611 -0.45801807 0.36414608
[6] -0.95167954 -1.96734801 1.77910267 -0.51803260 0.91834117
途中省略
[91] 0.76621199 0.66828262 1.04878517 0.12996552 -0.81528159
[96] 2.47874580 -0.39178534 0.73668553 1.81568251 1.36909985totoro (2010-04-24 (土) 00:33:22)
Rで多次元項目反応理論(多次元IRT)または多次元カテゴリカル因子分析をしたいのですが、パッケージはありますでしょうか?
解析例などを示しているページがありましたら、教えていただけると幸いです。
itok (2010-04-19 (月) 20:46:34)
VistaでR-2.8.0を使っております。最近latticeを勉強しております。
教科書の例題に以下のようなヒストグラムがありました。data(Chem97, package = "mlmRev") library(lattice) histogram(~gcsescore | factor(score), data = Chem97)
出力をみると、縦軸目盛が、1行目では右に、2行目では左についています。
他の例題をみても、このように互い違いになるのが標準設定のようです。
希望としては、両方の行で、縦軸目盛を左に統一したいのですが、可能でしょうか。
ヘルプを読んでみましたが、どのパラメータを設定すればよいのかわかりませんでした。
どうかよろしくおねがいいたします。
histogram(~gcsescore | factor(score), data = Chem97, scales=list(alternating=FALSE))scales パラメータの alternating 要素についての説明をご参照ください。
にわか (2010-04-18 (日) 00:35:23)
主成分分析のスコアプロットで、データのグループごとに色分けして表示させたいのですが、可能でしょうか?
irisで
.PC <- princomp(~PL+PW+SL+SW, cor=TRUE, data=iris)
scoreplot(.PC,labels="names",cex=0.7,col=2)
このようにラベル表示はできるのですが、グルーピングが分かりやすいようにデータの1:50と51:100、101:150を色分けして表示できるとありがたいのです。
どなたかご教示頂けたら助かります。
よろしくお願いします。
Kita (2010-04-14 (水) 15:33:17)
SASの場合、GLM解析を行った場合、Least Square Mean(LSMEAN)が計算されるとしていますが、RでLSMEANを求めるためにはどうすればいいのですか?
よろしくお願いします。
> a <- rnorm(100)
> b <- rnorm(100)
> res <- glm(b ~ a)
> summary(res)
Call: glm(formula = b ~ a)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.41666 -0.58843 0.03412 0.62652 1.93350
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -0.04376 0.09761 -0.448 0.655
a 0.07966 0.10726 0.743 0.459
(Dispersion parameter for gaussian family taken to be 0.9520184)
Null deviance: 93.823 on 99 degrees of freedom
Residual deviance: 93.298 on 98 degrees of freedom
AIC: 282.85
Number of Fisher Scoring iterations: 2
> names(res)
[1] "coefficients" "residuals" "fitted.values"
[4] "effects" "R" "rank"
[7] "qr" "family" "linear.predictors"
[10] "deviance" "aic" "null.deviance"
[13] "iter" "weights" "prior.weights"
[16] "df.residual" "df.null" "y"
[19] "converged" "boundary" "model"
[22] "call" "formula" "terms"
[25] "data" "offset" "control"
[28] "method" "contrasts" "xlevels"
> sum(res$residuals^2)
[1] 93.2978> a <- rnorm(100)
> b <- rnorm(100)
> c <- rnorm(100)
> res <- glm(b ~ a*c)
> summary(res)
Call:
glm(formula = b ~ a * c)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.26867 -0.54240 -0.08402 0.54603 2.49951
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.03420 0.08595 0.398 0.692
a -0.12119 0.09144 -1.325 0.188
c 0.06217 0.08400 0.740 0.461
a:c 0.04987 0.08823 0.565 0.573
(Dispersion parameter for gaussian family taken to be 0.7136645)
Null deviance: 70.126 on 99 degrees of freedom
Residual deviance: 68.512 on 96 degrees of freedom
AIC: 255.97
Number of Fisher Scoring iterations: 2
これで交互作用項a*cが出ています。-- Saito 2010-04-14 (水) 20:25:55Saito (2010-04-08 (木) 23:03:40)
下のほうで、adapt関数以外で重積分をする方法についてで質問した者です。
少し前の話ですので、新しく質問をさせていただきました。
あれから色々と勉強して、以下のようなサイトを見ながらやっていたのですが、どうにも巧くいきません。
http://www.okada.jp.org/RWiki/?RcmdrPlugin%C4%B6%C6%FE%CC%E7
http://www.okada.jp.org/RWiki/?%BB%E4%C5%AA%A5%D1%A5%C3%A5%B1%A1%BC%A5%B8%BA%EE%C0%AE%CB%A1
http://www.okada.jp.org/RWiki/?Windows%A4%C7R%A4%CE%A5%D1%A5%C3%A5%B1%A1%BC%A5%B8%A4%F2%BA%EE%C0%AE%A4%B9%A4%EB
まず今回はすでにあるtarファイルを使いますので、最初から作る必要はないと考え、adaptパッケージのtarファイルをこのサイトから直接C直下にダウンロードしました。そしてコマンドプロンプトにcd c:\ R CMD INSTALL adapt_1.0-4.tar.gzとしましたが、以下のようなメッセージが出てしまい先に進めません。
c:\>R CMD INSTALL adapt_1.0-4.tar.gz * installing to library 'C:\Users\Saito\Documents/R/win64-library /2.11' * installing *source* package 'adapt' ... ** libs DLLフャ... x86_64-w64-mingw32-gfortran -O2 -c adapt.f -o adapt.o make: x86_64-w64-mingw32-gfortran: Command not found make: *** [adapt.o] Error 127 ... ョケ ERROR: compilation failed for package 'adapt' * removing 'C:\Users\Saito\Documents/R/win64-library/2.11/adapt' * restoring previous 'C:\Users\Saito\Documents/R/win64-library /2.11/adapt'
これはフォートランの命令が読めない、というメッセージなのでしょうか?文字化けも気になるのですが・・・。ちなみにPathは、c:\>Path PATH=c:\Rtools\bin;c:\Rtools\perl\bin;c:\Rtools\MinGW\bin; C:\Program Files (x86)\HTML Help Workshop; C:\Program Files\R \R-2.11.0alpha-x64\bin; C:\Program Files (x86)\MiKTeX 2.8\miktex \bin; C:\Windows\system32;C:\Windows;C:\Windows\System32\Wbem; C:\Windows\System32\WindowsPowerShell\v1.0\となっています。
ちなみにOSはWindows7、Rのバージョンは、R-2.11.0alpha-x64となっています。何か64bit版特有のことをしないといけないのでしょうか。fortranは持っていませんので、買えと言われても少し困ってしまいます。
自分では手詰まりになってしまって、解決できませんでした。もし何か解決策をご存知でしたら、教えていただけると幸いです。
どうぞよろしくお願いいたします。
c:\>Path PATH=C:\mingw\mingw-w64-crt;C:\mingw\bin;C:\msys\1.0\bin;c:\Rtools\bin;c:\Rtools\perl\bin; c:\Rtools\MinGW\bin;C:\Program Files (x86)\HTML Help Workshop;C:\Program Files\R\R-2.11.0alpha-x64\bin; C:\Program Files (x86)\MiKTeX 2.8\miktex\bin;C:\Windows\system32;C:\Windows;C:\Windows\System32\Wbem; C:\Windows\System32\WindowsPowerShell\v1.0\と、少しづつ汚くなってきています…。
もし何かお気づきの点がありましたら、ご助言いただけると幸いです。 -- Saito 2010-04-09 (金) 00:12:37
c:\Rtools\MinGW\binの中にはgfortranというファイルは置いてあるのですが…。試しに、x86_64という名前のフォルダをC直下に作って、w64というフォルダをさらにその中に作り、そしてmingw32をその中に作ったあと、c:\Rtools\MinGW\binの中にあった、gfortranを持ってきたのですが、それでもダメでした。もちろんパス設定を変えて再起動したあとです。
PATH=C:\x86_64\w64\mingw32;C:\mingw\mingw-w64-crt;C:\mingw\bin;C:\msys\1.0 \bin;c:\Rtools\bin;c:\Rtools\perl\bin;c:\Rtools\MinGW\bin;C:\Program Files (x86)\HTMLHelp Workshop;C:\Program Files\R\R-2.11.0alpha-x64\bin;C:\Program Files (x86)\MiKTeX 2.8\miktex\bin;C:\Windows\system32;C:\Windows;C:\Windows \System32\Wbem;C:\Windows\System32\WindowsPowerShell\v1.0\すみません、いまだに解決していません。まだ何かお気づきの点がありましたら、ご助言ください。 -- Saito 2010-04-14 (水) 14:34:13
C:\Users\Saito>R R version 2.11.0 alpha (2010-04-06 r51611) Copyright (C) 2010 The R Foundation for Statistical Computing ISBN 3-900051-07-0 RヘAゥRネ\tgEFAナAuョSノウロリvナキB フノ]ヲホAゥRノアトzzキアニェナォワキB zzフレラノヨオトヘA'license()'「ヘ'licence()'ニヘオトュセウ「B Rヘスュフv」メノ、ッvWFNgナキB レオュヘ'contributors()'ニヘオトュセウ「B ワスARRフpbP[Woナィナpキロフ`ョノツ「トヘ 'citation()'ニヘオトュセウ「B 'demo()'ニヘキホfンアニェナォワキB 'help()'ニキホICwvェoワキB 'help.start()'ナHTMLuEUノwvェンワキB 'q()'ニヘキホRIケオワキB > rnorm(10) [1] -0.8424220 -0.4483672 -1.2258821 0.1038380 -0.8283102 -0.5972562 [7] 0.1248794 -1.9883918 0.2761847 1.2740648 >ただ、gfortranとやると、
C:\Users\Saito>gfortran gfortran: no input filesと出てしまいます。Pathがうまくつながっていないのでしょうか? -- Saito 2010-04-14 (水) 15:45:19
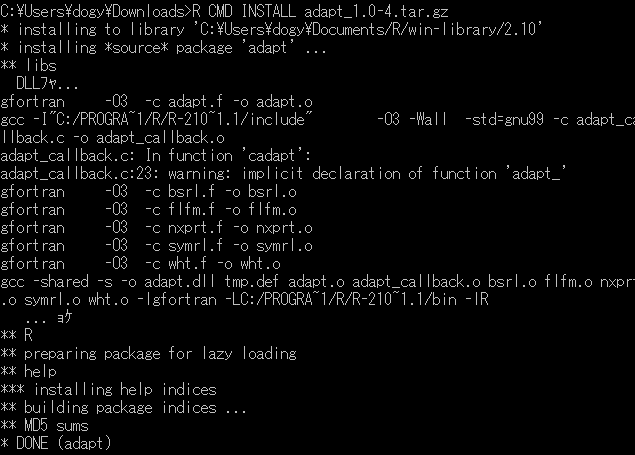
C:\Users\Saito\Downloads>R CMD INSTALL adapt_1.0-4.tar.gz * installing to library 'c:/PROGRA~2/R/R-210~1.1/library' G[F G[FfBNgヨフCXg[ツェワケ 'c:/PROGRA~2 /R/R-210~1.1/library'一応河童の屁さんの言うとおり、Rtoolsを再インストールして、パスはいじらずに(ただし以下のPathのように自動生成される分は放置)、ダウンロードに移動して、R CMD INSTALL adapt_1.0-4.tar.gzと命令したのですが…。
PATH=c:\Rtools\bin;c:\Rtools\perl\bin;c:\Rtools\MinGW\bin;c:\Program Files (x86)\R\R-2.10.1\bin;C:\Windows\system32;C:\Windows;C:\Windows\System32\Wbem; C:\Windows\System32\WindowsPowerShell\v1.0\環境はWindows7なのですが、もしよろしければうまく行った動作環境を教えていただけませんか? -- Saito 2010-04-14 (水) 20:13:44
KM (2010-04-07 (水) 16:28:46)
相関行列を類似度行列としてクラスタ解析したいと考えています。
hclust()にas.dist(x)という形で読み込みたいのですが、hclustに入力する距離行列は非類似度行列(値が小さい因子から統合される)とのことです。
−1x相関係数を非類似度として読み込んでみましたが、hang=-1にすると因子ラベルが0に揃ってしまいます。
以下の2点いずれかができれば解決すると思うのですが、いかがでしょうか?
・類似行列のクラスタ解析ができる方法を実行
・hclustでラベルを揃える位置を0以外の位置に指定する
よろしくお願いします。
> (r <- cor(iris[,1:4]))
Sepal.Length Sepal.Width Petal.Length Petal.Width
Sepal.Length 1.0000000 -0.1175698 0.8717538 0.8179411
Sepal.Width -0.1175698 1.0000000 -0.4284401 -0.3661259
Petal.Length 0.8717538 -0.4284401 1.0000000 0.9628654
Petal.Width 0.8179411 -0.3661259 0.9628654 1.0000000
> (d <- as.dist(2*(1-r)))
Sepal.Length Sepal.Width Petal.Length
Sepal.Width 2.23513957
Petal.Length 0.25649245 2.85688021
Petal.Width 0.36411775 2.73225187 0.07426914
> plot(hclust(d), hang=-1)
あ〜る (2010-04-07 (水) 11:19:57)
Rのoptim関数と同じ関数をjavaで計算したく、javaのlibraryで探しています。
また、javaで完結しているコード(他の言語を呼び出したりしない)を探しています。
実際に計算したいのは、L-BFGS-B法ですが、javaのlibraryでは見つけられませんでした。
もしご存知の方がいらっしゃったらご教示ください。
よろしくお願いいたします。
kojiro_i619 (2010-03-31 (水) 13:50:28)
お世話になります。中澤先生の本から、以下gnm <- readShapePoly("gunma.shp") gnmdata <- gnm$att.data gnmpoly <- map2SpatialPolygons(gnm, region.id=att(gnm, "region.id")) ## <−これがうまくいきません。 gunmadata <- gnmdata gunmapoly <- gnmpoly aged <- read.delim("agedprop.txt") gunmadata <- merge(aged, gunmadata, sort=F, by="JCODE") DD <- gunmadata$AP2006 classes <- cut(DD, seq(min(DD), max(DD), length=5), include.lowest=T) cols <- topo.colors(4) plot(gunmapoly, col=cols[ordered(classes)], xlab="", ylab="", axes=F) legend(max(x)-0.3*(max(x)-min(x)), min(y)+0.1*(max(y)-min(y)), legend=names(table(classes)), cex=0.6,fill=cols) title("群馬県市町村の65歳以上高齢者割合4区分(2006年)") text(x, y, gunmadata$CITY1, cex=0.5, pos=1, offset=0)などと、作ったのですが、途中の命令が、ないようです。
見本のコードでも、教えていただければ、幸いです。
rbegginer (2010-03-30 (火) 21:27:26)
いつも勉強させていただいております。
以下のプログラムについて、高速化のコツ、ヒントなどをいただけないでしょうか。
以下のようなプログラムで、1千万の母集団から5サンプルの抽出を1000回行い、外れ値を除去して平均値を計算する、ということを行っております。
5サンプルが終わったら6,7,8...100とサンプルサイズを変えてまた平均値を計算...というようにしています。さらに基準や数値を変えて実行しようと思っています。
実はサンプル抽出を1000回ではなく10000回行いたいのですが、このプログラムが大変時間がかかり、5000回でやってみると50分程度かかります。
時間がかかっていると部分、高速化できるような書き方がありましたら教えていただけないでしょうか
こちらのTipsでforは時間がかかるという記述を読みましたが、sapplyにする方法も思いつかず悩んでおります。
どうぞよろしくお願いします。環境はWindowsVista, R2.10.1です。pop <- round(rnorm(10000000, mean=300, sd=20)+(300*rexp(10000000))) ssz <- c(5,6,7,8,9,10,15,20,25,35,50,100) # 抽出するサンプルの数 nrecv <- 0 nremv <- 0 for(j in 1:length(ssz)) { for(i in 1:1000){ svct <- sample(pop, ssz[j]) gmean <- mean(svct) gsd <- sd(svct) cfv.u <- gmean+(3*gsd) # 外れ値のカットオフポイント設定 cfv.l <- gmean-(3*gsd) # 外れ値のカットオフポイント設定 tmp <- svct tmp[tmp>cfv.u] <- NA # 外れ値除去 tmp[tmp<cfv.l] <- NA # 外れ値除去 svct.el <- tmp nrecv[i] <- mean(svct.el, na.rm=T) # 外れ値除去後サンプルの平均 } nremv[j] <- mean(nrecv) # 1000個の平均値の平均 }
prog3 <- function()
{
set.seed(666)
# pop <- round(rnorm(10000000, mean=300, sd=20)+(300*rexp(10000000))) これを止める
ssz <- c(5,6,7,8,9,10,15,20,25,35,50,100) # 抽出するサンプルの数
n <- length(ssz)
m <- 5000
nrecv <- numeric(m)
nremv <- numeric(n)
covct3 <- 1
for(j in 1:n) {
pop <- matrix(round(rnorm(ssz[j]*m, mean=300, sd=20)+
(300*rexp(ssz[j]*m))), ssz[j]) # こっちにする
gmean <- colMeans(pop)
gsd <- sd(pop)
cfv.u <- gmean+(covct3*gsd) # 外れ値のカットオフポイント設定
cfv.l <- gmean-(covct3*gsd) # 外れ値のカットオフポイント設定
for (i in 1:m) {
svct <- pop[,i]
svct <- svct[cfv.l[i] < svct & svct < cfv.u[i]]
nrecv[i] <- mean(svct)
}
nremv[j] <- mean(nrecv) # m 個の平均値の平均
}
return(nremv)
}
> system.time((ans <- prog3()))
ユーザ システム 経過
7.881 0.127 8.041
> ans
[1] 527.4018 530.2227 530.0385 530.0244 528.1573 530.5734
[7] 525.2133 524.3569 523.8537 520.2583 518.8018 516.8923kojiro_i619 (2010-03-29 (月) 13:10:05)
CD−ROMの内容をロードするWEBサイトがありましたら、教えてください。
にわか (2010-03-28 (日) 00:06:36)
winXPでR2.10.1を使っています。
線形モデルの結果にaov、anovaを用いた分散分析を行うと、anova行うとエラーがでます。aovではでません。
いろんな所(たとえばhttp://www1.doshisha.ac.jp/~mjin/R/15.html)に、anovaで行う事例が載っていますので、当方の何かに問題があると思うのですが、見当がつきません。
漠然とした質問で恐縮ですが、どのようなところに問題ありそうでしょうか。
よろしくご教示いただけたら幸いです。
> anova(lm(count~spray, data=InsectSprays))
Analysis of Variance Table
Response: count
Df Sum Sq Mean Sq F value Pr(>F)
spray 5 2668.8 533.77 34.702 < 2.2e-16 ***
Residuals 66 1015.2 15.38
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1jiro (2010-03-25 (木) 23:11:55)
OS: mac osx 10.6.2( intel mac core 2 duo )
R ver: 2.10.1(32bit、64bit ともにインストール)
PostgreSQL ver: 8.3.1
上記の環境にて、R言語からPostgreSQLに接続しようとしています。
DBIのver 0.2-5 を install.package(DBI) コマンドにてインストールし、シェルからコマンドをたたき、RPostgreSQL をインストールしました。$> sudo R CMD INSTALL RPostgreSQL_0.1-6.tar途中、
rawToChar(magic[1:5])中で警告がありました: 文字列をnullに切り詰めました: 'Rpo\0\0'という警告がでたもののインストールには成功したようです。
さて、Rのコンソールから、> library( RPostgreSQL )と実行すると下記のメッセージが表示され、パッケージのロードに失敗します。
64ビット版で失敗しているようなので、この問題の解決方法か、32bit 版の RPostgreSQL のインストール方法をお教え頂けないでしょうか。
よろしくお願いします。Error in dyn.load(file, DLLpath = DLLpath, ...) : ~ 共有ライブラリ '/Library/Frameworks/R.framework/Resources/library/RPostgreSQL/ libs/x86_64/RPostgreSQL.so' を読み込めません dlopen(/Library/Frameworks/R.framework/Resources/library/ RPostgreSQL/libs/x86_64/RPostgreSQL.so, 6): Symbol not found: _PQbackendPID Referenced from: /Library/Frameworks/R.framework/Resources/library/ RPostgreSQL/libs/x86_64/ RPostgreSQL.so Expected in: flat namespace in /Library/Frameworks/R.framework/Resources/library/RPostgreSQL/libs/x86_64/RPostgreSQL.so エラー: 'RPostgreSQL' に対するパッケージもしくは名前空間のロードが失敗しました
にわか (2010-03-24 (水) 16:07:09)
標記の題名で11でお世話になった者です。
クリアに解決しましたので、投稿しておきます。
説明変数がマトリックスの場合には、protect関数(?) のI()を使えば、yarnのデータ構造のように、列毎に別々に変数を指定せずにまとめることができ、しかもデータフレームとしておけるようです。
例)y <- c(2, 4, 6) z <-matrix(1:12, 3, 4) mydata <- data.frame(Y=y, Z=I(z))以上、お世話になりました。
rbeginner (2010-03-20 (土) 14:07:10)
いつも勉強させていただいています。
コンソールに表示される命令と出力を全て保存する関数はあるのでしょうか?
メニューからファイル -> ファイルを保存 を選ぶとlastsave.txtというファイルとして保存されますが、これをコードで行いたいと考えています。
理想はsink(... append=T, split=T) のように逐一保存したいのですが、sink関数だと出力しか保存されないようです。
どうぞよろしくお願いいたします。
saka (2010-03-19 (金) 13:29:49)
GLMでガンマ分布とlogリンク関数を使って,将来の魚の漁獲量の予測値とその予測値の予測区間を求めたいのですが,予測区間をどのように求めたら良いのか分かりません。
推定値の信頼区間ではなく,それより広い範囲になることが多い予測区間の方です。
来年の予測漁獲量は○トンくらいだけど,95%の確率で○〜○トンの範囲になるということを知りたいのです。
GLMで解析するデータは下のようなものです。
これに,例えば新たに説明変数1,2,3がそれぞれ(12,5,1500)が得られたときに,予測漁獲量を「predict」を使って求めるところまではできるのですが,予測区間の求め方を教えてください。
よろしくお願いします。目的変数 説明変数1 説明変数2 説明変数3 7553 11.1 0.6 2240 14897 13.0 1.6 1292 30957 12.6 6.2 972 35056 9.7 26.2 2655 17436 11.4 9.4 547 4656 14.0 0.2 820 6879 12.3 0.0 1223 41853 13.9 4.4 2043 16308 12.1 4.8 1875 11293 11.8 3.0 1363 3964 10.2 9.8 236 7993 13.1 0.5 1236 6748 11.5 0.2 2144 3522 12.2 0.0 758 15395 10.5 7.6 1601 7567 11.3 6.0 1175 5823 10.7 4.4 880以上です。
> data <- data.frame(
+ y = c(7553, 14897, 30957, 35056, 17436, 4656, 6879, 41853,
+ 16308, 11293, 3964, 7993, 6748, 3522, 15395, 7567, 5823),
+ x1 = c(11.1, 13, 12.6, 9.7, 11.4, 14, 12.3, 13.9, 12.1, 11.8,
+ 10.2, 13.1, 11.5, 12.2, 10.5, 11.3, 10.7),
+ x2 = c(0.6, 1.6, 6.2, 26.2, 9.4, 0.2, 0, 4.4, 4.8, 3, 9.8,
+ 0.5, 0.2, 0, 7.6, 6, 4.4),
+ x3 = c(2240, 1292, 972, 2655, 547, 820, 1223, 2043, 1875,
+ 1363, 236, 1236, 2144, 758, 1601, 1175, 880)
+ )
>
> data.glm <- glm(y ~ x1 + x2 + x3, family = Gamma(log), data = data)
>
> data.prd <- predict(data.glm, newdata=data.frame(x1=12, x2=5, x3=1500),
+ se.fit=TRUE)
>
> exp(data.prd$fit)
1
13082.80
> exp(data.prd$fit - 1.96 * data.prd$se.fit)
1
10336.17
> exp(data.prd$fit + 1.96 * data.prd$se.fit)
1
16559.29にわか (2010-03-17 (水) 23:31:15)
ある植物を植えたときに、処理Aと処理Bで活着率に差があるかどうかを、時期を変えて調べた次のようなデータがあります。
各時期に処理A,Bとも30株ずつ植えたうち、表の株数が活着し、残りは枯死しました。
<各30個体を植えたときの活着した株数>
時期1 時期2 時期3 時期4
処理A 14株 13株 10株 15株
処理B 15株 14株 13株 16株
この場合、処理A、Bのどちらが活着に有効かを検定する場合、各時期ごとには活着の成功か失敗かという問題だと思うので、
prop.test(c(10,19),c(30,30))
というように比率の検定をすればいいと思うのですが、
全体で見る場合、単に合計で 52/120 と 58/120 の比率の検定をするのか、時期を反復と見て対応のあるt検定を行ってよいのか悩んでいます。
あるいは、「対応のある比率の検定」のようながあるのでしょうか。
どなたかご教示下さい。
よろしくお願いします。
Saito (2010-03-13 (土) 15:10:03)
あちこち調べ、自分でも試したのですが、わからなかったので質問させてください。
最近(といっても数ヶ月前)にCRANからadaptパッケージが消えたようです。
今まで重積分をするときにはadaptを使って積分していたので、消えた後も過去のRのバージョンのlibraryからadaptを持ってきて使っていました。
しかし、2.10.0以降になると以下のようにre-installを求められます。> library(adapt) エラー: package 'adapt' was built before R 2.10.0: please re-install it
re-installしようにもCRANにないのでできません。そこで仕方なく他の方法で重積分ができないか探したのですが見当たりませんでした。
どなたか、解決策をご存知でしたらご教授願えないでしょうか。
なお、環境はWindows 7、x64 R-2.11.1 Pre-releaseです。
初心者 (2010-03-10 (水) 00:24:41)
prcompを使用して固有値・固有ベクトルを算出しているのですが、どうも結果が意図したものと異なってしまいます。
・Rの出力> A <- matrix(c(0, -4, 4, 1, 4, -3, 1, 2, -1), nrow=3) > A [,1] [,2] [,3] [1,] 0 1 1 [2,] -4 4 2 [3,] 4 -3 -1 > prcomp(A, scale=F, center=F) Standard deviations: [1] 5.579673e+00 9.312648e-01 1.184667e-16 Rotation: PC1 PC2 PC3 [1,] -0.7093545 -0.6210515 -0.3333333 [2,] 0.6431273 -0.3767530 -0.6666667 [3,] 0.2884500 -0.6872788 0.6666667・参考にしたURL
http://www004.upp.so-net.ne.jp/s_honma/urawaza/eigenvector.htm
このページによると固有値は0,1,2となるはずなのですが、何か使い方等で間違っているのでしょうか?
Windows XPで R2.8.1 を使用しています。
> # prcomp(A, scale=F, center=F) で実行されること
> A <- matrix(c(0, -4, 4, 1, 4, -3, 1, 2, -1), nrow=3)
> B <- t(A)%*%A/(nrow(A)-1)
> ans <- eigen(B)
> sqrt(ans$values) # Standard deviations:
[1] 5.579673e+00 9.312648e-01 2.267999e-08
> ans$vectors # Rotation:(列単位で符合は任意)
[,1] [,2] [,3]
[1,] 0.7093545 0.6210515 -0.3333333
[2,] -0.6431273 0.3767530 -0.6666667
[3,] -0.2884500 0.6872788 0.6666667初心者 (2010-03-10 (水) 00:04:04)
Windows VistaのR-2.10.1です.(Rの他のバージョン,他のOSでもうまく動いて欲しいです)
例えば,「何章」や「何節」もしくは「番号のみ」(いずれも1〜2桁で,全角の数字の可能性もあります)から番号だけにするようなとき,section <- c("1", "23", "4部", "56部", "7章", "89章") section <- c(section, "1", "23", "4節", "56節") sub("([[:digit:]]{1,2})[^[:digit:]]?", "\\1", section)でしてみましたが、結果が
[1] "1" "23" "4部" "56" "7章" "89" "1" "23" "4節" "56"のようになってしまいました.
どのようにしたら,うまく番号だけ抜き出せるのでしょうか? また何が原因なのでしょうか?? 宜しくお願いいたします.
R初心者 (2010-03-09 (火) 10:36:33)
R-2.10.1をWindows Vistaで使用しています。mylist <- list(alph=letters, Alph=LETTERS, num=as.character(0:9)) grep("A", mylist, ignore.case=FALSE) # (1) grep("C", mylist, ignore.case=FALSE) # (2) grep("c", mylist, ignore.case=FALSE) # (3)ここで、(1)と(2)などpatternが(3)の "c" 以外のgrepの挙動は私の望んだものとなっています。
しかし、(3)のgrepの挙動がおかしい(私が望んでいるものではない)です・・・。
本来ならば 1 が帰ってきて欲しいのですが、 c(1,2,3) が帰ってきてしまいます。
1 と帰ってくるような手立てはございますでしょうか? また、これはバグなのでしょうか?
grep以外の関数での解決方法でも結構です。よろしくお願いいたします。
lgrep<-function (pattern, x, ignore.case = FALSE, extended = TRUE, perl = FALSE,
value = FALSE, fixed = FALSE, useBytes = FALSE, invert = FALSE) {
unlist(lapply(mylist, function(x) !(!length(grep(pattern,
x, ignore.case, extended, perl, value, fixed, useBytes,
invert))))) }
lgrep("c",mylist,ignore.case=FALSE)
alph Alph num
TRUE FALSE FALSE> lgrep<-function (pattern, x, ignore.case = FALSE, extended = TRUE, perl = FALSE,
+ value = FALSE, fixed = FALSE, useBytes = FALSE, invert = FALSE) {
+ unname(which(sapply(mylist, function(x) length(grep(pattern,
+ x, ignore.case, extended, perl, value, fixed, useBytes,
+ invert)))==TRUE)) }
> #
> lgrep("A", mylist, ignore.case=FALSE) # (1)
[1] 2
> lgrep("C", mylist, ignore.case=FALSE) # (2)
[1] 2
> lgrep("c", mylist, ignore.case=FALSE) # (3)
[1] 1sapply(mylist,function(x,y) length(grep(y,x))>0,y="A")#上の場合 seq(mylist)[sapply(mylist,function(x,y) length(grep(y,x))>0,y="A")]#下の場合とか。でも、length(grep(y,x))>0は美しくないね。 -- akira 2010-03-09 (火) 16:04:30
a <- c("A", "B", "CD")
y <- 3:9
x <- data.frame(a=(1:4), b=c("a","b","c","d"), value=c(TRUE, FALSE, FALSE, TRUE))
x <- list(a=a, num=y, data=x)
# 関数の定義
lgrep <- function(pattern, x, ...,
classes = "ANY", deflt = NULL,
how = c("unlist", "replace", "list" )) {
rapply(x, function(x, pattern, ...) grep(pattern, x, ...),
classes=classes, deflt=deflt, how=how, pattern=pattern, ...=...)
}
lgrep("\\<A\\>", x, how="list", deflt=NA)
structureですが、やっぱりよくわかりません・・・。なぜ、data.frameは data.frame(...) 全体が検索対象にならないのか・・・。なぞが深まりました。 -- R初心者 2010-03-09 (火) 20:25:04lapply(x, function(x,y) apply(as.matrix(x),2,function(x,y) grep(y,x),y=y), y="A")こんな感じかな。もっと入れ子構造ならrapplyを使うのでしょうか? -- akira 2010-03-10 (水) 07:52:08