![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
初心者のための R および RjpWiki に関する質問コーナー
過去の記事のアーカイブ。
きむ (2010-03-08 (月) 00:50:28)
いつも勉強させていただいてます。
毎度、基本的なことですみませんが、質問させてください。
現在、以下のような処理をしようとしています。
具体的には、dataA(45万個のデータ)のxとdataB(2000個程度のデータ)のyをidというフラグで関連づけて簡単な処理(zを求める)をしたいと考えています。
(「if(length(y)==0) y <- 0」は該当しないidがほとんどであり、この場合にnumeric(0)が帰ってくるため、このようにしてみました。)
ii <- 450000 z <- numeric(ii) for(i in 1:ii){ y <- dataB[dataB$id == dataA$id[i],"y"] if(length(y)==0) y <- 0 ifelse(y < dataA$x[i], z[i] <- 0, z[i] <- y-dataA$x[i]) }
ただ、45万回のforループを処理するため、かなり時間がかかります。
dataAをdataBの共通分(5万個程度)のみ抽出し、処理してからdataAにmergeしようかとも思いましたが、メモリ不足でmergeできませんでした。
applyファミリー等を使えば、forを内部化し、処理を高速化することが可能でしょうか?
プログラミングのテクニックについて、アドバイスいただければ助かります。
基本的な質問で申し訳けありませんが、よろしくお願いいたします。
mesh.id m5 GL 1 S035E005-21-3-3 21 100 2 S035E005-21-3-3 22 100 3 S035E005-21-3-3 23 100dataBの内容(データ数2063):FL2が先ほどの例のyです
mesh.id FL2 1 S035E010-14-1-2 21.36078 2 S035E010-14-1-4 20.57547 3 S035E010-14-2-1 20.80305ちなみに、実際のプログラムです:dataAがm5GL,dataBがm25Vshです。1つのm25Vshが25個のm5GLに対応しているのでm5GLのうちの使用分は51575個です。 -- きむ 2010-03-08 (月) 10:09:13
kk <- length(m5GL$mesh.id)
FL <- numeric(kk)
for(i in 1:kk){ #5mメッシュGLと比較してメッシュ別FLを算定
FL2 <- m25Vsh[m25Vsh$mesh.id == m5GL$mesh.id[i],"FL2"]
if(length(FL2)==0) FL2 <- 0
ifelse(FL2 < m5GL$GL[i], FL[i] <- 0, FL[i] <- FL2-m5GL$GL[i])
}ユーザ システム 経過 8379.59 15.28 8453.30
> test <- function()
+ {
+ z <- numeric(ii)
+ for(i in 1:ii){
+ y <- dataB[dataB$id == dataA$id[i],"y"]
+ if(length(y)==0) y <- 0
+ ifelse(y < dataA$x[i], z[i] <- 0, z[i] <- y-dataA$x[i])
+ }
+ return(z)
+ }
> set.seed(666)
> ii <- 450000
> dataA <- data.frame(id=sample(ii), x=rnorm(ii))
> num <- 2000
> dataB <- data.frame(id=sample(ii, num), y=rnorm(num))
> system.time(z <- test())
ユーザ システム 経過
80.553 0.488 80.596
> test2 <- function()
+ {
+ z <- numeric(ii)
+ for(i in 1:ii){
+ y <- dataB[dataB$id == dataA$id[i],"y"]
+ if(length(y)==0) y <- 0
+ z[i] <- y
+ }
+ z <- ifelse(z < dataA$x, 0, z-dataA$x) # これはループの外で
+ return(z)
+ }
> system.time(z2 <- test2())
ユーザ システム 経過
62.656 0.984 63.291
> all.equal(z, z2) # 答えが同じになることを確かめよう!
[1] TRUEEmacs超初心者 (2010-03-07 (日) 04:44:03)
Emacs 23 on Windowsを先日から使い始めました。
若干、ここで質問するべき事項かどうか微妙なところではございますが、他で質問する場所が見当たりませんので、質問させてください。
ESSとauto-completeを使用しているのですが、ESSモード時にいちいちM-x auto-complete-modeと打たなくてはなりません。(global-auto-complete-mode t)や
(add-hook 'ess-mode-hook '(lambda () (auto-complete-mode)))をしてみましたが、自動でオンになりませんでした。
ESSモード時もしくは.Rファイルを開いたら、自動でオンにするための何か良い解決方法などはございますでしょうか?
(setq ess-mode-load-hook
'((lambda ()
(require 'auto-complete)
(when (boundp 'ac-modes)
(setq ac-modes
(append ac-modes
(list 'ess-mode ))))
(global-auto-complete-mode t)
)))
などと書けば、auto-complete.el自体を編集しないでもOKです。-- 2010-03-08 (月) 12:03:02(when (boundp 'ac-modes)
(setq ac-modes
(append ac-modes
(list 'ess-mode))))
でうまくいきました。ac-modesがキーワードなんですね。ありがとうございましたm(_ _)m -- Emacs超初心者 2010-03-08 (月) 16:27:23にわか (2010-03-04 (木) 00:18:15)
winXPで2.10.1を使っています
NIRのデータでPLS回帰をしようと思っていろいろ調べているところですが、パッケージplsのサンプルデータyarnのデータ構造がよくわからないので、どなたか教えてください。
NIRのデータが波長ごとに列変数になっているように見え、表示させると列名がNIR.1 NIR.2・・・NIR.268 density trainのようになっています。
ところがnames(yarn)をしますと、
[1] "NIR" "density" "train"
で、列変数が3つしかなく、NIR.1〜NIR.268が全てNIRに代表されています。
これはどういう構造のデータフレームなのでしょうか。また、どのようにすればこういう構造のデータフレームが作れるのでしょうか。
よろしくご教示のほどお願いします。
y <- c(1, 2, 3) x <- matrix(c(1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12), nrow=3, ncol=4) attr(x, "dimnames") <- list(NULL, NULL) ZZZ <- list(NIR=x, a=y)これで一応、plsr(a ~ NIR, 2, data=ZZZ・・・・) のように、NIRだけでNIR.1からNIR.4を指定することができます。ただ、このZZZをデータフレームとすることが出来ません。根本的に間違っている様な気がします・・・ NIRデータをpls回帰に使われておられる方、データの作り方をご教示いただけないでしょうか。よろしくお願いします。 -- にわか 2010-03-04 (木) 14:03:24
cex (2010-03-03 (水) 18:54:40)
CRANからRColorBrewerをインストールしたのですが,色の分割数が9まででいまひとつ使い勝手が良くありませんでした.
そこで,多数(100程度)の分割に耐えうるカラーパレットを追加するようなRのパッケージはありますでしょうか?
もしご存じの方がいらっしゃれば,ご教示お願いします.
まるこ (2010-03-02 (火) 20:14:55)
R初心者です。
Rのバージョンは2.10.1、OSはVISTAです。
クラスター分析がしたくて、data <- read.table("xxx.csv", header=TRUE, sep=",") d <- as.dist(data) ans <- hclust(d, method="ward") plot(ans, hang=-1)というスクリプトを教えていただきました。
データはあらかじめ作り、「カレントディレクトリ」に入れるようにという指示でした。
「カレントディレクトリ」というのは、Rが入っているフォルダという意味でしょうか?
D\Program Files\R-2.10.1 や、その中の「bin」や「doc」に保存して上記のスクリプトを実行しましたが、エラーになりました。
エラーメッセージは、In file(file, "rt") : ファイル 'rensyuu.csv' を開くことができません: No such file or directory > d <- as.dist(data) 以下にエラー as.vector(x, mode) : cannot coerce type 'closure' to vector of type 'any' > ans <- hclust(d,method="ward") 以下にエラー hclust(d, method = "ward") : オブジェクト 'd' がありません > plot(ans,hang=-1) 以下にエラー plot(ans, hang = -1) : オブジェクト 'ans' がありませんとなります。
それで、CSVファイルの保存場所が間違っているのだと思い、1行目をdata <-read.table(file.choose(), header=TRUE, sep=",")として、ファイルを選び、実行しました。
すると、またエラーメッセージが出ました。警告メッセージ: 1: In storage.mode(m) <- "numeric" : 強制変換により NA が生成されました ~ 2: In as.dist.default(data) : 正方行列ではありません ~ > ans <- hclust(d,method="ward") 以下にエラー hclust(d, method = "ward") : 外部関数の呼び出し(引数 11) 中に NA/NaN/Inf があります > plot(ans,hang=-1) 以下にエラー plot(ans, hang = -1) : オブジェクト 'ans' がありません過去ログで、データを新しいブックに貼り付けたらNAが消えたという方がいたので
それもやってみましたが、解決しません。
CSVファイルの保存場所の件と、2番目の方法でエラーにならないためにはどうしたらいいか、どちらのことでも結構ですので、ご存じの方にアドバイスをいただきたいと思います。
よろしくお願いいたします。
森の熊五郎 (2010-03-01 (月) 23:54:09)
下記のプログラムを使って、修理の件数予測をしようと思っています。
手法は簡単で、過去の製品の前年度比実績推移をもとに直近のデータも
過去と同じ推移になると想定して予測をします。
2002年に出したAモデルから2007年に出したFモデルを例に作りました。修理件数は2005年から現在までのデータがCSVのファイルにあります。
Aモデルに比べFモデルは60ヶ月遅れて出しました。2002年のモデルの2005年の
修理データは出荷後3年のデータですので、2007年モデルにとっては2010年、つまり今年の状況にちかいだろうという想定をし、ラグ関数で時間をシフトし、それぞれの結果をグラフで見て検討しています。
せっかくの計算結果をcsvに落とし、みんなに見せようと思って、プログラムを作りましたが、このまま、実行するとせっかくタイムラグ関数を使ってシフトしたにもかかわらず、書き込まれたCSVにはタイムシフト前の状態で書き込まれます。当たり前といえば当たり前なのですが、タイムラグがかかったまま
CSVに書き込む方法はありますか?ご教示ください。repair <- read.csv("Repair2005_10.csv", header=TRUE) N2002A <- ts(repair$X2002A, start=c(2005, 4), frequency=12) N2003B <- ts(repair$X2003B, start=c(2005, 4), frequency=12) N2004C <- ts(repair$X2004C, start=c(2005, 4), frequency=12) N2005D <- ts(repair$X2005D, start=c(2005, 4), frequency=12) N2006E <- ts(repair$X2006E, start=c(2005, 4), frequency=12) N2007F <- ts(repair$X2007F, start=c(2005, 4), frequency=12) R2002A <- lag(N2002A/lag(N2002A, k=-12), k=-64) R2003B <- lag(N2003B/lag(N2003B, k=-12), k=-52) R2004C <- lag(N2004C/lag(N2004C, k=-12), k=-40) R2005D <- lag(N2005D/lag(N2005D, k=-12), k=-28) R2006E <- lag(N2006E/lag(N2006E, k=-12), k=-16) R2007F <- lag(N2007F/lag(N2007F, k=-12), k=- 4) w7 <- rep(1, 7)/7 MDA <- filter(R2002A, filter=w7, sides=2) MDB <- filter(R2003B, filter=w7, sides=2) MDC <- filter(R2004C, filter=w7, sides=2) MDD <- filter(R2005D, filter=w7, sides=2) MDE <- filter(R2006E, filter=w7, sides=2) MDF <- filter(R2007F, filter=w7, sides=2) MRESULT <- data.frame(MDA, MDB, MDC, MDD, MDE, MDF) write.csv(RESULT, "RESULT200510.csv", row.names=TRUE)
> MRESULT=data.frame(MDA,MDB) > write.csv(RESULT,"RESULT200510.csv",row.names=TRUE) 以下にエラー inherits(x, "data.frame") : オブジェクト 'RESULT' がありませんとなります。つまり,期待する結果が得られないといっているのは,ファイルに書き出すものが違っているだけなんじゃないですか?
> set.seed(666)
> repair <- data.frame(X2002A=1:120, X2003B=1001:1120)
> head(repair)
X2002A X2003B
1 1 1001
2 2 1002
3 3 1003
4 4 1004
5 5 1005
6 6 1006
> N2002A <- ts(repair$X2002A, start=c(2005, 4), frequency=12)
> N2003B <- ts(repair$X2003B, start=c(2005, 4), frequency=12)
> R2002A <- lag(N2002A/lag(N2002A, k=-12), k=-64)
> R2003B <- lag(N2003B/lag(N2003B, k=-12), k=-52)
>
> w7 <- rep(1, 7)/7
> MDA <- filter(R2002A, filter=w7, sides=2)
> MDB <- filter(R2003B, filter=w7, sides=2)
> MRESULT <- data.frame(MDA, MDB)
> head(MRESULT)
MDA MDB
1 NA NA
2 NA NA
3 NA NA
4 5.444898 1.011952
5 3.944898 1.011940
6 3.278231 1.011928きむ (2010-02-18 (木) 19:05:21)
いつも勉強させていただいております。
モデルを用いたクラスター分析をしようとパッケージ「mclust」をCLANよりダウンロード、インストールしたのですが、library(mclust)で呼び出したときに、以下が表示されます。by using mclust, or by using any other package that invokes mclust, you accept the license agreement in the mclust LICENSE file and at http://www.stat.washington.edu/mclust/license.txtそこで、上記サイトに行って「license.txt」ファイルをダウンロードしてきたのですが、それをどのように設定したら良いのかがわかりません。
(とりあえず上記ライセンスでテストし、使えそうであれば正式な費用負担を考えています。←パッケージにも有償のものがあるのを今回初めて知りました。)
これまで、パッケージのライセンスがファイル形式のものを使用したことがなく、途方にくれています。
設定方法をご存知の方がおられましたら、教えてください。
よろしくお願いします。
くま (2010-02-18 (木) 10:17:57)
これまで正常に起動していたRcmdr上で、dev.off()を実行すると、突然エラー:cannot shut down device 1 (the null device)が出るようになってしまいました。インストールしているRcmdr 1.4-10/R 2.8.1でもRcmdr 1.5-4/R 2.10.1でも同じエラーが出ます。また、再起動しても同じです。OSはWindows 7 Home Premiumです。完全にお手上げの状態です。対処方法をご教示いただけると有り難いです。
ken (2010-02-16 (火) 22:07:15)
クラスタ解析をRで実施するにはいくつかの関数があるようですが、デンドログラムのプロット後さらにrect.hclustで加工したいためhclustの利用を考えています。
hclustでデンドログラムをプロットする際に要素を列の平均の大きい順にしたいのですが、どうしてもわからないため質問させていただきます。
私が行っている操作は以下の通りです。set.seed(567) x <- round(matrix(rnorm(50),ncol=5),3) d <- dist(x) cl <- hclust(d,method="complete") plot(cl, sub="none", main="hclust Dendrogram", hang=-1)目的としているプロットは次の操作で得られています。
しかし、rect.hclustで加工ができないので悩んでいます。dc <- as.dendrogram(cl) Colv <- colMeans(x) ddc <- reorder(dc, Colv) plot(ddc, main="as.dendrogram Dendrogram")何卒、アドバイスをいただけますようお願いいたします。
ざーさい (2010-02-15 (月) 08:43:19)
ある疾患に罹患した人のID8桁のリストがあり、罹患した人そうでない人のIDのリスト(1ID複数レコード)のうち罹患人は1をそうでない人には0を変数として割り付けたいのですが、どうすればいいでしょうか?
> data <- data.frame(id = as.character(10000001:10000010))
> id.list <- c("10000002", "10000005", "10000007")
> data$dis <- 0
> data$dis[data$id %in% id.list] <- 1
> data
id dis
1 10000001 0
2 10000002 1
3 10000003 0
4 10000004 0
5 10000005 1
6 10000006 0
7 10000007 1
8 10000008 0
9 10000009 0
10 10000010 0
もっと簡単な方法もあるかも -- surg 2010-02-15 (月) 15:12:51くま (2010-02-12 (金) 06:41:57)
RはXPで使っていましたのである程度は分かっているつもりでしたが、この度Windows 7にインスト―ルして使ったところ、aovやlmを実行すると、
関数"is"を見つけることができませんでした
というエラーメッセージが出て計算できなくなってしまいました。どうすれば、よろしいでしょうか。対策をご教示いただけるとありがたいです。
なお、操作はRcmdr上から行っており、その他の計算は支障なくできています。
Rは初心者 (2010-02-11 (木) 00:04:12)
修理受付の予測をするために、Rを勉強を始めました。
「Rによる時系列分析入門」を片手に悪戦苦闘の毎日です。
修理の件数は、季節変動があり、週変動もあります。
すなわち、
�修理は冬より夏に多く、
�火曜日にピークがきて週末にかけて台数が落ち込む
という傾向があります。
これは
�夏場の温度によって部品が壊れやすくなる
�お店に修理に出すのが週末で物流TATを考えると、工場にくるのが火曜日ごろになる
ということが原因です。
日毎の修理受付件数データを蓄積し、Rを使って予測までをして見ようと思っています。
月次のデータをもとに季節変動を抽出する方法、例えば、月次データをTESTDATA.csvに入れて、REP = read.csv("TESTDATA.csv", header=TRUE) RNUM = ts(REP$num, start=c(2008, 4), frequency=12) bunkai = decompose(RNUM, type="multiplicative") plot(bunkai)として季節変動を分解する方法までは勉強しました。
これを日次データをもとに、週変動と季節変動を求める方法を検討して,悩んでいます。上記のように、ts関数はRNUM = ts(REP$num, start=c(2008, 4), frequency=12)のように、年・月しか入れることができず、
Start=c(2008, 1, 4)のように年月日をいれることができない。
また日次でやると、祝日(修理職場も休みなので入荷がゼロ)となり、データ欠落が出て、かつその影響が翌日にでるような場合、それを補正する方法がわかりません。
予測まではまだまだですが、このような場合、どのように分析をしたらよいのかをアドバス頂けたらと思います。
nat (2010-02-07 (日) 02:37:21)
こんにちは。R初心者です。いつも拝見させていただいています。
ある式を最小化する2つのパラメータをoptimを使って求めようとしています。
2つのパラメータx1, x2のうち、x2はx1によって最適解の範囲が制限されるため、うまくいきません。f <- function(par, B, u) { x1 <- par[1] x2 <- par[2] (x1 / B + x1 / (B - x1) ) - (x1^u + x2^u) }おおよそ以下のような値を代入します。
B <- 10e+5 u <- -0.7 Q <- B / 10f を最小化し、かつx1 + x2 <= Qとなるようなx1, x2を出してきてほしいのですが、どのようにして「かつx1 + x2 <= Q」の部分を組み込めばよいのかがわかりません。
constrOptimなども試しましたが、よくわかりませんでした。optim(fn=f, par=c(0, 1), B1=B1, u=u, lower=c(0, 0), upper=c(Q, Q), method="L-BFGS-B")このやり方だと、「かつx1 + x2 <= Q」の部分が満たされない時があります。
ご教授よろしくお願いします。
> f <- function(par, B, u) {
+ Q <- B / 10
+ x1 <- par[1]
+ x2 <- par[2]
+ if((x1 + x2) > Q) 1e+10
+ else (x1 / B + x1 / (B - x1)) - (x1 ^ u + x2 ^ u)
+ }
>
> optim(c(1, 1), f, B = 10e+5, u = -0.7)
$par
[1] 1.542165e-37 1.258702e+00
$value
[1] -5.865545e+25
$counts
function gradient
501 NA
$convergence
[1] 1
$message
NULL
-- surg 2010-02-07 (日) 08:22:17> f <- function(par) {
+ x1 <- par[1]
+ x2 <- par[2]
+ if (x1+x2 > Q) x2 <- (Q-x1)*0.9 # 例えばこんな風にしてみる
+ (x1 / B + x1 / (B - x1) ) - (x1^u + x2^u)
+ }
> B <- 10
> u <- 0.7
> Q <- B / 10
> optim(c(0,1), f)
$par
[1] 0.3143118 0.6856882
$value
[1] -1.148777
$counts
function gradient
113 NA
$convergence
[1] 0
$message
NULLsyo (2010-02-06 (土) 17:11:34)
Rにてテキストマイニングの勉強をしている初心者なのですが、Rで語に対するTF-IDF判定や重要度判定を行いたいと考えています。Rでそのような事を行うためには、どのようなツールが最適でしょうか?初歩的な質問ですが、ご助言お願い致します。
yoou (2010-02-05 (金) 01:15:42)
Rで学ぶデータマイニングで、自宅のvistaを使い勉強してるのですが、 install.packages("relimp")でJapan(Tokyo)を選ぶと以下のエラーメッセージが出ます。--- Please select a CRAN mirror for use in this session --- 警告:unable to access index for repository ftp://ftp.ecc.u-tokyo.ac.jp/CRAN/bin/windows/contrib/2.4 download.packages(pkgs, destdir = tmpd, available = available, 中で警告がありました: no package 'relimp' at the repositoriesどうしたら解決できるでしょうか。
mittyo (2010-02-03 (水) 09:48:44)
はじめて投稿します。
RでGRASSのデータを使いたくて、spgrass6をインストールしようとしましたが、うまくいきませんでした。
エラーは以下の通りです。Error in dyn.load(file, DLLpath = DLLpath, ...) : 共有ライブラリ '/root/R/i686-pc-linux-gnu-library/2.10/rgdal/libs/rgdal.so' を読み込めません /root/R/i686-pc-linux-gnu-library/2.10/rgdal/libs/rgdal.so: undefined symbol: pj_get_ellps_ref Error : package 'rgdal' could not be loaded ERROR: lazy loading failed for package ‘spgrass6’rgdal.soに問題がありそうなので、Rでlibrary("rgdal")を実行したところ、
> library("rgdal") 要求されたパッケージ sp をロード中です Error in dyn.load(file, DLLpath = DLLpath, ...) : 共有ライブラリ '/root/R/i686-pc-linux-gnu-library/2.10/rgdal/libs/rgdal.so' を読み込めません /root/R/i686-pc-linux-gnu-library/2.10/rgdal/libs/rgdal.so: undefined symbol: pj_get_ellps_ref エラー: 'rgdal' に対するパッケージもしくは名前空間のロードが失敗しました > Error in dyn.loadError in dyn.loadと同様のエラーが出ました。
rgdalを入れなおそうと思い、install.packages("rgdal", dependencies=TRUE)を実行し、再度
library(rgdal)、install.packages("spgrass6", dependencies=TRUE)を実行しましたが、同様の結果でした。
環境ですが、OS:Vine Linux 5.0 R:version 2.10.0 (2009-10-26)です。
ご教示のほどよろしくお願い致します。
* Installing *source* package ‘rgdal’ ...
gdal-config: gdal-config
checking for gcc... gcc -std=gnu99
checking for C compiler default output file name... a.out
:
SNIP
:
** building package indices ...
* DONE (rgdal)
ダウンロードされたパッケージは、以下にあります
‘/tmp/RtmpvHL96d/downloaded_packages’
'.Library' 中のパッケージの HTML 索引を更新します
> library("rgdal")
要求されたパッケージ sp をロード中です
Error in dyn.load(file, DLLpath = DLLpath, ...) :
共有ライブラリ '/usr/local/lib/R/library/rgdal/libs/rgdal.so' を読み込めません
/usr/local/lib/R/library/rgdal/libs/rgdal.so: undefined symbol: pj_get_ellps_ref
エラー: 'rgdal' に対するパッケージもしくは名前空間のロードが失敗しましたNote: proj/conus not foundの部分が気になるのですが… -- mittyo 2010-02-04 (木) 03:05:50
sui (2010-01-30 (土) 10:00:23)
いつもお世話になり、ありがとうございます。
下記につきまして長くなりますが、ご教示の程、よろしくお願いいたします。HT <- rep(0:1, c(10, 20)) cHT <- factor(HT, levels=0:1) group <- c("A","B","A","B","A","B","A","B","A","B","A", "B","A","B","A","B","A","B","A","B","A","B", "A","B","A","B","A","B","A","B") df <- data.frame(HT, cHT, group)上記のようなデータフレームを仮定します。(HT=1は高血圧あり、HT=0はなし)
列の要素が整数の場合(列:HT)は目的とするgroup毎の高血圧ありの度数とカイ2乗検定のP値を得ることが出来ました。> for(i in 1) { # 解析する列の入力 df <- df # データフレーム名の入力 g<-group # group の所にグループ変数の列名 cat(names(df)[i], by(df[, i], g, sum), (chisq.test(table(factor(df[, i], levels=0:1), df$g)))$p.value) } HT 10 10 0.6985354 # group 毎の高血圧ありの度数とカイ2乗検定の P 値最終の目標は class() 関数で列の要素を判別して if を用い、列の要素が連続変数の時は平均値と t 検定の P 値を、カテゴリカルデータであれば上記のように group 毎の高血圧ありの度数とカイ2乗検定の P 値を出力する事です。そこで初めからカテゴリカルデータに変換された cHT を用いて同様の出力を得たいと考えました。しかし、カテゴリカルデータに変換した cHT なら by(df$cHT, group, summary) とすれば HT ありの人数は分かりますが同時になしの人数まで出力されてしまいます。
カテゴリカルデータを用いて上記のように group 毎の高血圧ありのみの度数とカイ2乗検定の P 値(例数が少ないので fisher.test の方が良いかもしれませんが)を出力する方法があればご教示頂きたいと思います。よろしくお願いいたします。(R version 2.8.1)
require(Hmisc) summary( group ~ HT + cHT, data=df, method='reverse', test=T)デフォルトで continuity correction 無しの カイ2乗検定をします.連続値の場合は平均値+偏差とか,中央値と quartiles とかいろいろ設定を選べます.
> table(HT, group)[2,] A B 10 10 > table(cHT, group)[2,] A B 10 10次は,データによって検定法を選択して実行することですね。「連続変数なら t 検定」,「カテゴリー変数ならカイ二乗検定」の2通りでよいのですね。カテゴリー変数は必ず factor になっていると仮定できる。つまり,HT のように整数値で表されているカテゴリー変数は考えなくて良いのですね。上に続けて,以下の通り。
> df$x <- rnorm(30)
> head(df)
HT cHT group x
1 0 0 A -0.64007308
2 0 0 B 0.34494342
3 0 0 A -0.59577331
4 0 0 B 0.05745206
5 0 0 A -1.02755942
6 0 0 B 0.41386607
> for (i in c(2, 4)) {
+ x <- df[, i]
+ if (class(x) == "factor") {
+ tbl <- table(x, group)
+ cat(colnames(df)[i], tbl[2,], chisq.test(tbl)$p.value, "\n")
+ } else if (class(x) == "numeric") {
+ cat(t.test(x ~ group)$p.value, "\n")
+ } else {
+ stop("error")
+ }
+ }
cHT 10 10 0.6985354
0.3210783
こんなもんでしょうかね。 -- 河童の屁 2010-01-30 (土) 19:32:01森の熊太郎 (2010-01-28 (木) 15:22:19)
現在、コールセンタの利用者のリピータ状況を調べたいと思っています。
一人ひとりにユニークな番号(会員番号:重複なし)をつけています。
月に何万件と問い合わせがきます。問い合わせのたびにログを1件づつ残しており、そのデータが蓄積されています。ただ件数が多いので、Excelでは1月分を1区切りでファイルを作り管理しています。
それらのファイルをもとに、例えば1月 2月 3月 4月 X0011 X23243 X3454 X46738 X0023 X43534 X3456 X78987のように横に月、たてにログ毎の会員番号を記載した表を作り、ここから、リピータの登場の程度を調べたいと思います。
コール数 ユーザ数 累計コール数 累計ユーザー数 1月 23456 19876 23456 19876 2月 25677 20343 49133 34020のように累計ユーザ数を調べたい。
つまり、ユーザは同月内でも複数回コールをし、また翌月もコールをする傾向があり、コールセンタとして、ユーザの局在性がどれだけあるのかを調べたいと思います。
Excel2007があれば、ピボットテーブルで簡単に出来るかと思いますが、Rでもこのようなことは出来ますか?(Excel2007は業務用になく、結果としては出来ずにおり、困っております)
sui (2010-01-22 (金) 20:37:37)
初めて投稿いたします。Rは始めたばかりで初歩的な質問かもしれませんがよろしくお願いします。(R version 2.8.1)a <- c(1:10) b <- c(11:20) c <- c(21:30) d <- c(31:40) group <- c("X", "X", "X", "X", "X", "Y", "Y", "Y", "Y", "Y") df <- data.frame(a, b, c, d, group)以上のようなデータフレームを仮定します。 グループXとY間でパラメータ a, b, c, d の平均値に差があるかどうかを検定する場合(上のデータの分布は正規分布ではありませんが)、t.test(a ~ group)、t.test(b ~ group)、t.test(c ~ group)と順番に関数を実行していけば結果は得られます。しかし、実際のデータではパラメーターが50程度あるため一括して結果を得たいと考えています。どのような関数(apply family? ←変数が2つでも使用できるのでしょうか)もしくはプログラム(forを用いたプログラム?)を用いればいいのでしょうか?ご教示いただければ幸いです。
> for (i in 1:4) print(t.test(df[,i]~df$group))
Welch Two Sample t-test
data: df[, i] by df$group
t = -5, df = 8, p-value = 0.001053
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-7.306004 -2.693996
sample estimates:
mean in group X mean in group Y
3 8
以下略for (i in 1:4) {
cat("=================\n")
cat("variable:", names(df)[i], "\n")
print(t.test(df[,i]~df$group))
}T-A (2010-01-22 (金) 00:29:41)
何度もすみません。
先ほど正規乱数を母集団として標本を抽出してその平均値を出すこと何回も繰り返すコマンドを質問したものです。
母集団とその標本平均の集団の分散が等しいものがほしいのですが、どうすればよいでしょうか・・・
x <- rnorm(100)
y <- (x-mean(x))/sd(x)
w <- 5*y+50
のようにして分散が25、平均50のデータを作って
そこから標本平均の集団も分散が25に近似するように抽出することは可能なのでしょうか。
replicate(200, mean(sample(w, 10)))
のようにして母集団から標本を抽出して平均値を求めることを200回繰りかえして、そこから分散が25に近似するように100個抽出することは可能でしょうか。
よろしければご教授ください。
w <- rnorm(200, 50,sd=5) h <- replicate( 5, sample(w,10, replace=F) ) hh <- t(t(h)/apply(h,2,sd)*sd(w))
Mame (2010-01-21 (木) 16:57:38)
たびたびすみません。
下でRserveについて質問したものです。
(まだ解決に至っておりませんが、できましたら報告します)
パッケージのarulesを利用して、アソシエーション分析をしたいのですが、リスト形式のデータをトランザクション形式に使用するとエラー(voidEval failed)が出てしまいます。
参考にしているのは、金明哲先生の
http://www1.doshisha.ac.jp/~mjin/R/200611_40.pdf
です。
書いたコードは、Rconnection c=new Rconnection((args.length>0)?args[0]:"127.0.0.1"); c.voidEval( "library(arules)" ); //データを作ります RList l = new RList(); l.add( new REXP( new String[] {"パン","牛乳","ハム","果物"} ) ); l.add( new REXP( new String[] {"ソーセージ","ビール","オムツ"} ) ); l.add( new REXP( new String[] { "弁当","ビール","オムツ","タバコ"} ) ); l.add( new REXP( new String[] {"弁当","ビール","オレンジジュース","果物"} ) ); //リストの作り方がよくわからなかったので //データフレームにしてらかリストに変換しました //下記ではエラーが出ました。 //String m = "data1 <-list(c(\"パン\",\"牛乳\",\"ハム\",\"果物\"),"+ "c(\"パン\",\"オムツ\",\"ビール\",\"ハム\"),"+ "c(\"ソーセージ\",\"ビール\",\"オムツ\"),"+ "c(\"弁当\",\"ビール\",\"オムツ\",\"タバコ\"),"+ "c(\"弁当\",\"ビール\",\"オレンジジュース\",\"果物\"))"; //c.voidEval( m ); c.assign( "data1", REXP.createDataFrame(l)); c.voidEval( "data1 <- list(data1)"); //REXP x = c.eval( "class(data1)" ); //System.out.println( x ); <= listとでます。 //この下でエラー(voidEval failed)になります。 c.voidEval( "data.tran <- as(data1,\"transactions\")");以上です。
よろしければご教授のほどよろしくお願いいたします。
松田紀之 (2010-01-21 (木) 14:59:28)
PrefixSpanをRで組み始めたのですが,うまく再帰的に書けずにいます.パッケージにもなさそうです.どなたかRで動かせる実用的なプログラムをご存知でしたら教えてください.
aoi (2010-01-21 (木) 11:47:48)
たびたびすみません。よろしくお願いします。
条件に一致する文字列を取り出したいと思っています。
例えば> exex_2 [,1] [1,] "a b dxe" [2,] "abcde" [3,] "a1v2 c4" [4,] "a1 b2 c3" [5,] "banana ringo kiwi orange kodama"のような文字列の行列があったときに、スペース区切りで一番はじめの要素だけを取り出す、2番目だけの要素を取り出す、3番目の要素だけを取り出すということを行いたいと思っています。
一番はじめの要素を取り出すのは> sub(" .*","",exex_2) [,1] [1,] "a" [2,] "abcde" [3,] "a1v2" [4,] "a1" [5,] "banana"で出来たのですが、2番目、3番目の要素を取り出すことが出来ません。
目標としては2番目の要素でしたら[,1] [1,] "b" [2,] [3,] "c4" [4,] "b2" [5,] "ringo"3番目の要素でしたら
[,1] [1,] "dxe" [2,] [3,] [4,] "c3" [5,] "kiwi"のように取り出せたら、と考えています。
Rの正規表現やHelpを読ませていただいたりしてしばらく考えたのですがうまく出来なかったので、すみませんがご教授いただければと思います。
またsessionInfo()~ R version 2.8.1 (2008-12-22) i386-apple-darwin8.11.1
です。よろしくおねがいします。
> sapply(strsplit(exex_2, " "), "[", 1) [1] "a" "abcde" "a1v2" "a1" "banana" "5" > sapply(strsplit(exex_2, " "), "[", 2) [1] "b" NA "c4" "b2" "ringo" NA > sapply(strsplit(exex_2, " "), "[", 3) [1] "dxe" NA NA "c3" "kiwi" NA > sapply(strsplit(exex_2, " "), "[", 4) [1] NA NA NA NA "orange" NA > sapply(strsplit(exex_2, " "), "[", 5) [1] NA NA NA NA "kodama" NA > sapply(strsplit(exex_2, " "), "[", 6) [1] NA NA NA NA NA NA
Mame (2010-01-21 (木) 10:09:14)
はじめて投稿させていただきます。
R、RServeともに初心者ですがよろしくお願いします。
現在、RServeをつかってグラフの画像を生成したいのですが、うまくいきません。
・RServeのjava-oldの中のexsample を参考にしています。
・plot は、画像が生成され中にプロットがされていました。
・histogram は、画像そのものは生成されるのですが、白の背景だけでヒストグラムは表示されませんでした。
・Rのコマンドラインからは、histogramで画像を生成することができます。
■環境
R version 2.10.1
Rserve version 0.6.1
Eclipse version 3.5.1
OS:Windows7(windows2000でも同じ結果でした)
ご教授のほど、よろしくお願いいたします。
■以下ソースコードimport org.rosuda.JRclient.*; public class HelloWorld { public static void main(String args[]) { try { Rconnection c=new Rconnection((args.length>0)?args[0]:"127.0.0.1"); //histogramを使うために読み込みます c.voidEval( "library(lattice)" ); c.voidEval("x <- iris"); REXP xp=c.eval("try(jpeg(\"test.jpg\"))"); if (xp.asString()!=null) { System.out.println("Can't open jpeg graphics device:\n"+xp.asString()); REXP w=c.eval("if (exists(\"last.warning\") && length(last.warning)>0) names(last.warning)[1] else 0"); if (w.asString()!=null) System.out.println(w.asString()); return; } //下のplot は描画できます。 //c.voidEval("plot(Sepal.Length, Petal.Length, data=x,col=unclass(Species))"); //背景が白い画像だけが出来上がります。 c.voidEval( "histogram(~ Sepal.Length, data=x)" ); c.voidEval("dev.off()"); c.close(); } catch(RSrvException rse) { System.out.println("Rserve exception: "+rse.getMessage()); } catch(Exception e) { System.out.println("Something went wrong, but it's not the Rserve: "+ e.getMessage()); e.printStackTrace(); } } }
T-A (2010-01-21 (木) 01:49:07)
R初心者の者です。
あまりの初心者でコマンドのし方がわからないのでよろしければご教授ください。
正規乱数100個を母集団として作り、大きさ10の標本を非復元抽出で取り出し、その標本平均を求めることを100回繰り返すコマンドをするにはどうすればよいでしょうか?
x <- rnorm(100)
y <- sample(x,10)
mean(y)
ここまではたどり着けたんですが、次に進めず困っています。
こんな感じでmean(y)を100個ほしいんですが・・・
kum (2010-01-16 (土) 19:57:41)
主成分分析の結果をbiplot(PC)で表すと第1主成分が横軸、第2主成分が縦軸になりますが、それを(1,3),(2,4)といったように別の主成分で図を書くようにはできないでしょうか?
editでbiplotの中身をそれらしく変更したりしましたができませんでした。
どなたかご教授ください。
> biplot3 <- stats:::biplot.princomp > biplot3 <- edit(biplot3)として
> function (x, choices = 1L:3L,scale = 1, pc.biplot = FALSE,suffix=suffix, ...) > biplot.default(t(t(scores[, choices])/lam), t(t(x$loadings[,suffix]) * lam), ...)とbiplot.princompを変更後
> suffix <- 1:100 > biplot3(PC,suffix=suffix)としましたが [以下にエラー t(x$loadings[, suffix]) : 選択の長さは 2 でなければなりません] と出てしまいました。 -- 2010-01-16 (土) 21:37:14
choices <- c(1, 3) biplot2(PC$scores[,choices], PC$loadings[,choices], suffix=suffix)のようにすればよいのですから。biplot3 に choices 引数を追加してやれば,使いやすくなるでしょう。ついでに,biplot.princomp がやっているスケール変更も biplot3 の初期部分でやるようにしてしまえばよいでしょうね。 -- 河童の屁 2010-01-16 (土) 22:06:35
a <- princomp(iris[ ,1:4]) # 動作確認用 princomp オブジェクトを作成 以下の2図を比較してちゃんと動くことを確認 biplot(a, choices=c(1,3)) # 本来の biplot.princomp で全ケースを描画 biplot3(a, choices=c(1,3), suffix=c(23, 101, 123)) # 3 ケースだけ描画ちょっと長くなるけど -- 河童の屁 2010-01-16 (土) 23:31:05
biplot3 <- function (x, choices = 1L:2L, scale = 1, pc.biplot = FALSE, # change
var.axes = TRUE, col, cex = rep(par("cex"), 2),
xlabs = NULL, ylabs = NULL, expand = 1, xlim = NULL, ylim = NULL,
arrow.len = 0.1, main = NULL, sub = NULL, xlab = NULL, ylab = NULL,
suffix = NULL, # add 前の定義はちょっとまずかったので訂正
...)
{
# add start # biplot.princomp の部分,一部変更
if (length(choices) != 2)
stop("length of choices must be 2")
if (!length(scores <- x$scores))
stop(gettextf("object '%s' has no scores", deparse(substitute(x))),
domain = NA)
if (is.null(suffix)) # biplot.princomp に追加
suffix <- 1:nrow(scores)
loadings <- x$loadings # 変更
lam <- x$sdev[choices]
if (is.null(n <- x$n.obs))
n <- 1
lam <- lam * sqrt(n)
if (scale < 0 || scale > 1)
warning("'scale' is outside [0, 1]")
if (scale != 0)
lam <- lam^scale
else lam <- 1
if (pc.biplot)
lam <- lam/sqrt(n)
x <- t(t(scores[, choices])/lam) # biplot.default の引数 x
y <- t(t(loadings[, choices]) * lam) # biplot.default の引数 y
# add end
# これ以降は biplot.default
n <- nrow(x)
p <- nrow(y)
:
# text(x, xlabs, cex = cex[1L], col = col[1L], ...) # 以下のように変更
text(x[suffix,], xlabs[suffix], cex = cex[1L], col = col[1L], ...) # change
:G-T (2010-01-15 (金) 11:58:25)
プログラム言語をRで初めて学んでいます。
かなりの初心者なので、質問内容に至らない点があるかと思いますが、どうかご指導ください。
今回牧山先生の解説(http://www1.doshisha.ac.jp/~mjin/R/200709_50.pdf)を読みながら、地図の作成を学んでいるのですが、
その際に使われるmaptoolsパッケージの"read.shape"という関数が、
「エラー: 関数 "read.shape" を見つけることができませんでした」と表示され、何故か使うことが出来ません。
きちんとmaptoolsパッケージを呼び出してから実行していますし、maptoolsパッケージに入っている他の関数"readShapePoly"は使うことが出来ました。
これはmaptoolsパッケージが中途半端にしかインストールできていないということなのでしょうか?
それを確認・修正する方法をご教授いただければ幸いです。
※アプリケーションが古いのかと思い、再度Rをversion 2.10.1にインストールし直しましたが、同じ症状が見られます。
※念のため使用環境を掲載いたします。sessionInfo()
R version 2.10.1 (2009-12-14)
i386-pc-mingw32
locale:
[1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932
[3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C
[5] LC_TIME=Japanese_Japan.932
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] maptools_0.7-29 lattice_0.17-26 sp_0.9-47 foreign_0.8-38
loaded via a namespace (and not attached):
[1] grid_2.10.1 tools_2.10.1
library(maptools)
Map<-readShapePoly("C:/GISdata/gunma.shp")
plot(Map)aoi (2010-01-14 (木) 12:38:13)
いつも丁寧に答えてくださり本当にありがとうございます。
拙い質問で申し訳ないのですが、よろしくお願いします。
grep関数で文字列の検索を行っています。
糖鎖の構造についての文字列の検索を行っているのですが、
例えば以下のような構造だったら
Fuca1-2Galb1-4GlcNAcb1-2Mana1-3(Fuca1-2Galb1-4GlcNAcb1-2Mana1-6)Manb1-4GlcNAcb1-4GlcNAcb-Sp20
末端の構造、つまり最初のFucだけ取り出したいと思っています。
上記のような構造でしたらgrep("^Fuc",mixmix_rownames)のように出すことが出来るのですが
ただ、grepの正規表現にひっかかってしまって
例えば
a-D-Gal-Sp8
だと
Galを出したいのですがgrep("^Gal",mixmix_rownames)で出すことが出来ません。
また、
[3OSO3][6OSO3]Galb1-4[6OSO3]GlcNAcb-Sp0
のような構造でもGalを出したいのですが、出すことが出来ません。
grep("[3OSO3]",mixmix_rownames,fixed=TRUE)にすると正規表現を無視することが出来るので、文字列が一致するものを取り出せるのですが、
そうすると末端構造(最初のGal)だけを取り出すことが出来ません。
ちなみに> sessionInfo() R version 2.8.1 (2008-12-22) i386-apple-darwin8.11.1です。
よろしくお願いします。
> mixmix_rownames <- c(
+ "Fuca1-2Galb1-4GlcNAcb1-2Mana1-3(Fuca1-2Galb1-4GlcNAcb1-2Mana1-6)Manb1-4GlcNAcb1-4GlcNAcb-Sp20",
+ "a-D-Gal-Sp8",
+ "[3OSO3][6OSO3]Galb1-4[6OSO3]GlcNAcb-Sp0"
+ )
>
> grep("^([A-z]-|\\[\\w*\\])*Fuc", mixmix_rownames, perl=TRUE)
[1] 1
> grep("^([A-z]-|\\[\\w*\\])*Gal", mixmix_rownames, perl=TRUE)
[1] 2 3
>
「これでは網羅できていない」に100カノッサ -- surg 2010-01-14 (木) 14:35:58grep("^Man",mixmix_rownames,value=TRUE)
[1] "Mana1-3(Mana1-6)Manb1-4GlcNAcb1-4GlcNAcb-Sp13"
[2] "Mana1-2Mana1-2Mana1-3Mana-Sp9"
:
[19] "Mana1-3(Galb1-4GlcNAcb1-2Mana1-6)Manb1-4GlcNAcb1-4GlcNAcb-Sp12"
[20] "[3OSO3][6OSO3]Manb1-4[6OSO3]GlcNAcb-Sp0"
[21] "b-D-Man-Sp8"
この内1から19は今も出せているのですが、20と21は出せていないので、どうすれば良いのか、という問いです。よろしくお願いします。hassyy (2010-01-14 (木) 04:09:07)
長いタイトルで申し訳ありません。よろしくお願い申し上げます。
Windows版R(ver2.9.0)にRODBC_1.3-1をインストールして作業しております。
RからAccess2003で作成したmdb内のクエリの結果に接続して、計算を行っています。
下記のように記載し、問題なくクエリに接続できて、必要なデータを入手することができるところまでは確認しました。conndb <- odbcConnectAccess("Data.mdb") dat <- sqlQuery(conndb, "select * from [query1]")上記例のquery1の他に同じレイアウトのクエリが数種類あり、(query2, query3,,,,,query11)、それらについても同様に読み込み、同じ処理を行いたいと思っています。
conndb <- odbcConnectAccess("Data.mdb") list <- c(query1, query2,,,,query11) for (i in list){ dat <- sqlQuery(conndb, "select * from [i]") ** datに対する処理 ** }のような感じでソースをかけるとよいと思ったのですが、上記のような書き方では動かないようでした。
どのようにすればよろしいのでしょうか?
conndb <- odbcConnectAccess("Data.mdb")
list <- 1:11
for (i in list){
tablename<-paste("query",i,sep="")
qtext<-paste("select * from ",tablename,sep="")
dat<-sqlQuery(conndb,qtext)
#** datに対する処理 **
}kum (2010-01-13 (水) 21:52:01)
失礼かと思いますが、わからない点があるので連続で投稿させていただきます。
例えばcsvファイルに100個のデータがあるとして、biplotで表すとそのまま100個のデータが示されますが、これを特定のデータ(最初の10個、途中の20個など)だけを表示するようにはできないのでしょうか?
ご教授ください。
> ans.prcomp <- prcomp(iris[,1:4]) # 例として iris データを使うと > suffix <- c(45, 6, 78, 90, 99, 113, 145) # 描画したい行(指定の難易度はさまざま) > biplot(ans.prcomp$x[suffix,], ans.prcomp$rotation, xlabs=suffix) # xlabs を使わないと通し番号になってしまう
> PC <- princomp(~A+B+C+D+E+F+G+H, cor=TRUE,data=X) > suffix <- c(2,3,4,5,6,7,8,9,10) > biplot(PC$x[suffix,], PC$rotation, xlabs=suffix)としたのですが「エラー dimnames(x) <- list(xlabs, dimnames(x)2L) : NULL に対して属性を設定しようとしました」と出てしまいました。prcompでなくprincompを使っていることがエラーの原因でしょうか? -- kum 2010-01-13 (水) 23:27:36
> suffix <- 2:10 > biplot(PC$scores[suffix,], PC$rotation, xlabs=suffix)としたところ「以下にエラー 1L:p : 引数の長さが0です」と出てしまいました。 -- kum 2010-01-14 (木) 00:42:13
> biplot(PC$scores[suffix,], PC$loadings, xlabs=suffix)と書いたところ表示できました。熱心にご教授くださって感謝します。 ・・・しかし、「biplot(PC$scores[suffix,], PC$loadings, xlabs=suffix)」で表示される範囲が「biplot(PC)」、つまり全データを示した図の中でどこにあるかがわかるのが望ましいのですが、そうすることはできないのでしょうか。 -- kum 2010-01-14 (木) 09:13:29
まず、元のプログラムを別の名前にコピーして
biplot2 <- stats:::biplot.default
プログラムを編集する
biplot2 <- edit(biplot2)
この後、編集ウインドウで以下の2か所を追加・変更
function (x, y, var.axes = TRUE, col, cex = rep(par("cex"), 2),
xlabs = NULL, ylabs = NULL, expand = 1, xlim = NULL, ylim = NULL,
arrow.len = 0.1, main = NULL, sub = NULL, xlab = NULL, ylab = NULL,
suffix=suffix, # 追加
:
# text(x, xlabs, cex = cex[1L], col = col[1L], ...) 変更
text(x[suffix,], xlabs[suffix], cex = cex[1L], col = col[1L], ...)
編集されたプログラムを保存する。
実際に関数を呼び出すのは以下のようにする。関数名、引数の指定の仕方に注意。
biplot2(PC$scores, PC$loadings, suffix=suffix)kum (2010-01-13 (水) 18:03:45)
初めまして,kumと申します。
研究で主成分分析を行うことになったのでRを導入し,結果も得ることができました。
そこで,biplot(PC)で描かれた図を保存してレジュメに貼ろうとしたのですが保存の仕方がわかりません。
このwikiの「R による EPS 画像の作成とその LATEX への取り込み」を参考に
Y <- biplot(PC) や Y <- PC として data(Y) としたとことろ「データセット 'Y' がありません」と 出てしまいました。
どなたか解決法をご教授ください。
parade (2010-01-13 (水) 17:44:36)
潜在特性モデル・項目反応理論のパッケージltmを使って、ある集団Aの反応データから、項目パラメータを出しました(2パラメータ)。
このパラメータを使って、別の集団Bの成員の能力値を推定したいのですが、どのようにすれば良いかご存じの方いらっしゃいますでしょうか。
また、初めに求めたパラメータのうちのいくつかだけを使って、別の集団の成員の能力値を推定する、という場合にはどのようにアレンジするかについても、お教えいただければ幸いです。
どうぞよろしくお願いいたします。
gomen (2010-01-11 (月) 16:47:33)
自由評価(0〜100点)のレビューサイトで、Aさん平均点60点で100作=計6000点と、Bさん平均点80点で75作=計6000点で同じになります。
ただし、この時の各作品の平均点の上昇に対する
影響力はBさんの方がAさんより凄く高くなります。
簡単に書くと、同じ審査員なのに影響力が凄く違うと不平等なので解消したい。
でも、処理方法をマスター出来そうに無いので誰か協力してください。
目的、本当の価値が知りたい。
自由点数だと平均点60の70点(少しおもしろいに相当する)と平均点80の70点(駄作)が同じになってしまうのです。そこで0〜101位に換算すれば、それがどのぐらいの価値なのかストレートに導き出せると言う事です。
(現実の差 平均点60で100作=計6000点と平均点80で100作=計8000点、この時の差2000点を平均点60に足すと100点満点の作品数を100作中で約50作も増加可能なのです。)
以下、理論
Aさんは平均点60点なので60点の作品というのは平凡な作品に相当します。
仮に推定順位50位とします。
Bさんは平均点80点なので80点の作品というのは平凡な作品に相当します。
同じく推定順位50位にするべきと思います。
後は最高点と最低点ですが、ABどちらにしても、それが推定順位での最高位と最低位に相当します。仮に推定順位1位と推定順位100位にします。
処理方法のルール
後は点数の書かれたボールを、その0〜50位の仮想ゲージと50〜101位の仮想ゲージに並べて、接点の順位を割り出して、最後に同点数の処理をすれば推定順位に変換完了となります。
(同様の分離により、32分割する事も可能と思いますが、補正が大変なのと、点数配分が極端な審査員に有利に働くので、最大の誤差である50位=個人の平均点のみとします。)
点数の種類が少ない時(5〜8個以下と思う)は0〜101位の仮想ゲージを使う。
点数の種類が1種類の時は50位にしかならないので無効とします。
使用データー
CSVファイルから、会員IDはあるけれど会員番号が振られて無い。
作品数1万ぐらい、会員数1.5万ぐらい、総カルテ数50万ぐらい
初心者 (2010-01-10 (日) 23:14:19)
「回数」が被説明変数になるデータをポアソン回帰と負の二項回帰で分析したいと考えております。Rを使ったこれらの分析方法をお教え願えませんでしょうか。できればRコマンダーでのやり方を教えていただけると大変ありがたいです。宜しくお願いします。
勉強中 (2010-01-07 (木) 18:00:10)
よろしくお願いします。
アンケートを実施して、結果をEXCELにまとめる際に、マルチアンサーを
1つのカラムに 「2,3,5」 のようにまとめているとします。
例えばid Q1 Q2 Q3 1 2 1,3,7,9,10 1 2 3 2,5 1 3 4 3 1 4 1 4,6 1 5 2 6,7 2 6 3 10 2 7 3 3,4 2 8 4 2,5,6,7,8,9 2 9 1 1,2,4 2 10 3 5,7 2こういった際に、Rでクロス集計は可能でしょうか?
まだアンケート実施前で、できればRでやりたいのですが、集計の期限が決まっているため、事前の可・不可を知っておきたいです。
R 2.9.2
windows XP sp3
よろしくお願いします。
Q2 <- data.frame(t(sapply(a$Q2, function(s) {
val <- numeric(10)
sufix <- as.integer(unlist(strsplit(as.character(s), ",")))
val[sufix] <- 1
return(val)
})))
実行後の Q2 は以下の通り
X1 X2 X3 X4 X5 X6 X7 X8 X9 X10
1 1 0 1 0 0 0 1 0 1 1
2 0 1 0 0 1 0 0 0 0 0
3 0 0 1 0 0 0 0 0 0 0
4 0 0 0 1 0 1 0 0 0 0
5 0 0 0 0 0 1 1 0 0 0
6 0 0 0 0 0 0 0 0 0 1
7 0 0 1 1 0 0 0 0 0 0
8 0 1 0 0 1 1 1 1 1 0
9 1 1 0 1 0 0 0 0 0 0
10 0 0 0 0 1 0 1 0 0 0Tiana (2010-01-05 (火) 17:39:26)
はじめまして,留学生のTianaです.
lm関数を使って重回帰解析を行っています.
質問の便宜上,以下の回帰式を使用させていただきます.
z=c+ax+by
ただし,zは応答変数,x,yは説明変数,a,b,cは回帰係数です.
lm(z〜x+y,data=dataset1)のようにすれば,dataset1に基づいた最適解(二乗誤差を最小にするようなa,b,c)が求まりますが,データセットが変われば最適解ではなくなります.
求めたいのは,各々のデータセットの最適解ではないかもしれませんが,複数のデータセットそれぞれの二乗誤差の和が最小になるようなa,b,cです.
この問題を解決する方法はどなたかご存知でしょうか?
日本語が下手で失礼な文面はあったかもしれませんが,教えていただければとても幸いです.
よろしくお願い致します.
alldata <- rbind(dataset1, dataset2, dataset3) lm(z〜x+y,data=alldata)のようにする。
> # テストデータとして,このようなデータセットを作り
> data1 <- structure(list(x = c(47.8, 48.2, 62.6, 31.1, 43.7, 60, 38.3,
+ 48.4, 59.4, 60.4), y = c(65.5, 34.7, 57.5, 46.3, 46, 60.9, 34.9,
+ 50.3, 58.5, 45.4), z = c(62, 52.2, 57.1, 46.3, 35.1, 51.7, 31.6,
+ 48.8, 64.9, 50.3)), .Names = c("x", "y", "z"), row.names = c(NA,
+ -10L), class = "data.frame")
> #
> data2 <- structure(list(x = c(56.1, 43.3, 39.5, 52.5, 27.2, 55.2, 49.6,
+ 56.1, 62.5, 58), y = c(49.6, 46.8, 54.3, 38.4, 34.5, 66.3, 54.1,
+ 65.2, 49.9, 41), z = c(60.1, 45, 44.1, 31.2, 38.2, 60, 50.4,
+ 56.7, 64.2, 50.2)), .Names = c("x", "y", "z"), row.names = c(NA,
+ -10L), class = "data.frame")
> #
> data3 <- structure(list(x = c(50.5, 50.7, 60.8, 49.5, 46.1, 61, 27, 57.8,
+ 39.5, 57), y = c(60.4, 55.3, 58.1, 32.6, 62.3, 43.5, 33.9, 53.5,
+ 46.3, 54.1), z = c(39.7, 51.7, 62.1, 36.2, 54.5, 56.8, 31.8,
+ 53.7, 51.7, 61.8)), .Names = c("x", "y", "z"), row.names = c(NA,
+ -10L), class = "data.frame")
> #
> # 残差平方和の和を求めるために,このような関数を定義して
> func <- function(par)
+ {
+ c <- par[1]
+ a <- par[2]
+ b <- par[3]
+ return(sum(
+ (data1$z-c-a*data1$x-b*data1$y)^2+
+ (data2$z-c-a*data2$x-b*data2$y)^2+
+ (data3$z-c-a*data3$x-b*data3$y)^2
+ ))
+ }
> #
> # このように optim 関数を使えばば,解が求まります
> ans1 <- optim(c(1,1,1), func)
> options(digits=20)
> ans1$par # 順に,Intercept, a, b の値
[1] 5.1494284959399153 0.4486484068519486 0.4484184855761603
> #
> # 全部のデータを一緒にして lm をやるとちょっと違う解になります
> ans2 <- lm(z~x+y, rbind(data1, data2, data3))
> ans2$coefficients # 順に,Intercept, a, b の値
(Intercept) x y
5.1698189750573782 0.4486579549173459 0.4480422838232404
# しかし,違いが出るのは収束判定が甘いからというのが一つの原因
# 以下のようにすれば,全部のデータを一緒にして lm をやった場合の解に近づきます
> ans3 <- optim(c(1,1,1), func, control=list(reltol=1e-14))
> ans3$par
[1] 5.1698211304708774 0.4486577795757830 0.4480424597122442
図に描いてよく考えれば,別々のデータセットに同じ回帰平面をあてはめて誤差の平方和の和を最小にするのと,データセットをまとめて一つの回帰平面を当てはめたときの誤差平方和は同じものですよね。つまり,全部のデータセットを一緒にして,lm をやればそれがあなたが求めたい解になっているのだと思いますよ。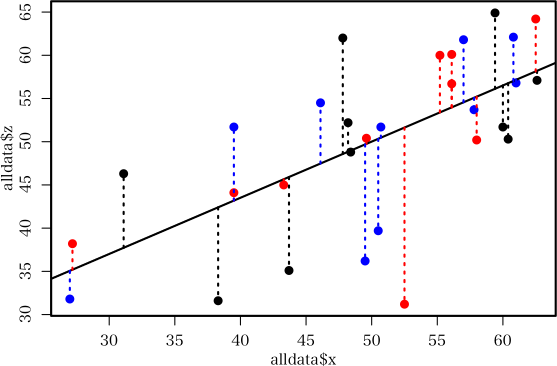
> data1 <- structure(c(60.7, 63.8, 62.8, 59.5, 50, 54.3, 50.2, 65.5, 82.9,
+ 118.2, 142.7, 101.8, 78.8, 51.7, 56.3, 90.1, 125.9, 166.8, 182.6,
+ 179, 134.6, 100.3, 69.1, 39.4, 31.2, 26.8, 26.8, 31.2, 31.2,
+ 31.2, 33.6, 36, 40.8, 47.6, 51.2, 42.7, 29.2, 32.4, 34.8, 41.6,
+ 50.8, 60.2, 67.8, 72.9, 60.4, 27.6, 34, 26.7, 21.3, 27.5, 23.3,
+ 23.8, 27.5, 25, 23.5, 27.8, 38.7, 33.9, 56.5, 21.5, 24.4, 22.9,
+ 25.2, 32.5, 29.2, 20.7, 37.1, 27.5, 19.3, 22, 14.6, 10.6), .Dim = c(24L,
+ 3L), .Dimnames = list(NULL, c("z", "x", "y")), .Tsp = c(1, 24,
+ 1), class = c("mts", "ts"))
>
> data2 <- structure(c(60.9, 43.5, 42, 55.3, 61.4, 61.4, 85.4, 113.6, 132,
+ 107.4, 79.8, 85.4, 74.2, 47.1, 66.5, 73.7, 98.7, 159.1, 154.5,
+ 128.9, 95.7, 65.5, 45, 34.8, 28.4, 28.4, 34.8, 37.6, 34.8, 34.4,
+ 45.6, 49.4, 51.2, 42.8, 46.4, 44, 29.2, 24.4, 24, 33.6, 43.2,
+ 62.7, 64.9, 40.4, 32.9, 14.8, 14, 18.4, 19.5, 18.1, 25.1, 19.2,
+ 22.4, 22.9, 20.1, 29.5, 46.2, 31.3, 33, 26.4, 23.9, 20.7, 10.8,
+ 13.7, 17.7, 34.3, 45.8, 38.2, 30.2, 20.4, 12.1, 9.1), .Dim = c(24L,
+ 3L), .Dimnames = list(NULL, c("z", "x", "y")), .Tsp = c(1, 24,
+ 1), class = c("mts", "ts"))
>
> data1
Time Series:
Start = 1
End = 24
Frequency = 1
z x y
1 60.7 31.2 21.3
2 63.8 26.8 27.5
3 62.8 26.8 23.3
略
23 69.1 34.0 14.6
24 39.4 26.7 10.6
>
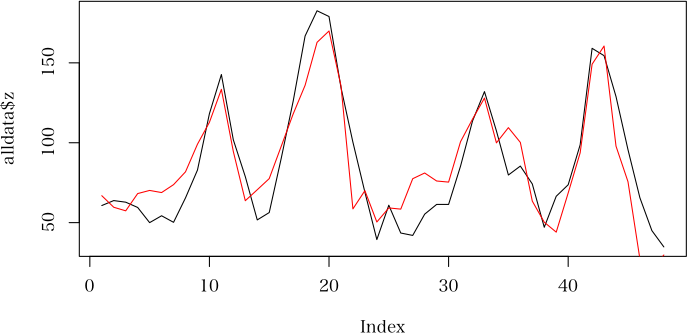
> alldata <- data.frame(rbind(data1, data2))
> ans.lm <- lm(z ~ x+y, alldata)
> plot(alldata$z, type="l")
> lines(fitted.values(ans.lm), col="red")
> summary(ans.lm)
Call:
lm(formula = z ~ x + y, data = alldata)
Residuals:
Min 1Q Median 3Q Max
-35.4932 -14.6937 0.3991 9.1139 41.6834
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -19.1196 8.7029 -2.197 0.0332 *
x 2.3930 0.2396 9.986 5.43e-13 ***
y 0.5314 0.3407 1.560 0.1259
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 18.07 on 45 degrees of freedom
Multiple R-squared: 0.7988, Adjusted R-squared: 0.7898
F-statistic: 89.32 on 2 and 45 DF, p-value: < 2.2e-16
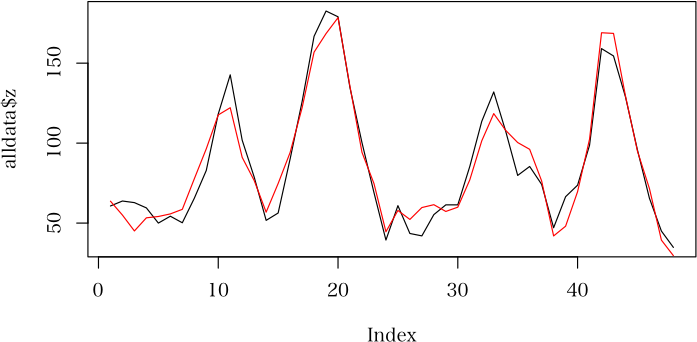
d <- data.frame(diag(24)[,-1])
colnames(d) <- paste("H", 1:23, sep="")
alldata <- rbind(data.frame(data1, d), data.frame(data2, d))
ans.lm <- lm(z ~., alldata)
plot(alldata$z, type="l")
lines(fitted.values(ans.lm), col="red")
summary(ans.lm)
結果
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -1.6278 16.9593 -0.096 0.9244
x 1.6980 0.3435 4.943 6.05e-05 ***
y 0.5798 0.4462 1.299 0.2073
H1 -4.8058 14.1831 -0.339 0.7379
H2 -12.3011 14.2281 -0.865 0.3966
H3 -11.8486 14.2191 -0.833 0.4136
H4 -13.1716 14.3102 -0.920 0.3673
H5 -10.1022 14.2485 -0.709 0.4858
H6 -10.4521 14.5318 -0.719 0.4796
H7 2.0627 15.2537 0.135 0.8937
H8 6.3584 18.0933 0.351 0.7286
H9 18.7776 16.0356 1.171 0.2541
H10 4.0706 18.9812 0.214 0.8322
H11 7.7339 14.9545 0.517 0.6102
H12 14.5447 14.2227 1.023 0.3176
H13 -9.8345 14.1444 -0.695 0.4942
H14 2.6706 14.1637 0.189 0.8522
H15 6.2902 14.4251 0.436 0.6670
H16 20.5260 15.3697 1.335 0.1954
H17 44.2918 18.1046 2.446 0.0229 *
H18 33.4838 21.1097 1.586 0.1270
H19 40.3405 17.7626 2.271 0.0333 *
H20 23.2166 15.3865 1.509 0.1456
H21 36.2390 14.4325 2.511 0.0199 *
H22 10.1858 14.6057 0.697 0.4929
H23 -5.2729 15.0943 -0.349 0.7302
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 14.12 on 22 degrees of freedom
Multiple R-squared: 0.9399, Adjusted R-squared: 0.8717
F-statistic: 13.77 on 25 and 22 DF, p-value: 1.880e-08
ろんどん (2010-01-04 (月) 13:01:14)
R 2.10.0にて対応分析をしようとしています。
エラーが出て、エラーの意味がよくわかりません。
エラーの意味と、対策方法を教えて頂きたく思います。
データは、9個の物に対して18の質問項目について答えてもらったデータです。以下の様なデータです。8 8 6 7 6 6 4 6 9 6 7 6 6 6 7 4 7 6 2 1 2 7 1 1 1 1 6 0 0 1 7 0 2 0 0 6 0 0 0 2 0 0 0 0 13 0 0 0 11 0 2 1 0 9 1 5 7 5 4 3 1 5 4 0 1 0 2 0 0 2 3 8 1 3 3 8 3 5 2 4 9 2 3 4 5 3 4 3 3 8 0 0 0 2 0 1 0 2 9 6 5 2 0 2 2 9 10 2 0 0 0 0 0 0 0 2 5 0 1 1 1 1 0 1 1 4 1 1 1 2 1 1 1 2 2 2 2 3 4 7 7 9 9 10 2 2 2 7 2 4 3 3 7 1 2 3 3 2 2 2 2 4
以下プログラムです。> library(ca) 要求されたパッケージ rgl をロード中です > w<-read.table("C:/deta1.txt") > Sdata<-matrix(0,18,9) > for(i in 1:18){Sdata[,i]<-w[,i]} 以下にエラー `[.data.frame`(w, , i) : undefined columns selected > cor<-ca(Sdata,9) > x<-matrix(0,27,18) > x[1:18,1:18]<-cor$rproj 以下にエラー x[1:18, 1:18] <- cor$rproj : 置き換えるべき項目数が,置き換える数の倍数ではありませんでした > x[19:27,1:18]<-cor$cproj 以下にエラー x[19:27, 1:18] <- cor$cproj : 置き換えるべき項目数が,置き換える数の倍数ではありませんでした > plot(x[,3],x[,4],type="n") > text(x[,3],x[,4],labels=c("A","B","C","D","E","F","G","H","I","J","K","L","M", "N","O","P","Q","R","1","2","3","4","5","6","7","8","9")) > abline(0,0,0,0)ご教授よろしくお願い致します。
library(ca)
w <- read.table("C:/deta1.txt")
Sdata <- w # 代入する意味はないと思うが
cor <- ca(Sdata, 9)
x <- rbind(cor$rowcoord, cor$colcoord)
plot(x[, 3], x[, 4], type="n")
text(x[, 3], x[, 4], labels=c(LETTERS[1:18], 1:9)) # ここはこういう風に書くほうがよい
abline(h=0, v=0) # 要するに座標軸を描きたかった?
また,ca の plotメソッドが使えるのだから,一番簡単にやりたかったら,plot(ca(w), c(3, 4)) だけで,添付のようなきれいな図が描けますよ。V1〜V9 は変数名,1〜18 は行名なので,自分でちゃんと定義すれば定義したとおりに図に描けますよ。? plot.ca でオンラインヘルプを読んでください。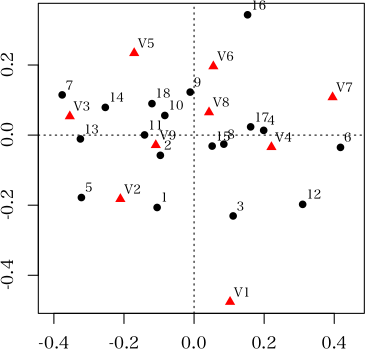
MiK (2009-12-29 (火) 09:28:09)
Rを使ったバイオインフォマティクスに関する質問です、Rの質問でないと思われた方にはごめんなさい。
IPI(International Protein Index)のデータベース内のipi番号からそのタンパク質が持つドメインを検索しようとしています。ここでは簡単にするためにipi番号からドメインデータベースのpfamの番号を返すプログラムを作ることとします。
私の知っている範囲では、BiomRtパッケージの中からgetBMを使うことができます。例えば以下のように、getBM(attributes = c("ipi","pfam"), filters = "ipi", value="IPI00883220", mart=useMart("ensembl",dataset="ggallus_gene_ensembl"))この場合、
ipi pfam IPI00883220 PF04818という値が返ってきます。これはIPI00883220がipiと同時にensembleのデータベースにも存在するから正常に働きます。
しかし、たまに正常に働かないipi番号が存在します。
例えばvalue="IPI00883261"に変えて上記のプログラムを動かすとipi pfam IPI00883261 PF01111という値が期待されますが、実際には
[1] ipi pfam <0 rows> (or 0-length row.names)と返ってきてしまいます。
これは、IPI00883261がipiデータベースには存在するが、ensembleのデータベースには存在しないため正常に働かないと考ました。というのは、BiomRtはensembleのデータベースにアクセスして値を返しているからです。
そこで、この問題を回避するために直接ipiのデータベースを参照できる方法を知りたいと思っています。それを知っている方がいたらぜひご教授ください。よろしくお願いいたします。
> set.seed(11111)
> data.base <- data.frame( # データフレームの例(1万行)
+ ipi=sprintf("IPI%08i", sample(99999999, 10000)),
+ pfam=sprintf("PF%05i", sample(99999, 10000)))
> head(data.base) # こんな風な構造になっている(する)
ipi pfam
1 IPI50144832 PF35844
2 IPI97023278 PF04626
3 IPI78760034 PF42298
4 IPI90222583 PF08148
5 IPI81417775 PF11722
6 IPI79989218 PF66366
> ipi <- c("IPI43502178", "IPI55223826", "IPI46947973",
+ "IPI98703034", "IPI49743044") # 5 個の ipi コードに対する pfam コードを得る
> lapply(ipi, function(x) data.base[data.base[,1] == x,]) # これだけでよい。。。
[[1]]
ipi pfam
7733 IPI43502178 PF34214
[[2]]
ipi pfam
3813 IPI55223826 PF36706
[[3]]
ipi pfam
1138 IPI46947973 PF39932
[[4]]
ipi pfam
6900 IPI98703034 PF00650
[[5]]
ipi pfam
1835 IPI49743044 PF48089はぜ (2009-12-22 (火) 11:56:00)
初心者です。
投稿ミス等ありましたらご容赦ください。
横軸に日付、縦軸に数量をとった折れ線グラフを作成しています。
とりあえずグラフはできたのですが、調査期間中毎日データをとっているわけではではないので、
データのない期間があると、その期間の前後の点が線で結ばれてしまい、本来データのない期間にも関わらずそこにデータがあるように見えてしまいます。
データのない期間は数量0として扱いたいのですが、どなたかいい方法をご存知でしょうか?
元データには、データのない日付は入力されていません。
OSはWindowsXP、Rヴァージョンは2.10.0です。
よろしくお願いします。
x <- structure(c(14601, 14602, 14603,
14605, 14606, 14607, 14608, 14609, 14610,
14615, 14616, 14617, 14618, 14619,
14621, 14622, 14623), class = "Date")
y <- c(23L, 24L, 44L, 25L, 13L, 12L, 11L, 13L, 29L,
45L, 30L, 42L, 39L, 38L, 31L, 13L, 19L)
plot(x, y, xaxt="n")
axis.Date(1, x, format="%m-%d")
for (i in 1:(length(x)-1)) {
if (x[i+1]-x[i] == 1) segments(x[i], y[i], x[i+1], y[i+1])
}
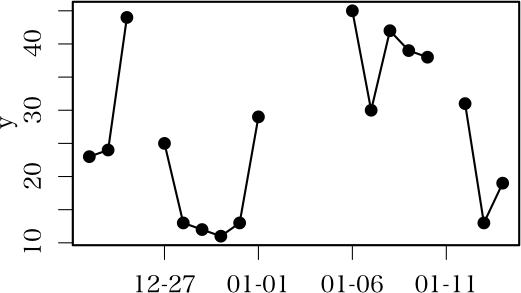
xx<-seq(min(x),max(x),"day") yy<-rep(NA,length(xx)) yy[xx%in%x]<-y plot(xx,yy,type="b") # 欠損値は線で結びません.で良いのです. -- 2009-12-22 (火) 13:01:47
ana <- d.sub[d.sub$SP== "a",]
ana.sub <- data.frame(tapply(ana$DATE, ana[, c("Date","DRCT")], length))
ana.sub[is.na(ana.sub)] <- 0
ana.sub$total <- ana.sub$d + ana.sub$u
ana.sub$DATE <- as.Date(rownames(ana.sub))
rownames(ana.sub) <- 1:nrow(ana.sub)
ana.sub$ID <- rownames(ana.sub)
cumulative.t <- c()
cum.t <- 0
for(i in 1:nrow(ana.sub)) {
d.sub <- ana.sub[ana.sub$ID == i,]
sub.t <- d.sub$total
cum.t <- cum.t + sub.t
cumulative.t <- c(cumulative.t, cum.t)
}
ana.sub$cum.t <- cumulative.t
plot(ana.sub$DATE, ana.sub$cum.t, col ="red", type ="l", lty = 2, lwd=2,
axes = FALSE,las =1,
xlim = c(as.Date("2009-08-01"), as.Date("2009-12-31")),
ylim = c(0, max(ana.sub$cum.t)*1.1),
ylab ="", main = "")
lines(ana.sub$DATE, ana.sub$total, col = "blue")
lines(ana.sub$DATE, ana.sub$d, col ="orange")
lines(ana.sub$DATE, ana.sub$u, col = "green")
axis(1, at = as.Date(v.date), labels = F, pos = 0)
axis(2, las = 1, pos = as.Date("2009-08-01")) -- [[はぜ]] &new{2009-12-22 (火) 14:10:42};> head(t) [1] "2009-08-01 05:20:30 JST" "2009-08-01 06:10:34 JST" "2009-08-01 07:23:30 JST" [4] "2009-08-01 10:50:19 JST" "2009-08-01 22:41:56 JST" "2009-08-02 07:09:01 JST"これを日付単位に何件であったか集計する。と。
d <- substr(t, 1, 10)それを table 関数で集計するだけ。では?
> table(d)
d
2009-08-01 2009-08-02 2009-08-04 2009-08-05 2009-08-08
5 6 5 4 3
以下略
あとは,先ほどのコメントにある方法で,table(d) を縦軸(頻度)として,折れ線グラフを描くだけでしょう。横軸は,names(table(d)) で取れます(観察のなかった日付は出てきません)。GYOU (2009-12-21 (月) 23:47:04)
こんにちは。
Rには行列を時計回りや反時計回りに回転する関数は無いのでしょうか?
例えば1 2 3 4という行列を時計回りに90度回転して
3 1 4 2という具合です。
過去ログを探しましたが、グラフの文字列を回転する話ばかりで、行列の回転は見当たりませんでした。自作するしか無いのでしょうか?
m <- matrix(1:16, 4, byrow=T)
n <- m
for(i in 1:ncol(m)){ n[i,] <- rev(m[,i]) }
for(i in 1:ncol(m)){ n[,i] <- rev(m[i,]) }ai (2009-12-14 (月) 23:45:42)
抽象的な質問で申し訳ありませんが,plot(ほにゃらら)とコマンドを入力すると,次の図を見るためには<Return>キーを押して下さい:と連続して表示される4つの図をepsに保存したいのですが,
par(mfrow=c(2, 2))を使っても,最初の図しか保存されません。
どうしたらすべての図,または,3番目といったひとつの図をepsに保存できますか?
x <- rnorm(100)
y <- rnorm(100)
a <- lm(y~x)
postscript("test%i.ps", width=300, height=200, onefile=FALSE)
plot(a)
dev.off()> x <- rnorm(100) > y <- rnorm(100) > a <- lm(y~x) > plot(a) ページ変更を確認するまで待機中... # Windows 版でも同じプロンプトだったと思うけど、 ページ変更を確認するまで待機中... # 変わったのだろうか ページ変更を確認するまで待機中... # スクリーン・デバイスの上に、 ページ変更を確認するまで待機中... # 「クリックまたはENTERキーを押すと次のページに移ります」と出てる > devAskNewPage() # FALSE なのに聞いてくる [1] FALSEいつから変わったのかな。 -- 河童の屁 2009-12-15 (火) 15:54:23
lemon (2009-12-14 (月) 13:44:20)
kesvmでplot(data.ksvm,data=x.cl[,1:2])の出力結果の意味が良く分かりません。
図の▲や●はデータを示すと思われるのですが、図の右側の濃淡がどのような意味を持っているのか分かりません。
説明が載っているところがあればお教え下さい。
TAK (2009-12-14 (月) 11:12:06)
R初心者です。
マザーズなどの上場企業のデータから、ダイナミックパネルデータ分析を行おうと思うのですが,以下のようなプログラミングでエラーが出てきてしまいます。何が原因なのか教えていただけないでしょうか?よろしくお願いいたします。
元データ(サービス.txt)firm yr ROE PCM 対数資産合計 対数売上高合計 流動比率 負債比率 自己資本比率 無形資産比率 ジャスダック マザーズ ヘラクレス 2003 2004 2005 2006 2007 2008 4 2005 8.14 0.826719577 2.894869657 3.480581787 158.13 146.08 40.64 0.25477707 1 0 0 0 0 1 0 0 0 4 2007 20.76 2.829175122 3.47070443 3.923088515 205.42 83.72 54.41 0.50744249 1 0 0 0 0 0 0 1 0 4 2004 37.79 3.044496487 2.85672889 3.329397879 170.66 143.73 41.09 0.139082058 1 0 0 0 1 0 0 0 0 4 2006 38.67 3.814822174 3.197556213 3.70182693 159.65 133.83 42.77 0.57106599 1 0 0 0 0 0 1 0 0 5 2008 5.2 2.394227616 3.875697762 3.484157424 111.38 421.96 15.49 0.013313806 1 0 0 0 0 0 0 0 1プログラム
library(plm) SA <- read.table("サービス.txt", header=T) SA <- plm.data(SA, index=c("firm", "yr")) attach(SA) form1 <- ROE~対数資産合計+対数売上高合計+流動比率+負債比率+自己資本比率+無形資産比率 emp.gmmSA = pgmm(dynformula(form1, lag=list(1,0,0,0,0,0,0)), data=SA, effect="twoways", model="twosteps", gmm.inst=~ROE,lag.gmm=c(2,99))表示されるエラー
以下にエラー FUN(X[[4L]], ...) : 添え字が許される範囲外です。
出口 (2009-12-10 (木) 19:32:48)
1ファイル内に47都道府県別のデータが混在しているものを、それぞれのエリア別(都道府県)に分けたく、以下のようなプログラムを記述しましたが、for構文の利用の仕方が違っているため、ファイル分割できません。
下記のプログラムですと、ToDohFuKenが47のものだけが、x.iのデータフレームとして作成されます。for ( i in 1:47) { x.i<- subset(Gr.Area, ToDohFuKen==i) # Gr.Areaは、作業するもののデータフレーム # ToDohFuKenは、Gr.Areaデータフレーム内に存在する変数名 }期待する成果としては、X1、X2、X3、・・・X47と、47のデータフレームができあがることですが、ご教授いただけないでしょうか?
(Gr.Area <- data.frame(x1=rnorm(47), x2=rnorm(47), ToDohFuKen=1:47))
(d <- split(Gr.Area, Gr.Area$ToDohFuKen))
どうしても x1, x2, ... のようにしたければ
for ( i in 1:47) {
eval(parse(text=sprintf("x%i<- subset(Gr.Area, ToDohFuKen==i)", i)))
}
一行でやるなら
eval(parse(text=sprintf("x%i<- subset(Gr.Area, ToDohFuKen==%i)", 1:47, 1:47)))迷える黒い羊 (2009-12-10 (木) 17:59:04)
たぶん初歩的な問題(データ上の問題)だとは思うのですが、manovaの計算の際、下記のエラーが出ます。
エラーの意味をご教授願えませんでしょうか?以下にエラー `contrasts<-`(`*tmp*`, value = "contr.Treatment") : 関数 "is" を見つけることができませんでした
> attach(all_lda)
> all_lda_ma<-manova(cbind(A,B)~species)
以下にエラー `contrasts<-`(`*tmp*`, value = "contr.Treatment") :
関数 "is" を見つけることができませんでしたset.seed(11111)
all_lda <- data.frame(A=rnorm(83), B=rnorm(83),
species=rep(factor(c("n", "p")), c(61, 22)))
attach(all_lda)
all_lda_ma <- manova(cbind(A, B) ~ species)
summary(all_lda_ma)
detach(all_lda)
以下のような結果が表示されると思うんですけど
Df Pillai approx F num Df den Df Pr(>F)
species 1 0.019969 0.81504 2 80 0.4463
Residuals 81> is エラー: オブジェクト 'is' がありません
R初心者 (2009-12-10 (木) 10:09:13)
R初心者です。
Rで「組合せの数」を求める関数かパッケージをご教示ください。
Excelのcombin関数に相当するものがありますでしょうか。
数学記号でnCr(n:総数,r:抜き取り数)と表示されるものです。
どうぞ,よろしくお願いします。
sakura (2009-12-10 (木) 06:32:15)
素朴な初心者質問です。具体的でなくて済みません。平均値に相乗平均を使うRのパッケージ(ライブラリー)って、あるのかなぁ??ということと、もし使うとするとどんな手順(組み込ませ方)があるのでしょう???という質問のですが・・・。よろしくお願いします。
geomean <- function(x, ...) exp(mean(log(x), ...)) 使用例 > geomean(c(2,6,3,9)) [1] 4.242641 > geomean(c(1,2,3, NA), na.rm=TRUE) [1] 1.817121
takahashi (2009-12-09 (水) 21:02:16)
初心者です。よろしくお願いします。
例えばx <- c(1:14) y <- c(-8, -13, -16, -15, -12, -6, 0, 9, 15, 33, 48, 46, 32, 24)と言った、曲線(に近いデータ)があったとします。
この曲線で、xが0から14までを20等分された値、つまりx0 = 0, x1= 0.7, x2 = 0.14, x3= 0.35・・・からx20 = 14の時のそれぞれのyの値(y0, y1, y2, y3, ・・・y20)を得るにはどうしたら良いでしょうか。
医学の循環器の研究で、各心拍の長さがことなることと、データを得る機器によって、1秒間あたりのフレーム数が異なるため、得られたデータを何等分かし、時相を一致させる必要があるため、この様な処理が必要となりました。
またデータの曲線は、関数で表せないものが多いです。
凄く単純なことに思えますが、どうしても良いプログラムができません。よろしくお願いします。
x <- 1:14
y <- c(-8, -13, -16, -15, -12, -6,
0, 9, 15, 33, 48, 46, 32, 24)
ans <- spline(x, y)
ans2 <- spline(x, y,
xout=seq(0, 14, length=21))
plot(x, y)
lines(ans$x, ans$y)
points(ans2$x, ans2$y, pch=19,
col=2, cex=0.7)
legend("bottomright",
c("観察値", "予測値"),
pch=c(1,19), col=c("black", "red"))
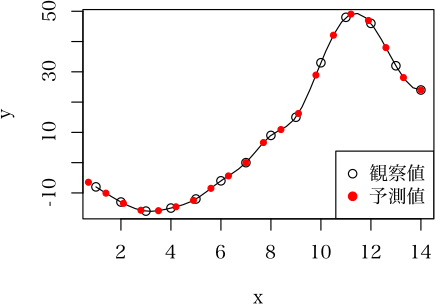
のざわ (2009-12-09 (水) 01:10:31)
お世話になります。
R-2.10.0でOSはopensolaris2009.06です。
以下のようなプログラムを作り実行させるとError in unserialize(node$con) : error reading from connectionと、こんなエラーが帰ってきて、並列計算できません。
何が悪いのでしょうか?library(snow) cl <- makeCluster(4, type = "SOCK") test.p <- function(a,b) { return((a+b)) } clusterExport(cl, "test.p")
はぜ (2009-12-08 (火) 16:32:16)
R始めたばかりです。
excelで作ったデータシートをcsv形式に変換させてread.csv("ファイル名")で読ませたのですが、なぜかデータ(数値、日付、文字列)の一部がNAになってしまいます。
元のexcelファイル、csvファイルとも調べましたが空白にしているセルはありません。
nrow("ファイル名")で行数を調べたところ、行数が実際のデータ数より多いので、データの一部がNA化するだけでなく、データが全く入力されていない末尾の空白行何行かもNAとして認識しているようです。
OSはWindowsXP、Rヴァージョンは2.10.0です。
どなたか解決策をご存知でしたらよろしくお願いします。
mako (2009-12-08 (火) 14:15:32)
いつも参考にさせていただいています。
現在、ニューラルネットパッケージnnetの関数(nnet)を用いてニューラルネット構造(重み)を学習し、その構造(重み)を用いて別の環境(fortran等)で再現したいと考えています。
nnetを用いて学習はできるのですが、出力される重みwtsの構造がわからなくて困っています。入力層が1、中間層が4、出力層が1(例えば以下の例)の場合、重みwtsは13個作成されますが、、どういう順番(どこの接続)の値なのでしょうか?(そもそもなぜ13個になる?4(入力と中間)+4(中間と出力)+1(出力?)=9では?)P <- matrix(sample(seq(-1,1,length=100), 100, replace=FALSE), ncol=1) target <- P^2 result <- nnet(P, target, size=4, maxit=100) y <- predict(result, P) plot(P,y, col="blue", pch="+") points(P,target, col="red", pch="x")また、WEKAによるニューラルネットワーク機能で描けるようなネットワーク構造を描画するパッケージがあるでしょうか?
ヘルプやリファレンス等調べましたが見つけられませんでした。
このような質問をするところではないかも知れませんが、ご存知の方がおられたら助けてください。よろしくお願いいたします。
> summary(result) a 1-4-1 network with 13 weights options were - b->h1 i1->h1 7.08 -7.27 b->h2 i1->h2 -1.15 -6.52 b->h3 i1->h3 7.54 7.06 b->h4 i1->h4 1.85 -8.71 b->o h1->o h2->o h3->o h4->o 14.84 -6.58 6.52 -9.10 -6.20 > result$wts [1] 7.081986 -7.272869 -1.148621 -6.524123 7.536072 7.062696 [7] 1.854337 -8.707560 14.843569 -6.577558 6.517180 -9.097420 [13] -6.203486
Saito (2009-12-07 (月) 21:57:38)
いつもお世話になっております。
色々と調べ、試行錯誤しましたが、上手くできないので質問させてください。
あるプログラムを走らせていて、ある時間が経過しても終了しない場合、それを終了させるにはどのようにすればよいでしょうか?
自分ではstopifnot()、proc.time()、breakを上手く使えばよいのかな、と思ったのですが、上手くいきません。例えばstopifnot(rnorm(1e+07) proc.time()[3] > 1)のような感じで、rnormに1秒以上かかるようなら、プログラムを中断させたいのですが…。ただ、proc.timeもRを起動してからの時間ですので、少し工夫がいるとは思うのですが…。
もしどなたか方法をご存知でしたら、ご教授願えないでしょうか?
> system.time(rnorm(1e8))[1] user.self 20.63くらいかかるようです。この状況で上記のコードを試すと、1秒を超えても計算が中断されません。きっちり20秒程度計算しているようです。
prog <- function()
{
s <- proc.time()[3]
repeat {
if (proc.time()[3]-s > 10) return(rnorm(100))
}
}
prog()修士論文執筆中 (2009-12-06 (日) 12:53:08)
はじめまして.修士論文執筆中と申します.
ある時からRの32bitバージョンが起動しなくなってしまいました.
私はマックブックで OS X ver.10.6.2,CPUは2.4GHz Intel Core 2 Duo,メモリは2GB,という状況で使っています.
本来ならば32bitでしか起動しないはずですし,こないだまでそうだったのですが,ある時から急に64bitでしか起動しなくなってしまいました.
Rcmdrをインストールしようにもbitがあわないからか全く起動せず非常に困っています.
ちなみにR64bitからは次のようなデータを得られました.
sessionInfo()
R version 2.10.0 (2009-10-26)
x86_64-apple-darwin9.8.0
locale:
[1] ja_JP.UTF-8/ja_JP.UTF-8/C/C/ja_JP.UTF-8/ja_JP.UTF-8
attached base packages:
[1] stats graphics grDevices utils datasets methods base
どうすれば32bitで起動するようになるでしょうか?
アドバイスをお願いいたします.
Mac OS X GUI Version Build Download Mac OS X GUI rev. 5528 for R 2.10.x tiger-Deployment R-GUI-5528-2.10-tiger-Deployment.dmg Mac OS X GUI rev. 5528 for R 2.11.x tiger-Deployment R-GUI-5528-2.11-tiger-Deployment.dmg Mac OS X GUI rev. 5528 for R 2.10.x leopard-Leopard R-GUI-5528-2.10-leopard-Leopard.dmg Mac OS X GUI rev. 5528 for R 2.10.x leopard-Leopard64 R-GUI-5528-2.10-leopard-Leopard64.dmg Mac OS X GUI rev. 5528 for R 2.11.x leopard-Leopard R-GUI-5528-2.11-leopard-Leopard.dmg Mac OS X GUI rev. 5528 for R 2.11.x leopard-Leopard64 R-GUI-5528-2.11-leopard-Leopard64.dmg
悩みの多い熊五郎 (2009-12-05 (土) 21:51:34)
修理受付データを分析して、解約を減らす方法を検討しています。修理には多くの部品が使われております。高額でもお客様は納得されれば修理しますし、安価でも解約になるケースがあり、どの部品が修理部材に使われると解約につながるのかを統計的に調べたいと思っています。Rをつかって、どの部品が交換されると解約される可能性が高いなのでの分析をすることが出来るのでしょうか?交換する部品は1個以上で、修理案件ごとに個数が異なります。
データとしては以下のようなデータが集計されています。修理受付No 部品1 部品2 部品3 部品4 部品5 修理価格 修理解約 001 PartA PartB 1000 修理 002 PartA PartZ PartC 4500 修理 003 PartV PartA PartB 3000 解約 004 PartD PartS PartB 6000 解約 ・・・・・・・・・・・・・・・・・・・・・・・・・・・・・・・・・ 999 PartZ 8000 修理この調査をし対策をしたいと考えております。アドバイスをお願いします。
修理受付No PartsA PartsB PartsC ... PartsZ 修理価格 修理解約 001 1 1 0 ... 0 1000 修理 002 1 0 1 ... 1 4500 修理 003 1 1 0 ... 0 3000 解約 004 0 1 0 ... 0 6000 解約 : 999 0 0 0 ... 1 8000 修理また,修理か解約かはどのパーツが使われたかだけに依存しているとも思えません(修理日数とか担当者の親切さとかなんとかにも関係する)ので,そのあたりのデータも集めておく方が良いでしょう。
sibu (2009-12-03 (木) 17:11:21)
こんにちは、私はネットワーク分析でテキストマイニングを行っているものなのですが。
テキストデータを形態素解析し、MLTPを用いて名詞に限定してbigramを求め、その結果を整形しネットワークマップ図にしようとこころ見たのですが。> at<-read.csv("c:/temp/a.csv",head=FALSE) Warning message: In read.table(file = file, header = header, sep = sep, quote = quote, : 'c:/temp/a.csv' の readTableHeader で不完全な最終行が見つかりましたというエラーが出てしまい、データを出力しようとしても
> at[1:30,] [1] ミマ\021爍ア <NA> <NA> <NA> <NA> <NA> <NA> [8] <NA> <NA> <NA> <NA> <NA> <NA> <NA> [15] <NA> <NA> <NA> <NA> <NA> <NA> <NA> [22] <NA> <NA> <NA> <NA> <NA> <NA> <NA> [29] <NA> <NA>という風になり、データを解析できません
なぜこのようなエラーが出るのでしょうか? ご意見お聞かせください、お願いします。
Saito (2009-12-01 (火) 17:53:41)
いつもお世話になっています。
過去の投稿、ネットを確認しましたが、見当たらなかったので質問させてください。
例えばlibrary(lattice) contourplot(volcano)で表示される四方の軸の目盛りを内側表示したいのですが、どのようにすればよいでしょうか。
plotであれば、plot(1, 1, tcl=0.5)とでもすればできるのですが。contourplotがやっている描画方法がよくわからず、うまく解決できませんでした。どなたか方法をご存知の方がいましたらご教授願えないでしょうか。
ちなみにR-2.9.2、WindowsXPです。
どうぞよろしくお願い致します。
おむすび (2009-11-30 (月) 21:26:10)
行列をつくって,それぞれの値を順番に置き換えたいのですが,どうしたらいいでしょう。m<-matrix(c(2, 5, 1, 5, 10, 2, 10, 20, 5),nrow=3)たとえば,rankでやると,
m2<-matrix(rank(m),nrow=3)これを1から7にしたいのです。
ついでに,orderだと順番が違うのはなぜですか?
> m2
[,1] [,2] [,3]
[1,] 2 3 4
[2,] 3 4 5
[3,] 1 2 3
1から5でした。すみません。 -- おむすび 2009-11-30 (月) 22:07:31> order(c(4,2,1,5,3)) [1] 3 2 5 1 4という例を考察すればよいでしょう。一番小さい1は3番目,2番目に小さい2は2番目,3番目に小さい3は5番目,4番目に小さい4は1番目,5番目に小さい(一番大きい)5は4番目ということ。
> x <- c(1,2,2,3,3,3,3,4,5) > rank(x, ties.method="average") # これが,デフォルト(平均順位) [1] 1.0 2.5 2.5 5.5 5.5 5.5 5.5 8.0 9.0 > rank(x, ties.method="first") [1] 1 2 3 4 5 6 7 8 9 > rank(x, ties.method="random") [1] 1 2 3 4 7 5 6 8 9 > rank(x, ties.method="max") [1] 1 3 3 7 7 7 7 8 9 > rank(x, ties.method="min") # これが世間でよく行われる方法? [1] 1 2 2 4 4 4 4 8 9とにかく,オンラインヘルプをよくお読みになることをおすすめします。
> m2
[,1] [,2] [,3]
[1,] 2 3 4
[2,] 3 4 5
[3,] 1 2 3
こんなのになりませんか? -- おむすび 2009-11-30 (月) 22:29:53> m <- matrix(c(2, 5, 1, 5, 10, 2, 10, 20, 5), nrow=3)
> values <- as.numeric(names(table(m)))
> m2 <- m
> for (i in 1:length(values)) {
+ m2[m2 == values[i]] <- i
+ }
> m2
[,1] [,2] [,3]
[1,] 2 3 4
[2,] 3 4 5
[3,] 1 2 3kobayashi (2009-11-29 (日) 22:19:04)
以下のようにデータを作り、エクセルのシリアル値であるDay(今回は例として適当に定義)を日付データ(newDay)にしてx軸をプロットします。Day <- c(33000, 36000, 39000) Value1 <- 1:3 Value2 <- 4:6 x <- data.frame(Value1, Value2) newDay <- as.Date(Day, origin="1900/01/01") plot(newDay, x[, 1], type="l") matplot(newDay, x[, 1], type="l") > x Value1 Value2 1 1 4 2 2 5 3 3 6 > newDay [1] "1990-05-09" "1998-07-26" "2006-10-12"すると、plotではx軸は日付データになってくれるのですが、matplotではx軸が変な値になってしまいます。(変換前のシリアル値にもなっていません)
原因及び解決方法を教えていただけないでしょうか。
matplot(newDay, x[, 1], type="l", xaxt="n") axis(1, at=newDay, label=as.Date(newDay, origin="1970/01/01")) # 単に,axis(1, at=newDay, label=newDay) でよいようだ。。。
sisisi (2009-11-25 (水) 10:20:58)
始めまして、私はテキストマイニングについて勉強しているものです。
最近は大学の講義データの講師の音声データと授業のスライドデータをテキスト化したものをテキストマイニングしようとしていますが、なかなか思うようにデータが集まりません。
このような異なる二つのデータをテキストを分析するときはどのような分析方法がよいのでしょうか?
どうかご意見お聞かせ下さい。
kao (2009-11-24 (火) 15:00:05)
Rで、soft independent modeling of class analogy(SIMCA)分析を行いたいです。
パッケージがあるのであれば、教えていただけないでしょうか。
よろしくお願いします。
tsuyoshi (2009-11-21 (土) 18:25:42)
初心者です
Rで、トランスログ生産関数分析をしたいです。
どなたかご存知でしたら教えていただけると幸いです。
柳沢 (2009-11-20 (金) 03:16:30)
こんにちは.windows で R 2.9.0 を使っています.
例えば plot(c(1,22)) を描いたとします.そのとき y-axis の 5, 10, 15, 20 のところに tick mark が現れます. この tick mark を傾けることは可能ですか? それが「良いグラフィックス」かどうかは別問題としてください.
plot(c(1,22), yaxt='n') text(rep(1,length(z<-axTicks(2))), z, '|', srt=-45)
が一番求めるものに近いのですが,xaxs='i' としない場合, 上記の text の中の x軸の値がいくつになるのかが分かりません.
どうぞよろしくお願いします.
plot(c(1,22), yaxt='n')
axis(2,tick=F)
par(xpd=T)
text(rep(par()$usr[1]-strwidth("|"),length(axTicks(2))),axTicks(2),"|",srt=45)
par(xpd=F)
こんな感じは参考になるかも -- 2009-11-20 (金) 11:15:52R初心者 (2009-11-18 (水) 00:58:33)
as.Date("2009-11-1")の1ヶ月後や2ヶ月後をRで計算できないものでしょうか?
1日後だったら、as.Date("2009-11-1")+1で簡単に計算できるのですが、1ヶ月後は
tail(seq(as.Date("2008-1-1"), len=2, by="1 month"), 1)
しか思いつかなかったので、質問させていただきました。
この方法だと、as.Dateの部分が日付のベクトルだとうまくいかなくなります。
何か良い方法があれば教えていただければと存じます。
> test <- function(str)
+ {
+ str2 <- tail(seq(as.Date(str), len=2, by="1 month"), 1)
+ cat(sprintf("%s %i\n", str2, difftime(as.Date(str2), as.Date(str))))
+ }
> test("2009-01-01")
2009-02-01 31
> test("2009-01-31") # 一ヶ月後は31日後になっている
2009-03-03 31
> test("2009-02-01") # 一ヶ月後は28日後になっており,
2009-03-01 28 # 2月になっての一ヶ月後は1月最終日からの一ヶ月後より早い
日付にベクトルを与えられないというのは本質的なものではなくて,以下のように対処すればよいだけでしょう。
> func <- function(str)
+ {
+ sapply(str, function(str2) as.character(seq(as.Date(str2), len=2, by="1 month")[2]))
+ }
> func(c("2009-01-01", "2009-01-31", "2009-02-01"))
2009-01-01 2009-01-31 2009-02-01
"2009-02-01" "2009-03-03" "2009-03-01"
あるいは,as.POSIXct を使うと,引数がベクトルの場合でも指定日数後の日付を得ることができます。
> as.POSIXct(c("2009-01-01", "2009-02-01"))+60*60*24*30
[1] "2009-01-31 JST" "2009-03-03 JST"aoi (2009-11-17 (火) 17:35:07)
いつも丁寧に教えてくださり、本当にありがとうございます。
実験データをもとにしてヒートマップとデンドログラムを書いています。大変単純な質問で恐縮なのですが、距離をユークリッドやマンハッタンではなくコサイン距離で書いてみたいと思っていて、一応は書くことができたのですが、デンドログラムが細かく分かれすぎてしまい、分岐の最後までpdfにしても書くことが出来ません。(heatmapにしてもhclustでplclustにしても同様です。)
heatmap 関数のmargins=c(5,5)も変更してみたり、pdfのサイズを大きくしたりしているのですがうまくいきません。もっと最後の分岐まで書きたいです(現状では長過ぎて裏返ってしまっています)良い方法がありましたら、教えていただければと思います。
ちなみにコサイン距離の関数は
http://rbloger.blog51.fc2.com/blog-entry-26.html
より利用させていただきcosine.function <- function(x) { # 列ベクトルのコサイン距離の計算 col.similarity <- matrix(NA, ncol=ncol(x), nrow=ncol(x)) # コサイン距離を格納する行列 for (i in 1:ncol(x)) { for (j in 1:ncol(x)) { # コサイン距離の算出 col.similarity[i, j] <- (x[, i] %*% x[, j])/(sqrt(sum(x[, i]^2))*sqrt(sum(x[, j]^2))) } } rownames(col.similarity) <- colnames(x) # 列名の付与 colnames(col.similarity) <- colnames(x) # 行名の付与 return(col.similarity) # コサイン距離の行列を返す }転置してheatmapの関数に組み込むようにheatmap関数を変更しています。
また> sessionInfo() R version 2.8.1 (2008-12-22) i386-apple-darwin8.11.1です。よろしくお願いします。
Answer1 Answer2 Answer3 A 1 2 3 B 5 10 15 C 89 135 78 D 26 49 42 E 36 70 30 F 167 216 144 G 125 143 89 H 145 229 1110 I 13 5 11 J 58 61 49 K 77 91 25といったようなデータでこれでA,B,C・・・に当たる物が糖鎖で Answer1,Answer2・・・に当たる物がサンプルでこれが400行、300列くらいあります。
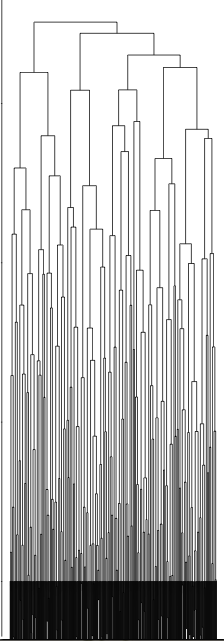
set.seed(123456789)
x <- matrix(sample(0:1, 400*300, replace=TRUE), 400) # 400行,300列のデータファイル
pdf("dendrogram.pdf", width=150, height=6) # width, height を適当に設定(詰まっていれば大きく)
plot(hclust(as.dist(cosine.function(x))), hang=-1)
dev.off()
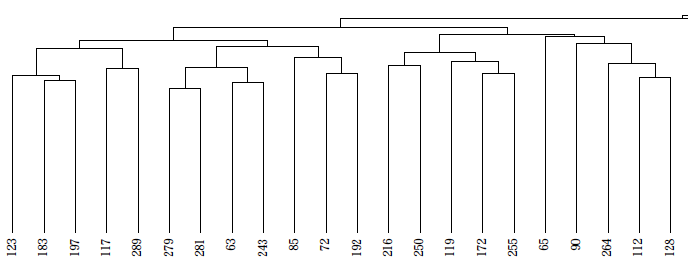
x <- read.table("mixmix.txt", header=TRUE, sep="\t")
pdf("dendrogram2.pdf", width=100, height=5)
plot(hclust(as.dist(cosine.function(t(x[,-1])))), hang=-1)
dev.off()
なお,蛇足ではあるが,ブログから参照したという関数は無駄が多いというか稚拙なプログラムなので,以下のようにした方が300倍ほど速い。
> cosine.function2 <- function(x)
+ {
+ x <- as.matrix(x)
+ ss <- 1/sqrt(colSums(x^2))
+ col.similarity <- t(x) %*% x*outer(ss, ss)
+ colnames(col.similarity) <- rownames(col.similarity) <- colnames(x)
+ return(col.similarity)
+ }
> system.time(a1 <- cosine.function(t(x[,-1]))) # 二重の for ループを使っているので遅い!
ユーザ システム 経過
14.962 0.070 14.894
> system.time(a2 <- cosine.function2(t(x[,-1]))) # 圧倒的な速度
ユーザ システム 経過
0.046 0.002 0.048
> all.equal(a1, a2) # 言うまでもないが,同じ結果
[1] TRUEb.b. (2009-11-17 (火) 14:49:43)
Height ~ SI * (1 + exp (8.285 -1.22 * log(60 + 16.195) - log(SI))) / (1 + exp(8.285 -1.22 * log(Age + 16.195) - log(SI)))という非線形関数で、AgeとHeightが分かっている場合に、SIの値を求める方法を知りたいのですが、どのようにすればよいのでしょうか。
> root <- function(Height, Age) {
+ uniroot(function(SI) SI * (1 + exp(8.285 -1.22 * log(60 + 16.195) - log(SI))) /
+ (1 + exp(8.285 -1.22 * log(Age + 16.195) - log(SI))) -
+ Height, c(0.01, 1e10))
+ }
> root(8.889316, 50)
$root # これが答え
[1] 10
$f.root
[1] -1.202766e-08
$iter
[1] 5
$estim.prec
[1] 6.103516e-05
Height, Age, SI のうちの任意の 2 変数の値を与えれば,残りの 1 変数の値を求めることができるようにするには,stats パッケージの power.t.test などに参考になるプログラム例があります。 -- 今日は寒いぞ河童の屁 2009-11-17 (火) 16:01:29
> root <- function(Height=NULL, Age=NULL, SI=NULL) {
+ Height.function <- quote(SI * (1 + exp(8.285 -1.22 * log(60 + 16.195) - log(SI))) /
+ (1 + exp(8.285 -1.22 * log(Age + 16.195) - log(SI))))
+ if (sum(sapply(list(Height, Age, SI), is.null)) != 1) stop("NULL は 1 変数のみ")
+ if (is.null(Height)) {
+ Height <- eval(Height.function)
+ }
+ else if (is.null(Age)) {
+ Age <- uniroot(function(Age) eval(Height.function)-Height, c(1, 1000))$root
+ }
+ else if (is.null(SI)) {
+ SI <- uniroot(function(SI) eval(Height.function)-Height, c(0.01, 1e10))$root
+ }
+ return(c(Height=Height, Age=Age, SI=SI))
+ }
> root(Height=8.889316, Age=50) # SI を求める
Height Age SI
8.889316 50.000000 10.000000
> root(SI=10, Age=50) # Height を求める
Height Age SI
8.889316 50.000000 10.000000
> root(Height=8.889316, SI=10) # Age を求める
Height Age SI
8.889316 50.000013 10.000000 akira (2009-11-16 (月) 21:34:21)
library(survival)のcoxphで作成したモデルを使ってpredict.coxphで生存期間を予測したいと思うのですが、type="expected"を出力する方法が分からず、困っています.基本的なことなのでしょうが、教えていただけるとうれしいです> fit <- coxph(Surv(time, status) ~ age + ph.ecog + strata(inst), lung) #このモデルを使って > head(lung) inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss 1 3 306 2 74 1 1 90 100 1175 NA 2 3 455 2 68 1 0 90 90 1225 15 3 3 1010 1 56 1 0 90 90 NA 15 4 5 210 2 57 1 1 90 60 1150 11 5 1 883 2 60 1 0 100 90 NA 0 6 12 1022 1 74 1 1 50 80 513 0 > predict(fit,type="lp") #これは線形回帰の係数と思ってます > predict(fit,type="expected") #これはモデルに使ったデータを回帰した生存期間ですよね? > predict(fit,newdata=lung[1,]) #これはtype="lp"を返しますが... # predict(fit,newdata=lung[1,],type="expected") #生存期間は返してくれないtype="lp"からリスクスコアは出せると思うのですが、log h(t) = a + b1x1 + b2x2 + ... + bnxn の時間tは出せないものでしょうか?
そもそも、理解を間違っているのでしょうか?
type: the type of predicted value.
Choices are the linear predictor ("lp"),
the risk score exp(lp) ("risk"),
the expected number of events given the covariates and follow-up time ("expected"),
and the terms of the linear predictor ("terms").# lungのinst==1だけで動作を理解してみました.
x <- subset(lung,subset=inst==1,select=c("time","status","age","ph.ecog"))
cxfit <- coxph(Surv(time, status) ~ age + ph.ecog,x) # Coxモデルを作る
all(exp(predict(cxfit,type="lp")) == predict(cxfit,type="risk")) # log(lp) = riskのこと
predict(cxfit,type="terms") # log h(t) = a + b1x1 + b2x2 の b1x1 と b2x2 の項
predict(cxfit,type="expected") # ここの返り値で思考が止まってしまいます.
ヘルプの「the expected number of events given the covariates and follow-up time (‘"expected"’)」を見てtype="exprected"はeventの数は時間tにおけるstatusを返すと思い込んでいました… ← ここが分かっていない# で、predict.coxphで1つ目のデータを推定してみました. y <- x[1,] predict(cxfit,y,type="lp"); predict(cxfit,type="lp")[1] #ほぼ同じ値 # predict(cxfit,y,type="risk"); predict(cxfit,type="risk")[1] #これもほぼ同じ値 predict(cxfit,y,type="terms"); predict(cxfit,type="terms")[1,] #これもほぼ同じ値 # predict.coxphはlp, risk, termをそれぞれ推定することは理解できましたCox比例ハザードはlog h(t) = a + b1x1 + b2x2 + ... + bnxn だから時間tに依存せずにlog h(t)が出るんですよね…そうすると、生存期間を臨床情報で多重回帰した式から生存期間は求められないような気がしてきました…どうでしょうか?-- akira 2009-11-17 (火) 09:20:11
library(survival)
x <- subset(lung,subset=inst==1,select=c("time","status","age","ph.ecog"))
y <- x[1,]
cxfit <- coxph(Surv(time, status==1) ~ age + ph.ecog,x) # Coxモデルを作る
predict(cxfit,y,type="risk") # yのstatusが「1」となるリスク
5
1.027764
y$status
[1] 2
#「predict(cxfit,y,type="risk")が1より大きいが、y$statusは「2」なのでpredictと一致しない」
と思っていたのですが -- akira 2009-11-17 (火) 21:44:32sibu (2009-11-16 (月) 15:01:39)
私はネットワーク分析でテキストマイニングを行おうとしているのですがー structure(.External("dotTclObjv", objv, PACKAGE = "tcltk"), class = "tclObj") : [tcl] bad screen distance "R_call 01C0BFF8 1.38998e+213raph".というエラーが出て分析することができないのです。
Rについては初心者でこのエラーが何を意味するかわかりません
どうかこのエラーの意味を教えてください。
ちなみに今回のネットワーク分析ではigraphとtcltkを使っています。
sakura (2009-11-15 (日) 18:37:09)
epiRにあるcmh.testとmantelhaen.test(stats)では微妙に結果が違うのは何故でしょうか?
cmh.test {lawstat} R Documentation
The Cochran-Mantel-Haenszel Chi-square Test
Description
This function performs the Cochran-Mantel-Haenszel (CMH) procedure. The CMH
procedure tests independence of two variables in each stratum, conditionally
on the levels of a third variable. This procedure is widely used in various
law cases, in particular, on equal employment and discrimination, as well in
biopharmaceutical studies (Agresti, 2002; Gastwirth, 1984; Hall et al., 1999).
mantelhaen.test {stats} R Documentation
Cochran-Mantel-Haenszel Chi-Squared Test for Count Data
Description
Performs a Cochran-Mantel-Haenszel chi-squared test of the null that two
nominal variables are conditionally independent in each stratum, assuming that
there is no three-way interaction.
単に,オンヘルプをコピーしただけの河童でした。> cmh.test(blackhire
Cochran-Mantel-Haenszel Chi-square Test
data: blackhire
CMH statistic = 145.840, df = 1.000, p-value = 0.000, MH Estimate =
0.477, Pooled Odd Ratio = 0.639, Odd Ratio of level 1 = 1.329, Odd
Ratio of level 2 = 0.378, Odd Ratio of level 3 = 0.508, Odd Ratio of
level 4 = 0.357, Odd Ratio of level 5 = 0.209, Odd Ratio of level 6 =
0.412, Odd Ratio of level 7 = 0.250, Odd Ratio of level 8 = 0.820
> mantelhaen.test(blackhire)
Mantel-Haenszel chi-squared test with continuity correction
data: blackhire
Mantel-Haenszel X-squared = 145.2466, df = 1, p-value < 2.2e-16
alternative hypothesis: true common odds ratio is not equal to 1
95 percent confidence interval:
0.4225550 0.5395037
sample estimates:
common odds ratio
0.4774621
カイ二乗値が 145.84 と 145.2466 の違いなんですね??たいへんだあ> mantelhaen.test(blackhire,correct=FALSE)
Mantel-Haenszel chi-squared test without continuity correction
data: blackhire
Mantel-Haenszel X-squared = 145.8403, df = 1, p-value < 2.2e-16
alternative hypothesis: true common odds ratio is not equal to 1
95 percent confidence interval:
0.4225550 0.5395037
sample estimates:
common odds ratio
0.4774621
このような確認は,必要だと思う人(ユーザ)が自分でやることかなあ。どこかに書いておいてくれればよいことでしょうけど。そのようなものがないときは,こんな風にすれば,確認できるでしょうという筋道の例示としては存在意義があるのかなあ? -- 推測力に乏しい河童の屁 2009-11-15 (日) 19:47:59きむ (2009-11-13 (金) 19:14:43)
少しづつRを使い始めています。
過去のQ&AやTipsを見たのですが、見つけきれませんでしたので、教えてください。
以下の様な2つのデータフレームがある場合、DB1(の行名)のうち、DB2(の行名)に含まれるものだけ抽出して新しいデータフレームDB3を作成するにはどのようにすれば良いでしょうか?
(例の場合、a,c,eだけのデータフレームにしたい)DB1 <- as.data.frame(matrix(1:10,ncol=2)) rownames(DB1) <- letters[1:5] DB2 <- as.data.frame(matrix(101:110,ncol=2)) rownames(DB2) <- letters[c(1,3,5,7,9)]行名ではなく、要素であれば参照する方法はQ&Aで確認できたのですが、、行名の引用方法がわかりません。行名を要素に追加してからハンドリングすれば良いのかも知れませんが、行名を直接引用する方法があれば教えてください。
(ついでに、)同様に、行名でDB1に、DB2の値をリスト参照・追加して、DB3を作成することは可能でしょうか?
(DB1のa,c,eにDB2の要素値を追加する:EXCELのVLOOKUPのイメージ)
以上、
かなり基本的な質問かと思いますが、よろしくお願いいたします。
> DB1 V1 V2 a 1 6 b 2 7 c 3 8 d 4 9 e 5 10 > DB2 W1 W2 c 102 107 g 104 109 i 105 110 a 101 106 e 103 108 > db1 <- data.frame(ID=rownames(DB1), DB1) > db2 <- data.frame(ID=rownames(DB2), DB2) > db3 <- merge(db1, db2) > rownames(db3) <- db3[,1] > db3 <- db3[-1] > db3 # 正しい答えは,これです V1 V2 W1 W2 a 1 6 101 106 c 3 8 102 107 e 5 10 103 108
motok (2009-11-13 (金) 14:37:25)
あるイベントが発生した時刻を "2009.11.13 14:32" の形で1行に1個づつ記録したデータファイルがあります。同じ時刻(1分以内)にイベントが発生すると、その時刻が2個3個と記録されます。
このデータファイルから例えば5分刻みのイベント発生頻度を数えてヒストグラムにしたいのですが、ちょっと手も足も出ていません。mydata <- read.table("test.dat", sep=",") names(mydata) <- c("event") attach(mydata) x <- strptime(event, format="%Y.%m.%d %H:%M")として文字列を時刻オブジェクト(?)にしたところで立ち往生しています。
なにかヒントを頂けるととてもうれしいです。
# FAQでないことをいのりつつ。
as.POSIXct("2009-11-13 12:45:35 JST") + sample(10, 5, replace=TRUE)
のようにするほうが,再現性を保証するためにはよいだろうけど。 -- 河童の合羽も雨に濡れ 2009-11-13 (金) 16:30:22
> x <- sort(Sys.time()-sample(3600, 2000)-10000, replace=TRUE) > head(x) # 以下のようなフォーマットです(このほかにも許容されるフォーマットがある) [1] "2009-11-13 12:45:35 JST" "2009-11-13 12:45:36 JST" "2009-11-13 12:45:38 JST" [4] "2009-11-13 12:45:39 JST" "2009-11-13 12:45:41 JST" "2009-11-13 12:45:43 JST" > y <- difftime(x[-1], x[-2000], units="secs") # 時間の差をとる(単位は sec で) > head(y) Time differences in secs [1] 1 2 1 2 2 1 > hist(y, breaks=c(seq(0, 20, by=1))) # 階級幅はお好きなように
主成分分析初心者 (2009-11-10 (火) 17:03:08)
Rコマンダーを使用し、主成分分析を行ってます。手順は、コマンダーで、エクセルを読み込んで、主成分分析を行い、寄与率をエクセルにコピペしている初心者です。
変数が多くなり(40変数)どれが、有効な変数なのか、苦戦しております。
現在は、コマンダーで出た、Dataset <- sqlQuery(channel = 1, select * from [Sheet3$]) .PC <- princomp(~変数1+変数2+・・・・+変数40, cor=TRUE, data=Dataset) unclass(loadings(.PC)) # component loadings .PC$sd^2 # component variances summary(.PC) # proportions of variance remove(.PC)をもとに、エディターで、
princomp(~○○, cor=TRUE, data=Dataset)○○部分を書き換えやっていましたが、40変数より39、38・・・とやっていこうとすると膨大なパターンを行わなくてはいけず、困っています。
出来ましたら、組み合わせ的に、40変数より、変数39の時の計算方法39通りでの、第一主成分の寄与率が、保存できる方法がありましたら、どなたか教えて頂きたく、思います。よろしくお願いいたします。
set.seed(123456789) # 毎回同じデータが得られるように,乱数の初期値を設定
d <- matrix(rnorm(4000), 100) # 100x40のデータ行列(データフレームでもよい)
r <- cor(d) # すべての変数を使って相関係数行列を計算しておく
sapply(1:40, # 1 番目から 40 番目の変数に対して順に以下を行う
function(i) {
r2 <- r[-i, -i] # i 番目の変数を除いたときの相関係数行列
v <- eigen(r2)$value # 固有値ベクトルを v に付値
return(v[1]/39) # 1 番目の固有値を 39 で割ったものが,第 1 主成分の寄与率
}
)
答えは以下のようになります
[1] 0.06285943 0.06326298 0.06333256 0.06192062 0.06229890 0.06145829
中略
[37] 0.06249882 0.06298219 0.06305921 0.06190447
なお,このプログラムは,元の 40 個の変数が欠損値を含まないことを前提にしています。欠損値を含むときには相関係数行列を求めるときに,欠損値を含まないデータだけを使って相関係数行列を求めるようにしてください。set.seed(987654321) # 乱数初期化
nv <- 10 # 変数の個数(ほどほどにしないと,死んでも終わらない)
d <- matrix(rnorm(100*nv), 100) # 100xnv データ行列
r <- cor(d) # 元の相関係数行列
var.no <- paste("V", 1:nv, sep="") # 変数番号(どの変数が使われた分析か示すために)
n <- 2^nv # 組み合わせの数
bincomb <- matrix(FALSE, nrow=n, ncol=nv) # 組み合わせの生成
for (j in 1:nv) {
bincomb[, j] <- rep(c(rep(FALSE, n/2^j), rep(TRUE, n/2^j)), length = n)
}
bincomb <- bincomb[-1,] # どの変数も使われない場合を除く
n <- n-1
result <- numeric(n) # 結果の保存
vars <- character(n) # 分析に使った変数名の保存
for (i in 1:n) {
sel <- bincomb[i,] # どの変数が使われたかの論理ベクトル
vars[i] <- paste(var.no[sel], collapse=",") # 変数名記録
if (sum(sel) >= 2) {
result[i] <- eigen(r[sel, sel])$val[1]/sum(sel) # 結果の記録
}
else {
result[i] <- NA
}
}
RESULT <- data.frame(vars, result) # 結果をデータフレームにして,
o <- order(RESULT$result, decreasing=TRUE) # 寄与率の大きい順に並べ替えて,
RESULT[o,] # 表示
以下のようなものが1023行も出ます
vars result
72 V4,V7 0.6079310385256367
260 V2,V8 0.5864172084814746
514 V1,V9 0.5793448970957535
36 V5,V8 0.5781238621539512
272 V2,V6 0.5730279383691933
:
959 V1,V2,V3,V5,V6,V7,V8,V9,V10 0.1507698641888030
1015 V1,V2,V3,V4,V5,V6,V8,V9,V10 0.1506910833840152
1023 V1,V2,V3,V4,V5,V6,V7,V8,V9,V10 0.1392082155345871
以下ゴミの行が変数の個数分続く初心者 (2009-11-08 (日) 11:41:34)
R + eclipse + StatET を導入して、無事、eclipse 上で R のコードを実行できるようになったのですが、
ブレークポイントを置いてデバッグするということはできません。
(ブレークポイントはおけるのですが、デバッグモードで R を起動できません)
これは設定が足りないだけなのでしょうか、それともそこまで StatET がサポートしていないのでしょうか。
R : 2.10.0, eclipse : 3.5.1, StatET : 0.8.0
よろしくお願いします。
きむ (2009-11-05 (木) 19:38:58)
かなりの初心者です。
windowsXPでRのver2.9.0を仕様しています。
データフレームのラベル設定に関する質問です。
いくつかのデータフレームを結合して一つのデータフレームにしようとしています。その際に、基のデータフレーム(名)を持った形で作成したいのですが、どのようにデータフレームを作成すれば良いでしょうか?
イメージしているのは、例えばライブラリ[kohonen]のサンプルデータ(nir)です。
spectra,composition, temperature, trainingといったカテゴリー?の下に個々にデータセットを有する形となっています。
(data(nir)とすることで、nir$spectra等でのハンドリングが可能)
例えば、以下のA、BというデータフレームからCを作成する場合に、どのような手順で処理を行えば上記例のようなデータフレームが作成できるのでしょうか?
(ex C$A,C$Bという形でハンドリングしたい)
(データセットのラベルはC$A.a, C$A.b, のように)
A <- data.frame(a=c(1, 2, 3), b=c(0, 0, 0))
B <- data.frame(a=c(4, 5, 6), b=c(1, 1, 1))
C <- cbind( A, B )
単純に上記のようにデータフレームを作成・結合し、paste関数とcolname関数を用いて(A.a, A.b,B.a,B.b)を与えてみたりしたのですが、うまくいきません。
(そもそも上記「nir」はdata関数が使えますが、上記で作成した「C」は使えません。)
(←作成したデータフレームをデータセットとして設定しないといけないのかもわかりませんが、その方法がわかりません。)
以上、
かなりまとまりのない質問ですみません。要は「nir」のようなデータフレームを作成する手順がわかりません。よろしくお願いします。
library(kohonen) data(nir) class(nir)nirのようなlistがつくりたい、ということであれば、
A <- data.frame(a=c(1, 2, 3), b=c(0, 0, 0)) B <- data.frame(a=c(4, 5, 6), b=c(1, 1, 1)) (C <- list(A=A, B=B))でよろしいかと。 それとも、きむさんが必要としてるのは
C <- cbind(A, B)
names(C) <- c("A.a", "A.b", "B.a", "B.b")
みたいなものなのかな。
nirのような「list」が得たいのか、C$A.a, C$A.b…のようなラベルが付いた「data.frame」が得たいのか、ちょっと分からないですけれども。 -- 2009-11-05 (木) 20:19:33> A <- data.frame(a=c(1, 2, 3), b=c(0, 0, 0)) # コメントが,遅かりし? > B <- data.frame(a=c(4, 5, 6), b=c(1, 1, 1)) > C2 <- data.frame(A, B) # データフレームの結合には,data.frame を使う,そうすれば,,,, > C2 # のように,同じ名前の列名は修飾されるので,識別可能となる a b a.1 b.1 1 1 0 4 1 2 2 0 5 1 3 3 0 6 1 > C2$a # C2$a と [1] 1 2 3 > C2$a.1 # C2$a.1 は,別物なんだ [1] 4 5 6 # でもね,その違いは些細なものなんだから,そんなのに依存して区別するのも考えもの
coro (2009-10-31 (土) 01:50:23)
初心者です。このような質問をするのは恥ずかしい限りです。
windowsXPでversion2.3.0を使用しております。
作成した曲線と任意のX軸と曲線との垂直交点の値(Y値)を求める方法を探しております。
例えば、V <- c(84,80,76,68,59,50,44,38,35,33,32,34,37,41,49,63,73,81) plot(V,type="b") abline(v=12.5)上記にてX軸12.5のときのY値を算出するにはどのようにすればよいのでしょうか?細かいグリッドを付けて交点を探したりもしましたが、計算できる方法がありましたらお教え頂きたく投稿致しました。
> x <- 1:18 > V <- c(84,80,76,68,59,50,44,38,35,33,32,34,37,41,49,63,73,81) > plot(x, V,type="p") > ans <- spline(x, V, n=100) > lines(ans, col="red") > spline(x, V, n=100, xout=12.5) $x [1] 12.5 $y [1] 35.44528 > library(splines) > ans2 <- interpSpline(x, V) > predict(ans2, 12.5) $x [1] 12.5 $y [1] 35.44523 attr(,"class") [1] "xyVector"
Saito (2009-10-29 (木) 14:57:19)
いつもお世話になっています。
今日は並列計算中の途中経過を表示(例えば全体の何%終わったか)させる方法についてご教授いただければ、と思います。例えば、普通のfor()だと、M <- NULL for (i in 1 : 1000) { M[i] <- rnorm(1, i, 10) cat(paste((i/1000)*100, "%"), "\n") }とでもすれば、今、全体の何%ほど完了しているのか見ることができます。しかし、並列計算になってくると上手くいきません。以下に上手くいっていないサンプルコードを示します。
library(rlecuyer) library(snow) f <- function(n){ a <- rnorm(1, n, 1) b <- rnorm(1, n, 2) return(c(a=a, b=b)) } cl <- makeSOCKcluster(c("localhost", "localhost")) clusterSetupRNG (cl,seed=12) clusterExport(cl, "f") system.time(ff <- parSapply(cl, 1:10, function(x){return(f(1)) cat(paste((x/25)*100, "%"), "\n") }) )計算結果はきちんと保存されていくのですが、進行具合が上手く表示されないようです。
どなたか、このような場合に上手く進行具合を表示させる方法をご存知でしたら、ご教授いただけないでしょうか。
なお、環境はR-2.10.0、WindowsXP(SP3)です。どうぞよろしくお願いいたします。
system.time(ff <- parSapply(cl, 1:10,
function(x){
cat(paste((x/25)*100, "%"), "\n")
return(f(1))
})
)cl <- makeSOCKcluster(c("localhost", "localhost"), outfile="cat.txt")
ただ、欲をいえば、合計が100%になるような表示だと見易くて良いです。system.time(ff <- parSapply(cl, 1:10,
function(x){
cat(paste((1/10)*100, "%"), "\n")
return(f(1))
})
)
のようにしていますが、これだとcat.txtの中身は
starting worker for localhost:10187 Type: EXEC 要求されたパッケージ rlecuyer をロード中です Type: EXEC Type: EXEC 10 % 10 % 10 % 10 % 10 %となってしまい、10%の数を数えなければ進行状況がわかりません。
cl <- makeSOCKcluster(c("localhost", "localhost"), outfile="cat3.txt")
clusterSetupRNG (cl,seed=12)
clusterExport(cl, "f")
system.time(ff <- parSapply(cl, 1:10, function(x){
cat(paste((x/10)*100, "%"), "\n")
return(f(1))
})
)
そのcat3.txt内の出力結果。
starting worker for localhost:10187 Type: EXEC 要求されたパッケージ rlecuyer をロード中です Type: EXEC Type: EXEC 60 % 70 % 80 % 90 % 100 %ただ、60%からスタートしているのが気にかかります。片方のコアの結果のみ出力しているのでしょうか? それと、試行回数を増やすと、なんだか変な出力になってしまうようです。以下に例を示します。
system.time(ff <- parSapply(cl, 1:100,
function(x){
cat(paste((x/100)*100, "%"), "\n")
return(f(1))
})
)
結果。-- Saito 2009-10-29 (木) 17:44:38
starting worker for localhost:10187 Type: EXEC 要求されたパッケージ rlecuyer をロード中です Type: EXEC Type: EXEC 51 % <snip> 50 % 100 %
require(snow)
cl <- makeCluster(2)
clusterSetupRNG (cl,seed=12)
pbar.open<-function(cl)
{
dummy<-winProgressBar(paste(cl$host,cl$rank),"0%",0,100,0)
close(dummy) # これはおまじない
assign("pb",
winProgressBar(paste(cl$host,cl$rank, "!!下にも居るよ!!"),
"0%",
0, 100, 0),
envir=.GlobalEnv)
NULL
}
pbar.close<-function(cl)
{
close(get("pb",envir=.GlobalEnv))
NULL
}
clusterExport(cl, "pbar.open")
clusterExport(cl, "pbar.close")
f <- function(n){
a <- rnorm(1, n, 1)
b <- rnorm(1, n, 2)
return(c(a=a, b=b))
}
clusterExport(cl, "f")
dummy<-clusterApply(cl,cl,pbar.open)
ff <- parSapply(cl,
1:10,
function(x){
setWinProgressBar(pb,
(x/10)*100,
label=paste((x/10)*100,"%"))
Sys.sleep(5)
return(f(1))
})
clusterCall(cl,pbar.close)
stopCluster(cl)
おまじない以外は自分で調べてみてください.R使用開始3日目 (2009-10-29 (木) 13:24:28)
Rを使用して3日目の初心者です。よろしくお願いします。
得られたデータについて、Rでクロス集計を行い、その結果をExcelに移し、まとめたいと考えています。data<-read.table("sample.csv", header=T, sep=",") data[,3] <- factor(data[,3], levels=1:5, labels=c("AG", "CO", "EX", "NEUR", "OP")) data[,2] <- factor(data[,2], levels=1:2, labels=c("男", "女")) cross(2, 3, data, latex=F)以上、クロス集計はできたのですが、その結果をRコンソールからコピー&ペーストでExcelに貼り付けようとすると、1行の毎の情報が1つのセルに情報が入ってしまい、情報が区切られていません。
person sex AG CO EX NEUR 合計 男 7 6 7 8 28 % 25.0 21.4 25.0 28.6 100.0 女 4 9 5 4 22 % 18.2 40.9 22.7 18.2 100.0 合計 11 15 12 12 50 % 22.0 30.0 24.0 24.0 100.0この表示のまま、つまり、各セルに「男」や「合計」、「24.0」などが入るように、Excelに情報を移したいのですが、どのような方法をとればいいでしょうか?
環境は、「Windows XP sp3」「R 2.9.2」です。
出来る範囲で色々と試してみたのですが、元々プログラム言語に触れたことがなく、分かりませんでした。
どうぞ、よろしくお願いします。
河童も驚いた (2009-10-28 (水) 22:57:27)
以下のようなプログラムで,abcde しか描かれない。plot((1:10)^2) text(4, 50, "abcde") text(4, 40, " fghij") text(4, 30, "xyz 123") text(c(4, 6, 8), 20, paste("Var", 1:3))文字列に空白が含まれる場合には,描かれない(描かれているけど見えない??)
> sessionInfo() R version 2.10.0 Patched (2009-10-27 r50238) i386-apple-darwin9.8.0 locale: [1] ja_JP.UTF-8/ja_JP.UTF-8/C/C/ja_JP.UTF-8/ja_JP.UTF-8 attached base packages: [1] stats graphics grDevices utils datasets methods baseなんということだ。
R version 2.10.0 Patched (2009-11-02 r50295) :途中省略 [R.app GUI 1.30 (5510) x86_64-apple-darwin9.8.0]
R version 2.10.0 Patched (2009-11-03 r50305), Mac OS X 10.6.1 (Snow Leopard) OS バージョンが 0.1 上がった? > plot(1:10) > text(5, 5, "123 def + ")
> plot(1:10)
> metric <- function(str) {
+ cat("argument:", deparse(substitute(str)), "strwidth:", strwidth(str),
+ "strheight:", strheight(str), "\n")
+ }
> metric("123_abc") # 描画される
argument: "123_abc" strwidth: 1.436108 strheight: 0.2790818
> metric("123 abc") # 描画されない
argument: "123 abc" strwidth: 1.375176 strheight: 0.2790818
> metric("123 abc\n") # 描画される
argument: "123 abc\n" strwidth: 1.375176 strheight: 0.7209
> metric("あいう_えお") # 描画される
argument: "あいう_えお" strwidth: 2.006757 strheight: 0.2790818
> metric("あいう えお") # 描画されない
argument: "あいう えお" strwidth: 1.945824 strheight: 0.2790818
> metric("あいう えお\n") # 描画される
argument: "あいう えお\n" strwidth: 1.945824 strheight: 0.7209akira (2009-10-28 (水) 18:10:27)
いつもありがとうございます.乱数について教えて下さい.
測定値のシミュレーションをしようとしています.
測定値は100個程度の正規分布を想定していますが、測定限界の関係で上限が0、下限が60,000になります.
この状態で、データを測定値に応じて送別化して、各層でSDを振った擬似データを作りたいと思っています.
rnormで平均とSDを指定した正規分布をとれますが、上限と下限を指定できません.
そもそも、上限と下限がある時点で正規分布でないのですが、測定には有効域があるので上限と下限を指定したいのです.
良い方法をご存じないでしょうか?
my.rnorm <- function(n,mean,sd,lower,upper){
x <- vector()
while(length(x)<n){
x <- c(x,rnorm(n,mean=mean,sd=sd))
x <- x[x>lower & x<upper]
}
return(x[1:n])
}
> my.rnorm(100,30,15,0,60)
[1] 18.737474 14.982537 12.871060 35.218585 46.118110 26.359055 57.167014
[8] 43.737251 17.558190 37.044597 28.312282 27.937564 37.606587 45.872538
[15] 31.474119 39.678433 22.517348 1.820924 21.478013 19.614942 7.276308
:
[85] 27.630476 53.156905 10.630811 32.732432 27.359767 30.165729 54.583836
[92] 14.693563 56.358438 17.895591 34.016179 6.088306 36.830447 30.897142
[99] 30.521995 29.281466
測定の性質にもよると思いますが、上限下限の外側は、正規分布に従えば存在しても測定不能で、測定不能なデータが欠損した状態が実測データであると考えれば、単純に削れるような。。。> set.seed(999)
> x <- rnorm(10000) # 標準正規乱数でよい
> r <- range(x) # 発生された乱数の最小値・最大値を求めて
> w <- diff(r) # 範囲を求めて
> MIN <- 100 # 希望する最小値
> MAX <- 60000 # 希望する最大値
> y <- (x-r[1])/w*(MAX-MIN)+MIN # 範囲が [MIN, MAX] になるように線形変換
> summary(y)
Min. 1st Qu. Median Mean 3rd Qu. Max.
100 23730 29370 29470 35320 60000> set.seed(1000)
> library(msm)
> x <- rtnorm(10000, mean=30000, sd=20000,lower=0, upper=60000)
> summary(x)
Min. 1st Qu. Median Mean 3rd Qu. Max.
7.26 18600.00 29880.00 30030.00 41630.00 59980.00
> hist(x)
aor (2009-10-27 (火) 00:25:10)
いつもお世話になっております。
carパッケージのAnova関数を利用し、以下のようなコードで多変量検定を行いました (コード自体は青木先生の掲示板より拝借しました) 。
この結果の一部を取り出したいのですが、どのようにすればよいか教えていただけないでしょうか。library(car) hsb2 <- read.table('http://www.ats.ucla.edu/stat/R/faq/hsb2.csv', header=T, sep=",") result <- Manova(lm(cbind(write,read)~female+math+science+socst, hsb2)) summary(result)このコードを実行すると、例えば
Multivariate Tests: female Df test stat approx F num Df den Df Pr(>F) Pillai 1 0.169885 19.851322 2 194 1.4335e-08 *** Wilks 1 0.830115 19.851322 2 194 1.4335e-08 *** Hotelling-Lawley 1 0.204653 19.851322 2 194 1.4335e-08 *** Roy 1 0.204653 19.851322 2 194 1.4335e-08 *** ---といったような多変量統計量が出力されます。このうち、"test stat"の部分を取り出してベクトルに格納したいのですが、これがうまくいきません。
str(result), あるいはstr(summary(result))で見ても、”Sum of squares and products for the hypothesis:”しか見当たらず、この結果しか取り出すことができません。
結果を別のベクトルオブジェクトに入れておくと後から使いやすいので何とか取り出したいと思っています。よい方法をご存知の方がいましたらよろしくお願いいたします。
環境はR2.9.2, Windows Vistaです。
hemuhemu (2009-10-26 (月) 18:26:30)
いつもお世話になっております。
クロス集計を行った結果で因子分析を行いたいのですが、「これらの初期値からは最適化ができません」とのエラーが帰ってきてしまいます。
どういう場合に上記のエラーが帰ってくるのか、また、何か対処法はあるのかを教えていただけませんでしょうか?
エラーメッセージ、因子分析について調べてみたのですが、よく分かりませんでした。
Rは、R version 2.9.1 i386-pc-mingw32で、windowsXPを使用しております。
どうぞよろしくお願い致します。
a b c d e f g h i j 1 1 0 0 0 12 0 0 0 5 0 2 0 0 2 0 0 0 2 0 0 0 3 0 0 0 0 0 0 0 1 0 1 4 0 8 0 0 2 0 0 0 0 0 5 0 0 1 0 0 0 7 0 0 0 6 1 0 0 0 0 0 0 0 2 0 7 0 0 0 2 0 0 0 0 0 1 8 0 2 0 0 0 0 1 0 9 0 9 0 0 10 0 0 0 0 0 0 0 10 0 0 0 0 3 4 0 0 0 0
-- hemuhemu 2009-10-26 (月) 18:55:41
ひどい初心者 (2009-10-25 (日) 18:14:23)
R言語にワイルドカード(どんな文字列にもマッチする記号)のようなものはありますか?
tefu (2009-10-23 (金) 16:27:11)
非常に初歩的なことだと思うのですが、調べてもわからなかったので質問します。x <- rnorm(100) hist(x) title("abc")上記のようなプログラムを実行するとヒストグラムのタイトル部分に"histgram of x" と "abc" が重なってしまい見にくいのですが、解決する方法はありますでしょうか?ご教示願います。
hist(x, main="")
title("abc")
ではなく,
hist(x, main="abc")とするほうがよいことがわかります。 -- おそかりし?河童の屁 2009-10-23 (金) 17:30:20
Ackin (2009-10-19 (月) 17:22:50)
例> A <- data.frame(a=c(1, 2, 3), b=c(0, 0, 0)) > AA <- data.frame(aaa=A[1], bbb=c(4, 5, 6)) > AA a bbb 1 1 4 2 2 5 3 3 6上記例で、AAに既存データフレームのデータをもってきて、ラベル名はaaaにしたいのですが、どうするのでしょうか?似た質問で既存データフレームのラベル名を変更するにはどうしたら良いでしょうか?
桜 (2009-10-16 (金) 23:08:37)
x1,x2 というふたつのダミー変数を用いて、回帰分析を行いました。
x1, x2 の値によってできる4つのグループ内の分散の逆数を重みとして、重み付回帰分析をしたいのですが、重みをつけても結果が変わりません。
何を間違えているのかわかりません。ご教示いただけたら幸いです。
よろしくお願いします。
でっちあげの例:x1 <- rep(c(0,1),c(8,18)) x2 <- rep(c(0,1,0,1),c(3,5,10,8)) set.seed(190) y <- c(rnorm(3,10,2),rnorm(5,11,4),rnorm(10,12,3),rnorm(8,10,4)) w <- 1/ c(rep(var(y[1:3]),3), rep(var(y[4:8]),5), rep(var(y[9:18]),10), rep(var(y[19:26]),8) )
lm(y~x1*x2) Call: lm(formula = y ~ x1 * x2) Coefficients: (Intercept) x1 x2 x1:x2 10.202 3.143 1.070 -5.014
lm(y~x1*x2, weights=w) Call: lm(formula = y ~ x1 * x2, weights = w) Coefficients: (Intercept) x1 x2 x1:x2 10.202 3.143 1.070 -5.014
> lm(y~x1+x2)
Call:
lm(formula = y ~ x1 + x2)
Coefficients:
(Intercept) x1 x2
12.4062 0.2772 -2.4567
> lm(y~x1+x2, weights=w)
Call:
lm(formula = y ~ x1 + x2, weights = w)
Coefficients:
(Intercept) x1 x2
11.857 1.220 -2.129> summary(lm1)$coeff
Estimate Std. Error t value Pr(>|t|)
(Intercept) 10.202096 1.991574 5.1226306 3.916815e-05
x1 3.142593 2.270743 1.3839488 1.802549e-01
x2 1.069892 2.519164 0.4247011 6.751793e-01
x1:x2 -5.014352 3.003911 -1.6692745 1.092330e-01
(Intercept) は x1==0 & x2==0 の時の y の平均なのですが、その Std. Error である 1.9915714 という値は y[x1==0 & x2==0] となんらかのシンプルなつながりがあるのでしょうか?
cbind(rep(1,length(x1)),x1,x2,x1*x2)などを見てみましたが、よく分かりませんでした。
mean(y[x1==0 & x2==0]) [1] 10.20210 > sd(y[x1==0 & x2==0]) [1] 2.927438
森の熊五郎 (2009-10-15 (木) 12:02:28)
ts関数を使う場合、毎月・または毎四半期のデータを入力することになりますが、日々のデータを入れて、7日間での周期で変動するパラメータについて、季節変動抽出をdecompose関数を使って分解したいと考えましたが、日々のデータでの入力がts関数では出来ずに困っています、他の方法があるのでしょうか?ご教示下さい。
> set.seed(999) > (x <- jitter(rep(c(1, 2, 3, 4, 2, 0, -1), 10), amount=1)) [1] 0.77814275 2.16612144 2.18933138 4.70526246 2.57349352 -0.76131548 [7] -0.78710601 0.16191381 1.78154483 3.23894478 3.57137765 2.10224548 途中省略 [61] 2.98281213 0.55632492 -0.54765611 1.88545396 2.14234596 2.99028707 [67] 4.72009442 2.73918408 -0.19494635 -1.33750080 > (y <- ts(x, frequency=7)) Time Series: Start = c(1, 1) End = c(10, 7) Frequency = 7 [1] 0.77814275 2.16612144 2.18933138 4.70526246 2.57349352 -0.76131548 [7] -0.78710601 0.16191381 1.78154483 3.23894478 3.57137765 2.10224548 途中省略 [61] 2.98281213 0.55632492 -0.54765611 1.88545396 2.14234596 2.99028707 [67] 4.72009442 2.73918408 -0.19494635 -1.33750080 > (z <- decompose(y)) $seasonal Time Series: Start = c(1, 1) End = c(10, 7) Frequency = 7 [1] -0.7309683 0.5398884 1.1755496 2.2610667 0.8231408 -1.7139546 [7] -2.3547226 -0.7309683 0.5398884 1.1755496 2.2610667 0.8231408 途中省略 [61] 0.8231408 -1.7139546 -2.3547226 -0.7309683 0.5398884 1.1755496 [67] 2.2610667 0.8231408 -1.7139546 -2.3547226 $trend Time Series: Start = c(1, 1) End = c(10, 7) Frequency = 7 [1] NA NA NA 1.551990 1.463957 1.409018 1.558963 1.396979 [9] 1.329658 1.304173 1.413051 1.419239 1.532508 1.403111 1.556131 1.436136 途中省略 [57] 1.644785 1.678867 1.847450 1.884422 2.136606 2.083784 2.096797 2.104237 [65] 2.069433 1.962109 1.849274 NA NA NA $random Time Series: Start = c(1, 1) End = c(10, 7) Frequency = 7 [1] NA NA NA 0.892205746 0.286395450 [6] -0.456378671 0.008654032 -0.504096936 -0.088001448 0.759222105 途中省略 [61] 0.023065117 0.186495890 -0.289730356 0.512184743 -0.466975924 [66] -0.147371514 0.609753665 NA NA NA $figure [1] -0.7309683 0.5398884 1.1755496 2.2610667 0.8231408 -1.7139546 -2.3547226 $type [1] "additive" attr(,"class") [1] "decomposed.ts" > plot(z)
plot(z, xlab="")
axis(1, at=1:70, labels=as.Date(1:70*7, origin="2009/04/15"), cex.axis=0.7,
line=7.5)
aor (2009-10-10 (土) 14:50:22)
いつもお世話になっております。par(bty = "l") plot(1:10, xaxt = "n")上のようなグラフで、y軸の目盛を描画せず、x軸のラインだけを描画する方法はありますか?
par(bty = ...)での指定は2本以上描画されますし、また例えば
plot(1:10, axes = F) axis(1, labels = F)等であとからaxix関数を使用しても、x軸の左右の余白 (?) が切れてしまい、また目盛も描かれるようです。
以下は補足ですが、最終的にはpar(mfrow = c(1, 2)) par(mar = c(4,2,4,0)) par(oma = c(4,4,4,4)) par(bty = "l") plot(1:10, xlab = "condition1") plot(1:10, xlab = "condition2")というように複数のグラフをつなげて作成し、condition2のグラフにy軸を表示しないようにしたいと考えています。そのため試行錯誤しており、condition2のグラフでaxes = Fと指定して、後からaxisでx軸を書き足そうとしたのですが、プロット領域の両端までラインが描画されないため、まずプロットするときにx軸のラインのみを描画する方法を探しているところです。自分でも、何かすごく回り道をしている気がするのですが…
よい方法をご存知の方、いらっしゃいましたらよろしくお願いします。
環境はWindows Vista, R2.9.2です。
X <- 1:10 plot(X, axes = FALSE) axis(1, labels = FALSE, tck = 0, at = range(X))とすれば、Xの範囲に線が引かれます。 正確にはXの最小・最大点にtickを打つように指定しつつ、tick自体は長さを0にして描かれないようにしています。
X <- 1:10 LIM <- c(-10, 15) # 線を引きたい範囲 plot(X, axes = FALSE, xlim = LIM) axis(1, labels = FALSE, tck = 0, at = LIM)とすればOKでは。
par(mfrow = c(1, 2), mar = c(4,2,4,0), oma = c(4,4,4,4), bty = "l")
par(mfrow = c(1, 2), mar = c(4,0,4,0), oma = c(4,4,4,4), bty = "l") LIM <- c(0.5, 10.5) plot(1:10, xlim = LIM, xlab = "Condition1") plot(1:10, axes = F, xlim = LIM, xlab = "Condition2") axis(1, labels = F, tck = 0, at = LIM) # まずラインを引いて、 axis(1) # 目盛を描画ご指導感謝いたします。-- aor 2009-10-10 (土) 22:42:27
gonzu (2009-10-05 (月) 18:48:51)
いつもありがとうございます.
1000回同じ条件で繰り返しを行い、その95%の信頼区間を求めてグラフに表そうとしているのですが、上手くいかず悩んでいます.
mymonte <- function(n) { count <- 0 for(i in 1:1000) { X <- runif(704)*50 Y <- runif(704)*50 XY <- data.frame(X, Y) XY <- data.frame(XY, Dataset[, c(2, 12)]) w <- owin(c(0, 50), c(0, 50)) Rpp <- as.ppp(XY[, 1:2], w, fatal=FALSE) mark1 <- XY$DBH.02. mark2 <- XY$sort Rpp1 <- Rpp %mark% mark1 Rpp2 <- Rpp1 %mark% mark2 KC <- Kmulti(Rpp2, (mark1 <= 5) & (mark1 >= 1) & (mark2 == "ヤブムラサキ"), (mark1 > 5) & (mark2 == "シラキ"), correction="isotropic") par(new=TRUE) count <- count+1 } return(count/n) } plotmeans(sqrt(iso/pi)-r ~r, connect=F, data=KC)上記のよう実行しますと、平均値だけがプロットされて信頼区間は表れない図になります。countの使い方がおかしいのかとも思いますが….
どなたかご教授お願いいたします。
使用環境はR version 2.9.2 (2009-08-24) i386-pc-mingw32です.よろしくお願いいたします.
for(i in 1:1000){
X<-runif(704)*50
Y<-runif(704)*50
XY<-data.frame(X,Y)
XY<-data.frame(XY,Dataset[,c(2,12)])
w<-owin(c(0,50),c(0,50))
Rpp<-as.ppp(XY[,1:2],w,fatal=FALSE)
mark1<-XY$DBH.02.
mark2<-XY$sort
Rpp1<-Rpp %mark% mark1
Rpp2<-Rpp1 %mark% mark2
KC<-Kmulti(Rpp2,(mark1<=5)&(mark1>=1)&(mark2=="ヤブムラサキ"),
(mark1>5)& (mark2=="シラキ"),correction="isotropic")
par(new=T)
plot(KC, sqrt(iso/pi)-r ~ r,ylim=c(-5,5)) # L関数のグラフ
}
としていました。これを実行しますと、KCが回数を重ねるたびに更新されていくようなので、countにKCの結果を残して信頼区間を求めようとしています(うまくいってませんが…)。 -- gonzu 2009-10-06 (火) 20:30:23きりん (2009-10-05 (月) 11:09:19)
下の方と同様な内容で恐縮ですが、質問させてください.
(私もいつも同じような問題で悩むものですから)
下の方と同様の例ですが、# testdata.txt "文字","英数","値1","値2" "1","12345","a",10,1.4142 "2","00123","b",100,1.732 "3","02","0",1000,3.1415(私の扱うフィルも列名に【”】が付いています.)
これを、read.table(file="testdata.txt", sep=",", as.is=T)で読み込むと、
文字 英数 値1 値2 1 12345 a 10 1.4142 2 123 b 100 1.7320 3 2 0 1000 3.1415のように、読み込んでしまうのですが、ヘルプを読むとデフォルトで「quote="\"'"」となっているので1列目と2列目は文字列として読み込むことを期待しています.が、そうなっていません.
(「as.is=T」をつけなければファクターに変換されますが.この場合は「as.is=T」を取ってもファクターになっていませんから、そもそも文字としては認識されていない???)
下の方の様に「quote=""」とすると、これはquotingを無効にすることのようでX.文字. X.英数. X.値1. X.値2. "1" "12345" "a" 10 1.4142 "2" "00123" "b" 100 1.7320 "3" "02" "0" 1000 3.1415のようになるのは、ある意味納得できます.列名に「X.」とかがつくのはそういう仕様なのかもしれません.
ちなみに> tmp <- read.table(file="testdata.txt", sep=",", as.is=T, quote="") > tmp$文字 [1] "\"12345\"" "\"00123\"" "\"02\""となって、値自体に【”】が入っています.クオート文字として認識されませんから、文字列の一部として認識されています.(ま、これをgsubで削除すればいいのかもしれませんが・・・)
私も、列数が少ない場合や、デフォルトでnumericで読まれるような列を文字として読み込む場合は、【colClasses】引数を使用しています.
ですが、異なったファイル毎に、いちいちcolClassesを指定するのは面倒ですし(特に列数が500とか1000とかの場合)、ファイルを作成する場合に、【”】でクオートされた列は文字列として読まれることを期待してファイルを作成しています.
このように、【”】でくくられた列(値)を文字列として読み込まないのは、どういうことなのでしょうか?scanのヘルプも読みましたが、【”】でくくられた列(値)を文字列として読み込まないとは読めません.英語をうまく訳せていないのかもしれませんが・・・
どなたかご教示いただけると幸いです.
> x <- readLines("testdata.txt", n=2)
> l1 <- unlist(strsplit(x[1], ","))
> l2 <- unlist(strsplit(x[2], ","))
> (Quotes <- grep('\".*\"', l2))
[1] 1 2 3
> colClasses <- rep("numeric", length(l2)) # 基本的には数値として読む
> colClasses[Quotes] <- "character" # ダブルクオートが2つある列は,文字列として読む
> colClasses # 実際どんなふうになるか
[1] "character" "character" "character" "numeric" "numeric"
> (d <- read.table("testdata.txt", header=TRUE, colClasses=colClasses, sep=","))
文字 英数 値1 値2
1 12345 a 10 1.4142
2 00123 b 100 1.7320
3 02 0 1000 3.1415
> sapply(d, class) # 期待したものになっていることの確認
文字 英数 値1 値2
"character" "character" "numeric" "numeric"> default.stringsAsFactors() [1] TRUEとなります.で「as.is = !stringsAsFactors」ですから、options()?でdefault.stringsAsFactors()を替えていなければ、「as.is=F」 がデフォルトではないでしょうか?で「Note that this is overridden by as.is and colClasses」ですから、今回「as.is=T」を指定していますので、文字列として認識された列はそのまま、文字列になるのではないのでしょうか?かつ、今回の例では「as.is=F」と指定しても
> read.table(file="testdata.txt", sep=",", as.is=F)[,1] [1] 12345 123 2となって、ファクターとしても認識してくれません.こういうことをakiraさんはおっしゃっているのではなくて? 今頃で申し訳ないのですが
> sessionInfo() R version 2.8.1 (2008-12-22) i386-pc-mingw32です. -- きりん 2009-10-05 (月) 14:59:18
> class(type.convert("00123", as.is=T)) # as.is に関わらず integer
[1] "integer"
> class(type.convert("00123", as.is=F))
[1] "integer"
> class(type.convert("A", as.is=F)) # 数値ではないものは,as.is により変わる
[1] "factor"
> class(type.convert("A", as.is=T))
[1] "character"
> class(type.convert("TRUE", as.is=T)) # 文字列でも,TRUE/FALSE は別
[1] "logical"
> class(type.convert("TRUE", as.is=F)) # as.is に関わらず,どっちも logical
[1] "logical"read.table2 <-
function (file, header = FALSE, sep = "", quote1 = "\"'",
quote = "\"'", dec = ".", #「quote1」引数を追加
row.names, col.names, as.is = !stringsAsFactors, na.strings = "NA",
colClasses = NA, nrows = -1, skip = 0, check.names = TRUE,
fill = !blank.lines.skip, strip.white = FALSE, blank.lines.skip = TRUE,
comment.char = "#", allowEscapes = FALSE, flush = FALSE,
stringsAsFactors = default.stringsAsFactors(), encoding = "unknown")
・・・
first <- scan(file, what = "", sep = sep,
quote = quote1, #「quote1」引数に変更
nlines = 1, quiet = TRUE, skip = 0, strip.white = TRUE,
blank.lines.skip = blank.lines.skip, comment.char = comment.char,
allowEscapes = allowEscapes, encoding = encoding)
・・・
for (i in (1:cols)[do]) {
data[[i]] <- if (is.na(colClasses[i]) & as.is[i] &
(length(grep("^\".*\"$", data[[i]][1])) == 1) )
gsub("\"$", "", gsub("^\"", "", data[[i]]))
else if (is.na(colClasses[i]))
type.convert(data[[i]], as.is = as.is[i], dec = dec,
na.strings = character(0))
> read.table2(file="testdata.txt", sep=",", as.is=T, quote="")
string eisu val1 val2
"1" 12345 a 10 1.4142
"2" 00123 b 100 1.7320
"3" 02 0 1000 3.1415冷蔵庫 (2009-10-02 (金) 16:22:13)
いつもお世話になっています。R初心者です。
read.tableのquoteの挙動、データの取り込みについて教えていただきたいのですが、
例えば、以下のデータがtestdata.txtとしてあった場合、
(1行目が列名、1列目が行名です。列名、行名にそれぞれ「””」が含まれています。)#testdata.txt
"文字","英数","値1","値2" "1","12345","a",10,1.4142 "2","00123","b",100,1.732 "3","02","0",1000,3.1415
データの文字と数値を区別して取得したく、testresult <- read.table(file="testdata.txt", sep = "," , as.is = T, quote= "")
で取り込むと、読み込んだデータの列名が「X.列名.」になってしまい、
行名も「"行名"」になってしまいます。X.文字. X.英数. X.値1. X.値2. "1" "12345" "a" 10 1.4142 "2" "00123" "b" 100 1.7320 "3" "02" "0" 1000 3.1415
この列名の「X. - .」、行名の「" - "」が付かないように読み込みたいのですが、
ここで、quote=""を除くと、「00123」(文字)が「123」(数字)として認識されてしまいます。文字 英数 値1 値2 1 12345 a 10 1.4142 2 123 b 100 1.7320 3 2 0 1000 3.1415
列名に「X. - .」、行名に「" - "」を付けず、
データ内容を元のとおりに取得する方法は何かありますでしょうか?
Rは、
R version 2.9.1
i386-pc-mingw32
で、windowsXPを使用しております。
どうぞよろしくお願い致します。
> (x <- read.table(file="testdata.txt", sep = "," , header=TRUE,
+ colClasses=c("character", "character", "character", "integer", "numeric")))
文字 英数 値1 値2
1 12345 a 10 1.4142
2 00123 b 100 1.7320
3 02 0 1000 3.1415
> sapply(x, class)
文字 英数 値1 値2
"character" "character" "integer" "numeric"初心者S (2009-09-30 (水) 16:52:45)
R2.4.1を使っている初心者Sです.
以下のようなデータに対して,非線形回帰をおこないました.データに対するモデル式の当てはまりのよさを決定係数で評価したいと考えておりますが,summary()では,算出されないようです.もし,決定係数の具体的な算出方法がおわかりでしたら,ご教示頂けないでしょうか?よろしくお願い申し上げます.# データ Delay<-c(3,6,12,24,36) Value<-c(85,75,50,40,30) # nlsでの非線形回帰 fm <- nls(Value ~ 100 /(1 + k * Delay), start=list(k=0)) summary(fm)
> logLik(fm) 'log Lik.' -12.80497 (df=2) > AIC(fm) [1] 29.60994
akira (2009-09-29 (火) 10:35:07)
いつもありがとうございます.
for-loopで処理を回すと、ときどきエラーが出ます.エラー発生時にif処理を加えたいのですが、エラーを検出する方法が分かりません.
具体的にはLDAでcolinearにならなければpredict、colinearならNAを返したいです.
できればLDAに特化しない方法にしたいので、warnings()の返り値をとろうと思ったのですが、うまく行きません.
LDAで良い例をかけないので、代替コードを書きます.> x <- NULL > for(i in c(1,-4,5,-6,3)) x <- c(x,log(i)) Warning messages: 1: In log(i) : 計算結果が NaN になりました 2: In log(i) : 計算結果が NaN になりました > x [1] 0.000000 NaN 1.609438 NaN 1.098612NaNになる場合に
> x [1] 0.000000 -100 1.609438 -100 1.098612になるようにするイメージです.
try('処理A','エラー処理')
でいいと思うのですが、'エラー処理'が少し長いスクリプトで、かと言って関数やsourceで呼び出す程でもないものだったりして、
if('処理A'==OK){
'処理B'
'処理C'
}else{
'エラー処理A'
'エラー処理B'
}
みたいにしたいな、と。try('処理A';'処理B';'処理C','エラー処理A';'エラー処理B')
みたいな方法もあるのでしょうか? -- akira 2009-09-29 (火) 13:19:47x <- NULL
dat<-c(1,-4,5,-6,3)
len<-length(dat)
for(i in 1:len){
cmd<-paste("x <- c(x,log(dat[",i,"]))",sep="")
eval(parse(text=cmd))
}
dat2<-NULL
len2<-length(warnings())
for(j in 1:len2){
cmd2<-paste("dat2[",j,"]<-as.character(warnings()[[",j,"]][[2]])[[3]]",sep="")
eval(parse(text=cmd2))
}
dat2<-as.numeric(dat2)
x[dat2]<-"-100"
xoptions(warn=2) # 警告をエラーに設定するのがミソ x<-NULL for(i in c(1,-4,5,-6,3)) x <- c(x,tryCatch(log(i),error=function(x)-100))
R初学者 (2009-09-28 (月) 14:38:06)
以前にも同じような質問をさせていただきましたが、再度質問させて頂きます。
plot()やhist()関数でグラフを作った後に、x軸の下にmtext()を用いて図の説明等を加えたいと思っています。
その際、説明の中にイタリックが一部混ざります。
そのイタリックを表示するために、expression(italic())を用いているのですが、改行を行った際に、イタリックに指定した部分だけがずれてしまいます。
どのようにすれば正しく改行されるのでしょうか?
以下に例を貼っておきます。> par(mar=c(7, 5.3, 2.5, 1)) > hist(rnorm(1000), main="例", ylab="頻度") > mtext(expression(paste("x軸ラベルの下に左揃えで表示するのですが、\nこのように文の途中にイタリック体を入れて", + italic("italic"),"改行すると\nうまく改行ができません")), + side=1, line=5,font=2,adj=0)よろしくお願いいたします。
par(mar=c(8, 5.3, 2.5, 1))
hist(rnorm(1000), main="例", ylab="頻度")
texts<-list(expression("x軸ラベルの下に左揃えで表示するのですが、"),
expression(paste("このように文の途中にイタリック体を入れて",
italic("italic"),
"改行すると")),
expression("うまく改行ができません"))
lapply(seq(length(texts)),function(i)mtext(texts[[i]],side=1,line=i+3,font=2,adj=0))par(mar=c(8, 5.3, 2.5, 1))
hist(rnorm(1000), main="例", ylab="頻度")
mtext(expression("x軸ラベルの下に左揃えで表示するのですが、"),side=1,line=4,font=2,adj=0)
mtext(expression(paste("このように文の途中にイタリック体を入れて",
italic("italic"),
"改行すると")),side=1,line=5,font=2,adj=0)
mtext(expression("うまく改行ができません"), side=1,line=6,font=2,adj=0)(2009-09-26 (土) 16:12:33)
Rコマンダーを使用し、クラスター分析を行ってますが、デンドログラムでのラベル表示順に、テキストとして、ラベルを出力したいのですが、どのようにすると良いのでしょうか。どなたか教えていただけると助かります。cutreeを使用し、出力しても、左から準でなく、デンドログラムと一致させることが困難です。 -- クラスタ検討中の初心者 2009-09-26 (土) 11:10:59
> set.seed(3141592)
> (dataset <- data.frame(x=rnorm(10), y=rnorm(10),
+ Country=paste("国", letters[1:10], sep="-")))
x y Country
1 0.19317799 1.3714874 国-a
2 -0.04847455 -0.5333877 国-b
3 -0.21257424 0.6863163 国-c
4 0.93021111 -0.7932223 国-d
5 -0.65881763 -0.7391611 国-e
6 -1.08777535 0.7393223 国-f
7 1.33396058 0.3414277 国-g
8 -0.52538553 -0.2207230 国-h
9 0.45192105 -0.6031778 国-i
10 -0.20926002 1.2376127 国-j
> obj <- hclust(dist(dataset[1:2]))
> str(obj) # これをやって,どんなものが返されるか確認しましょ!
List of 7
$ merge : int [1:9, 1:2] -1 -2 -5 -3 -4 -6 3 -7 6 -10 ...
$ height : num [1:9] 0.424 0.505 0.535 0.796 1.013 ...
$ order : int [1:10] 6 3 1 10 7 5 8 4 2 9
$ labels : NULL
$ method : chr "complete"
$ call : language hclust(d = dist(dataset[1:2]))
$ dist.method: chr "euclidean"
- attr(*, "class")= chr "hclust"
> plot(obj, hang=-1, label=dataset$Country)
> as.character(dataset$Country[obj$order]) # 結局,こうやればいいのですよ
[1] "国-f" "国-c" "国-a" "国-j" "国-g" "国-e" "国-h" "国-d" "国-b"
[10] "国-i"
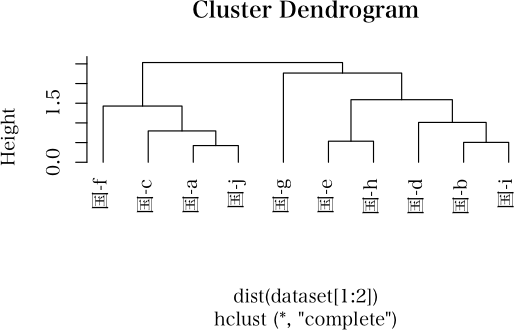
sakura (2009-09-23 (水) 21:34:26)
> tapply(<file_name>$age, list(sex=<file_name>$<cate_valiable>, <cate_valiable>=<file_name>$sex), + mean, na.rm=TRUE) <cate_variable> sex male female AvAw 53.58940 56.82658 V_over 60.60800 63.74648 W_over 52.92593 58.81119 Zoo 56.63345 62.18116上記のスクリプトを変更して、meanの横に、sd やパーセンタイル値を表示させるにはどうしたらいいのでしょうか?
例えば、<cate_variable>~ sex male female~ mean sd mean sd~ AvAw 53.58940 10.34567 56.82658 10.98765 V_over 60.60800 12.45678 63.74648 10.98765 W_over 52.92593 11.56789 58.81119 10.98765 Zoo 56.63345 13.78901 62.18116 10.98765のように出力させるには???
> set.seed(7460) # 試してみるときのために
> n <- 200
> d <- data.frame(
+ age=sample(factor(1:5), n, replace=TRUE),
+ sex=sample(factor(1:2), n, replace=TRUE),
+ x=rnorm(n))
> a <- tapply(d$x, list(d$sex, d$age), # sex, age の組み合わせごとに,
# データ数b,平均値,標準偏差を求める(その他,どんなことでも)
+ function(z) {
+ return(c(sum(!is.na(z)), mean(z), sd(z)))
+ })
> t(matrix(unlist(a), 3)) # 各行が,factor のどのような組み合わせか,表示すべし
[,1] [,2] [,3] # 各列は,データ数,平均値,標準偏差の順
[1,] 23 -0.244357375 1.0565744
[2,] 25 0.005392937 1.0587101
[3,] 30 -0.173957371 1.0238300
[4,] 28 0.127966034 0.9435006
[5,] 20 0.075841321 1.1881929
[6,] 13 0.253818609 0.9067395
[7,] 17 0.106300283 0.9716491
[8,] 17 0.115522521 1.0345919
[9,] 13 -0.025486074 0.6829240
[10,] 14 0.042842295 1.0857778
> array(unlist(a), dim=c(3,5,2)) # array 関数で表示するも宜しかろう
, , 1 # sex = 1
[,1] [,2] [,3] [,4] [,5] # age = 1 - 5
[1,] 23.0000000 25.000000000 30.0000000 28.0000000 20.00000000 # データ数
[2,] -0.2443574 0.005392937 -0.1739574 0.1279660 0.07584132 # 平均値
[3,] 1.0565744 1.058710098 1.0238300 0.9435006 1.18819286 # 標準偏差
, , 2 # sex = 2
[,1] [,2] [,3] [,4] [,5]
[1,] 13.0000000 17.0000000 17.0000000 13.00000000 14.00000000
[2,] 0.2538186 0.1063003 0.1155225 -0.02548607 0.04284230
[3,] 0.9067395 0.9716491 1.0345919 0.68292405 1.08577777 R初学者 (2009-09-16 (水) 19:36:26)
グラフ(今回は棒グラフ)をつくる際に、x軸のラベルを以下の例のように、
二段構成にしたいと思っていますが、どうすればよいのかわかりません。
male femalespecies name
色々と調べてはみたのですが、はっきりとした答えがなかったもので・・・。
よろしくお願いいたします。
> hist(rnorm(10000), + xlab="一行目の後にバックスラッシュとnを置けば,\n改行されるので,その後は二行目になる")
old <- par(mar=c(5, 5.3, 2.5, 1))
hist(rnorm(1000),
main="main においても,\n同じテクニックが使えるよ",
ylab="頻度\nmarの設定が必要になることもある",
xlab="一行目と二行目の")
mtext("文字サイズやフォントを変えるときなどはこんなやり方も",
side=1, line=3.8, cex=0.7, font=2)
おじさんの悩み (2009-09-15 (火) 01:24:36)
現在、あるサービスにおいてWEBにてQ&A集を作ることを検討しています。
Q&Aを作るにあたり、特定の期間モニターに質問をして頂き、その内容を分析しました。その結果、質問の上位20位までの累積質問率が下記の通りになりました。このような分布をもとに、全質問に対し、7割ないし8割の回答を目標にするには何件のQ&Aを用意すればよいかの検討をしています。
指数近似や多くの関数を元に最小ニ乗法を使って合わせこみをしましたが、うまくいきません。Rを使って、この問題を解くのに有用なモジュールないし手法はありますでしょうか?
0 0
1 5.9
2 11.1
3 15.7
4 19
5 21.9
6 24.5
7 26.9
8 29.2
9 31.5
10 33.3
11 34.9
12 36.5
13 38.1
14 39.7
15 41.2
16 42.6
17 44
18 45.4
19 46.7
20 48
> x <- 0:20 > y <- c(0, 5.9, 11.1, 15.7, 19, 21.9, 24.5, 26.9, + 29.2, 31.5, 33.3, 34.9, 36.5, 38.1, 39.7, 41.2, + 42.6, 44, 45.4, 46.7, 48) > d <- data.frame(x=x, y=y) > res <- nls(y~SSasymp(x, a, b, c), data=d) > summary(res) Formula: y ~ SSasymp(x, a, b, c) Parameters: Estimate Std. Error t value Pr(>|t|) a 55.76073 1.44109 38.694 <2e-16 *** b 1.55679 0.55181 2.821 0.0113 * c -2.40868 0.05779 -41.678 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 0.7988 on 18 degrees of freedom Number of iterations to convergence: 0 Achieved convergence tolerance: 1.721e-06 > plot(d$x, d$y, xlim=c(0, 70), ylim=c(0, 60), + pch=19, cex=0.5) > abline(h=res$m$getPars()[1], lty=3, col="blue") > x2 <- seq(0, 70, by=0.1) > d2 <- data.frame(x=x2) > y2 <- predict(res, newdata=d2) > lines(x2, y2, col="red")
> y <- c(0, 5.9, 11.1, 15.7, 19, 21.9, 24.5, 26.9,
+ 29.2, 31.5, 33.3, 34.9, 36.5, 38.1, 39.7, 41.2,
+ 42.6, 44, 45.4, 46.7, 48)
> y2 <- log(diff(y))
> x2 <- log(1:20)
> plot(y2 ~ x2, pch=19, cex=0.5, main="べき乗則(広義のジップの法則)--両対数グラフ",
+ xlab="順位の対数", ylab="頻度の対数")
> ans <- lm(y2 ~ x2)
> abline(ans, col="red")
> ans
Call:
lm(formula = y2 ~ x2)
Coefficients:
(Intercept) x2
1.9725 -0.5749 # 傾きが -0.579 なので,広義のジップの法則に従っているといえる
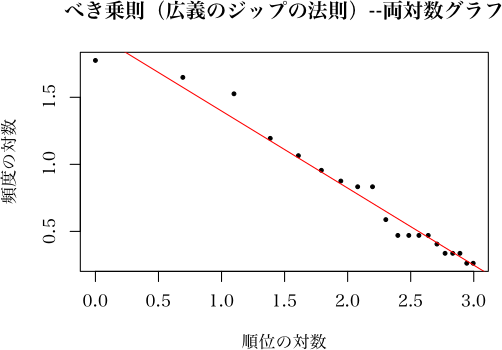
> x2 <- log(1:100)
> y2 <- ans$coefficients[1]+ans$coefficients[2]*x2
> plot(exp(y2), pch=19, cex=0.5,
+ main="1〜20までの頻度から予測した結果--ロングテール",
+ xlab="順位", ylab="頻度")
> points(diff(y), col="red", pch=19, cex=0.5)
> legend("topright", legend=c("実測値", "予測値"), pch=c(19, 19),
col=c("red", "black"))
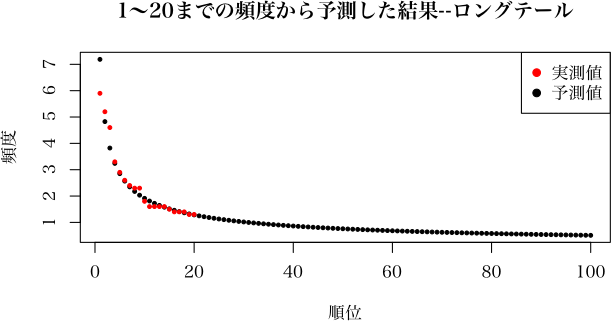
R初心者くん (2009-09-13 (日) 03:40:01)
お世話になっています。Rcomanderについて質問させてください。
3列のデータがあったとします。その最も左の列にサンプルの名称が、残り2列はそのサンプルのデータとします。
そのサンプル名をRcomanderを利用してつくったクラスタリングの図に載せたいのですができません。ただ、数値ならば可能です。
ご存知の方がいらっしゃたらご教授願えないでしょうか
お願いします。
HClust.1 <- hclust(dist(model.matrix(~-1 + Petal.Length+Petal.Width+Sepal.Length+Sepal.Width, iris)) , method= "ward") plot(HClust.1, main= "Cluster Dendrogram for Solution HClust.1", xlab= "Observation Number in Data Set iris", sub="Method=ward; Distance=euclidian", labels=iris$Species)

yuri (2009-09-09 (水) 10:56:26)
10万行ほどのファイルを読み込んで作業をしますが、新しく一列増やし(場所はどこでも良い)、列名をindexとして通し番号を付けたいのです。その際、あらかじめ自分で行数を確認して付けるのではなく、自動で行数を認識させて、自動的に必要な分だけの通し番号を付けるような関数はありますでしょうか。
にぽぽ(2009-09-08 (火) 20:21:00)
初級者にすら到達していない者です。
統計解析をRで行いなさい、と突然上の者から指示され、こちらのサイト等を見せていただいて相当あれこれやってみたのですが、もうどうしようもなくて、こちらに書かせていただきました。
R2.9.0です。
下の表のような表があります。
縦軸が商品、横軸が年齢層です。
縦軸横軸ともに10項目以上ありますA B C D E F・・・ イ 0 1 0 0 0 0 ロ 1 0 0 0 0 0 ハ 0 0 1 1 1 0 ・ ・ ・縦軸の相関関係を表わそうとigraphでやってみたのですが、どうにもうまくいきません。
shouhin<-matorix(c(0,1,0,1,0,0,0,0,1,0,0,1,0,0,1,0,0,0・・・),nrow=13,ncol=7)と書いて
library(igraph)として、行き詰ってしまいました。
そもそも、相関関係という言葉が正しいのかもわからないのですがやりたいことは、例えば縦軸の商品イとハは、横軸の50歳代に人気だから近しい商品である、とグラフでわかるようにしたいのです。
どの関数を使ったら、目的のグラフを描画することができるでしょうか。
お力をお貸しいただければ嬉しいです。
> (d <- structure(list(A = c(0L, 1L, 0L), B = c(1L, 0L, 0L), C = c(0L,
+ 0L, 1L), D = c(0L, 0L, 1L), E = c(0L, 0L, 1L), F = c(1L, 0L,
+ 0L)), .Names = c("A", "B", "C", "D", "E", "F"), class = "data.frame",
+ row.names = c("イ", "ロ", "ハ")))
A B C D E F
イ 0 1 0 0 0 1
ロ 1 0 0 0 0 0
ハ 0 0 1 1 1 0
> library(MASS)
> library(maptools)
> (ans <- corresp(d, nf=2))
First canonical correlation(s): 1 1
Row scores:
[,1] [,2]
イ -8.732638e-17 -1.4142136
ロ -2.121320e+00 0.7071068
ハ 7.071068e-01 0.7071068
Column scores:
[,1] [,2]
A -2.121320e+00 0.7071068
B -2.019203e-16 -1.4142136
C 7.071068e-01 0.7071068
D 7.071068e-01 0.7071068
E 7.071068e-01 0.7071068
F -9.637845e-17 -1.4142136
> layout(matrix(1:2, 1))
> plot(ans$cscore)
> pointLabel(ans$cscore[,1], ans$cscore[,2], rownames(ans$cscore), offset=1)
> plot(ans$rscore)
> pointLabel(ans$rscore[,1], ans$rscore[,2], rownames(ans$rscore), offset=1)
> layout(1)
ぼう (2009-09-08 (火) 09:07:07)
お世話になります。
たとえば下記のようなグループ名をもつ行列dfがありまして(reshapeパッケージで2つのカテゴリーを行名にcastしたものです)これをアルファベットと数字に分割して別々に列の値に入れたいと思っています。
欠損が無ければstrsplit(rownames(df),"-")とunlistを組み合わせて簡単にできそうですが、ところどころ値のないカテゴリーがありうまく処理できずにいます。
アドバイスいただければ幸いです。
これをdf <- data.frame(1:6) rownames(df)<-c( "A-I", "A-II", "A-",#←こんな風に欠損がところどころある。 "B-I", "B-II", "-III" )こうしたい
|c1|c2 |value| |A |I |1| |A |II |2| |A |NA |3| |B |I |4| |B |II |5| |NA|III|6|
ans <- sapply(rownames(df), function(str) {
a <- unlist(strsplit(str, "-"))
len.a <- length(a)
if (len.a == 1) { # "-" の後がない
return(c(a, NA))
} else if (a[1] == "") { # "-" の前がない
return(c(NA, a[2]))
} else { # 完全
return(a)
}
})
(df2 <- data.frame(c1=ans[1,], c2=ans[2,], value=df[,1]))tefu (2009-09-07 (月) 21:17:18)
初歩的な質問で申し訳ありません。
ある関数function(x)のxに、毎回違う数字を入れて何度も繰り返したい場合、どのような方法があるでしょうか?
挿入する数字に規則性はなく、数百回単位で繰り返したいのですが、効率的な方法はありますか?
aor (2009-09-06 (日) 19:11:53)
以前はfactor関数についてお世話になりました。
aov関数で求められる平方和について教えていただきたく、投稿いたします。
Jonathan Baronによる解説 を読んで疑問が出てきました。
上記の解説、Unbalanced (Nonorthogonal) Designsのセクションによると
SPSSで平方和のタイプを2と設定すれば、aov関数の結果と一致する、とあります。
aovで求められる平方和はタイプ2、ということなのでしょうか。
現在、SPSS、SASを使用できる環境ではなく、検証できておりません。
aovでの平方和についてご存知の方、あるいは検証方法についてのヒントでも
いただけたら幸いです。よろしくお願いいたします。
環境はR2.9.2、windows vistaです。
なお、平方和について不勉強で、色々検索し、中澤先生の解説, 岸本先生の解説 などを参考にしました。
anova関数ではタイプ1、という記述は色々なところで散見されるのですが、 aov関数でどのように行っているかは (自分としては) 不明です。
また、筑波大学の井関先生の作成された分散分析関数 により、
平方和をタイプ2に指定して検証してみましたが、aov関数の結果と一致するようです。
以下はJonathan Baronの解説で記載されていた混合計画の分散分析のコードです。##Jonathan Baronの解説でのaov関数を使うコード (少し修正を加えました) #データ生成 data1<-c(49,47,46,47,48,47,41,46,43,47,46,45,48,46,47,45,49,44,44,45,42,45, 45,40,49,46,47,45,49,45,41,43,44,46,45,40,45,43,44,45,48,46,40,45,40,45,47,40) ### 交互作用ありにしたいときのデータ (これは私が加えたものです) ####data1<-c(49,10,46,47,48,47,41,46,43,5,46,5,48,46,47,45,100,44,44,45,42,45, ####45,40,49,46,47,45,49,45,41,43,44,46,100,40,45,43,44,45,48,46,40,45,10,45,10,40) ####data1<-c(49,10,46,47,48,47,41,46,43,5,46,5,48,46,47,45,100,44,44,45,42,45, ####45,40,49,46,47,45,49,45,41,43,44,46,100,40,49,10,46,47,48,47,41,46,43,5,46,5) ## 行列化 (データ確認のため) matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2"))) # aov用のデータフレーム作成 Hays.df <- data.frame(rt = data1, subj = factor(rep(paste("subj", 1:12, sep=""), 4)), shape = factor(rep(rep(c("shape1", "shape2"), c(12, 12)), 2)), color = factor(rep(c("color1", "color2"), c(24, 24)))) # 非釣合型のデータにするため、grp変数を付け加える Hays.df$grp <- factor(rep(c(1,1,1,1,1,1,1,1,2,2,2,2), 4)) # aovで分析 aovres <- aov(rt ~ grp*color*shape + Error(subj/(color+shape)), data=Hays.df) summary(aovres) ## anovakun (井関先生の分散分析関数) でも検証するためにデータフレームに ## 分散分析関数を読み込めるようにしないと動作しません。 datkun <- data.frame(matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2")))) grp <- c(1,1,1,1,1,1,1,1,2,2,2,2) datkun <- data.frame(cbind(grp, datkun)) # anovakunで分析 anovakun(datkun, "AsBC", 2, 2, 2, type2=T)以上の分析で、結果が一致したため、aov関数の平方和について疑問を抱いたしだいです。
どうぞよろしくお願いいたします。
maru (2009-09-05 (土) 10:40:39)
過去の売り上げから今後の売り上げ予測とか
このような時系列の予測が出来るのでしょうか?
INA (2009-09-04 (金) 23:58:36)
みなさま、どうぞよろしくお願いいたします。
2つ以上の引数を用いる関数(corなど)を、tapplyで扱えずに苦心しております。
ある産業分類(200種類くらい)に含まれる個店データ約20万件があり、以下のような構造です。
DataSet
個店ID,産業分類,従業者数,販売額,設立年
000001,01_パン小売業,10,10000,11
000002,09_豆腐小売業,4,1200,2
000003,14_金物小売業,20,25000,4
: : : : :
これらを「産業分類別に」分類,集計しようと考えています。
その際、tapply関数を用いて、各産業ごとの集計を簡単にしようと考えました。
例えば産業別の販売額合計については、> tapply(DataSet$販売額,DataSet$産業分類,sum)で、産業分類別に問題なく集計できました。
しかし「販売額と従業者数の相関関係」を見ようとした場合、
>cor(DataSet$従業者数,DataSet$販売額)
のように、複数の引数を指定する必要があります。
これをtapplyを用いて、産業分類別に処理しようとしたところ、うまくいかないのです。
たとえば> tapply(DataSet,DataSet$産業分類,cor(DataSet$従業者数,DataSet$販売額)) 以下にエラー match.fun(FUN) : 'cor(DataSet$従業者数,DataSet$販売額)' は関数、文字、またはシンボルではありません > tapply(list(DataSet$従業者数,DataSet$販売額),list(DataSet$産業分類),cor) 以下にエラー tapply(list(UDataSet$従業者数,DataSet$販売額), : 引数は同じ長さでなければなりませんのようになります。
産業分類が多種類であることや、データが頻繁に追加・削除されることから、相関係数等を求める操作をなるべく簡単にしておきたいのです。
version 2.9.2、WinXP SP3上で動作中です。
なにぶんにも未熟でございまして、よい方法をご存知の方、お手数とは存じますが、どうぞよろしくお願いいたします。
sapply(split(DataSet, DataSet$産業分類), function(x) cor(x$従業者数, x$販売額))
sapply(split(DataSet[,3:4],DataSet[,2]), function(x) cor(x)[1,2])
sapply(levels(iris$Species), function(x) {
y <- iris$Species==x
cor(iris$Petal.Width[y], iris$Petal.Length[y])
})akira (2009-09-01 (火) 19:30:02)
いつもありがとうございます。
ベクトルで「死」を指定するとエラーになるのですが、対処法はありますでしょうか?WinXP R-2.9.1 Patchedの場合 Rgui.exeならOK >c("死") #OK R.exeだとエラー >c("死") #NG c(invalid multibyte character in mbcs_get_next +) + Ubuntu9.04 R-2.9.1 の場合 >c("死") #OKとなります。
R.exeだけで生じます。エンコードとか関係するのでしょうか?
とりあえず、Ubuntuで対応できますが、少し気持ち悪いです。
aor (2009-08-31 (月) 21:33:00)
初めて投稿させていただきます。
200行30列からなるデータフレーム (dat) の第4列から第24列までを因子変数にしたいと考えています。
以下のようなコードではうまくいきませんでした。factor(dat[4:24]) factor(dat[,4:24]) for (i in 4:24){ datf[i] <- factor(dat[i]) }上3つのコードは以下のような警告がでます。
以下にエラー sort.list(unique.default(x), na.last = TRUE) : 'x' は 'sort.list' に対してはアトミックでなければなりません。 'sort' をリストに対して呼び出しましたか? sapply(dat[,4:24], factor) sapply(dat[4:24], factor)上2つは警告は出ませんが、classがcharacterになります。
現在は以下のように、個々の変数にfactorを適用しています。dat[,4] <- factor(dat[,4]) dat[,5] <- factor(dat[,5] .... dat[,24] <- factor(dat[,24]これを範囲指定してまとめて行いたいのですが、よい方法をご存知の方、よろしくお願いいたします。
なお、R2.9.1, WindowsVista環境です。
set.seed(123) # 再現性のあるデータを作るため
dat <- data.frame(matrix(sample(4, 6000, replace=TRUE), 200)) # テスト用のデータフレーム
datf <- dat # 変換後のデータフレーム
for (i in 4:24) {
datf[,i] <- factor(dat[,i]) # カンマの有無に注意
}
sapply(datf[,4:24], class)
これも基本です。データフレーム x において,x[1] と x[,1] には違いがあり,x[1:2] と x[1:2] には違いがない。x[1] でないと駄目な場合,x[,1] でないと駄目な場合,どちらでもよい場合という3つの場合があるので注意。たとえば,上の datf[,i] は datf[i] でもよいなど。sapply などを使う場合も同じように,どの記述法でなければならないかに注意。set.seed(123) # 再現性のあるデータを作るため dat <- data.frame(matrix(sample(4, 6000, replace=TRUE), 200)) # テスト用のデータフレーム datf <- dat # 変換後のデータフレーム datf[4:24] <- lapply(dat[4:24], factor) sapply(datf[4:24], class)
なんび (2009-08-31 (月) 17:33:24)
Metaforを使って、メタアナリシスを行い、フォレストプロットを描くのに、
forest(xxxx) というコマンドではデータが下からプロットされ、
自動的に「study1」「study2」と試験名がふられますが、
データを上からプロットさせる方法はありますか。
「study1」ではなく、自分のデータ(SOC)を描きいれる方法がありますか。
以下のようなプログラムを作っています。
library(metafor)
dat<-scan("./zom.prn",
list(SOC="",ai=0,n1i=0,ci=0,n2i=0),
skip=0,nlines=30,sep="")
zompeto<-rma.peto(ai,n1i,ci,n2i, slab=NULL, subset=NULL, data=dat,add=c(1/2,0), to=c("only0","none"), level=95, digits=4)forest(zompeto)
> dat<-scan("./zom.prn",
+ list(SOC="",ai=0,n1i=0,ci=0,n2i=0),
+ skip=0,nlines=30,sep="")
以下にエラー file(file, "r") : コネクションを開くことができません
追加情報: Warning message:
In file(file, "r") :
ファイル './zom.prn' を開くことができません: No such file or directory
+ zompeto<-rma.peto(ai,n1i,ci,n2i, slab=NULL, subset=NULL, data=dat,
+
+ add=c(1/2,0), to=c("only0","none"), level=95, digits=4)
以下にエラー eval(expr, envir, enclos) : オブジェクト 'ai' がありません
+
> forest(zompeto)
以下にエラー forest(zompeto) : オブジェクト 'zompeto' がありません
例えば,forest.default の example についていえば,データを逆順に並べ直せば,下から描かれるとちょうどあなたの希望通りになるでしょう。また,ラベルも,自分で定義すればその通り描かれると思いますよ。
forest.default の example の実行結果と,以下のコードの実行結果を比べてみましょう。ね。
dat.bcg2 <- dat.bcg[rev(dat.bcg[,1]),] # 大元のデータを逆順に並べる
dat <- escalc(measure="RR", ai=tpos, bi=tneg, ci=cpos, di=cneg, data=dat.bcg2)
slab <- paste(dat.bcg2$author, dat.bcg2$year)
slab[13] <- "Foo Bar 2009" # ラベルの書き換えというか,データに自分のものを加えればよいだけでは?
forest(dat$yi, dat$vi, slab=slab,
atransf=exp, xlab="Risk Rates (log scale)")x <- data.frame(a=c(5,2,3,2,1), b=c(4,3,2,1,5)) y <- x[nrow(x):1,] # とすればよい実際にやって調べて見るなり。データの扱い方の基本あるよ。
taipapa (2009-08-23 (日) 21:45:22)
お世話になります.
次のようなデータを分析してます.コントロール28人,疾患群25人で,各人は脳画像の4ヶ所(A,B,C,D)からXというデータを取ります.知りたいのは,コントロールと疾患群でXの値が違うかどうか,違うならA,B,C,Dのうちどこの場所でちがっているのか,ということです.治療の有無はC or Pで,場所はlocationで表し,各個人はsubjectIDを振ってます.また,Xは0から1の間の値を取ります.> madata CorP X location subjectID 1 control 0.708 A 1 2 control 0.648 A 2 3 patient 0.638 C 3 4 control 0.547 D 4 5 patient 0.632 B 5 6 control 0.723 C 6 ......というかんじです.
各A,B,C,Dは同一人物の場所なので,subjectIDでrandom effectとし(こういう表現で良いか分かりませんが),以下のような線形混合モデルを2つ作成しました.model1 <- lme(X ~ CorP*location, random= ~ 1| subjectID, data) model2 <- lme(X ~ CorP*location, random= ~ location| subjectID, data)基本的なことですが,書式がよく分からず,私の想定したモデル(同一人物からのXの値はlocationが異なってもfixed effectではなくrandom effectとする)を正しく表しているのはどちらなのでしょうか? model2だとおもっているのですが...
また,defaultでは,restricted log-likelihoodの最大化を行うようになっていますが,私が見た資料ではlog-likelihoodの最大化を指定していました.この使い分けはどうすれば良いのでしょうか?
根本的に間違っているところもあるかもしれませんが,ご教示いただければ有り難いです.
ID CorP A.X B.X C.X D.X 1 control 0.708 0.648 0.648 0.547 2 control 0.648 0.632 0.632 0.723 3 patient 0.638 0.708 0.708 0.632 4 ***
dodoria (2009-08-20 (木) 02:34:47)
順序ロジスティック回帰や多項ロジスティック回帰で、変量効果は指定できるのでしょうか??
Hiro (2009-08-16 (日) 02:02:40)
パッケージ"gdata"をロードしたいのですが、
パッケージインストーラーでインストールして、
パッケージマネージャでロード済みをチェックしても、
以下の出力が表示されてロードすることができません。
library(gdata)
Error in loadNamespace(i1L, c(lib.loc, .libPaths())) :'gtools' という名前のパッケージはありません エラー: 'gdata' に対するパッケージもしくは名前空間のロードが失敗しました
これはどういった不具合なのでしょうか。
ご回答いただければと幸いです。
beerdog (2009-08-14 (金) 23:16:42)
はじめまして。Windows版のR2.9.1を現在は使っています。
内蔵のエディタで、タブのコントロールはできるのでしょうか?
デフォルトで変な(?)桁数のようなので変更したいのですが。
教えていただけますでしょうか?
Gyo (2009-08-14 (金) 21:02:56)
いつもお世話になっております。
Rの超初心者なのですが、グラフィックの方法について、
質問させていただけますでしょうか。
以前のnekoさんのご質問内容に似ているのですが、、
latticeパッケージにあるwireframe関数で画像を描画した後、
その底面に同じ座標軸で図を描写したいのですが、
なにか方法はありますでしょうか?
lattice本のスクリプト集、以前のnekoさんへの回答内容を参考にさせていただいたのですが、
wireframe関数の場合、どのようにしたらいいか分かりませんでした。
ご教授いただければ幸いです。よろしくお願いいたします。wireframe(volcano, drape=T, col.regions=rgb(1, 0, 0, 0.1), col=rgb (1,1,1,0), panel = function(x, y, ..., subscripts){ panel = panel.wireframe(x, y, ..., subscripts) panel.points(20,20,col=2,cex=2) #多分ここに何かが入ると思うのですが。。 })Rは、
R version 2.9.1
i386-pc-mingw32
で、windowsXPを使用しております。
どうぞよろしくお願い致します。
> panel.3d.contour <-
function(x, y, z, rot.mat, distance,
nlevels = 20, zlim.scaled, ...)
{
add.line <- trellis.par.get("add.line")
panel.3dwire(x, y, z, rot.mat, distance,
zlim.scaled = zlim.scaled, ...)
clines <-
contourLines(x, y, matrix(z, nrow = length(x), byrow = TRUE),
nlevels = nlevels)
for (ll in clines) {
m <- ltransform3dto3d(rbind(ll$x, ll$y, zlim.scaled[2]),
rot.mat, distance)
panel.lines(m[1,], m[2,], col = add.line$col,
lty = add.line$lty, lwd = add.line$lwd)
}
}
> wireframe(volcano, zlim = c(90, 250), nlevels = 10,
aspect = c(61/87, .3), panel.aspect = 0.6,
panel.3d.wireframe = "panel.3d.contour", shade = TRUE,
screen = list(z = 20, x = -60))
詳細は省きますが,この ltransform3dto3d()関数でzlim.scaled[1]と変更して作成されたグラフを参考にしてください.後はltransform3dto3d()関数のヘルプを丁寧に読んで,自力で解決してください.ちなみにSarkarの原書は,『Rによる多変量グラフィックス入門』などと言う題名で,近くシュプリンガー社より翻訳が出版されます(正確な時期はまだ分かりません).EPS (2009-08-11 (火) 21:36:13)
Cox比例ハザードモデルで幾つかの独立変数(20程度)について単変量解析を行う予定です。
dataframeA B C D F G duration status1 88 50 43 143 72 14 12 0
2 130 86 40 98 64 8 16 1
3 138 100 28 122 72 11 18 1一つ一つのパラメータについてresult.A<-coxph(Surv(duration,statsu)~A, dataframe)と言ったように解析を繰り返しているのですが、それぞれの独立変数の単変量の結果を一括して出力する方法はないのでしょうか?
for文やsapply関数を用いれば可能なのでしょうか?
ご教授いただければ幸いです。よろしくお願いいたします。
EDI (2009-08-08 (土) 01:58:58)
いつも、setwd()をつかって、使っているコンピュータ上に作業ディレクトリーを作り、その中にあるプログラムファイルを動かし、同じディレクトリーに結果を保存しています。
しかし、頻繁に別のコンピュータで作業したりしているので、これに不便を感じています。
そこで、ネットワーク上の別のコンピュータに保存されているプログラムを使ったり、結果を書き出したり、する方法が分かれば大変助かると思っています。
質問としては、作業ディレクトリーとして同じネットワーク上の別のコンピュータのディレクトリーを指定する方法のやり方を教えていただけないでしょうか。
Saito (2009-08-04 (火) 16:11:47)
いつもお世話になっております。
ヘルプドキュメント、RSiteSearch()、ネット検索しましたが、見当たらないので質問させてください。環境はWindowsXP、R-2.9.1です。
回帰をするときのオフセット項について質問です。普通、オフセットは係数が推定されない項であるので、Rの例えばglm()だとglm(y ~ 1, offset=foo) glm(y ~ 1 + offset(foo))などとすると思います。しかし、使っている関数によっては引数でoffsetがついていないときがあります。例えば、私は今library(spBayes)のspGLM()という関数を使って解析をしていたのですが、引数にoffsetが見当たりません。そこで、offset()を使って(上記の二つ目のコードのように)オフセットを考慮しようとしたのですが、全く考慮されないようです。以下にサンプルコードをしめします。
library(spBayes) library(MASS) ##Generate binary data n <- 50 coords <- cbind(runif(n,1,100),runif(n,1,100)) phi <- 3/50 sigma.sq <- 1 R <- sigma.sq*exp(-phi*as.matrix(dist(coords))) w <- mvrnorm(1, rep(0,n), R) x <- as.matrix(rep(1,n)) beta <- 0.1 p <- 1/(1+exp(-(x%*%beta+w))) y <- rbinom(n, 1, prob=p) ##Collect samples beta.starting <- coefficients(glm(y~x-1, family="binomial")) beta.tuning <- t(chol(vcov(glm(y~x-1, family="binomial")))) n.samples <- 50000 set.seed(1) #オフセットなしの場合 m.1 <- spGLM(y~1, family="binomial", coords=coords, starting= list("beta"=beta.starting, "phi"=0.06,"sigma.sq"=1, "w"=0), tuning= list("beta"=beta.tuning, "phi"=0.1, "sigma.sq"=0.1, "w"=0.01), priors= list("beta.Normal"=list(0,10), "phi.Unif"=c(0.03, 0.3), "sigma.sq.IG"=c(2, 1)), cov.model="exponential", n.samples=n.samples, sub.samples=c(25000,n.samples,10), verbose=TRUE, n.report=500) > print(summary(mcmc(m.1$p.samples))) Iterations = 1:2501 Thinning interval = 1 Number of chains = 1 Sample size per chain = 2501 1. Empirical mean and standard deviation for each variable, plus standard error of the mean: Mean SD Naive SE Time-series SE (Intercept) -0.571 0.4431 0.008861 0.04042 sigma.sq 1.138 0.6225 0.012448 0.07147 phi 24.928 18.0839 0.361605 2.17077 2. Quantiles for each variable: 2.5% 25% 50% 75% 97.5% (Intercept) -1.3557 -0.8788 -0.6007 -0.2938 0.3712 sigma.sq 0.1825 0.6351 1.1654 1.5494 2.3773 phi 10.0896 11.9748 17.1254 32.0178 77.8743 #オフセットを考慮した場合 m.1 <- spGLM(y~1 + offset(rep(1, length(y))), family="binomial", coords=coords, starting= list("beta"=beta.starting, "phi"=0.06,"sigma.sq"=1, "w"=0), tuning= list("beta"=beta.tuning, "phi"=0.1, "sigma.sq"=0.1, "w"=0.01), priors= list("beta.Normal"=list(0,10), "phi.Unif"=c(0.03, 0.3), "sigma.sq.IG"=c(2, 1)), cov.model="exponential", n.samples=n.samples, sub.samples=c(25000,n.samples,10), verbose=TRUE, n.report=500) > print(summary(mcmc(m.1$p.samples))) Iterations = 1:2501 Thinning interval = 1 Number of chains = 1 Sample size per chain = 2501 1. Empirical mean and standard deviation for each variable, plus standard error of the mean: Mean SD Naive SE Time-series SE (Intercept) -0.571 0.4431 0.008861 0.04042 sigma.sq 1.138 0.6225 0.012448 0.07147 phi 24.928 18.0839 0.361605 2.17077 2. Quantiles for each variable: 2.5% 25% 50% 75% 97.5% (Intercept) -1.3557 -0.8788 -0.6007 -0.2938 0.3712 sigma.sq 0.1825 0.6351 1.1654 1.5494 2.3773 phi 10.0896 11.9748 17.1254 32.0178 77.8743となり、offset()を入れても入れなくても、結果が同じになってしまうようです。このような個々のパッケージについて質問してしまって申し訳ありません。もし、どなたか、解決方法をご存知でしたら、こ教示願えれば幸いです。
どうぞよろしくお願い致します。
(2009-08-03 (月) 17:55:38)
data.frameは長さが同じものを結合するときにしか使えません。
長さが違う場合はどうすればよいですか
経過月数 品名a 品名b --- 品名Z
1 0 1 --- 2
2 1 3 --- 5
: : : --- :
N 2 NA --- NA
N+1 0 NA --- NA
まあ、こんな風。-- 河童の屁 2009-08-03 (月) 22:56:11gonzu (2009-07-28 (火) 17:06:17)
パッケージspatstatのK関数を何回か繰り返して求めて、その結果をまとめてグラフに表そうとしています。試に5000cm×5000cmのプロット上に、乱数を発生させて円を作り、5回計算を繰り返すようにしました。
しかし、for(i in 1:5) { w<-disc(1000,c(runif(1)*5000,runif(1)*5000),mask=FALSE) plot(w) xy<-tennenrin.csv pp<-as.ppp(xy,w,fatal=FALSE) K<-Kest(pp,correction="isotropic") } matplot(K,type="l")このようにすると、5本ではなく1本のラインしかグラフに表れず、困っています。何か方法がありましたら、ご教授お願いします。
使用環境は> sessionInfo() R version 2.7.2 (2008-08-25) i386-pc-mingw32です。よろしくお願いします。
mymonte<-for(i in 1:10){
X<-runif(704)*50
Y<-runif(704)*50
XY<-data.frame(X,Y)
XY<-data.frame(XY,Dataset[,c(2,12)])
w<-owin(c(0,50),c(0,50))
Rpp<-as.ppp(XY[,1:2],w,fatal=FALSE)
mark1<-XY$DBH.02.
mark2<-XY$sort
Rpp1<-Rpp %mark% mark1
Rpp2<-Rpp1 %mark% mark2
KC<-Kmulti(Rpp2,(mark1<=5)&(mark1>=1)&(mark2=="ヤブムラサキ"),(mark1>5)& (mark2=="シラキ"),
correction="isotropic")
par(new=T)
plot(KC, sqrt(iso/pi)-r ~ r,ylim=c(-5,5)) # L関数のグラフ
}森林 (2009-07-28 (火) 11:47:09)
テキストファイルの1行目にあるタイトルを読み込み、図などに表示したいのですが、 データは、dat <- read.table(fname, header=T,skip=1)とすればよいけれど、タイトルのみを読むために、
title<-read.table(fname,nrows=1)
とすれば、
titleV11 1号試験地
となります。そこで、
text(x,y,title)
とすると、"1"のみが表示され、"1号試験地"が表示されません。
もっと適切な命令があると思いますが、R-tipsには記載されていないので、おたずねします。よろしくお願いします。
taipapa (2009-07-26 (日) 18:47:05)
初めて投稿します.説明変数を脳の画像上のある値(x1,x2,x3,x4と一人から複数箇所)とし,応答変数を高次脳機能検査(WAISRなど複数,仮にy1,y2,y3,y4)としてその関係を調べようとしています.すべて連続量です.応答変数がひとつなら重回帰で良いかと思うのですが,応答変数も複数の場合はどのような手法が最善なのでしょうか?現在は,それぞれの高次脳機能検査の結果をひとつずつ応答変数として,複数の画像上の値を説明変数としてlmで重回帰をやり,stepAICやwleで,最小のAICを求めて最善モデルを出してます.アドバイスいただければ幸いです.
tatsu (2009-07-25 (土) 23:35:19)
いつもありがとうございます。本当に初心の初心者の質問ですが。ご存知のとおり、円グラフを描くにはpie(x)というコードを使いますが、グラフ上に実際の割合値を載せることは出来ますか。(ラベルなら表示出来ますようですが)よろしくお願いします。
> x <- 1:5 > y <- letters[1:5] > z <- paste(paste(y,round(100*x/sum(x),1),sep=" "),"%",sep="") > z [1] "a 6.7%" "b 13.3%" "c 20%" "d 26.7%" "e 33.3%" > pie(x,labels=z)
x <- 1:5
z <- sprintf("%.1f", 100*x/sum(x))
pie(x,labels=z)Saito (2009-07-23 (木) 22:09:38)
いつもお世話になっています。似た質問や説明ページがないか捜したのですが見当たらなかったので質問させてください。
例えば、f(x, y)=exp(0.2*x + 0.3*y + sin(2*x) + cos(y) + 1)といった形の関数をxは0〜20まで、yは0〜10まで積分して、その体積を求めようと思ったらどのようにすればよいのでしょうか。以下のようなコードでは動きません。> library(adapt) > f <- function(x, y){ > + exp(0.2*x + 0.3*y + sin(2*x) + cos(y) + 1) > + } > adapt(2, lo=c(0, 0), up=c(10, 20), functn=f) 以下にエラー function (x, y) : 要素 2 は空です; '*' の引数リストの評価されていた部分は: (0.3, y)
integrateやlibrary(adapt)、library(MASS)に含まれるadaptやareaは勉強したのですが、いまひとつ使いこなせません。もしどなたかこのような二重積分の方法がわかる方がいましたら、ご教授していただけないでしょうか。
なお、環境はR-2.9.1、WindowsXPです。
どうぞよろしくお願い致します。
f <- function(x){
exp(0.2*x + 0.3*x + sin(2*x) + cos(x) + 1)
}
adapt(4, lo=c(0, 0, 0, 0), up=c(10, 20, 10, 20), functn=f)
これでも答えは出ますが、間違っている気がします。値がものすごく大きいです…。
えーっと…、まだ読み落としている引数があるのでしょうか…。読み落としている箇所だけでも教えていただけないでしょうか…。-- Saito 2009-07-24 (金) 11:34:52f <- function(X){
x <- X[1]
y <- X[2]
exp(0.2*x + 0.3*y + sin(2*x) + cos(y) + 1)
}
adapt(2, lo=c(0, 0), up=c(10, 20), functn=f)
むむ、合っているのか不安です…。二次元以上になると正解例を作るのもスキル不足で巧くいきません…。検算ができていないので、どなたか分かる方がいましたらこれで合っているのかご教授していただけると幸いです。-- Saito 2009-07-24 (金) 15:53:12> f1 <- function(x) x
> (x1 <- integrate(f1,0,1))
0.5 with absolute error < 5.6e-15
> f2 <- function(x) x[1]*x[2]
> (x2 <- adapt(2,c(0,0),c(1,1),funct=f2))
value relerr minpts lenwrk ifail
0.25 7.38275e-08 165 73 0gonzu (2009-07-22 (水) 15:56:20)
はじめまして。
パッケージspatstatでのK関数の繰り返しについて質問させていただきます。
使用環境はR version 2.7.2 (2008-08-25) i386-pc-mingw32です。
データセットに5000cm×5000cmプロット内の樹木の位置情報(x,y)を持つtennenrin.csvを入れてw<-owin(c(0,1000),c(0,1000)) plot(w) xy<-tennenrin.csv pp<-as.ppp(xy,w,fatal=FALSE) K<-Kest(pp) plot(pp) plot(K)このようにするとプロット内の左下1000cm×1000cm内でのK関数が算出されるのですが、このwindowをランダムに発生させる方法がわかりません。
試しに同じ面積のwindowを作ろうとして、Excelで乱数を999組発生させてRにデータとして取り込み、そこからwindowの(x,y)の抽出を繰り返そうとしてみましたがmeasurement<-read.delim("clipboard") measurement > n<-999; m<-1 > x<-1:n > sample(x,m,replace=TRUE) [1] 469となりうまく(x,y)で抽出することができません。どなたかご教授お願いします。
P <- sample(0:4000, 2, rep=TRUE) w <- owin(P[1]+c(0,1000),P[2]+c(0,1000))
mito (2009-07-22 (水) 15:11:08)
いつも丁寧に教えてくださりありがとうございます。
heatmapである物質とある物質の反応値を書いていてrecallとprecisionを
出したいのでheatmapの中の行と列ごとの木を必要なグループ数、もしくは分割の高さを与えて、幾つかのグループに分割したいと思っています。
hclustの場合はcutree()で出来たのですが、ヒートマップだと中でこのhclustが使われていることは分かったのですが、ここからどう取り出せば良いのか分かりません。hclust()内にheatmapの
時と同じ変数を入れても並び順が異なっているので困っています。(ヘルプをみるとheatmapは平均値を利用していることと、行と列とでクラスタリングをしているからだと思うのですが・・・)
ちなみに、ヒートマップはmixmix <- read.table("mixmix.txt",header=TRUE,sep="\t",row.names=1) mixmix_numeric <- apply(mixmix,c(1:2),as.numeric) mixmix_matrix <- matrix(mixmix_numeric,nrow=377,ncol=291, dimnames=list(rownames(mixmix_numeric), colnames(mixmix_numeric))) mixmix_scale <- scale(mixmix_matrix) mixmix_scale_numeric <- apply(mixmix_scale,c(1:2),as.numeric) mixmix_scale_euclid <- dist(mixmix_scale_numeric,method="euclid") pdf("hyoujunka_honto.pdf",paper="special",width=37,height=35,pointsize=5) heatmap(mixmix_scale_numeric,hclustfun=(function(mixmix_scale_euclid) hclust(mixmix_scale_euclid,method="ward")), col=c("#000000","#a31782","#2353a9","#00a765","#5ac493","#bbdd22","#fff300", "#ff8d00","#fb4000","#fc7574")) dev.off()で書いています。
また> sessionInfo() R version 2.8.1 (2008-12-22) i386-apple-darwin8.11.1です。よろしくお願いします。
桃太郎 (2009-07-17 (金) 16:38:54)
はじめまして。
超初心者の質問ですみません。
パッケージインストールがうまくいかず困っています。
環境は> sessionInfo() R version 2.7.0 (2008-04-22) i386-pc-mingw32です。パッケージ"gstat"を使用したくインストールし読み込んだところ
>library(gstat)~ エラー: パッケージ 'sp' が 'gstat' によって要求されましたが、見つけられませんでした ~ 追加情報: Warning message: パッケージ 'gstat' はバージョン 2.7.2 の R の下で造られましたと返ってきてしまいました。
パッケージ'sp'がインストールされていないようでしたので「パッケージのインストール」からサイトミラーを選択しspのインストールを行うと>local({pkg <- select.list(sort(.packages(all.available = TRUE))) + if(nchar(pkg)) library(pkg, character.only=TRUE)}) > utils:::menuInstallPkgs() URL 'ftp://ftp.ecc.u-tokyo.ac.jp/CRAN/bin/windows/contrib /2.7/sp_0.9-28.zip' を試しています ftp data connection made, file length 844848 bytes 開かれた URL downloaded 825 Kb パッケージ 'sp' は無事に開封され、MD5 サムもチェックされました 警告: パッケージ 'sp' の前のインストールを取り除くことが出来ませんでした ダウンロードされたパッケージは、以下にありますと警告が返ってきてしまい、うまくインストールが出来ません。
何が原因なのでしょうか?
また"sp"がインストールされれば"gstat"は読み込めるのでしょうか?
どなたかご教授よろしくお願いいたします。
mona (2009-07-15 (水) 18:47:18)
はじめまして。
Rの棒グラフの書き方について質問させてください。
正の数値をbarplotで水平方向に表示させると、
x軸の0から棒が伸びていきますが、
この伸びていく原点を変更したいのです。
たとえば10をその原点にとり水平方向に棒グラフを書くと、
データが5の場合は左方向に、15の場合は右方向に
10から棒が伸びていくように描きたいと思っています。
どなたかご教授よろしくお願いします。
shift <- 10 ; barplot(c(0,5,10,15,20)-shift, horiz=T, offset=shift)
neko (2009-07-14 (火) 19:18:50)
はじめまして。
Rの超初心者なのですが、描画の方法について、質問させてください。
levelplot関数で画像を描画した後に、更に重ねて描画したいのですが、どのような方法があるのでしょうか?
どなたか教えていただけますでしょうか。
使用環境は、sessionInfo() R version 2.8.1 (2008-12-22) i386-pc-mingw32です。どうぞよろしくお願い致します。
> library(lattice)
> levelplot(volcano, panel = function(x,y,..., subscripts)
{
panel = panel.levelplot(x, y, ..., subscripts)
panel.points(20,20,col=2,cex=2 )
})
Latticeの訳本,図版のレイアウトの関係でちょっと手間取っているようです.とにかく図版の数が多いので.> 駅前旅館さんichigo (2009-07-14 (火) 18:37:07)
追加で質問させていただきます
東京・埼玉のシェープファイルをダウンロードし、ワーキングディレクトリに配置します.library(maptools) tokyohx <- readShapeSpatial("tokyohk.shp") plot(tokyohx) saitamax <- readShapeSpatial("saitamak.shp") plot(saitamax, add=T)隣り合う複数の県(東京、埼玉、神奈川など)を考え,任意の点を与えた時に,その内部の地点かどうかを判断したいと思っています.
下の「in.polygon」関数では、頂点や境界上の点はFALSEを返すので、東京と埼玉の境界上にある点は、東京でも埼玉でもないと判断されてしまいます.
このような場合、東京と埼玉をあわせた外枠?を取得すれば上記のような現象は発生しないと思うのですが、
二つのシェイプデータからそのようなデータを作成することは可能でしょうか?
また、日本全土のデータ(japank.shp)を用いれば、複数の県をまとめて扱う事ができるかとも考えたのですが、
日本全土のデータから、任意の点をを東京・埼玉の内部かどうかを判断する事は可能なのでしょうか?
どうぞよろしくお願いいたします.
ichigo (2009-07-14 (火) 12:32:07)
ShapeFileライブラリの「ShapeFileデータ(地域境界・地域メッシュ)」を ありがたく使わせていただいております.
いろいろ調べていたところ、「R Book」に解説が載っているRmapライブラリは
Version2.0以上には対応していないようなので、maptoolsを使ってごにょごにょやろうとおもっております.上記ページから東京の島無しのシェープファイルをダウンロードしてワーキングディレクトリに配置します.
library(maptools) tokyohx <- readShapeSpatial("tokyohk.shp") plot(tokyohx)これで東京の外枠が描画できます.
さて、質問なのですが、ある任意の地点の緯度経度を与えてその地点が東京都内部の地点なのか
どうかを判定することがこのデータを使ってできますでしょうか?
できるとすればどのような処理内容になるでしょうか? もしよければご回答くださると助かります.
例えば以下のような緯度・経度です.> chiten_x <- c(35.677822, 139.771458) > chiten_x [1] 35.67782 139.77146 > sessionInfo() R version 2.8.1 (2008-12-22) i386-pc-mingw32 locale: LC_COLLATE=Japanese_Japan.932;LC_CTYPE=Japanese_Japan.932; LC_MONETARY=Japanese_Japan.932;LC_NUMERIC=C;LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base other attached packages: [1] maptools_0.7-25 sp_0.9-36 foreign_0.8-29 loaded via a namespace (and not attached): [1] grid_2.8.1 lattice_0.17-17よろしくお願いします.
tokyohx@polygons[[1]]@Polygons[[1]]@coords
tmp <- data.frame(tokyohx@polygons[[1]]@Polygons[[1]]@coords) library(sgeostat) in.polygon(139.771458, 35.677822, tmp$X1, tmp$X2) [1] TRUE in.polygon(139.771458, 40.677822, tmp$X1, tmp$X2) [1] FALSE
point.in.polygon(139.771458, 35.677822, tmp$X1, tmp$X2)
nosyan (2009-07-13 (月) 16:12:45)
Rでavi,wmvなどの動画ファイルを読み込む方法はありますでしょうか?
パッケージを探してみましたが、該当するものが見つからなかったので、ここで質問しました。
(2009-07-13 (月) 10:17:34)
こんにちは。
とても基本的なことで質問させてください。
使っているものはR version 2.6.2 (2008-02-08) です。
test.tableを使おうとすると
(a<-test.table(*****))
エラー: 関数 "test.table" を見つけることができませんでした
とでてきます。
パッケージなどをインストールしなおしたりしたのですが、
どうしても見つからないとエラーがでてしまいます。
対処法をご存知の方がいらっしゃいましたらよろしくお願いします。
名無しさん (2009-07-13 (月) 03:59:00)
いつもお世話になっております。
昔使っていたC言語のくせがとれず、"Rらしい"書き方がまだできないでいる初心者です。
以下のようなプログラムを"Rらしく"書くにはどのようにしたらよいでしょうか?
例えば、[1,] 1 2 4という長さ3のベクトルvecがあったときに
[,1] [,2] [,3] [1,] 0 1 9 [2,] 1 0 4 [3,] 9 4 0各ベクトル要素の差の2乗を求めた(添え字が対応している)3*3のマトリックスmatを求めるとすると、
for(i in 1:3){~ for(j in 1:3){ mat[i,j] <- ( vec[i] -vec[j] ) ^ 2 } }とすると求まりますが、とてもC言語的です。
Rらしい書き方の例を教えてください。
お願いします。
> outer(vec,vec,FUN="-")
[,1] [,2] [,3]
[1,] 0 -1 -3
[2,] 1 0 -2
[3,] 3 2 0
> outer(vec,vec,FUN="-")^2
[,1] [,2] [,3]
[1,] 0 1 9
[2,] 1 0 4
[3,] 9 4 0
# いっぺんで済ませたかったら
> ds = function(x,y) (x-y)^2
> outer(vec,vec,FUN=ds) # outer(vec,vec,FUN=function(x,y) (x-y)^2) でも可
[,1] [,2] [,3]
[1,] 0 1 9
[2,] 1 0 4
[3,] 9 4 0onen (2009-07-09 (木) 16:34:26)
はじめまして。R超初心者です。調べてみてもわからなかったので書き込ませていただきます。
まず背景状況は以下の通りです。
OS→Mac OS X 10.5.7sessionInfo()
R version 2.9.0 (2009-04-17)
i386-apple-darwin8.11.1
locale:
ja_JP.UTF-8/ja_JP.UTF-8/C/C/ja_JP.UTF-8/ja_JP.UTF-8
attached base packages:
[1] stats graphics grDevices utils datasets methods
[7] base
以上。source('myfunc/myfunc.R')をやってみたのですが、
「構文解析中に不正なマルチバイト文字列がありました (1行) 」
と表示されてしまいます。
どなたか解決策ご教授お願いいたします。
レイン (2009-07-09 (木) 13:06:52)
はじめまして。R初心者でして調べてみてもわからなかったので書き込ませていただきます。
デンドログラムを作成して、クラスターを分類したのですが、各クラスターの中を占める語句を検索する方法はありますでしょうか?それぞれのクラスターがどのような語句を重要視してみたのかがわからないのです。
よろしくお願いします。
なんび (2009-07-08 (水) 17:26:29)
metaforをつかってメタアナリシスを行いたく、
丹後俊郎先生著「メタアナリシス入門」のデータを使ってシミュレーションしたところ、
オッズ比がマイナスの値となってでてきました。
どこがおかしいのかどうしてもわからないので教えてください。
structure(list(ai = c(3, 7, 5, 102), bi = c(35, 107, 64, 1431
), ci = c(3, 14, 11, 127), di = c(36, 102, 82, 1303)), .Names = c("ai",
"bi", "ci", "di"))
library(metafor)
dat<-scan("./blocker_abcd.txt",
list(ai=0,bi=0,ci=0,di=0),
skip=0,nlines=4,sep="")
blpeto<-rma.peto(ai,bi,ci,di, slab=NULL, subset=NULL, data=dat,add=c(1/2,0), to=c("only0","none"), level=95, digits=4)forest(blpeto)
パンチドランカー (2009-07-08 (水) 01:04:28)
Windows で、PHPの shell_exec 関数を利用して、Rを実行しているのですが、以下のように、大なり小なり記号があると、うまく実行できません。echo a=rnorm;b=a[a>=0.5]| rterm --slave代入"<="は"="に置き換えましたが、上の対策方法が分かりません。
どなたか、この解決法、ご教授していただけませんか?
% > echo 'a=rnorm(10);b=a[a>=0.5]; print(b)' | R --slave [1] 1.0513718 0.7390706 0.9937101
rscript -e "a<-rnorm(10);b<-a[a>=0.5]; print(b)"を実行したところ、OKでした。シングルクォテーションは駄目でした。 -- パンチドランカー 2009-07-09 (木) 21:05:23
Saito (2009-07-06 (月) 17:12:34)
いつもお世話になっております。
並列処理をしようと思うのですが、どうやら同一の乱数から結果を返すらしく、同じ数字が二度づつ出てきてしまいます。以下にサンプルコードを示します。library(snow) f <- function(n){ a <- rnorm(1, n, 1) return(a) } cl <- makeSOCKcluster(c("localhost", "localhost")) clusterExport(cl, "f") system.time(ff <- parSapply(cl, 1:10, function(x){f(1)})) >ff [1] -0.2169740 0.9034649 0.7919217 1.9640380 0.9430635 -0.2169740 [7] 0.9034649 0.7919217 1.9640380 0.9430635Rで並列計算を読み、rsprngというものが必要ということはわかりました。そこで上記サイトのリンク先からrsprngのzipファイルをダウンロードして、Rから読み込ませたのですが、
HTML パッケージ記述を更新 library(rsprng) 以下にエラー library(rsprng) : 'rsprng' は有効なインストール済みパッケージではありませんと出るだけで、rsprngが使えるようになった気配がありません。パッチを当てろと書いてあったので、パッチを探しましたが、zipファイルにどう当てるのか、当て方がわかりません。それにこれはMacのパッチのような気がします。どなたか、解決策をご存知の方がおりましたら、ご教授していただけないでしょうか。
なお、環境はWindowsXP、R-2.9.1、Core 2 Duoです。
どうぞよろしくお願い致します。
> library(snow)
> cl<-makeCluster(rep("localhost",2))
> clusterSetupRNG (cl,seed=123)
Loading required package: rlecuyer
[1] "RNGstream"
> clusterCall(cl,runif,2)
[[1]]
[1] 0.4340643 0.3185276
[[2]]
[1] 0.5240271 0.0738508
snowとrlecuyerを普通にインストールすれば上記は動作するでしょう.-- 2009-07-07 (火) 00:15:13f <- function(n){
a <- rnorm(1, n, 1)
b <- rnorm(1, n, 2)
return(list(a=a, b=b))
}
cl <- makeSOCKcluster(c("localhost", "localhost"))
clusterExport(cl, "f")
system.time(ff <- parSapply(cl, 1:10, function(x){f(1)}))
> t(ff)
a b
[1,] 0.8602452 -0.9136815
[2,] 1.220000 1.192357
[3,] 0.5558699 2.945972
[4,] -0.07316578 -0.5083378
[5,] 0.9687442 -1.506602
[6,] 0.8602452 -0.9136815
[7,] 1.220000 1.192357
[8,] 0.5558699 2.945972
[9,] -0.07316578 -0.5083378
[10,] 0.9687442 -1.506602
この結果をa列b列についてそれぞれ、例えば平均値を求めたいとします。これがデータフレームであれば、
apply(t(ff), 2, mean)とでもすれば出来るのですが、list型では一発ではできません。一応次のようにすればできますが、すごく手間がかかってしまいます。
> mean(c(t(ff)[[1]], t(ff)[[2]]))#これを全てデータで打ち込めば、平均値は出ますが…。 [1] 1.040123 > mode(as.data.frame(t(ff)))#データフレームにも直らないようです。 [1] "list"もし、どなたか、このようなlist型のデータでの要約の仕方をご存知でしたら、ご教授していただけないでしょうか。どうぞよろしくお願い致します。 -- Saito 2009-07-07 (火) 16:59:29
mito (2009-07-03 (金) 16:35:00)
また、初歩的な質問で申し訳ありません。
heatmapで使えるhclustは階層型クラスタリングだけなのでしょうか。
平均を利用して書く、ということで非階層型クラスタリングのk-meansは使えないと考えてよいでしょうか。
kouda (2009-06-29 (月) 16:51:47)
いつも丁寧な返信をありがとうございます。
早速質問させていただきたいのですが、
列ごとに上下1,2パーセント切った状態で標準化を行うにはどうすればいいでしょうか。
標準化はscale関数に入れれば、列ごとに標準化は行っているのですよね?
外れていそうな値が多いので上下1,2パーセント切って標準化したいのですが、どのような操作を行えばいいのでしょうか。
足りない情報等ありましたら、教えていただければと思います。
よろしくお願いします。
使用環境はsessionInfo()
R version 2.8.1 (2008-12-22)
i386-apple-darwin8.11.1
です。よろしくお願いします。
> set.seed(1234); A <- matrix(sample(1:10,30,rep=TRUE),10,3)
> A
[,1] [,2] [,3]
[1,] 2 7 4
[2,] 7 6 4
[3,] 7 3 2
[4,] 7 10 1
[5,] 9 3 3
[6,] 7 9 9
[7,] 1 3 6
[8,] 3 3 10
[9,] 7 2 9
[10,] 6 3 1
# 列毎に上下10%をNA値に置き換え
> B <- A; for(i in seq(ncol(A))) {
x <- B[,i]
y <- (x<= quantile(x,0.1) | x >= quantile(x,0.9))
B[y,i] <- NA}
> B
[,1] [,2] [,3]
[1,] 2 7 4
[2,] 7 6 4
[3,] 7 3 2
[4,] 7 NA NA
[5,] NA 3 3
[6,] 7 9 9
[7,] NA 3 6
[8,] 3 3 NA
[9,] 7 NA 9
[10,] 6 3 NA
> scale(B)
[,1] [,2] [,3]
[1,] -1.8267086 0.9950898 -0.4572988
[2,] 0.6089029 0.5761046 -0.4572988
[3,] 0.6089029 -0.6808509 -1.1686525
[4,] 0.6089029 NA NA
[5,] NA -0.6808509 -0.8129757
[6,] 0.6089029 1.8330602 1.3210855
[7,] NA -0.6808509 0.2540549
[8,] -1.3395863 -0.6808509 NA
[9,] 0.6089029 NA 1.3210855
[10,] 0.1217806 -0.6808509 NA
attr(,"scaled:center")
[1] 5.750000 4.625000 5.285714
attr(,"scaled:scale")
[1] 2.052873 2.386719 2.811541
> colMeans(scale(B), na.rm=TRUE)
[1] 0.000000e+00 2.775558e-17 7.930164e-17
> var(scale(B)[,1], na.rm=TRUE)
[1] 1
> var(scale(B)[,2], na.rm=TRUE)
[1] 1
> var(scale(B)[,3], na.rm=TRUE)
[1] 1ono (2009-06-26 (金) 17:23:36)
単純な質問で恐縮です。(使用環境はmac OS 10.5.7, R version 2.9.0)
NAを含む行列を除くのは、na.omitやsubsetを使えば出来るのですが、NAを含む行のみを抽出する仕方を見つけることができないでいます。
以下に、パッケージに含まれているattenuのデータを使って示します。
DATA=attenu # The Joyner-Boore Attenuation Data(5列、182行のデータ)
names(DATA)#stationの列にNAが入っている行を抽出したいが、以下は全て失敗例#subset(DATA, station==NA)
subset(DATA, station=="NA")
subset(DATA, station=="")
subset(DATA, station=="<NA>")
subset(DATA, as.character(station)=="NA")
本当に単純な問題で申し訳ないですが、ご教授いただけると幸いです。
subset(DATA, subset=is.na(DATA$station)) subset(DATA, subset=!complete.cases(DATA))
Saito (2009-06-25 (木) 16:24:31)
いつもお世話になっております。
研究で普遍クリギングを使う予定であります。私の手元にあるデータの説明変数となる値は場所が近いほど相関があり、かつ様々な要因から影響を受けているように見えます。そのため、要因と自己相関を考慮できるモデルを考えていたところ、普遍クリギングという単語に思い当たり、Rでどのようにやるのか試してみました。しかし、これで本当に普遍クリギングとなっているのか、調べてもわかりませんでした。以下にサンプルコードを示します。library(fields) r1 <- runif(20, 0, 1) r2 <- runif(20, 0, 1) r3 <- runif(20, 0, 1) Lat <- r1 Long <- r2 val <- rep(c(1, 2, 3, 4), 5) Val <- val + r3 Area <- rep(c(1, 2, 3, 4), each=5) m <- matrix(0, 20, 4) for(j in 1 : 4) { for (i in 1 : length(Area)) { if (j==Area[i]) { m[i, j] <- 1 } else { m[i, j] <- 0 } } } colnames(m)=c("1", "2", "3", "4") D <- data.frame(Lat, Long, Val, m)#これが解析で使うデータ K <- Krig(x=data.frame(D$Lat, D$Long, D$X2, D$X3, D$X4), Y=D$Val) coef(K)#推定された係数値 G <- glm(Val~Lat + Long + as.factor(Area)) coef(G)#GLMによって得られた係数値。Krigで得られたものと値が似ている。
Krig()とは別な方法として、library(gstat)にあるgstat()を使う方法もあったのですが、こちらだと、独立変数のパラメータが出力されません。一方、Krigだと本当に普遍クリギングになっているのか不安です。もちろんgoogleやヘルプで調べ、"Gaussian random spatial processes"というものが理解への鍵ということはわかったのですが、私にとっては少し理論が難しく、やはりKrigの正体がつかめません。この関数はクリギングの面推定を行うらしいのですが、それはつまり普遍クリギングを行ってくれているのでしょうか?どなたかご存知の方がおられましたら、ご教授・助言等いただけると幸いです。また、何か思い違いをしているのかもしれません。それもご指摘いただければ幸いに思います。
なお、Rは2.9.0、OSはWindowsXPです。どうぞよろしくお願い致します。
# 恐らく(責任持ちません、自分でチェックして納得して下さい)
> coef.Krig(Krig(ozone$x, ozone$y, theta=20, m=1))
[,1]
[1,] 39.78066
# つまり当てはめ平均値曲面は定数 f(x,y)=39.78
> coef.Krig(Krig(ozone$x, ozone$y, theta=20, m=2))
[,1]
[1,] 4.022360e+01
[2,] 2.552514e-04
[3,] 1.338397e-02
# つまり当てはめ平均値曲面は平面 f(x,y)=4.02e+1+2.55e-04*x+1.34e-02*y
> coef.Krig(Krig(ozone$x, ozone$y, theta=20, m=3))
[,1]
[1,] 36.972752325
[2,] 0.033350055
[3,] 0.063710522
[4,] 0.011040245
[5,] 0.009597520
[6,] 0.004741821
# つまり当てはめ平均値曲面は2次曲面
# f(x,y)=37.0+0.033*x+0.063*y+0.011*x^2+0.0096*xy+0.0047*y^2library(fields) K2 <- Krig(data.frame(x1=ozone$x[, 1], x2=ozone$x[, 2],x3=(ozone$x[, 1])^2, x4=ozone$x[, 1]*ozone$x[, 2], x5=(ozone$x[, 2])^2), ozone$y, m=2) K3 <- Krig(ozone$x, ozone$y, m=3) coef(K2) coef(K3) set.panel(2, 2) plot(K2, main="m=2") x11() set.panel(2, 2) plot(K3, main="m=3") x1=ozone$x[, 1] x2=ozone$x[, 2] x3=(ozone$x[, 1])^2 x4=ozone$x[, 1]*ozone$x[, 2] x5=(ozone$x[, 2])^2 y=ozone$y summary(lm(y ~ x1 + x2 +x3 + x4 + x5))#線形回帰でも一応確認。m=3のときのr^2の値と似ている
七転八倒 (2009-06-25 (木) 00:47:34)
XPで2.7.1を使っています。
例えばトランプのようにA<2<3<...Q<Kのように自前で大小関係を定義したいのです。因子というものを使えばうまくいきそうなので試しているのですが、エラーがでてしまいます。
事前にA<2<3...<Kと順序つきで因子を指定したnum列を用い、suit num 1 S A 2 S 2 3 S 3 ... 52 C Kこのようなデータフレームを作りました。
これをランダムに並べ替えのち13枚取り出して、スートと番号にもとづいて並び替えようとxorder<-order(x$suit,pmax(x$suit,x$num)) x[xorder,]と入力したところ
以下にエラー mmm < each : これらの型の比較は未実装です 追加情報: Warning message: メソッド ("Ops.factor", "Ops.ordered") は "<" に対しては矛盾していますというエラーがでました。
pmax以下がないとうまくいきました。
これはどういう状態なのでしょうか?ご教授お願いしますm(_ _)m
> Lev <- c("1","2","3","C","B","A","x","z","y")
> (x <- ordered(c("A","2","2","C","y"), levels=Lev))
[1] A 2 2 C y
Levels: 1 < 2 < 3 < C < B < A < x < z < y
> order(x)
[1] 2 3 4 1 5
> x[order(x)]
[1] 2 2 C A y
Levels: 1 < 2 < 3 < C < B < A < x < z < y
> (y <- ordered(c("B","1","A","z","x"), levels=Lev))
[1] B 1 A z x
Levels: 1 < 2 < 3 < C < B < A < x < z < y
> order(x,y)
[1] 2 3 4 1 5
> x[order(x,y)]
[1] 2 2 C A y
Levels: 1 < 2 < 3 < C < B < A < x < z < yなつ (2009-06-22 (月) 19:13:22)
WindowsXPでR-2.8.1を使用しています。
Rを使って、Google Earthからカラー画像をキャプチャ→保存したいと思い調べています。
なお、Google Earth COM APIではモノクロ画像しかキャプチャできないようです。
調べた中で可能性がある?と思った方法は以下の二つです。
・CかC++でスクリーンキャプチャする関数を書き、dllとしてRにロードして使用する方法
CUIの関数はMinGWでビルドしてRでロードして使うことができたのですが、
GUIのプログラムはVCで少し囓った程度なので、
Win32API(w32api-3.13-mingw32-dev.tar)はDLしてきたのですが、
スケルトンすらビルドできませんでした。
そもそもMinGWでGUIプログラムをビルドしている例が見つけられなかったので
本当にできるのか自体がわかっていません。
・COMを通す方法
スクリーンキャプチャができるソフトウエアでCOMに対応したソフトウエアが見つけられませんでした。
上の方法にこだわってはいませんので
実現できそうな方法をご存知の方がいらっしゃいましたら、
大変お手数で恐縮なのですが、ヒントを頂けると助かります。
プログラム関連の用語の使い間違いや
根本的な思い違いがありましたら申し訳ありません。
あきやま (2009-06-20 (土) 14:07:03)
sinkを関数定義の中で使用するとうまくいきません。
RのバージョンはR-2.9.0,OSはWindowsXPです。
通常の使用方法a <- matrix(c(100,200,300,400),ncol=2) filename <- tempfile() sink(filename) chisq.test(a,correct=F) sink() file.show(filename)うまくいかなかった例
original_output <- function(){ a <- matrix(c(100,200,300,400),ncol=2) filename <- tempfile() sink(filename) chisq.test(a,correct=F) sink() file.show(filename) }うまくいった代わりの方法
original_output <- function(){ a <- matrix(c(100,200,300,400),ncol=2) filename <- tempfile() writeLines(capture.output(chisq.test(a,correct=F)),filename) file.show(filename) }sink関数の仕様でしょうか?
それとも使い方が間違っているのでしょうか?
どなたか教えていただきたいと思います。よろしくお願いします。
original_output <- function(){
a <- matrix(c(100,200,300,400),ncol=2)
filename <- tempfile()
capture.output(chisq.test(a,correct=F), file = filename)
file.show(filename)
}林 (2009-06-20 (土) 08:33:34)
貢献パッケージ内のクラスについて調べたいのですが、うまくいきません。
以下では、Hmiscのlabelledクラスにアクセスしようとしています。
> library(Hmisc) > age <- c(21,65,43); label(age) <- "Age in Years" > class(age) [1] "labelled" > (env <- environment(label)) <environment: namespace:Hmisc> > getClass("labelled", where=env) 以下にエラー getClass("labelled", where = env) : "labelled" は定義されたクラスではありません
ただ見るだけならばソースを読めばいいのですが、
最終的には継承して新しいクラスを作りたいと考えています。
しかしそれもうまくいきません。
> setClass("labelled2",contains="labelled") 以下にエラー reconcilePropertiesAndPrototype(name, slots, prototype, superClasses, : スーパークラス "labelled" の定義が, クラス "labelled2" の設定中にありません
クラス"labelled"の定義が見つけられていないのが原因というのは明らかなのですが、
どのように解決すればよいのでしょうか?
名前空間の問題だとは思うのですが…。where = envと指定までしているのになぜ見つからないのでしょうか?
なにか根本的な勘違いをしている気がします。よろしくお願いします。
環境は以下です。
> sessionInfo() R version 2.9.0 (2009-04-17) i386-pc-mingw32
> str(age) Class 'labelled' atomic [1:3] 21 65 43 ..- attr(*, "label")= chr "Age in Years"
(2009-06-17 (水) 14:59:45)
現在、生存時間解析に関連して、median survival timeの比較を検討しております。
treatment 4例、control 26例の計30例で、全例打ち切りなしで最後まで観察しております。
このとき、例えば survfit() コマンドでmedian survival timeとその信頼区間を求めることができますが、二つの群で有意差があるか検定するためのコマンドをご存知の型が折られたら、ご教授いただければ幸いです。
方法論としては
Brookmeyer, R. and Crowley, J. J. (1982b). A k-sample median test for censored data. Journal of the American Statistical Association, 77, 433-440.
あたりが思いつきますが、implementしたRのパッケージなどが分からず困っております。よろしくお願いいたします。
東福寺 (2009-06-16 (火) 23:25:58)
R2.9.0 をウィンドウズで使用しております。
とあるプログラムを走らせている中途に、下記のようなリストが出来上がってしまいました。(実際の数字ではありません。でっちあげた例です)実際はもっと非常に巨大なリストです。
a <- list(matrix(1:4,2), list(matrix(3:6,2), matrix(4:7,2)), matrix(5:8,2))
これを、length が4でそれぞれの element が 2x2 のマトリックスであるようなリストに簡単に変換する方法はないかと模索しております。
unlist(a, recursive=F)
が普通に思いつく方法なのですが、これだと、a1 と a3 を必要以上に分解してしまいます。
できあがりがlist(matrix(1:4,2), matrix(3:6,2), matrix(4:7,2), matrix(5:8,2))
となる方法をご教授願います。
FFFF (2009-06-16 (火) 16:18:39)
あるデータを読み込むと、
EX1=
WEIGHT HEIGHT
[1,] 50 150
[2,] 60 160
となっているのですが、これを通常の表記
EX2=
[,1] [,2]
[1,] 50 150
[2,] 60 160
とするにはどのようすればよろしいでしょうか?
よろしくお願い致します。
> EX1
WEIGHT HEIGHT
[1,] 50 60
[2,] 150 160
> colnames(EX1) <- NULL
> EX1
[,1] [,2]
[1,] 50 60
[2,] 150 160
> colnames(EX1) <- c("WEIGHT","HEIGHT")
> EX1
WEIGHT HEIGHT
[1,] 50 60
[2,] 150 160 もくず (2009-06-16 (火) 15:59:54)
いつもお世話になっています。
A= [,1] [,2] [,3][1,] A B C [2,] D E F
B= [,1] [,2] [,3][1,] G H I
の時、
C= [,1] [,2] [,3][1,] A/G B/H C/I [2,] D/G E/H F/I
となるようにするにはどのような計算式すればよいでしょうか?
C <- A/B
とすると
C= [,1] [,2] [,3][1,] A/G B/I C/H [2,] D/H E/G F/I
となってしまいます。
A/rbind(B,B) t(t(A)/B) scale(A,0*B,B) sweep(A,2,B,FUN="/")
すろん (2009-06-16 (火) 13:07:18)
いつもお世話になっております。
使用環境はR version 2.9.0 (2009-04-17) powerpc-apple-darwin8.11.1です。
以下のようなデータフレーム
> a a b c d 1 a 0.01905276 1.173279 3 2 b 0.80433105 1.154605 2 3 c 0.06782948 1.043263 NA 4 d 0.12807006 1.945755 1 5 e 0.35224305 1.224513 NA
から、subset()を用いて以下のように抽出して、新たなデータフレームを作ります。> a2 <- subset(a, !is.na(d)) > a2 a b c d 1 a 0.01905276 1.173279 3 2 b 0.80433105 1.154605 2 4 d 0.12807006 1.945755 1
この場合、データフレームa2の行数は3列で、length( a2$a )を実行すると返値は3になります。そして、さらなるデータフレーム> b a e 1 a 7.615926 2 b 2.504819 3 d 6.824558
と、上記のa2を結合しようとすることを考えます。この場合、merge()を使えば何の問題もなく二つのデータフレームを結合できます(以下の通り)。> a <- merge(a2,b) > a a b c d e 1 a 0.01905276 1.173279 3 7.615926 2 b 0.80433105 1.154605 2 2.504819 3 d 0.12807006 1.945755 1 6.824558
しかし、> levels(a$a) [1] "a" "b" "c" "d" "e"
とするとおわかりのように、元々のデータのレベルが残っています。
また、subsetを用いずにデータを抽出した場合も、結果は同様でした。a4 <- a[ !is.na(a$d),] > a4 a b c d 1 a 0.01905276 1.173279 3 2 b 0.80433105 1.154605 2 4 d 0.12807006 1.945755 1 > levels(a4$a) [1] "a" "b" "c" "d" "e"何とか、データフレームa2の列aのレベルを"a", "b", "d"にできないものかと思い、「レベルの消去」、「レベルの削除」、「subset, レベル」などでこちらのサイト内を検索いたしましたが、解決には至りませんでした。私の探したりないところもあるかもしれません。最初から各データフレームに、レベルを示す列を作っておくのが一番の得策かとは思います。しかし、どうにも気になります。もしsubset()等で得られたデータフレームの特定の列のレベルを、元データに依らない状態にする方法をご存じの方がいらっしゃいましたら、ご教示いただければと思います。
因みに、今はデータフレームを一度テキストファイル等に書き出してから再びRに読み込むという、実に場当たり的なことで対処しております。どうぞよろしくお願いいたします。
> df lb x y z 1 a 1 2 5 2 b 3 NA 3 3 c 2 3 2 4 d 1 2 3 5 e NA 5 2 6 f 4 4 1 > df2 <- subset( df, df["y"] == 2, drop = TRUE ) > df2 lb x y z 1 a 1 2 5 4 d 1 2 3 > levels(as.factor(df2$x)) [1] "1" # レベルは"1"のみに > levels( df2$lb ) [1] "a" "b" "c" "d" "e" "f" #削除した行のレベルも残っている上記のように,文字列の場合は最初のデータフレームのレベルを保持しているようです。気になったのでdf$xを最初からfactorにしてみたところ,以下のような結果になりました。
> df$x <- as.factor( df$x ) > df2 <- subset( df, df["y"] == 2, drop = TRUE ) > levels(df2$x) [1] "1" "2" "3" "4" > levels(as.factor(df2$y)) [1] "2" > levels(df2$lb) [1] "a" "b" "c" "d" "e" "f"factorの列のレベルも消去する方法を,もう少し考えてみます。-- すろん 2009-06-17 (水) 14:07:12
aabc (2009-06-14 (日) 22:30:00)
棒グラフの凡例から文字がはみ出てしまいます。
このHPとR Helpで隈なく調べてみたのですが、枠の大きさを変更する方法については見つからず、それで質問をさせて頂きました。
よろしければご教授下さい。
本体のversionはR-2.6.2です。
ご指摘を受けたので、具体的なコードを載せます(6/15(月)に追加編集)>df V1 V2 V3 1 2 15 23 2 13 50 135 3 36 124 371 4 105 214 679 5 161 301 684 6 150 222 403 7 120 152 196 8 65 80 91 9 41 43 30 10 47 32 18 >colnames(df) <- birth.number <- c("0人","1人","2人") #出生児数 >rownames(df) <- marry.at.age <- c("15~18歳","19~20歳","21~22歳","23~24歳", "25~26歳","27~28歳","29~30歳","31~32歳","33~34歳","35歳以上") >barplot(data.matrix(df),col=rainbow(10),horiz=TRUE,main="", xlab="標本数",ylab="出生児数") >legend(x=1500,y=2.3,marry.at.age,fill=rainbow(10), title="妻の\n結婚年齢",cex=0.8)枠そのものを変更できる方法はないでしょうか?
> barplot(data.matrix(df),col=rainbow(10),horiz=TRUE,main="",
xlab="標本数",ylab="出生児数")
>legend(x=1500,y=2.3, marry.at.age, fill=rainbow(10),
title=" 妻の結婚年齢 ", cex=0.8) #文字列の両端を1マス分空ける
>legend(x=750,y=2.3,marry.at.age, fill=rainbow(10),
title=" ", cex=0.8) #空白のスペース
ジェットラグ (2009-06-11 (木) 19:14:46)
既存のファイル(例えば"hoge.RData")をRguiで開いて作業中に,getwd()のように,このファイル名"hoge.RData"を取得する関数や方法はあるのでしょうか?ご教示いただければ幸いです.
RのバージョンはR-2.9.0,OSはWindowsXPです.
attach(NULL, name = "JetFunEnv")
assign("loadHistory",
commandArgs()[grep(".RData$",commandArgs(),ignore.case=T)],
pos="JetFunEnv")
assign("load",
function(file, envir= parent.frame()) {
assign("loadHistory",
c(get("loadHistory",pos="JetFunEnv"),file),
pos="JetFunEnv")
base:::load(file,envir)
},
pos="JetFunEnv")
見たいなのを, .Rprofileに仕込んで, `loadHistory'とタイプすると言うのでどう? ダブルクリックはcommandArgsに渡るし, loadはbase環境だし, そんでいいかと思うけど用途に合わせて発展させてみて下さい. -- 2009-06-11 (木) 22:26:51#ファイル中のオブジェクトにファイル名をコメントする関数の例
> Comment <- function(file.name) {
name <- load(file=file.name) # ファイルからオブジェクトを読み込む
A <- paste("comment(",name,")='from the file ",file.name,"'",sep="")
eval(parse(text=A)) # ファイル名をコメント属性に記録
B <- paste("save(",name,",file='",file.name,"')",sep="")
eval(parse(text=B)) } # 再びファイルに保存
> x <- 1:10
> save(x,file="x.data")
> rm(x)
> Comment("x.data")
> load("x.data")
> comment(x)
[1] "from the file x.data"> commandArgs() [1] "C:\\R\\bin\\RGui.exe" "C:\\temp\\hoge.RData"となるので,commandArgs()[2]で求めていたファイル名を取得できました.ただし,
> load("c:/temp/hoge.RData")
によってワークスペースをロードした場合には,commandArgs()ではファイル名を取得できないようです.この場合は,第二の方法のように,ファイル中のオブジェクトに該当ファイル名をコメント属性で記録しておくのが有効ですね.ご教示ありがとうございました. -- ジェットラグ 2009-06-16 (火) 20:10:33sh (2009-06-10 (水) 14:08:58)
'limma'パッケージがインストールできません。アドバイスをよろしくお願い致します。
> install.packages("limma",dep=T) Warning message: In getDependencies(pkgs, dependencies, available, lib) : package ‘limma’ is not available > sessionInfo() R version 2.9.0 (2009-04-17) i386-apple-darwin8.11.1
のの (2009-06-08 (月) 21:16:42)
次のデータyを,nls()関数で2次・3次多項式で近似し,それぞれ定数と係数を求めたいのですが,エラーメッセージが出て困っています(他のデータは,うまくいきます)。y<-c(0.4404,0.2764,0.1124,-0.0516,-0.2156,-0.3796,-0.5436,-0.7076) # データ~ x<-c(1:8) # 変数~ res1=nls(y~a+b*x,start=c(a=1,b=1),trace=T) # 1次式~これを実行すると,エラー「繰り返し数が最大値 50 を超えました」が出ます。
res2=nls(y~a+b*x+c*x^2,start=c(a=1,b=1,c=1),trace=T) # 2次多項式~ res3=nls(y~a+b*x+c*x^2+d*x^3,start=c(a=1,b=1,c=1,d=1),trace=T) # 3次多項式~これら2次式,3次式を実行すると,エラー「勾配が特異です」が出ます。
データは見た感じほぼ直線ですが,上記3つの式で近似して当てはまりの良さをAIC比較したいのですが,結果が得られません。
どなたか原因と対策をお教えいただけないでしょうか。どうか宜しくお願い致します。
バージョンは,R2.5.0,WindowsXP を使用しています。
> diff(y) [1] -0.164 -0.164 -0.164 -0.164 -0.164 -0.164 -0.164 > ye <- y+1e-3*rnorm(length(y)) #人工的な誤差を加えるのがコツ > res1=nls(ye~a+b*x,start=c(a=1,b=1),trace=T) # 1次式 310.7787600001795 : 1 1 7.94637207633663e-06 : 0.6045071713694352 -0.1639666027143474 > res2=nls(ye~a+b*x+c*x^2,start=c(a=1,b=1,c=1),trace=T) # 2次多項式 12261.1517016733 : 1 1 1 7.78438558419458e-06 : 6.040413965366944e-01 -1.636871378146956e-01 -3.105165551708922e-05 > res3=nls(ye~a+b*x+c*x^2+d*x^3,start=c(a=1,b=1,c=1,d=1),trace=T) # 3次多項式 604223.0598427523 : 1 1 1 1 7.6193699392589e-06 : 6.048664356480176e-01 -1.645455118395975e-01 1.939590111998957e-04 -1.666745679362869e-05
yuuuu (2009-06-05 (金) 02:55:35)
lda関数によって判別式の算出と算出に用いたデータ自体の判別は行えるのですが,その判別式を用いて別のデータの判別予測を行う方法がわかりません.
初歩的な質問かもしれませんが,どなたかお分かりの方いらっしゃいましたらよろしくお願いします.
mito (2009-06-03 (水) 17:29:59)
こんにちは。
すみません、初歩的な質問なのですが調べても分からなかったのでよろしくお願いします。
糖鎖とタンパク質(または血清やウィルスなど)の結合親和性を使ってヒートマップを
書いています。ヒートマップのクラスタがきれいに出ないので正規化しているのですが、実験の値(結合親和性)が明らかに外れているものがあり(実験ミスと思われる)、それらを棄却して正規化したいと思っています。
そこで列ごと(つまり一つのタンパク質に対する糖鎖の結合親和性)で実験の値が大きすぎるものと小さすぎるものをそれぞれ1〜2パーセント、または3個ずつくらい取り出して、それらを無視して正規化するということを列ごとに行いたいのですが、それが出来るような関数はあるでしょうか。もしあればその関数と利用方法、なければヒントを教えていただければと思います。
使用環境は> sessionInfo() R version 2.8.1 (2008-12-22) i386-apple-darwin8.11.1です。よろしくお願いします。
x <- matrix(sample(100,50),ncol=10)
y <- t(apply(x,1,function(x) scale(x,center=mean(sort(x)[-c(1,length(x))]))))
# 上下を1つずつ除いた場合
という意味でしょうか? -- akira 2009-06-03 (水) 19:35:10> ex_matrix Answer1 Answer2 Answer3 Answer4 A 101 120 70 35 B 153 162 88 46 C 89 135 78 24 D 26 49 42 17 E 36 70 30 16 F 167 216 144 71 G 125 143 89 65があったときに
> apply(ex_matrix,2,sort)
Answer1 Answer2 Answer3 Answer4
[1,] 26 49 30 16
[2,] 36 70 42 17
[3,] 89 120 70 24
[4,] 101 135 78 35
[5,] 125 143 88 46
[6,] 153 162 89 65
[7,] 167 216 144 71
を使ってこのソートした後の1行目と7行目の値を無視して正規化(Z値を各々出したい)したいのです。
1行目と7行目の値を列ごとに除外した平均と標準偏差が列ごとに出てほしいのです。-- mito 2009-06-04 (木) 18:01:20初心者 (2009-06-01 (月) 13:16:17)
はじめまして。
目的変数が1のデータが141個、0のデータが34個のデータセットもとに解析手法としてロジスティック回帰分析を用い、検証方法としてleave-one-outを行っています。
ですが、目的変数が1と0のデータの数に差があるため、glmパッケージの中のプログラミングを変更して、重みをつけようと考えています。どの部分に重みをつければいいのでしょうか?どなたかご存じの方はいらっしゃらないでしょうか?よろしくお願いします。
edora (2009-06-01 (月) 02:28:33)
"The R Book"を読んでRでシミュレーションをしてみようと思っています。
以下のように乱数を発生させるサンプルがあるのですがrunif(10000,0,1)の後の"*.Machine$integer.max"がなぜ付いているのかわかりません。これの意味はどういったものでしょうか?また得られる乱数の桁数はどう決まっているのでしょうか?---サンプル---- RNGkind("Super-Duper") # 10000個の擬似乱数を正の整数の範囲で生成してinitsというベクトルに付値 inits <- as.integer(runif(10000,0,1)*.Machine$integer.max)よろしくお願いします。
かわべ (2009-05-29 (金) 03:03:48)
多重比較検定をTukey法によって行っています.
青木先生のHP(http://aoki2.si.gunma-u.ac.jp/R/tukey.html)を参考に,source("http://aoki2.si.gunma-u.ac.jp/R/src/tukey.R", encoding="euc-jp") dat <- read.table("in.txt",header=T) tukey(dat$number,factor(dat$No))というふうに検定を行っていますが
t p 1:2 9.239636 0.000000e+00 1:3 12.311420 0.000000e+00 ・ 2:3 3.071784 1.206570e-02 ・ ・というようにp値が0になってしまうものがでてきます. サンプル数が大きすぎるため(各群サンプル数110,4群比較=440)p値がかなり小さくなっているのだと思いますが、このp値の指数をできるだけ大きい数字まで表示させることはできるのでしょうか。(例2.118100e-89というふうに)
かわぐち (2009-05-28 (木) 22:55:35)
生存分析を行い生存曲線をカプランマイヤー法で描こうと思っています。event <- c("y","y","y","y","y","n","y","y","n","y","y","y","y","n","n", "y","y","y","y", "n","y","n","y","y","y","y","y","n","n","y") time <- c(16,2,6,5,8,13,4,8,11,1,13,7,16,10,9,8,14,17,14,12,13,18,13,6,3, 15,17,16,14,8) dat <- Surv(time, event=="y") res <- survfit(dat) 以下にエラー survfit(dat) : Survfit requires a formula or a coxph fit as the first argumentとのエラーが出て分析できません。何が間違っているのでしょうか。
> library(survival)
要求されたパッケージ splines をロード中です
> event <- c("y","y","y","y","y","n","y","y","n","y","y","y","y","n","n",
"y","y","y","y", "n","y","n","y","y","y","y","y","n","n","y")
> time <- c(16,2,6,5,8,13,4,8,11,1,13,7,16,10,9,8,14,17,14,12,13,18,
13,6,3,15,17,16,14,8)
> dat <- Surv(time, event=="y")
> res <- survfit(dat)
> res
Call: survfit(formula = dat)
n events median 0.95LCL 0.95UCL
30 22 13 8 16
> plot(res)
2.9.0 では,
> dat <- Surv(time, event=="y") ~ 1としないとダメ.-- surg 2009-05-29 (金) 10:28:20
ぴ (2009-05-27 (水) 00:19:18)
すみません。どなたかこのやり方をご存知ではないでしょうか?
例えば、1から100までの数が順に並んでいると考えてください。
このとき、仮に20個の数字を一つのwindow sizeとしてこのグループとそれ以外の数との間でt-testを行います。更にそれを数を一つずつずらしていき、それぞれに対してt-testを行って、それぞれのp-valueを求めたいと考えています。
つまり、<比較A> [1-20]と [21-100]の間のt-test <比較B> [2-21]と [1& 22-100]の間のt-test <比較C> [3-22]と [1-2& 23-100]の間のt-test <比較D> [4-23]と [1-3& 24-100]の間のt-test <比較E> [5-24]と [1-4& 25-100]の間のt-test . . . <比較Z> [81-100]と [1-80]の間のt-testとして、それぞれのp-valueを求め、最終的にはその中で最も小さいp-valueを求めたいと考えています。
すみませんが、どなたかご存知ではないでしょうか?
お願いします。
x <- rnorm(100)
p <- rep(0,81)
for(i in 1:81){
g <- rep(0,100)
g[i:(i+19)] <- 1
p[i] <- t.test(x ~ g)$p.value
}
kddoi (2009-05-25 (月) 16:15:08)
棒グラフを作成しようとしております。しかし、個々の棒に付けるべき文字列が長いため、全ての名前を表示することができません。当初barplotのnames.argに名前を渡していたのですが表示できず、axis関数を使えばより細かく制御できるところまでは調べました。axisのR Documentationにother graphical parameters may also be passed as argumentsとあるので、srtなどを使って回転させようとしても駄目で、また文字の大きさをcexで変えようとしても駄目でした。以下がその様子を示す簡単なプログラムでVery long name 1から7までを全て表示することができません。pos <- barplot(c(1:7), ylim=c(0,8)) box() axis(1, at=pos, tick=FALSE, labels=paste("Very long name", 1:7))ようやくaxisの代わりにtext(pos, 0, paste("Very long name", 1:7), adj=-0.1, srt=-90, cex=0.7)とすることで全ての名前を表示させることができたのですが、場当たり的な対処の気がいたします。Rを使い初めて長くないため網羅的な検索ができていないので、もしより便利な方法があればお教えください。
ぽち (2009-05-24 (日) 00:47:22)
例えば、x <-c(1,2,3,4,5,6,8,9,10,11,12,13,15,16,17,19,20)~という配列があったとします。
ここから、番号が5個以上連続で続いているグループ(1,2,3,4,5など。4,5,6,8,9はこの場合ふくめません)が何個あるかを調べたいのですが、できるでしょうか?一通り検索で調べたのですが、検索ワードが悪いらしく分かりませんでした。よろしくお願いします。
> x <- c(1,2,3,4,5,6,8,9,10,11,12,13,15,16,17,19,20) > y <- rle(diff(x)) #差分の連続のしかたを調べる > z <- y$lengths[which(y$values == 1)] #差分1の連続する長さを得る > length(z[z >= 4]) #(基準-1)以上の要素数を得る [1] 2
初心者。。 (2009-05-22 (金) 15:43:22)
散布図で2種類のデータ群を色分けではなく、記号分けしたいのですが、どうすればよいのでしょうか?
例えば、Aのデータ群を○、Bのデータ群を×で表示したいのですが。。。
初歩的な質問ですが、よろしくお願いします。
> x <- cbind(matrix(sample(100,32),ncol=2,dimnames=list(1:16,c("A","B"))),
group=rep(c(1,4),8))
> plot(x[,1:2],pch=x[,3])
ということですか? -- akira 2009-05-22 (金) 19:03:38> x <- cbind(x=1:4,y=1:4) > plot(x,pch=x[,1])この意味をよく考えれば分かると思います。 -- akira 2009-05-23 (土) 18:09:45
よろしくお願いします。 (2009-05-22 (金) 14:24:41)
Rの初心者です。以下のようなデータを3次元(または4次元)表示したいのですが、コマンドの入力方法が分かりません。> x<-cbind(rnorm(700,mean=11,sd=10),rnorm(700,mean=28,sd=16), rnorm(700,mean=1,sd=1),rnorm(700,mean=14,sd=15))~ > y<-cbind(rnorm(600,mean=30,sd=50),rnorm(600,mean=27,sd=16), rnorm(600,mean=7,sd=1),rnorm(600,mean=16,sd=21))~どなたか、よろしくお願いします。
もうダメです。。 (2009-05-22 (金) 08:34:54)
ここでお尋ねしてよいのか迷いましたが、自分で考えてももう、どうしようもなく、時間がだけがどんどん過ぎてしましました。どなたか、助けて下さるとありがたいです。
SASの以下のプログラムをRにしなくてはいけません。
たくさん調べてみたのですが、私はどちらも初心者で、考えが及ばないのです。
以下は、Rで書くと、どのように書き直せるのでしょうか?proc tpspline data A~ model Y=(X1 X2 X3)/alpha=0.01; score data=A out=B output out=C Run;場違いな質問をしていたら申し訳ありません。
function ()
{
library(gss)
xx1 <- c(1,2,4,3,5,6,5,7,6,8)
xx2 <- c(5,2,1,9,3,1,2,3,9,5)
yy <- c(2,2,1,9,2,1,1,1,2,2)
data1 <- data.frame(x1 = xx1, x2 = xx2, y= yy)
data2 <- list(x12=cbind(xx1, xx2), y=yy)
anova1 <- ssanova0(y~1+x12, data=data2)
ex1 <- seq(from = min(xx1), to = max(xx1), by = 0.1)
ex2 <- seq(from = min(xx2), to = max(xx2), by = 0.1)
ex12 <- expand.grid(ex1, ex2)
data3 <- model.frame(~1+x12, list(x12=cbind(ex12[,1], ex12[,2])))
ey1 <- predict.ssanova0(anova1, data3, inc=c("1","x12"))
eymat1 <- matrix(ey1, ncol=length(ex2))
persp(ex1, ex2, eymat1)
}
SASのalphaというのは推定値の信頼区間を推定するための定数のようです。 ssanova0()を利用する場合は、上のRプログラムならanova1の中身を 利用すれば、推定値の信頼区間は容易に得られます。--竹澤 (2009-05-25 (月) 09:01:01)
青空 (2009-05-17 (日) 11:23:37)
Rは3年度ほど使わせてもらっていますが、最近VBAやC#用のDLLに変換できる機能があれば便利だなと思うようになりました。皆さんのご存知のとおり、MATLABはVBA用DLL作成機能がついていますが、Rはそのような機能がないのでしょうか?
sh (2009-05-14 (木) 23:04:29)
個数に比例するように面積を変化させたVenn Diagramを描くにはどうすればよろしいでしょうか?参考文献 http://d.hatena.ne.jp/kwg/20090202/p1
しまだ (2009-05-13 (水) 22:31:36)
Rまだまだ初心者です。筑波大のSDAMのようにデータ処理をRに任せるようなフロントエンドを自作するためのマニュアル等はあるのでしょうか?
初心者 (2009-05-11 (月) 00:30:22)
最近 R を使い始めた初心者です。
R でコードを書いて、デバッグしようと思うのですが、予め設定したブレークポイントで実行が中断して、1 行ずつ実行したり、その断面の状態の変数が分かる (VBA の VBE や Visual Studio でデバッグするときにある機能) ような開発環境はあるのでしょうか?
browser() でできることに近いのですが、browser() だと CUI ベースということと、知りたい変数名をいちいち入力するのが面倒なので、GUI ベースの開発環境を使えたりしたら教えていただければと存じます。
Eclipse 上で R を動かしたり、Meadow + ESS で試したりしたのですが、その ような機能はないように見えます。
そもそも、そのような開発をするほどの長さのコードを書かないものなのでしょうか?
焼津の半次 (2009-05-08 (金) 21:46:35)
Rで、一から画像を作成してPNG形式で保存したいのですが?Rでできるのでしょうか?
そのまま (2009-05-08 (金) 15:50:59)
一般化線形モデル(GLM)の分析について、以下のようなデータを用いて> d02$y [1] 59.61 33.27 20.27 10.18 11.77 12.88 23.30 14.15 5.10 0.79 > d02$x [1] 27.84 27.66 26.90 26.91 27.25 27.24 27.30 27.21 27.29 27.07AIC値を求めるとき、
> AIC(glm(y~x,family=poisson,data=d02)) [1] Inf Warning messages:~ 1: In dpois(y, mu, log = TRUE) : non-integer x = 59.610000 2: In dpois(y, mu, log = TRUE) : non-integer x = 33.270000 略 9: In dpois(y, mu, log = TRUE) : non-integer x = 5.100000 10: In dpois(y, mu, log = TRUE) : non-integer x = 0.790000のようなエラーが出ます。なぜでしょうか?教えてください。
> AIC(glm(as.integer(y)~x,family=poisson,data=d02)) [1] 98.2934 > AIC(glm(round(y)~x,family=poisson,data=d02)) [1] 93.20353
高橋 (2009-05-08 (金) 00:37:04)
本当に素人です。
Rで回帰分析を行うために、エクセルで”カンマで区切って数値”をして保存したあと、Rでそのファイルを読み込んだんですが、y値が認識できません。classで確認してみるとNULLになっています。何回やっても無駄でした。テキスト(例えば、矢野先生のRによる心理統計など)を見ながら、PATHやheader=TRUEなどちゃんと書いたと思います。何で認識しないんでしょうかね?教えてください。
maori (2009-05-06 (水) 17:21:29)
たびたび初歩的な質問をすみません。
糖鎖とタンパク質との反応のheatmapを書いていて、糖鎖の数が多くて見にくいのでヒートマップの行の名前として糖鎖の名前を順番通り取り出したいのですが、どのようにすればよいのか分かりません。
heatmapの中のlabRowがそれを示しているのだと思ったのですがどのように取り出せば良いのでしょうか。> z <- read.table("test.txt",header=TRUE,sep="\t",row.names=1) > z_numeric <- apply(z,c(1:2),as.numeric) > z_euclid <- dist(z_numeric,method="euclid") > heatmap(z_numeric,hclustfun= function(z_euclid) hclust(z_euclid,method="ward"))です。
また、ヒートマップの詳しい書き方について学べるページがありましたら、教えていただければと思います。
使用環境は> sessionInfo() R version 2.8.1 (2008-12-22) i386-apple-darwin8.11.1です。よろしくお願いします。
> label_z <- heatmap(z_numeric,hclustfun= function(z_euclid) hclust(z_euclid,method="ward"));labels(x$Rowv) character(0) > label_z $rowInd [1] 1 5 13 19 8 18 6 14 15 3 7 2 9 17 16 12 10 11 4 $colInd [1] 4 3 5 1 2 $Rowv NULL $Colv NULLとなってしまうのですが、何が原因でしょうか。どう解決すれば良いのでしょうか。 再び質問ですみません。-- maori 2009-05-06 (水) 20:52:23
hf (2009-05-06 (水) 13:01:44)
二項分布について,> dbinom(0,3,.5) [1] 0.125のように求めた値と,
> plot(dbinom(0:3,3,.5),xlim=c(0,5),ylim=c(0,.5))でx=0,1,2,3に対する値を求めてプロットしたものとを比べると,後者のグラフでは,横軸の1に対応する位置に上で求めた値0.125がプロットされます。結果として,x=0,1,2,3に対応する値が,グラフ上では(横軸の値)=1,2,3,4に対応する位置にプロットされ,1だけずれた位置に描かれます。
dbinom(x,n,p)でxに与えた値と,plot(dbinom(x,n,p))でグラフをプロットしたときの(横軸の値)とが一致するようにするには,どうすればよいでしょうか?
使用環境は以下の通りです。> sessionInfo() R version 2.8.0 (2008-10-20) i386-pc-mingw32よろしくお願いいたします。
tonton1225 (2009-05-04 (月) 16:14:57)
RからWinBUGSを呼び出し、下記のモデルファイルを読み込むとエラーが起きます。
エラーの内容はよく分からない記述になっていて、解読できません。
エラーは無駄に長く、コピペしても意味がないっぽいのでコピペしませんでした。
WinBUGSのバージョンは1.4.3、WindowsXPです。
なぜ、モデルファイルを読み込めないのかどなたか教えて下さい。
モデルファイルmodel { for(i in 1:N) { data[,i] ~ dmultinom(4,prob) } Tau <- 1 alpha1 ~ dnorm(0, Tau) alpha2 ~ dnorm(0, Tau) alpha3 ~ dnorm(0, Tau) alpha4 ~ dnorm(0, Tau) alpha <- ceiling(abs(c(alpha1,alpha2,alpha3,alpha4))) prob ~ ddirichlet(alpha) }WinBUGSを呼び出す
# パッケージの読込 library(MCMCpack) library(R2WinBUGS) # データの作成 Tau.real<-10 alpha.real<-rnorm(4, 0, Tau.real) alpha.real <- ceiling(abs(alpha.real)) prob.real<-rdirichlet(1,alpha=alpha.real) data<-rmultinom(n=100,size=4,prob=prob.real) N<-ncol(data) # 初期値と保存するパラメータを設定 in1<-in2<-in3<-list(alpha1=1,alpha2=1,alpha3=1,alpha4=1,prob=c(.25,.25,.25,.25)) inits<-list(in1,in2,in3) parameters<-c("alpha1","alpha2","alpha3","alpha4","prob") # R2WinBUGSを実行 sample.wb<-bugs( data, inits, parameters, model.file="model.bug.test.txt", debug=FALSE, n.chains=3, n.iter=10000, n.burnin=1000, codaPkg=TRUE, bugs.directory="C:/Program Files/WinBUGS14", working.directory=NULL )
incompatible copy BugsCmds.TextError [000003A1H] .beg INTEGER 831976856 .end INTEGER 16861596 .name ARRAY 256 OF CHAR "C:/Documents and Settings/sumiok" "C:/Documents and Settings/sumioka/My Documents/program/R/kubo/model.bug.test.txt", 0X, ..., 0X .pos INTEGER 35 .text TextModels.Model [01016F90H] .v Views.View [010182D0H] BugsCmds.Parse [00000470H] .name ARRAY 256 OF CHAR "C:/Documents and Settings/sumiok" "C:/Documents and Settings/sumioka/My Documents/program/R/kubo/model.bug.test.txt", 0X, ..., 0X .s BugsScanners.Scanner .char CHAR "]" .nextCh CHAR " " .real REAL 5.515143454889401E-308 .int INTEGER 1 .pos INTEGER 35 .type INTEGER 0 .len INTEGER 1 .string BugsScanners.String "i" "i", 0X, "ta", 0X, 0X, "hlet", 0X, "#", 0D4A8X, "#", 1B4BX, 3144X, 0X, 0X, 0D4ECX, "#", 0B64X, 3130X, 0D4C0X, "#", 0D4C0X, "#", 2653X, 3144X, 80D0X, 108X, 0D550X, 117X, 0X, 0X, 0D4D8X, "#", 0D4D8X, "#", 354FX, 3144X, 80D0X, 108X, 0D550X, 117X, 0X, 0X, 0D4FCX, "#", 625EX, 3131X, 0D880X, 104X, 0D4ECX, "#", 0B64X, 3130X, 0X, 0X, 2D60X, 32E7X, 2D74X, 32E7X, 14X, 0X, 0D51CX, "#", 4C97X, "õ", 0E290X, 102X, 2D60X, 32E7X, 2D74X, 32E7X, 14X, 0X, 0DBFCX, "#", 0D43EX, "#", 0D544X, "#", 4CE5X, "õ", 2D60X, 32E7X, 2D74X, 32E7X, 14X, 0X, 0E044X, "#", 0D55CX, "#", 1152X, 32E7X, " ", 0X, "!", 0X, 1FX, 0X, 0X, 0X, 0D554X, "#", 2050X, "ø", 0DBD0X, "#", 1D4X, "÷", 0D34X, 32E7X, 0X, 32E6X, 6150X, 7372X, "e#", 0D43EX, "#", 0D46CX, "#", 5270X, 3131X, 0X, 0X, 0D4ECX, "#", 0B64X, 3130X, 0X, 0X, 0X, 0X, 8A0X, 116X, 0DBFCX, "#", 0D43EX, "#", 0D494X, "#", 5270X, 3131X, 0X, 0X, 0D4ECX, "#", 0B64X, 3130X, 0X, 0X, 0X, 0X, 6E0X, 116X, 0DBFCX, "#", 0D43EX, "#", 0D4A8X, "#", 1B4BX, 3144X, 0X, 0X, 0D4ECX, "#", 0B64X, 3130X, 0D4C0X, "#", 0D4C0X, "#", 2653X, 3144X, 80D0X, 108X, 0D550X, 117X, 0X, 0X, 0D4D8X, "#", 0D4D8X, "#", 354FX, 3144X, 80D0X, 108X, 0D550X, 117X, 0X, 0X, 0D4FCX, "#", 625EX, 3131X, 0D880X, 104X, 0D4ECX, "#", 0X, 0X, 0X, 0X, 0X, 0X, 0X, 0X, 14X, 0X, 0D51CX, "#", 4C97X, "õ", 0E290X, 102X, 0X, 0X, 0X, 0X, 0X, 0X, 0X, 0X, 214X, 314BX, 0D558X, "#", 32D4X, "ø", 0DBD0X, "#", 1D4X, "÷", 0D538X, "#", 1D4X, "÷", 0DBCFX, "#", 3X, 0X, 10X, 0X, 1D4X, "÷", 0DBBCX, "#", 0X, 0X, 214X, 314BX .source POINTER NIL .textReader TextModels.Reader [010156C0H] .eot BOOLEAN FALSE StdInterpreter.CallProc [0000047AH] .a BOOLEAN FALSE .b BOOLEAN FALSE .c BOOLEAN FALSE .i Meta.Item .obj INTEGER 4 .typ INTEGER 0 .vis INTEGER 4 .adr INTEGER 854006166 .mod POINTER [32E60000H] .desc POINTER IllegalAddress000000BCH .ptr Meta.ArrayPtr [0100AD30H] .ext Kernel.ItemExt NIL .imported ARRAY 256 OF CHAR ""
以上が一部です。 -- tonton1225 2009-05-04 (月) 19:08:10
surg (2009-05-04 (月) 10:16:35)
質問というよりは報告ですが、strptime() で生成した POSIXlt オブジェクトの length が実際のサイズと関係なく「9」となっています.> (t <- strptime(paste("1-1-2001 9", 1:20, sep=":"), "%m-%d-%Y %H:%M")) [1] "2001-01-01 09:01:00" "2001-01-01 09:02:00" "2001-01-01 09:03:00" 略 [16] "2001-01-01 09:16:00" "2001-01-01 09:17:00" "2001-01-01 09:18:00" [19] "2001-01-01 09:19:00" "2001-01-01 09:20:00" > length(t) [1] 9そのため、データフレームへの結合で問題が起こります.
同じサイズのデータフレームには結合できません.> d1 <- data.frame(a=1:20) > d1$b <- t 以下にエラー `$<-.data.frame`(`*tmp*`, "b", value = list(sec = c(0, 0, 0, : replacement has 9 rows, data has 20サイズ9のデータフレームには一応結合できる(warning が出ますが).
> d2 <- data.frame(a=1:9) > d2$b <- t > d2 a b 1 1 2001-01-01 09:01:00 2 2 2001-01-01 09:02:00 略 8 8 2001-01-01 09:08:00 9 9 2001-01-01 09:09:00 Warning message: In format.data.frame(x, digits = digits, na.encode = FALSE) : 壊れたデータフレームです。列が切り詰められるか、または NA 値で埋められますデータフレーム作成時に入れておけば,問題なし.
> d3 <- data.frame(a=1:20, b=t) > d3 a b 1 1 2001-01-01 09:01:00 2 2 2001-01-01 09:02:00 略 19 19 2001-01-01 09:19:00 20 20 2001-01-01 09:20:00ちなみに、as.POSIXlt()を使っても同様.
> (t <- as.POSIXlt(paste("2001-01-01 9:1", 1:20, sep=":"))) [1] "2001-01-01 09:01:01" "2001-01-01 09:01:02" "2001-01-01 09:01:03" [4] "2001-01-01 09:01:04" "2001-01-01 09:01:05" "2001-01-01 09:01:06" 略 [16] "2001-01-01 09:01:16" "2001-01-01 09:01:17" "2001-01-01 09:01:18" [19] "2001-01-01 09:01:19" "2001-01-01 09:01:20" > length(t) [1] 9
Details
There are two basic classes of date/times.
Class "POSIXct" represents the (signed)
number of seconds since the beginning of
1970 as a numeric vector. Class "POSIXlt"
is a named list of vectors representing
sec
0-61: seconds
min
0-59: minutes
hour
0-23: hours
mday
1-31: day of the month
mon
0-11: months after the first of the year.
year
Years since 1900.
wday
0-6 day of the week, starting on Sunday.
yday
0-365: day of the year.
isdst
Daylight savings time flag. Positive if
in force, zero if not,
negative if unknown.
要するに,POSIXlt の要素は9個ということでしょう?
> t[1] [1] "2001-01-01 09:01:00" > cat(t[1]$sec,t[1]$min,t[1]$hour,t[1]$mday,t[1]$mon, t[1]$year,t[1]$wday,t[1]$yday,t[1]$isdst) 0 1 9 1 0 101 1 0 0何の問題もないように思います(ちなみに,確認した限りでは,R 1.8.1 でも同じく「9」を返します)
Details Both functions are generic: you can write methods to handle specific classes of objects, see InternalMethods.とありますので,「必要な length は,自分で書け」といっているような雰囲気?
> length(as.character(t)) [1] 20必要な情報はほとんどオンラインヘルプから得られると思いますよ。 -- [[ ]] 2009-05-04 (月) 16:51:27
> d1 <- data.frame(a=1:20) > d1$b <- t 以下にエラー `$<-.data.frame`(`*tmp*`, "b", value = list(sec = c(0, 0, 0, : replacement has 9 rows, data has 20はダメだけど、
> d1$b <- data.frame(t)$tなら大丈夫.-- surg 2009-05-04 (月) 20:16:50
u-kun (2009-05-01 (金) 16:23:35)
以前にR-2.8.1のですが、R-2.9.0をインストールしてR User Configurationを使用したところ、
expression()でmuや%*%等の表示がwindowsのグラフィックデバイスではできなくなりました。
muはmに%*%は'になってしまいます。
pdfに出力すればちゃんと表示されます。
R-2.8.1に戻しても表示されませんでした。
現在の使用環境はwindows XP sp2でRのバージョンは以下の通りです。R version 2.9.0 Patched (2009-04-28 r48429) i386-pc-mingw32 locale: LC_COLLATE=Japanese_Japan.932;LC_CTYPE=Japanese_Japan.932;LC_MONETARY=Japanese_Japan.932; LC_NUMERIC=C;LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods baseRの問題ではなく、私の使用環境の問題でしょうか?
maori (2009-04-28 (火) 21:47:55)
初めて質問させていただきます。
過去の質問等をみさせていただいてもよくわからなかったのでよろしくお願いします。
heatmapを書いているのですが、元になる値が(正規化はしています)とても近いので
マップの色が近くて困っています。
heatmapの内容を変えることにより調節できると思うのですが、helpをみてもgoogleで調べても
どうしたら良いか分かりません。
以下が私が入力したコマンドです。y <- read.table("standarddeviation.txt",header=TRUE,sep="\t",row.names=1) y_numeric <- apply(y,c(1:2),as.numeric) y_euclid <- dist(y_numeric,method="euclid") pdf("standarddeviation_ward4.pdf",paper="special",width=27,height=25,pointsize=25) heatmap(y_numeric,scale="column",hclustfun= function(y_euclid) hclust(y_euclid,method="ward"), margins=c(30,30),col=cm.colors(256),RowSideColors = rc,ColSideColors = cc, cexRow = 0.2 + 1/log10(10), cexCol = 0.2 + 1/log10(10)) dev.off()よろしくお願いします。
> x <- matrix(sample(100,200,re=T),ncol=4) > range(x) [1] 1 100 > heatmap(x) > heatmap(x,zlim=c(0,200))こういうことを言っているのでしょうか?でも個人的には、無理やり違うように見せるより、似たままでも十分情報を持つように思います。 -- akira 2009-04-29 (水) 00:01:00
> sessionInfo() R version 2.8.1 (2008-12-22) i386-apple-darwin8.11.1 locale: ja_JP.UTF-8/ja_JP.UTF-8/C/C/ja_JP.UTF-8/ja_JP.UTF-8 attached base packages: [1] stats graphics grDevices utils datasets methods baseでした。
akira (2009-04-28 (火) 19:31:45)
いつもありがとうございます。基本的事項で恐縮ですが、理由が良く分かりません。教えてください。> x <- data.frame("Alpha"=LETTERS[1:3],"Num"=1:3) > apply(x,2,is.numeric) # x$"Num"がTRUEにならないのはなぜ? Alpha Num FALSE FALSE > apply(x,c(1,2),is.numeric) # x$"Num"がTRUEにならないのはなぜ? Alpha Num [1,] FALSE FALSE [2,] FALSE FALSE [3,] FALSE FALSE > is.numeric(x$"Num") # x$"Num"だけ指定するとTRUE [1] TRUE > is.numeric(x$"Num"[1]) # x$"Num"だけ指定するとTRUE [1] TRUEとなります。
私の環境は> sessionInfo() R version 2.9.0 Patched (2009-04-24 r48395) i386-pc-mingw32 locale: LC_COLLATE=Japanese_Japan.932;LC_CTYPE=Japanese_Japan.932;LC_MONETARY=Japanese_Japan.932; LC_NUMERIC=C;LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods baseです。ちなみにWindowsのR-2.8.1 Patched、LinuxのR-2.9.0 patchedでも同じでした。
> apply(x,2,class)
Alpha Num
"character" "character"
> sapply(x,class)
Alpha Num
"factor" "integer"