![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
初心者のための R および RjpWiki に関する質問コーナー
参照は,個々の項目をクリック(質問への回答・コメントの参照も,個々の項目をクリック)
なっつ (2014-09-18 (木) 16:59:06)
お世話になります。
以下のようにdat中のNAを空文字に置換します。実際にはNAは数千個含まれますが、例として2つだけにしています。dat <- matrix(1:9000000, 3000, 3000) dat[1, 2] <- dat[2, 1] <- NA dat[is.na(dat)] <- ""この処理で目的は達成できるのですが、最後のdat[is.na(dat)] <- "" が単なる代入処理にしては計算時間が長いので、もっと短くならないかと思っています。よい方法がありましたらご教示ください。
宜しくお願いします。
> library(compiler)
> f <- function(x){x[is.na(x)] <- "";x}
> fc <- cmpfun(f)
> dat[1, 2] <- dat[2, 1] <- NA; system.time(f(dat))
ユーザ システム 経過
0.398 0.099 0.644
> dat[1, 2] <- dat[2, 1] <- NA; system.time(fc(dat))
ユーザ システム 経過
0.386 0.009 0.396dat <- matrix(1:9000000, 3000, 3000) dat[1, 2] <- dat[2, 1] <- NA system.time(dat[is.na(dat)] <- "") a <- 1:9000000 a[1] <- a[2] <- NA system.time(a[is.na(a)] <- "")
> dat <- matrix(1:9000000, 3000, 3000)
> dat[1, 2] <- dat[2, 1] <- NA
> system.time(dat[is.na(dat)] <- "")
ユーザ システム 経過
29.090 0.367 29.192
>
> a <- 1:9000000
> a[1] <- a[2] <- NA
> system.time(a[is.na(a)] <- "")
ユーザ システム 経過
3.954 0.039 3.961
>
> dat <- matrix(1:9000000, 3000, 3000)
> dat[1, 2] <- dat[2, 1] <- NA
> system.time(dat[is.na(dat)] <- "")
ユーザ システム 経過
3.949 0.012 3.930
>
> a <- 1:9000000
> a[1] <- a[2] <- NA
> system.time(a[is.na(a)] <- "")
ユーザ システム 経過
3.951 0.012 3.930> dat <- matrix(1:9000000, 3000, 3000)
> dat[1, 2] <- dat[2, 1] <- NA
> system.time(dat[is.na(dat)] <- "")
ユーザ システム 経過
22.318 0.184 22.573
> system.time(write.table(dat,"hoge.csv",sep=",",na="",row.names=F, col.names=F))
ユーザ システム 経過
5.096 0.108 5.214Back (2014-09-18 (木) 04:47:26)
Mac(OS10.9.4) でRを最近使用し初めました。エクセルのファイル(データは、サンプルグループが1から3あって、それぞれのグループの身長の平均を数値化したものを例として使用)を.csvのフォームでRに呼び込み、データを表示させ、View()を入れてみたのですが、毎回”Error in View(fev) : X11 dataentry cannot be loaded”メッセージが表示されます。他のファンクション(dim, names, tail, class)に関しては全く問題ありません。今の所、View()の時だけエラーが出ます。Windowsの場合は全く問題無く表示されたので、Mac特有の問題かと思うのですが、どなたか解決方法ご存知でしょうか?
初心者 (2014-09-02 (火) 23:16:29)
初めまして。R勉強中の者です。
lme4パッケージに含まれるlmerという関数で線形混合モデルを用いた解析を試みております。lmer(formula= Y ~ X1+X2+X3+X4+X5+(X1+X2+X3+X4+X5|Random))といった形で、ランダム効果を組み込んだ計算をさせたいのですが、
警告メッセージ: 1: In optwrap(optimizer, devfun, getStart(start, rho$lower, rho$pp), : convergence code 1 from bobyqa: bobyqa -- maximum number of function evaluations exceeded 2: In checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, : Model failed to converge with max|grad| = 33.3731 (tol = 0.002) 3: In checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, : Model failed to converge: degenerate Hessian with 4 negative eigenvaluesとなり、計算できません。
ランダム効果が複雑なため、計算を近似的に収束させるパラメータ(関数の最大数, 計算の繰り返し数)を指定すらばよいのかと思うのですが、このエラーからでは指定法が分からず悩んでおります。
尚、過去のバージョンでは、maxFN, maxIterという引き数で直接指定できたと情報を得ているのですが、現行版のパッケージ'lmer4'では、これらの書式が廃止されているようで、使えないようです。
ご存知の方がいらっしゃいましたら、ご教授いただければと思います。
lmer(formula= Y ~ X1+X2+X3+X4+X5+(X1+X2+X3+X4+X5|Random), control=lmerControl(optCtrl=list(maxfun=3000, maxiter=1000)))とする事で指定できました。まだエラーの嵐ですが、汗をかいてみたいと思います。 -- 初心者 2014-09-03 (水) 10:50:56
こまったちゃん (2014-08-28 (木) 07:38:58)
『Rによるやさしい統計学』でRを勉強しています。
「TukeyHSD(aov(統計テスト2~指導法2))」という関数を打ち込んだところ、TukeyHSD(aov(統計テスト2~指導法2)) 以下にエラー grepl("\n", lines, fixed = TRUE) : '<87><e5><b0>取ウ包シ<92>' に不正なマルチバイト文字がありますと表示されました。 ブログなどに書いてある対策を見ましたが、よくわかりませんでした。 誰かわかる方、いらっしゃいますか?
> 統計テスト2 <- iris[,1]
> 指導法2 <- iris[,5]
> TukeyHSD(aov(統計テスト2~指導法2))
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = 統計テスト2 ~ 指導法2)
$指導法2
diff lwr upr p adj
versicolor-setosa 0.930 0.6862273 1.1737727 0
virginica-setosa 1.582 1.3382273 1.8257727 0
virginica-versicolor 0.652 0.4082273 0.8957727 0> .Platform$OS.type; R.Version()$version.string
[1] "windows"
[1] "R version 3.1.1 (2014-07-10)"
> options(keep.source = FALSE)
> TukeyHSD(aov(統計テスト2~指導法2))
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = 統計テスト2 ~ 指導法2)
$指導法2
diff lwr upr p adj
versicolor-setosa 0.930 0.6862273 1.1737727 0
virginica-setosa 1.582 1.3382273 1.8257727 0
virginica-versicolor 0.652 0.4082273 0.8957727 0最近機械学習について興味をもちだしたもの (2014-08-26 (火) 13:27:23)
caretパッケージのチューニングパラメーター調整方法についてなのですが、最新のパッケージでは createGridがなくなりました。
expand.grid でやってる例が多いのですが、自分でパラメーターをふる場合、どうやって範囲をきめたら良いのでしょうか?
手法によって異なると思うのですが(例えば deep learning と randomForestの場合)
また createGridはなぜ無くなったのでしょうか?
http://topepo.github.io/caret/training.html
を読むと自動でパラメーターをふる関数が用意されてるみたいです。これが新しくできたから createGrid がなくなったのでしょうか?
よろしくお願い致します。
うどん (2014-08-24 (日) 17:55:11)
win7のR x64 2.15.0でパッケージurcaを使い、下記のサンプルの実行とsummaryにより得られた(略)内のtestと10pct等の数値を比較したく、取り出したいのですが、names()を実行するとs4であるとのエラーが出ました。s4を調べ本サイトで示されていましたスロットの内容を見ましたが、結果の取り出しに関するものがありませんでした。取り出し方法をお教えいただけるとありがたいです。
よろしくお願いいたします。library(urca) data(denmark) sjd <- denmark[, c("LRM", "LRY", "IBO", "IDE")] sjd.vecm <- ca.jo(sjd, ecdet = "const", type="eigen", K=2, spec="longrun", season=4) summary(sjd.vecm) (略) VValues of teststatistic and critical values of test: test 10pct 5pct 1pct r <= 3 | 2.35 7.52 9.24 12.97 r <= 2 | 6.34 13.75 15.67 20.20 r <= 1 | 10.36 19.77 22.00 26.81 r = 0 | 30.09 25.56 28.14 33.24 (略)
> str(sjd.vecm)
:
..@ cval : num [1:4, 1:3] 7.52 13.75 19.77 25.56 9.24 ...
..@ teststat : num [1:4] 2.35 6.34 10.36 30.09
> sjd.vecm@teststat
[1] 2.352233 6.342730 10.361950 30.087451
> sjd.vecm@cval
10pct 5pct 1pct
r <= 3 | 7.52 9.24 12.97
r <= 2 | 13.75 15.67 20.20
r <= 1 | 19.77 22.00 26.81
r = 0 | 25.56 28.14 33.24木偶 坊 (2014-08-20 (水) 10:04:08)
初めまして、教えてください。Windows 8, R version 2.9.2 を使用しています。
barplot()関数を使って処理しています。データ数を棒グラフ上に記述したいのですが、真ん中("centre")に記述できません。左("left")にも記述できません。
R version 3.1.1をダウンロードしてやってみましたが、同じです。変わりません。
ご教示をお願いします。setwd("f://Rdata") par(new = FALSE) par(mar = c(2, 4, 3, 0)) par(mgp = c(2, 0.8, 0)) par(lwd = 2) par(mfrow = c(1, 1)) par(cex = 1.2) par(ps = 14) par(xpd = FALSE) par(pty = "s") par(xaxs = "r") dta <- c(13278, 8860, 9082, 9435, 7222, 5431, 5091, 6193, 5557, 3716) main = "人口の多い都道府県(H25末資料)" par(bg = "white") y <- dta/10 xlab = "" ylab = "人口(万人)" ylim = c(0,1500) barplot(y, angle = 45, beside = FALSE, horiz = F, density = 16, col = rainbow(10), main = main, ylim = ylim, ylab = ylab, space = 0.1, width = 0.91, adj = 0.5, names = c("東京","大阪","神奈川","愛知","埼玉","北海道", "福岡","千葉","兵庫","静岡")) mn <- y x <- 1:10 text(x, mn, format(justify = "left", mn, adj = 0, trim = TRUE, width = 0), pos = 3, offset = 0.5, lty = 0, col = "blue") axis(2, at = NULL, labels = TRUE, tick = FALSE) rm(y, dta)
x <- barplot(y, 以下略 中略 mn <- y #x <- 1:10 使わないので,コメントアウト text(x, mn, 以下略。なお,label の format や offset も指定不要と思います。
kddoi (2014-08-19 (火) 17:38:52)
以前こちらのウェブサイトでお世話になりました。再度、困ったことがあり質問させてください。
行列の各列を複数のキーと見做してorderの引数として与え、ソートしたいと思っております。ただ、与えられる行列の列数が一定ではないため、任意の数の列数に対応したいのですが、そのような方法が分からずに苦労しております。
まず、次のようなことができることが最終目標です。
以下のプログラムではorderにa[,1],a[,2]を別々に与えているので上手く動いているのですが、これを関数として実装する際、引数の列数は可変を想定しております。
よって最終的には、a[,1]やa[,2]のように列を指定することなくorderが正しく動作することを目指しております(以下は最初からソートされている例です)。cov <- c(1, 1, 1, 2, 2, 2, 1, 2, 3, 1, 2, 3) dim(cov) <- c(6, 2) cov[order(cov[, 1], cov[, 2]), ]最初、orderが引数にリストを取ってくれないかと思ったのですが、「型 'list'('orderVector1' における)は未実装です 」と言われてしまいます。ただ、order自体が引数として「...」として複数の引数を受け取っているので、pairlistとして作成すれば何とか騙せないかと思ったのですが、この方針では今のところ上手く動かせて
おりません。
どうしても駄目ならば、文字列処理でcmd <- "" cmd <- paste("cov[order(cov[, 1]", sep="") for (i in 2:ncol(cov)) { cmd <- paste(cmd, ",cov[,", sep="") cmd <- paste(cmd, i, "]", sep="") } cmd <- paste(cmd, "),]", sep="") eval(parse(text=cmd))などと行うことも検討しているのですが、もう少しよい方法がありましたら何卒教えてくださりますよう、お願いいたします。
registar (2014-08-18 (月) 19:02:27)
DBに問い合わせるSQL文の中に、下記のソースのwhere id='8888'の様にシングルクオテーションが入ってる場合、そのシングルクオテーションを無視してSQL文を実効出来るようなコマンドはありませんか?お手数ですが御指導の程何卒宜しくお願い申し上げます。drv <- JDBC(rjdbc_driver, rjdbc_jar_file, rjdbc_id_quote) conn <- dbConnect(drv, rjdbc_url, rjdbc_user, rjdbc_passwd) dataframe_resource <- dbGetQuery(conn, "select id, aid, bid from xxx where id='8888'")他の言語と連携させてデータを取ってきているためシングルクオテーションが入ってしまっていてR側で無視したい状況です。rjdbc_driver, rjdbc_jar_file, rjdbc_id_quote, rjdbc_url, rjdbc_user, rjdbc_passwd には任意の値を入れてあります。
初心者 (2014-08-13 (水) 02:25:24)
glmの説明について調べると必ずと言って良いほど重回帰分析では応答変数に正規分布を仮定したが、glmでは応答変数に他の分布を仮定して云々と書かれています。
しかし重回帰分析の応答変数に何らかの分布を仮定しなくとも最小二乗法で係数推定ができるはずなので(最小二乗法で解を求める方法は何度か確認しました)正規分布の仮定をおく理由がわかりません。
質問1 仮定をおく理由を教えてください
質問2 誤差項は正規分布を仮定していますが上記と関係しているのでしょうか?
質問3 lmの結果に応答変数の正規性検定結果は含まれていませんでしょうか。
(2014-08-12 (火) 00:36:35)
大量の画像について、顔認識して顔の特徴分析をしようと思っています。
画像から顔の認識をして、目鼻口眉輪郭などの特徴量を計算するパッケージはありますでしょうか。
どうぞよろしくお願いします。
まさし (2014-08-08 (金) 09:45:40)
R言語でのfftw3のインストール方法を教えていただけないでしょうか?私もいくら調べても発見することができませんでした。よろしければインストール方法を教えていただければ幸いです。
$ sudo apt-get install libfftw3-devMacOSX (MacPorts)の場合
$ sudo port install fftw-3Windowsの場合は公式サイトにあるWindows Installation Notesを参考にされてはいかがでしょうか。私はWindowsユーザではないし、Windowsが手元にないので確認できませんが、Windows7 64bitの共有ライブラリ(*.dll)のコピー先をざっと検索してみたところ、ここに書いてあるように、あり得ないくらいトリッキーな仕様みたいです。これは、はまっちゃう人が続出しても仕方がない。Windowsユーザの常識かも知れないが、吃驚しました。 -- 2014-08-08 (金) 10:08:12
たけし (2014-08-07 (木) 10:14:22)
R言語にあるbiOps内のimgFFT()関数を用いて、あるパッケージRImageBook内のhouses.pngをフーリエ変換しようと試みたのですが、以下のエラーが発生しますhouses <-readImage(system.file("images/houses.png", package="RImageBook?")) housesb <- E2b(houses) housesbfft <- imgFFT(housesb) str(housesbfft) 以下にエラー imgFFT(housesb) : Sorry, fftw not available str(housesbfft) 以下にエラー str(housesbfft) : オブジェクト 'housesbfft' がありませんこのエラーからimgFFT()関数での処理で失敗していると判断しました
以下imgFFT()関数の処理内容ですfunction (imgdata, shift = TRUE) { if (!FALSE) stop("Sorry, fftw not available") imgmatrix <- array(imgdata) depth <- if (attr(imgdata, "type") == "grey") 1 else dim(imgdata)[3] width <- dim(imgdata)[2] height <- dim(imgdata)[1] res <- .C("fft_image", image = as.complex(imgmatrix), width = as.integer(width), height = as.integer(height), depth = as.integer(depth), PACKAGE = "biOps") imgdim <- c(height, width, if (depth == 3) depth else NULL) img <- array(res$image, dim = imgdim) if (shift) imgFFTShift(img) else img } environment: namespace:biOps> OS Windows7 64bit R インストールパッケージ:R 2.15.3 ImageMagick インストールパッケージ:ImageMagick-6.7.6-9-Q16-windows-dll.exe GTK2 インストールパッケージ:gtk2-runtime-2.16.6-2010-05-12-ash.exe EBImage インストールパッケージ:EBImage 3.12.0.zip RImageBook インストールパッケージ:バージョン 3.0.1 の R の下で造られたもの 参考にした本 Rで学ぶデータサイエンス デジタル画像処理 共立出版助言お願いいたします
ishi03 (2014-08-05 (火) 11:07:31)
R初心者のishi03と申します。
この質問の背景をご説明致します。
私は,理系出身ではありますが,これまで解析的なことを全く行ったことが無いのですが,以下のような関数を最大にするパラメータk1,k2を求める必要に迫られています。
関数は,f(k1,k2)=-N*(ln(4*pi)-N*(ln(d(k1,k2)))+k1*a+k2*b d(k1,k2)=( (4*pi)^(-1))*∫[0→2*pi]∫[0→pi]{sin[θ]*exp( (sin[θ]^2)*(k1*(cos[φ])^2+k2*(sin[φ])^2))}dθdφ k1≦k2≦0,N,a,bは事前に与えられている定数で定義されています。
k1,k2を求める上で,関数f(k1,k2)を最大にする必要があるので,Rのoptim関数を使用するのだろうと考え,以下に示すようなコードを記述してみたのですが,Nelder-Mead direct search function minimizer 以下にエラー optim(par = initial, fn = f, control = list(fnscale = -1, trace = TRUE)) : cannot coerce type 'closure' to vector of type 'double'との結果が返って参ります。
どうにか,こちらのk1,k2を算出することはできませんでしょうか。お教えいただけますと幸いです~。 以下に記述しましたコードを示します。# 事前に定義される定数(仮で設定) n <- 10 τmin <- 10 τint <- 5 # d(k1, k2)を関数で定義 library(cubature) d <- function(x, k1, k2) {sin(x[1]) * exp(((k1 *(cos(x[2]))^2 + k2 * (sin(x[2]))^2) * (sin(x[1]))^2)} d_int <- adaptIntegrate(c, c(0, 0), c(pi, 2 * pi)) # f(k1, k2)を関数で定義 # k1,k2 の条件を逸した場合は異常値を返すようにしてみました。(←良い方法があればお教え頂きたいです。)) f <- function(par) { k1 <- par[1] k2 <- par[2] if (k1 > k2 || k2 > 0) {return(-20000)} return(function(par) { k1 <- par[1] k2 <- par[2] N <- n a <- τmin b <- τint -N * log(4 * pi) - n * log(d_int) + k1 * a + k2 * b})} # 最大化する k1,k2 の算出 initial <- c(0, 0) opt <- optim(par=initial, fn=f, control=list(fnscale=-1, trace=TRUE))なお,使用環境はR version 3.1.1 (2014-07-10),win7の64bitになります。
大変申し訳ありませんが,宜しくお願い申し上げます。
# 事前に定義される定数(仮で設定)
n <- 10
τmin <- 10
τint <- 5
# d(k1, k2)を関数で定義
library(cubature)
d <- function(x, k1, k2)
# {sin(x[1]) * exp(((k1 *(cos(x[2]))^2 + k2 * (sin(x[2]))^2) * (sin(x[1]))^2)}
{sin(x[1]) * exp((k1 * cos(x[2])^2 + k2 * sin(x[2])^2) * sin(x[1])^2)}
# d_int <- adaptIntegrate(c, c(0, 0), c(pi, 2 * pi))
# f(k1, k2)を関数で定義
# k1,k2 の条件を逸した場合は異常値を返すようにしてみました。(←良い方法があればお教え頂きたいです。))
f <- function(par) {
k1 <- par[1]
k2 <- par[2]
if (k1 > k2 || k2 > 0) {return(-20000)}
# return(function(par) {
# k1 <- par[1]
# k2 <- par[2]
N <- n
a <- τmin
b <- τint
d_int <- adaptIntegrate(d, c(0, 0), c(pi, 2 * pi), k1=k1, k2=k2)$integral
-N * log(4 * pi) - n * log(d_int) + k1 * a + k2 * b
# })
}
# 最大化する k1,k2 の算出
initial <- c(-0.3, -0.2)
opt <- optim(par=initial, fn=f, control=list(fnscale=-1, trace=FALSE, maxit=10000))
optkamo (2014-07-31 (木) 12:34:11)
RHmmというパッケージにHMMFitという関数があり、その中身はHMMFitと入力すると見ることができるのですが、HMMFitの中にはBaumWelchという関数が使われており、その中身を見ようとすると、
> BaumWelch Error: object 'BaumWelch' not foundとなってしまい、関数が見つかりません。BaumWelchの中身を見る方法はありますでしょうか。もしくは作者のほうでパッケージ内の関数の中身を見えないようにする設定ができ、今回それに該当しているのでしょうか。
Google検索で似たようなケースがないか調べてみましたが、見つかりませんでした。ご教授よろしくお願いします。
名無し (2014-07-30 (水) 20:20:49)
c("123","abcd","xy") という文字列ベクトルに対して各要素の長さを5に統一し、不足部分は"*"で穴埋めしたいです。
処理後の文字列ベクトル c("**123","*abcd","***xy") formatCやsprintfについて調べて見ましたが、数値ベクトルのゼロサプレスはみつかるものの、文字列ベクトルのケースは見つかりませんでした。
ご教示お願いいたします。
> x <- c("123","abcd","xy")
> sapply(x, function(z) paste(substr("*****", 1, 5-nchar(z)), z, sep=""))
123 abcd xy
"**123" "*abcd" "***xy"
> x <- c("", "1", "12", "123", "1234", "12345", "123456", "123456789")
> sapply(x, function(z) paste(substr("*****", 1, 5-nchar(z)), z, sep=""))
1 12 123 1234 12345 123456 123456789
"*****" "****1" "***12" "**123" "*1234" "12345" "123456" "123456789"
どうですかね? -- 河童の屁は,河童にあらず,屁である。 2014-07-30 (水) 22:00:02> x <- c("123","abcd","xy","55555555")
> gsub(' ','*',sprintf("%5s",x))
[1] "**123" "*abcd" "***xy" "55555555"saka (2014-07-30 (水) 18:52:04)
Rstudio Server(CentOS, R version 3.0.2)を使い、mailRパッケージでメール送信をしたいと思っています。
以下のようなスクリプトで(メールアドレス等はダミー)、デスクトップのRstudio(win, Version 0.98.953, R version 2.15.1)だと実行できるのですが、Rstudio Serverだとエラーになります。
考えられる理由や解決法が分かれば、ご教授お願いできませんでしょうか。よろしくお願いします。send.mail(from = "sender@gmail.com", to = c("recipient1@gmail.com", "recipient2@gmail.com"), subject = "Subject of the email", body = "Body of the email", smtp = list(host.name = "smtp.gmail.com", port = 465, user.name = "gmail_username", passwd = "password", ssl = TRUE), authenticate = TRUE, send = TRUE)Rstudio Serverでのエラー表示
org.apache.commons.mail.EmailException: Sending the email to the following server failed : smtp.gmail.com:465 at org.apache.commons.mail.Email.sendMimeMessage(Email.java:1410) at org.apache.commons.mail.Email.send(Email.java:1437) at java.lang.reflect.Method.invoke(libgcj.so.10) at RJavaTools.invokeMethod(RJavaTools.java:386) Caused by: javax.mail.MessagingException: IOException while sending message; nested exception is: javax.activation.UnsupportedDataTypeException: no object DCH for MIME type text/plain; charset=ISO-8859-1 at com.sun.mail.smtp.SMTPTransport.sendMessage(SMTPTransport.java:1182) at javax.mail.Transport.send0(Transport.java:254) at javax.mail.Transport.send(Transport.java:124) at org.apache.commons.mail.Email.sendMimeMessage(Email.java:1400) ...3 more Caused by: javax.activation.UnsupportedDataTypeException: no object DCH for MIME type text/plain; charset=ISO-8859-1 at javax.activation.ObjectDataContentHandler.writeTo(libgcj.so.10) at javax.activation.DataHandler.writeTo(libgcj.so.10) at javax.mail.internet.MimeBodyPart.writeTo(MimeBodyPart.java:1593) at javax.mail.internet.MimeMessage.writeTo(MimeMessage.java:1839) at com.sun.mail.smtp.SMTPTransport.sendMessage(SMTPTransport.java:1134) ...6 more NULL Error: EmailException (Java): Sending the email to the following server failed : smtp.gmail.com:465
清水 (2014-07-28 (月) 23:01:43)
CentOSサーバ上でrApache環境を構築して、RプログラムをWeb環境で動作させています。
同環境下で、Apache POIライブラリ使用によるExcelファイル作成の必要性があったため、CRANのrJavaライブラリを使用しなければなりません。
そのままではrJavaライブラリをローディングできないので、http://www.r-bloggers.com/making-rapache-load-rjava/にあるように、rApache_rJava.confファイルを作成して、libjvm.soへのパスを通すことにより、library(rJava)によるライブラリのローディングが可能になりました。
しかし実際にライブラリを使用するためには.jinit()を発行する必要があり、これを記述したRページにアクセスした瞬間「このページは表示できません」となり、hs_err_pid#####.logに膨大なログが吐かれます。
Rソースは単純に以下のような内容で、環境変数の不足を疑って追加しても全く変化がありません。library(rJava) .jinit()1000行近くあるログの先頭は以下のような内容です。
# There is insufficient memory for the Java Runtime Environment to continue. # Native memory allocation (malloc) failed to allocate 2555904 bytes for committing reserved memory. # Possible reasons: # The system is out of physical RAM or swap space # In 32 bit mode, the process size limit was hit # Possible solutions: # Reduce memory load on the system # Increase physical memory or swap space # Check if swap backing store is full # Use 64 bit Java on a 64 bit OS # Decrease Java heap size (-Xmx/-Xms) # Decrease number of Java threads # Decrease Java thread stack sizes (-Xss) # Set larger code cache with -XX:ReservedCodeCacheSize= # This output file may be truncated or incomplete. # # Out of Memory Error (os_linux.cpp:2726), pid=13829, tid=140457446467552 # # JRE version: (7.0_51-b13) (build )~ # Java VM: Java HotSpot(TM) 64-Bit Server VM (24.51-b03 mixed mode linux-amd64 compressed oops) # Failed to write core dump. Core dumps have been disabled. To enable core dumping, # try "ulimit -c unlimited" before starting Java again --------------- T H R E A D --------------- Current thread (0x00007fbecf6e1000): JavaThread "Unknown thread" [_thread_in_vm, id=13829, stack(0x00007fff9ed8f000,0x00007fff9ee8f000)] Stack: [0x00007fff9ed8f000,0x00007fff9ee8f000], sp=0x00007fff9ee88fd0, free space=999k Native frames: (J=compiled Java code, j=interpreted, Vv=VM code, C=native code) V [libjvm.so+0x992f4a] VMError::report_and_die()+0x2ea V [libjvm.so+0x4931ab] report_vm_out_of_memory(char const*, int, unsigned long, char const*)+0x9b V [libjvm.so+0x81338e] os::Linux::commit_memory_impl(char*, unsigned long, bool)+0xfe V [libjvm.so+0x81383f] os::Linux::commit_memory_impl(char*, unsigned long, unsigned long, bool)+0x4f V [libjvm.so+0x813a2c] os::pd_commit_memory(char*, unsigned long, unsigned long, bool)+0xc V [libjvm.so+0x80daea] os::commit_memory(char*, unsigned long, unsigned long, bool)+0x2a V [libjvm.so+0x98e849] VirtualSpace::expand_by(unsigned long, bool)+0x1c9 V [libjvm.so+0x98e9cd] VirtualSpace::initialize(ReservedSpace, unsigned long)+0xcd V [libjvm.so+0x58a8e3] CodeHeap::reserve(unsigned long, unsigned long, unsigned long)+0x143 V [libjvm.so+0x420cf0] CodeCache::initialize()+0x80 V [libjvm.so+0x5a9605] init_globals()+0x45 V [libjvm.so+0x94ef8d] Threads::create_vm(JavaVMInitArgs*, bool*)+0x1ed V [libjvm.so+0x6307e4] JNI_CreateJavaVM+0x74 --------------- P R O C E S S ---------------"Unknown thread"が赤表示になっていてこれが原因と思われますが、対処法が全く不明です。
原因と対処法についてご教授願います。
macy (2014-07-27 (日) 18:00:29)
パッケージのマニュアル等を見ると、実行文の例文のところに# Not runという文字をよく見かけます。
このNot runはどういう意味でしょうか?
例文なので、実行するなという意味ではないような気がしたもので。
常識的なことかと思うのですが、教えて下さい。
Examples ## Not run: # 略 ## End(Not run)
初心者(R10日目) (2014-07-27 (日) 16:18:06)
以下のような文字ベクトルvがあるときに各要素の長さを取得する方法があれば教えてください。
v <- c("a","xyz","pq","")
各要素の長さとはc(1,3,2,0)を指します。
しまだ (2014-07-23 (水) 15:16:29)
ある水辺で飛来してくる水鳥をカウントし、データフレーム(data)に入れました。
中身は以下のとおり,indsは水鳥の個体数、my_time2は観測時刻です。inds my_time2 1 22 2013-03-13 08:00:00 2 6 2013-03-13 08:00:00 3 6 2013-03-13 08:16:00 4 13 2013-03-13 08:28:00 5 4 2013-03-13 08:32:00 6 2 2013-03-13 11:37:00 7 2 2013-03-13 11:45:00 8 3 2013-03-13 11:56:00 9 3 2013-03-13 12:05:00 10 3 2013-03-13 12:24:00 11 2 2013-03-13 15:47:00 12 2 2013-03-13 15:49:00 (以下、略)ここから(たとえば30分毎、あるいは1時間毎に個体数をまとめた)時系列グラフを作成する方法が分かりません。。。。histは時刻も使えるようですが・・・
x <- as.integer(data$my_time2) %/% 3600
num <- c(by(data$inds, x, sum))
time <- names(num) <- as.POSIXct(as.integer(names(num)) * 3600,
origin = "1960-01-01")
num
time
plot(time, num, type = "b")のびたき (2014-07-17 (木) 18:43:24)
初めまして、お世話になります。
以下のような条件判断で失敗しています。> a <- matrix(c(0.3, 0.15, 0.35, 0), ncol = 2, nrow = 2) > sum(a) == 0.8右辺も左辺もどちらも0.8なのでTRUEになるはずと期待しているのですが、FALSEが出力されます。
どのように直せばいいのかご教示ください。よろしくお願いいたします。
> a <- matrix(c(0.3,0.15,0.35,0),ncol=2,nrow=2)
> # 格納された16進表記
> sprintf("%A",sum(a))
[1] "0X1.9999999999999P-1"
> sprintf("%A",0.8)
[1] "0X1.999999999999AP-1"
> options(digits=22) # もうちょっと人間向けに
> sum(a)
[1] 0.7999999999999999333866
> 0.8
[1] 0.8000000000000000444089
これはFAQとしてR-FAQの7.31にあります.
たとえば
> TRUE == all.equal(sum(a), 0.8) [1] TRUEなどとすると良いでしょう -- なかま 2014-07-17 (木) 22:01:40
LOL (2014-07-10 (木) 09:51:51)
お世話になります。
数百万回for文を繰り返すような解析を行う必要が生じたため、WindowsからLinuxサーバー上で解析をすることになりました。
CPU数が2倍、コア数が4倍になるので大体8倍ぐらいの計算速度になるのかと予想したのですが、実際は2倍程度にしかなりませんでした。
とてもがっかりしたのですが、こういうものなんでしょうか?
SS (2014-07-02 (水) 09:26:06)
linux(CentOS)を使用しています。
パッケージをインストールする際に、インストールするディレクトリを指定したいのですが、どのようにすればよいのでしょうか。
また、間違ったフォルダにインストールしたとして、単にmvでパッケージを移動するだけではダメなのでしょうか。
どなたかご回答宜しくお願いします。
HY (2014-07-01 (火) 14:12:39)
過去の質問(未解決)にもありましたが、Rを使ってWilliams検定のP値を正確に出せません。
multcomp パッケージでの Williams 検定について
RでP値まで求めたいのですが、P値を計算できた方いらっしゃいますか?
以下は検証したデータです。入力データ bpdown <- data.frame( medicine=factor(rep(1:4, each=10)), sbpchange=c(153, 153, 152, 156, 158, 151, 151, 150, 148, 157, 158, 152, 152, 152, 151, 151, 157, 147, 155, 146, 153, 146, 138, 152, 140, 146, 156, 142, 147, 153, 137, 139, 141, 141, 143, 133, 147, 144, 151, 156)) #SimCompを使う方法 library(SimComp) SimTestDiff(data=bpdown, grp="medicine", resp="sbpchange", type="Williams", base=1, covar.equal=T) #[結果] # comparison endpoint margin estimate statistic p.value.raw p.value.adj #1 C 1 sbpchange 0 -9.700 -4.180 0.0002 0.0003 #2 C 2 sbpchange 0 -7.650 -3.806 0.0005 0.0008 #3 C 3 sbpchange 0 -5.367 -2.832 0.0075 0.0142 #multcompを使う方法 res1 <- aov(sbpchange ~ medicine, data=bpdown) library(multcomp) res2 <- glht(res1, linfct = mcp(medicine = "Williams")) summary(res2) #[結果] #Linear Hypotheses: # Estimate Std. Error t value Pr(>|t|) #C 1 == 0 -9.700 2.321 -4.180 < 0.001 *** #C 2 == 0 -7.650 2.010 -3.806 0.00111 ** #C 3 == 0 -5.367 1.895 -2.832 0.01410 *SASを使える方にお願いしてP値を計算していただいた場合、「毒性・薬効データの統計解析」のP64と同じ計算結果になります。
[SAS計算結果] OBS t wp wtc 1 -0.34471 0.63384 1.68830 2 -1.37884 0.95606 1.76560 3 -2.31242 0.99785 1.79073また、以下の青木先生のソースコードでt値は計算できましたが、得られたt値をpmvt関数にセットしても、やはり求めたいP値は計算できず...
ウィリアムズの方法による平均値の多重比較
のざわ (2014-07-01 (火) 11:35:21)
お世話になります。
たとえば以下を1,2,3,4,5こんな風にしたい
1,0,2,0,3,0,4,0,5,0あるいは
1,0,0,2,0,0,3,0,0,4,0,0,5,0,0と0を2個(あるいは複数)
これをforやwhileなどループを使わずに作る方法はありませんか?
x <- 1:5 c(rbind(x, numeric(length(x)))) c(rbind(x, numeric(length(x)), numeric(length(x))))とか
c(rbind(x, matrix(0, 1, length(x)))) c(rbind(x, matrix(0, 2, length(x)))) c(rbind(x, matrix(0, 10, length(x))))
pararel (2014-06-19 (木) 21:50:04)
Warning in install.packages : package ‘randomForest’ is not available (for R version 3.1.0)install.packages("randomForest")を実行すると上記の表示がされます。
R version 3.1.0でのrandomforest関数のパッケージ名を教えていただきたいと思います。
何卒よろしくお願い申し上げます。
ロビン (2014-06-08 (日) 14:01:41)
txt ファイルからNetCDF ファイルに変換したいのですが,Rを用いてできるでしょうか?
SK (2014-06-07 (土) 13:48:25)
はじめまして。semをまず動かしてみようと
http://www.okada.jp.org/RWiki/?R%A4%C7%B6%A6%CA%AC%BB%B6%B9%BD%C2%A4%CA%AC%C0%CF%A1%A6%B9%BD%C2%A4%CA%FD%C4%F8%BC%B0%A5%E2%A5%C7%A5%EB#j12acd36
の「潜在変数を仮定するモデル 」をそのまま実行させました。
およそ同じような結果になるのですが、summaryで表示される項目が全く異なります。
Goodness-of-fit indexやRMSEA index など、例示されているものがごっそり抜けています。
また、結果の最後のInterationが17ではなく15になります。
なにか設定とかが足りないでしょうか。
以下が結果です。> ans <- sem(model, cor(dat), 10) > summary(ans) Model Chisquare = 1.925355 Df = 5 Pr(>Chisq) = 0.8593742 AIC = 21.92536 BIC = -9.58757 Normalized Residuals Min. 1st Qu. Median Mean 3rd Qu. Max. -0.3976000 -0.0000002 0.0155300 0.1221000 0.2435000 0.6971000 R-square for Endogenous Variables v1 v2 v3 v4 v5 0.6297 0.0809 0.4573 0.0583 0.0506 Parameter Estimates Estimate Std Error z value Pr(>|z|) path1 0.7935558 0.4882341 1.6253592 0.10408604 v1 <--- F1 path2 -0.2843858 0.3849962 -0.7386717 0.46010637 v2 <--- F1 path3 -0.6762525 0.4525192 -1.4944171 0.13506663 v3 <--- F1 path4 -0.2414672 0.3860898 -0.6254172 0.53169727 v4 <--- F1 path5 0.2248359 0.3864879 0.5817413 0.56074096 v5 <--- F1 s1 0.3702693 0.6626860 0.5587401 0.57633909 v1 <--> v1 s2 0.9191248 0.4485423 2.0491376 0.04044867 v2 <--> v2 s3 0.5426826 0.5322169 1.0196644 0.30788765 v3 <--> v3 s4 0.9416935 0.4546109 2.0714275 0.03831887 v4 <--> v4 s5 0.9494489 0.4567576 2.0786712 0.03764758 v5 <--> v5 Iterations = 15 利用環境Windows7Pro64bit R i386 3.1.0 > sessionInfo() R version 3.1.0 (2014-04-10) Platform: i386-w64-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] splines stats graphics grDevices utils datasets methods base other attached packages: [1] sem_3.1-4 car_2.0-20 Rcmdr_2.0-4 loaded via a namespace (and not attached): [1] MASS_7.3-31 matrixcalc_1.0-3 nnet_7.3-8 tcltk_3.1.0 tcltk2_1.2-10 [6] tools_3.1.0
K (2014-06-02 (月) 16:32:32)
お世話になります。フィッシャーの判別分析を行うためMASSパッケージのldaを使用しています。以下のように1変数で判別式を求めるという処理をしているのですが、ldaが出力する判別クラスと、係数と定数項をldaから回収して作成した判別式による判別クラスが一致しません。
library(MASS) dat <- data.frame(value=c(9.43,9.43,9.47,9.50,9.60,9.62,9.62, 9.65,9.66,9.69,9.72,9.72,9.72,9.75,9.78,9.78,9.78,9.81), class=c(rep("A",5),rep("B",13))) z <- lda(class~., dat) dat <- cbind(dat, predict(z, dat)$class) colnames(dat)[ncol(dat)] <- "pred"datは以下のようになります。classは真のクラス、predはldaの判別クラスです。
value class pred 1 9.43 A A 2 9.43 A A 3 9.47 A A 4 9.50 A A 5 9.60 A B 6 9.62 B B 7 9.62 B B 8 9.65 B B 9 9.66 B B 10 9.69 B B 11 9.72 B B 12 9.72 B B 13 9.72 B B 14 9.75 B B 15 9.78 B B 16 9.78 B B 17 9.78 B B 18 9.81 B Bここで、変数をxとしたときの判別式は以下のように計算されるそうです。
#係数の算出 coeff <- z$scaling #定数項の算出 const <- apply(z$means %*% z$scaling, 2, mean)
0 = coeff*x - const x = const/coeff = 146.5764/15.26728 = "9.600692"
この結果から、"9.600692"を判別面としてpredのクラスが決められているはずなのですが、5番目のpredがAではなくBになっており、ldaの判別クラスと一致しません。なぜこのような不一致が生じるのでしょうか?
特定のパッケージのことで回答しづらいと思いますが、何でも構いませんのでご助言宜しくお願いします。
なお、係数や定数項の求め方は以下の書籍やホームページの記載によりました。
http://www.morikita.co.jp/books/book/536
http://homepage2.nifty.com/nandemoarchive/GLM/tahenryou_03_discrim.htm
http://entertainment-lab.blogspot.jp/2012/12/r.html
環境はWin 7, R 3.0.2, MASS 7.3-29です。
> library(MASS)
> dat <- data.frame(value=c(9.43,9.43,9.47,9.50,9.60,9.62,9.62,
9.65,9.66,9.69,9.72,9.72,9.72,9.75,9.78,9.78,9.78,9.81),
class=c(rep("A",5),rep("B",13)))
> z <- lda(class~., dat)
ここからデバッグ
> debug(predict)
> predict(z)
debugging in: predict(z)
debug: UseMethod("predict")
ng = 2
dm
LD1
A -2.5292791
B 0.9727997
prior
A B
0.2777778 0.7222222
x
value
1 9.43
2 9.43
3 9.47
4 9.50
5 9.60
略
dist <- matrix(0.5 * rowSums(dm^2) - log(prior), nrow(x), length(prior),
byrow = TRUE) - x[, 1L:dimen, drop = FALSE] %*% t(dm)
A B
1 -4.080144 4.0907860
2 -4.080144 4.0907860
3 -2.535536 3.4967058
4 -1.377080 3.0511458
5 2.484441 1.5659455
略
dist <- exp(-(dist - apply(dist, 1L, min, na.rm = TRUE)))
A B
1 1.000000e+00 0.0002827549
2 1.000000e+00 0.0002827549
3 1.000000e+00 0.0024001077
4 1.000000e+00 0.0119356523
5 3.991190e-01 1.0000000000
略
posterior <- dist/drop(dist %*% rep(1, ng))
A B
1 9.997173e-01 0.000282675
2 9.997173e-01 0.000282675
3 9.976056e-01 0.002394361
4 9.882051e-01 0.011794873
5 2.852645e-01 0.714735492
略
cl <- factor(nm[max.col(posterior)], levels = object$lev)
[1] A A A A B B B B B B B B B B B B B B
exiting from: predict(z)
$class
[1] A A A A B B B B B B B B B B B B B B
Levels: A B
$posterior
A B
1 9.997173e-01 0.000282675
2 9.997173e-01 0.000282675
3 9.976056e-01 0.002394361
4 9.882051e-01 0.011794873
5 2.852645e-01 0.714735492
略
$x
LD1
1 -3.38424667
2 -3.38424667
3 -2.77355554
4 -2.31553720
5 -0.78880937
略ay (2014-05-29 (木) 15:39:32)
MacのOS X MavericksでRを使用しています。
画像処理を行うため、biOpsが必要になったのですが、ターミナルで
install.packages("biOps", repos="http://cran.md.tsukuba.ac.jp/", type="source")
を入力したところ以下のようなメッセージが表示されました
sudo port install ImageMagick
♥ チョキチョキ,チョッキンと切り取り警告メッセージ: In install.packages("biOps", repos = "http://cran.md.tsukuba.ac.jp/", : パッケージ ‘biOps’ のインストールは、ゼロでない終了値をもちましたまた、biOpsのサイト(http://cran.r-project.org/web/packages/biOps/index.html)へ行ってみると
OS X Mavericks binaries: r-release: not available
と書かれていました。MavericksでのbiOpsの使用は不可能なのでしょうか?
ご存知の方いらっしゃいましたらよろしくお願いします。
$ sw_vers ProductName: Mac OS X ProductVersion: 10.9.3 BuildVersion: 13D65MacPortsでbiOpsパッケージに必要なPortを事前に入れておいてから、Rの中からbiOpsパッケージをインストールします。
> install.packages("biOps", repos="http://cran.md.tsukuba.ac.jp/",
type="source", configure.vars = 'LDFLAGS=-L/opt/local/lib')
URL 'http://cran.md.tsukuba.ac.jp/src/contrib/biOps_0.2.2.tar.gz'
を試しています
[snip]
* DONE (biOps)
[snip]
正常にインストールされれば、biOps.soは次のようになっています。
$ otool -L /Library/Frameworks/R.framework/Versions/3.1/
Resources/library/biOps/libs/biOps.so
/Library/Frameworks/R.framework/Versions/3.1/Resources/
library/biOps/libs/biOps.so:
biOps.so (compatibility version 0.0.0,
current version 0.0.0)
/opt/local/lib/libtiff.5.dylib (compatibility version 8.0.0,
current version 8.0.0)
/opt/local/lib/libjpeg.9.dylib (compatibility version 11.0.0,
current version 11.0.0)
/opt/local/lib/libfftw3.3.dylib (compatibility version 8.0.0,
current version 8.4.0)
/Library/Frameworks/R.framework/Versions/3.1/Resources/
lib/libR.dylib (compatibility version 3.1.0,
current version 3.1.0)
/System/Library/Frameworks/CoreFoundation.framework/
Versions/A/CoreFoundation (compatibility version 150.0.0,
current version 855.16.0)
/usr/lib/libSystem.B.dylib (compatibility version 1.0.0,
current version 1197.1.1)
もし、libtiff.5.dylibが表示されなければ、正常にインストールできたように見えても、正常にコンパイルできていないためにreadTiff()は使えません。libjpeg.9.dylibやlibfftw3.3.dylibも同様です。なお、MacPortsユーザは、外部ライブラリに依存するパッケージのインストールでいちいちCFLAGS, CPPFLAGS, CXXFLAGSを指定せずにすむように、/Library/Frameworks/R.framework/Resources/etc/Makeconfを書き換えておくと便利です。
$ diff /Library/Frameworks/R.framework/
Resources/etc/Makeconf{.orig,}
18c18
< CFLAGS = -Wall -mtune=core2 -g -O2 $(LTO)
---
> CFLAGS = -Wall -mtune=core2 -g -O2 $(LTO) -I/opt/local/include
20c20
< CPPFLAGS = -I/usr/local/include -I/usr/local/include/freetype2
-I/opt/X11/include
---
> CPPFLAGS = -I/usr/local/include -I/usr/local/include/freetype2
-I/opt/X11/include -I/opt/local/include
23c23
< CXXFLAGS = -Wall -mtune=core2 -g -O2 $(LTO)
---
> CXXFLAGS = -Wall -mtune=core2 -g -O2 $(LTO)
-I/opt/local/include
69c69
< LDFLAGS = -L/usr/local/lib
---
> LDFLAGS = -L/usr/local/lib -L/opt/local/libmao (2014-05-29 (木) 11:33:02)
お世話になります。以下のような行列の列1,2,3にcofの1,2,3番目の要素をそれぞれ掛け算したいと考えています。matrix(1:9,3,3) cof <- 2:4このとき、どのような処理をすればよいでしょうか。*や%*%を使ってみましたが、期待の結果が得られません。
宜しくお願いします。
> x <- matrix(1:9,3,3)
> cof <- 2:4
> t(t(x)*cof)
[,1] [,2] [,3]
[1,] 2 12 28
[2,] 4 15 32
[3,] 6 18 36永野 (2014-05-26 (月) 19:11:52)
Windows8でRstudioをインストールした後、実行しようとしたところ、「Fatal error:ERROR system error 5(アクセスが拒否されました)…」なるメッセージが表示され、起動することができませんでした。
どなたか対処方法をご存じの方がいらっしゃいましたら、ご指南のほどよろしくお願いいたします。
paul (2014-05-17 (土) 21:31:42)
お世話になります。
あるデータフレームの列を除こうとしています。
抜こうとしている列は数値で、その全値を足しこんだ総和が”0”のときの列を抜くようにデータフレームを再定義しようとしたときに、どうもうまくその列を抜けていません。以下のように実施しています。Base01 <- Base00[, (colSums(Base00[,14:50]) != 0)]colSumsで見たと結果で0となっている列が排除されていないようでした。
スマートな方法はありますでしょうか。
ちなみに、行を対象にした特定の行排除には以下のように実施して、期待通りの結果となりました。Base01 <- subset(Base01, rowSums(Base00[,14:50]) != 0)
> Base00 <- data.frame(a=1:5, b=-2:2, c=rnorm(5)) > Base00 a b c 1 1 -2 2.3755710 2 2 -1 -1.5356930 3 3 0 0.3075105 4 4 1 -2.2391591 5 5 2 -0.8341275 > Base01 <- Base00[, colSums(Base00[,1:3]) != 0] > Base01 a c 1 1 2.3755710 2 2 -1.5356930 3 3 0.3075105 4 4 -2.2391591 5 5 -0.8341275
☆ (2014-05-06 (火) 12:26:35)
お世話になっております。
質問項目のデータを用いて主成分分析を適用したかったのですが、順序尺度(6件法)であることも考慮してカテゴリカルな主成分分析を行いたいと考えました。
SPSSでは「カテゴリカル主成分分析」という分析方法が使えると伺ったのですが、現在持ちあわせておりません。
そこでR、できればRコマンダーで分析できれば、と考えたのですが手順がいまいち分かりませんでした。Rコマンダーのデフォルトでは相関行列を使うか・分散共分散行列を使うかの選択しかなかったため、項目が不等分散であったことも考慮して、まずはスピアマンの相関行列を作成しました。
この相関行列に主成分分析を適用したいと考えたのですが、Rコマンダーでそのようなことは可能でしょうか。できないようでしたら、Rでの解決方法もご指導いただけましたら幸いに存じます。
また、このような方法はカテゴリカルな主成分分析と言えるのでしょうか…
的外れな質問でしたら、大変申し訳ございません。
どうぞよろしくお願い申し上げます。
dengaku (2014-05-06 (火) 01:25:02)
お世話になります。
windows7で32bit版のR3.1.0を使用しております。
R3.1.0でscan関数に以下のような処理を行わせると、半角文字を処理する場合と、全角文字を処理する場合では、
出力結果が異なることに気がつきました。
どうも、区切り文字で区切る全角文字が1文字の場合、改行コードが加えられ、区切り文字で正しく要素毎に
分割されない場合があるようです。scan(text = "a,b,c", what="character", sep=",") Read 3 items [1] "a" "b" "c" scan(text = "ab,cd,e", what="character", sep=",") Read 3 items [1] "ab" "cd" "e" scan(text = "あ,い,う", what="character", sep=",") Read 1 item [1] "あ,い,う\n" scan(text = "あい,うえ,お", what="character", sep=",") Read 3 items [1] "あい" "うえ" "お\n" scan(text = "あい,うえ,おか", what="character", sep=",") Read 3 items [1] "あい" "うえ" "おか"R3.0.3以前では、上記のような全角文字を対象とした場合でも、半角文字の処理結果と同じように、改行コードが
加えられることなく、下記のように区切り文字で正しく区切られた結果が出力されておりました。
x <- scan(text="あ,い,う",what="character",sep=",")
Read 3 items
x
[1] "あ" "い" "う"
もし、私と同様の結果が再現されるようでしたら、教えていただきたいのですが、
全角文字を対象とした場合のR3.1.0のscan関数の出力結果は、正しい出力結果なのでしょうか。
それとも、バグなのでしょうか。
何卒、よろしくお願い致します。
> strsplit("a,b,c ",",")
[1] "a" "b" "c "
> strsplit("あ,い,う",",")
[1] "あ" "い" "う"
> strsplit("あい,うえ,お",",")
[1] "あい" "うえ" "お"まる (2014-04-30 (水) 09:32:08)
お世話になっております。
Rでエラーが出て、計算ができません。
IRTの段階反応モデル(grm)を行った際に、以下のエラーが出ました。以下にエラー log.pr[xj, ] : 添え字が許される範囲外ですこれは何を意味するエラーなのでしょうか。
ご教授願います。
> (a <- matrix(1:12, 3)); a[5,]
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12
以下にエラー a[5, ] : 添え字が許される範囲外です
a は3行4列なのに,5行目を参照しようとした。ということ。 -- 河童の屁は,河童にあらず,屁である。 2014-04-30 (水) 09:46:56しよう (2014-04-24 (木) 16:53:07)
a1, a2, a3....a10という変数があり、この中に文字列が格納されています。a1 <- c("AAA","BBB") a2 <- c("CCC","DDD") a3 <- c("EEE","FFF") a4 <- c("GGG","HHH") a5 <- c("III","JJJ") a6 <- c("KKK","LLL") a7 <- c("MMM","NNN") a8 <- c("OOO","PPP") a9 <- c("QQQ","RRR") a10 <- c("SSS","TTT")このとき、a1〜a10をすべて結合したいのですが、
c(a1,a2,a3,a4,a5,a6,a7,a8,a9,a10)と入力するのは醜いです。何かよい方法は無いでしょうか。
宜しくお願いします。
ans <- NULL
for (i in 1:10) {
eval(parse(text=paste("ans <- c(ans, a", i, ")", sep="")))
}
ans
いずれにしても,少なくとも,データを変数にではなく,リストや配列として用意すれば,まだましです。なぜ,そんな風に,変数を使わなければならなくなったのかをよく考えてみてください。J (2014-04-23 (水) 09:48:29)
お世話になります。Win7, R3.0.2を使用しています。
以下のように長さの異なるベクトルa,bをcbindすると、同じ長さになるようにベクトルaの要素が繰り返されます。a <- c(1, 2, 3) b <- c(4, 5, 6, 7, 8) cbind(a, b)ここで、繰り返されるaの要素を0またはNAにしたいのですが、何かよい方法は無いでしょうか。実際のデータでは10個ほどの長さの異なるベクトルをcbindしようとしているので、それに対応できるような方法を探しています。
お手数ですが、ご教示宜しくお願いします。
Cbind <- function(...) {
x <- list(...)
len <- max(sapply(x, length))
sapply(x, function(y) c(y, rep(NA, len - length(y))))
}
実行例
> Cbind(1:3, 20:24, 40:41)
[,1] [,2] [,3]
[1,] 1 20 40
[2,] 2 21 41
[3,] 3 22 NA
[4,] NA 23 NA
[5,] NA 24 NA
> Cbind(c(2, 3, 1, 5), c(4, 3, 6), c(9, 4, 2, 5, 3, 1), c(7, 6))
[,1] [,2] [,3] [,4]
[1,] 2 4 9 7
[2,] 3 3 4 6
[3,] 1 6 2 NA
[4,] 5 NA 5 NA
[5,] NA NA 3 NA
[6,] NA NA 1 NA> a1 <- 1:2; a2 <- 1:3; a3 <- 1:4
> maxl <- max(length(a1),length(a2),length(a3)); maxl
[1] 4
> length(a1) <- maxl; length(a2) <- maxl; length(a3) <- maxl;
> A <- cbind(a1,a2,a3); A
a1 a2 a3
[1,] 1 1 1
[2,] 2 2 2
[3,] NA 3 3
[4,] NA NA 4
> A[is.na(A)] <- 0; A
a1 a2 a3
[1,] 1 1 1
[2,] 2 2 2
[3,] 0 3 3
[4,] 0 0 4Cbind2 <- function(...) {
x <- list(...)
len <- max(sapply(x, length))
sapply(x, function(y) {length(y) <- len; y})
}クマ (2014-04-23 (水) 00:13:16)
MacでR version 3.1.0を使っておりまして、CSVで「○○」という時系列データを読み込みました。> dat <- read.csv("FCsuii.csv", na.strings="*")複数の変数がありますが、読み込みは成功しておりました。 そのあと変数のうちの一つ「kaiinsuu」を分析するためにパッケージ tseries_0.10-32をインストールして
> adf.test(kaiinsuu)を入力したところ
以下にエラー NCOL(x) : オブジェクト 'kaiinsuu' がありませんと出ました。
CSVデータは読み込めているが変数を関数で処理できない状態になっています・・・いったい、どんな作業が足りていないんでしょうか?ご教示いただければ幸いです。
よろしくおねがいいたします。
> with(dat, adf.test(kaiinsuu))
H (2014-04-14 (月) 18:58:53)
お世話になります。Windows 7上でRを使用しております。
R 2.15からR 3.1へのアップグレードをすると、一部のプログラム(*.rファイル)の実行が極端に遅くなってしまうことに気がつきました。R 2.15では1秒未満でしたが、R 3.1では10秒以上を要します。
遅くなってしまうのはこのようなこのようなプログラム(*.rファイル)です。少し大きめの数値ベクトルを定義し、そのベクトルの要素数をprint()するだけのプログラムです。
ベクトル部分を別ファイルに書き出しておいて、scan()すれば1秒程度で実行できます。ただ、できれば1ファイルのままの方が管理しやすいので、1ファイルのままで高速化する方法にお心当たりがございましたら、ご教示いただけないでしょうか。
どうぞよろしくお願いいたします。
なお、この現象はWindows 7 32bitと64bitの2つのPCで再現することを確認しました。。RGuiのR Consoleへのドラッグ&ドロップ(sourceコマンド)で、実行・テストしています。
nnet (2014-04-12 (土) 18:21:11)
Ubuntu 12.04 LTSで、
R version 3.1.0 (2014-04-10)を使用しています。
パッケージRSNNSをインストールしようとして、以下のように入力しました。install.packages("RSNNS")すると、次のようなメッセージが出て、インストールできません。
kr_io.cpp: メンバ関数 ‘krui_err SnnsCLib::krio_writeSiteDefinitions()’ 内: kr_io.cpp:930:25: エラー: 書式が文字列リテラルでは無く、かつ書式引数ではありません [-Werror=format-security] (中略) cc1plus: some warnings being treated as errors make: *** [kr_io.o] エラー 1 ERROR: compilation failed for package ‘RSNNS’コンパイルに失敗しているようですが、対処方法がわからず苦慮しています。
どなたか、対策をお教えいただければ幸いです。
> install.packages("RSNNS", type="source")
URL 'http://cran.md.tsukuba.ac.jp/src/contrib/RSNNS_0.4-4.tar.gz' を試しています
[snip]
kr_io.cpp: In member function ‘krui_err SnnsCLib::krio_writeSiteDefinitions()’:
kr_io.cpp:930: warning: format not a string literal and no format arguments
[snip]
installing to /Library/Frameworks/R.framework/Versions/3.0/Resources/library/RSNNS/libs
** R
** data
** demo
** inst
** preparing package for lazy loading
** help
*** installing help indices
** building package indices
** installing vignettes
** testing if installed package can be loaded
sh: rm: command not found
* DONE (RSNNS)
sh: rm: command not found
[snip]> library() ... Rcpp Seamless R and C++ Integration ... methods Formal Methods and Classes
$ lsb_release -dc Description: Ubuntu 13.10 Codename: saucy $ R --version | head -1 R version 3.1.0 beta (2014-03-28 r65330) -- "Spring Dance"nnetさんとはUbuntuのバージョンが違いますがRSNNSをインストールできています。
> packageVersion("Rcpp")
[1] ‘0.11.0’
> install.packages("RSNNS", .libPaths()[2], "http://cran.md.tsukuba.ac.jp")
URL 'http://cran.md.tsukuba.ac.jp/src/contrib/RSNNS_0.4-4.tar.gz' を試しています
[snip]
kr_io.cpp: In member function ‘krui_err SnnsCLib::krio_writeSiteDefinitions()’:
kr_io.cpp:930:25: warning: format not a string literal and no format arguments [-Wformat-security]
sprintf( buf, fmt_hdr1 );
[snip]
installing to /usr/lib/R/site-library/RSNNS/libs
** R
** data
** demo
** inst
** preparing package for lazy loading
** help
*** installing help indices
** building package indices
** testing if installed package can be loaded
* DONE (RSNNS)
[snip]
「DONE (RSNNS)」と表示されたらインストールできています。上記の通りkr_io.cppのところで「警告」はでますが、「エラー」はありません。なお、兵庫教育大のCRANミラーをデフォルトにしていますが現在止まっているので、筑波大学に変更するためにreposオプションをつけました。libオプションを.libPaths()[2]にしているのは、こちらの都合です。As it isでペーストするために、それらのオプションを削除せずにそのまま書き込んでいます。$ tar xvzf RSNNS_0.4-4.tar.gz $ cd RSNNS/src $ gedit MakevarsPKG_CPPFLAGSの下にでも「PKG_CXXFLAGS=-Wno-format-security」を追記して上書き保存。geditは終了。
$ cd - #元のディレクトリ戻る $ sudo R CMD INSTALL -l "/usr/local/lib/R/site-library" RSNNSこれでコンパイルが通ると思います。nnetさんは、上記で成功したら、是非、RSNNSの作者さんにkr_io.cppの不具合についてバグレポートしてあげてください。別のアプローチも紹介しておきます。Rのパッケージをコンパイルするときに使われるCXXFLAGSなどの環境変数は、/etc/R/Makeconfにあります。これを事前に編集します。
$ grep format-security /etc/R/Makeconf | sed '/^#/d' CFLAGS = -g -O2 -fstack-protector --param=ssp-buffer-size=4 -Wformat -Werror=format-security -D_FORTIFY_SOURCE=2 -g $(LTO) CXXFLAGS = -g -O2 -fstack-protector --param=ssp-buffer-size=4 -Wformat -Werror=format-security -D_FORTIFY_SOURCE=2 -g $(LTO) CXX1XFLAGS = -g -O2 -fstack-protector --param=ssp-buffer-size=4 -Wformat -Werror=format-security -D_FORTIFY_SOURCE=2 -gこのCXXFLAGSの「-Werror=format-security」を「-Wno-error=format-security」に書き換えると、RSNNSのコンパイルに成功しました。
sudo R CMD INSTALL -l "/usr/local/lib/R/site-library"
RSNNS * installing
*source* package ‘RSNNS’ ...
file src/Makevars’ has the wrong MD5 checksum
と、いきなり叱られ(ファイルサイズが当初と異なっているので、おっしゃるとおりなのですが)、それが理由かどうかわかりませんが、以前と同様にインストールは失敗しました。そこで、お示しいただいた第2の方法(Makeconf)をトライしました。すると、警告は相変わらず山のように出たものの、それ以降、コンパイルは更に進行し、「おおお・・・」と息を飲んで見つめていると、無事インストールに成功しました! 感謝です!解決策をお教えいただいた方、それ以外にもいろいろと一緒に悩んでいただいた方、皆様に感謝いたします。 折を見て(4月17日のUbuntuの次期バージョン14.04 LTSでの具合を確かめてからでしょうか)、RSNNSの作者さんへバグレポートを送ってみたいと思います。本当に、ありがとうございました。 -- nnet 2014-04-14 (月) 23:39:48お花見さん (2014-04-12 (土) 11:50:58)
ウエーブレットのパワースペクトルの色の説明で,色が1から0の値に対応していることが凡例で示されています.
この1から0の値は,何でしょうか?信頼係数とか有意水準と関係するのでしょうか?
taipapa (2014-04-07 (月) 14:01:38)
こちらでお尋ねすることかどうか分からないのですが,Q&A (初級者コース)の2013年1月〜7月の過去ログが削除されているようです.私の勘違いでしょうか,あるいはどこかにアーカイブされているのでしょうか?
Time (2014-04-06 (日) 12:03:05)
ggplot2 で x 座標、y 座標、時刻の情報を持ったデータを、散布図で描画しようとしています。
このとき、時刻情報をプロットの色、しかも色相環に準じた色でプロットしたいです。
例えば、0時が緑、6時が黄色、12時が赤、18時が青、24時が緑というパターンです。
ところが下記のように、色が補色に向かって変わっていくプロットは簡単にかけるのですが、なかなか色が元に戻るようにできません。
この目的を達成できるような関数があればお教え下さい。
よろしくお願い致します。require(ggplot2) data <- data.frame(Time <- 0:24, x <- rnorm(25, 0, 3), y <- rnorm(25, 0, 3)) (g <- ggplot(data, aes(x = x, y = y)) + geom_point(aes(colour = Time))) + scale_colour_gradient2(low = "green", mid = "red", high = "green")
data <- data.frame(Time = 0:24, x = rnorm(2。5, 0, 3), y = rnorm(25, 0, 3))
plot(x, y, col=rainbow(24)[as.integer(data$Time)], pch=19, xlim=c(-10, 5))
legend("bottomright", legend=0:23, col=rainbow(24)[1:24], pch=19, bty="n")
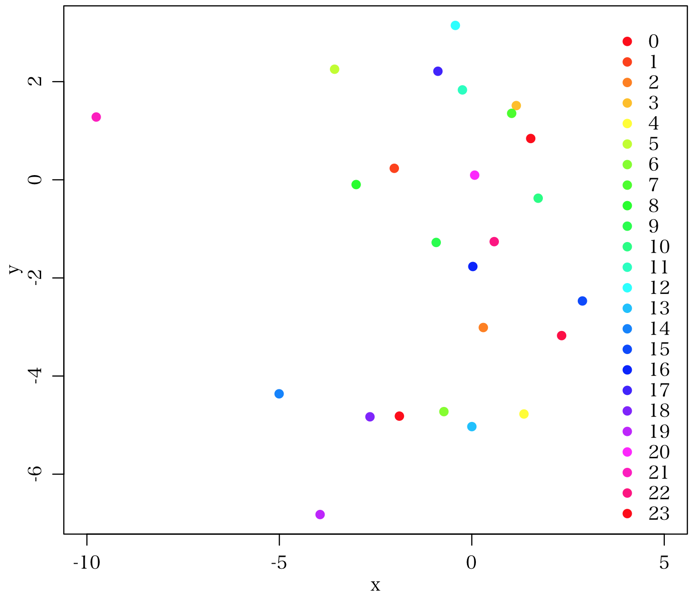
> col2rgb("green")
[,1]
red 0
green 255
blue 0
> rgb2hsv(r = 0, g = 255, b = 0)
[,1]
h 0.3333333
s 1.0000000
v 1.0000000
これでgreenのhの値が分かりました。これでぐるっと、hを回して色を作成します。
> k <- seq(from = 0.3333330, to = 1 + 0.3333330, length.out = 25)[1:24] > cols <- hsv(h = ifelse(k < 1, k, k - 1))この色を使って描画する。
> plot(y~x, data = data, col=cols[as.integer(Time)], pch=19, xlim=c(-10, 5))
> legend("left", legend=0:23, col=cols, pch=19, bty="n", ncol = 3)
これだと、「6時が黄色、12時が赤、18時が青」の要件は満たしていない。
> rgb2hsv(col2rgb(c("yellow", "red", "blue")))[1, ]
[1] 0.1666667 0.0000000 0.6666667
これらの値を中間に挟んでseq()で上のkを分割作成して結合する。> x <- LETTERS[1:3] > x [1] "A" "B" "C" > mean(x = 1:3) [1] 2 > x [1] "A" "B" "C" > mean(x <- 1:3) [1] 2 > x [1] 1 2 3
もみも (2014-04-04 (金) 17:13:02)
RのSLmiscをインストールしたいんですが。tar.gzだからなのかDLしRのフォルダにおいてもうまくインストールしてくれません。
k-means法のkを導き出すのにギャップ統計量を使った機械的な方法を採りたくてSLmiscが必要なのですが。
・SLmiscにかわるパッケージ
・SLmiscをどうにかインストールする方法
どなたか教えていただければ幸いです。
> source('~/SLmisc/R/kmeansGap.R', chdir = TRUE)
> source('~/SLmisc/R/gapStat.R', chdir = TRUE)
> x <- rbind(matrix(rnorm(150, sd = 0.1), ncol= 3), # man の examples の例
+ matrix(rnorm(150, mean = 1, sd = 0.1), ncol = 3),
+ matrix(rnorm(150, mean = 2, sd = 0.1), ncol = 3),
+ matrix(rnorm(150, mean = 3, sd = 0.1), ncol = 3))
>
> res <- kmeansGap(x = x, nstart = 10)
> plot(res)
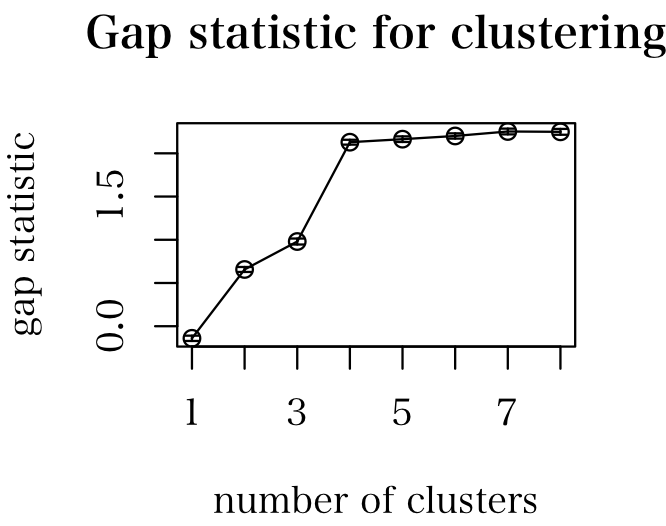
ヒスト (2014-03-31 (月) 10:30:10)
お世話になります。以下のように実線・ダッシュ・ドットの凡例を作りたいのですが、pchに指定できるのは一文字だけのようで、ダッシュを表現できません。plot(rnorm(10), type="l", lty="solid", ylim=c(-1, 1)) lines(rnorm(10), lty="dashed") lines(rnorm(10), lty="dotted") legend("topleft", pch=c("−", "--", "・"), c("A", "B", "C"), bg="white")何か良い方法はありませんでしょうか。WinXP、R3.02です。
宜しくお願いします。
sun (2014-03-28 (金) 10:10:06)
決定係数R2の値から直線性を判断しようと思っています.
R2の値がいくら以上は直線と決めたいんですが,その値の決め方に困っています.(例えばR2=0.97以上を直線とする.)
アドバイスをいただけたらうれしいです.
set.seed(123)
n <- 1000
layout(matrix(1:2, 2))
old <- par(mgp=c(1.6, 0.8, 0), mar=c(3, 3, 0.5, 0.5))
x <- rnorm(n, 50, 10)
y <- jitter(20 * x + 30, amount=100)
plot(x, y)
a <- lm(y~x)
abline(a, col=2)
text(80, 500, sprintf("R2 = %.5f", summary(a)$r.squared), pos=2)
y2 <- x^2
plot(x, y2)
a <- lm(y2~x)
abline(a, col=2)
text(80, 1000, sprintf("R2 = %.5f", summary(a)$r.squared), pos=2)
par(old)
layout(1)
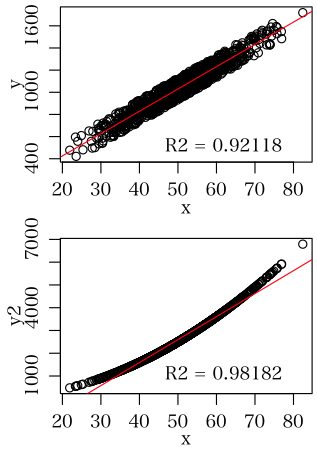
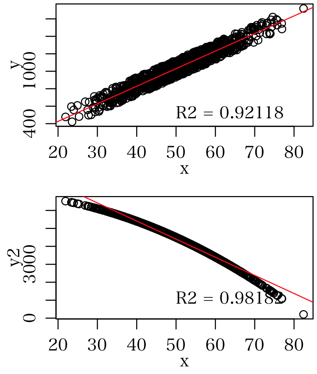
paul (2014-03-26 (水) 14:28:20)
ggplot2で凡例を出す所でつまずいております。
データフレームは下記です。head(testdf) time rl wl 1 2014-10-27 04:43:51 0.55 0.14 2 2014-10-27 05:03:53 0.98 0.07 3 2014-10-27 05:35:28 2.25 0.11 4 2014-10-27 06:17:47 0.48 0.22 5 2014-10-27 06:48:58 0.09 0.24 6 2014-10-27 07:21:01 0.25 0.21下記のように実行していますが、グラフは期待通りきちんと表示されますが凡例はでてきませんでした。さまざま文献で出てくるものと出てきてないものなどコードを見ましたがどのようにしたらでてくるのかがつかめておりません。
ggplot2のバージョンは0.9.3.1です。R version 3.0.2 (2013-09-25)です。ggplot(testdf, aes(time,rl)) + geom_line(color = 4, size = 1.5 , alpha=0.5) + geom_line(aes(y=wl),colour = 3, size = 1.5, alpha=0.5) + theme(axis.text.x = element_text(angle=90)) + xlab("Time") + ylab("ms") + ggtitle("Title Sample") + scale_x_datetime(breaks=date_breaks("30 min"),labels=date_format("%H:%M:%S"))何か指定が足りていないところなどありますでしょうか。
testdf <- data.frame(time = as.POSIXct(c("2014-10-27 04:43:51",
"2014-10-27 05:03:53",
"2014-10-27 05:35:28",
"2014-10-27 06:17:47",
"2014-10-27 06:48:58",
"2014-10-27 07:21:01")),
rl = runif(6), wl = runif(6))
library(ggplot2)
library(scales)
ggplot(testdf, aes(time)) +
geom_line(aes(y = rl, color = "4"), size = 1.5, alpha=0.5) +
geom_line(aes(y = wl, color = "3"), size = 1.5, alpha=0.5) +
theme(axis.text.x = element_text(angle=90)) +
xlab("Time") + ylab("ms") + ggtitle("Title Sample") +
scale_x_datetime(breaks=date_breaks("30 min"),labels=date_format("%H:%M:%S"))
melt()を使うなら次のように。
library(reshape)
t2 <- melt(testdf, id="time")
ggplot(t2, aes(time, value, colour = variable)) +
geom_line(size = 1.5, alpha=0.5) +
scale_colour_manual(values=c("green", "blue")) +
theme(axis.text.x = element_text(angle=90)) +
xlab("Time") + ylab("ms") + ggtitle("Title Sample") +
scale_x_datetime(breaks=date_breaks("30 min"),labels=date_format("%H:%M:%S"))
茎わかめ (2014-03-24 (月) 11:43:41)
同じフォルダ内にある同じ形式のファイルをlist.filesでリストアップして、そのファイルを1つ1つ開いて同じ処理をforで繰り返すコードを作りました。
しかし、そのファイルの中には開けないものがいくつもあり、(おそらくファイルが破損しているせいです。)そのたびに「cannnot open the connection」のエラーにひっかかって処理が途中で中断してしまいます。
もしそのエラーに引っかかってしまった場合は、そのファイルの処理はしないで、次のファイルの処理に進むという条件付けをしたいのですが、どのようにしたらよろしいでしょうか?> files <- list.files("フォルダのパス") > files2 <- unlist(files) > files3 <- paste("フォルダのパス", files2, sep = "/") > for (j in 1:length(files)) { > to.read <- file(files3[[j]], "rb") > ### 以下処理 ### > }の「to.read = file(files3j, "rb")」で中断してしまいます。
ご回答お願いします。
to.read <- try(file(files3[[j]], "rb"))
if (class(to.read) == "try-error") {
next
}
?tryCatchとか読んでいただければと。スカイツリー (2014-03-21 (金) 15:39:14)
csvファイルを読み込んで移動平均を計算します.このファイルはデータのないところは空欄になっています.
この場合,移動平均の命令をどのようにやればいいでしょうか.
このファイルのままやるとエラーが出てしまいます.
よろしくお願いします.
hokan <- function(x) {
a <- rle(is.na(x))
b <- cumsum(a$length)
for (i in seq_along(b)) {
if (i > 1 && a$values[i]) {
begin <- b[i-1]
end <- b[i]+1
width <- end-begin
delta <- (x[end]-x[begin])/width
x[(begin+1):(end-1)] <- x[begin]+delta*(1:(width-1))
}
}
return(x)
}
> x <- c(NA, 3, 2, NA, 5, 7, 6, 9, NA, NA, 20, NA, NA, NA, 12, NA)
> hokan(x)
[1] NA 3.00000 2.00000 3.50000 5.00000 7.00000 6.00000 9.00000 12.66667 16.33333
[11] 20.00000 18.00000 16.00000 14.00000 12.00000 NA$ cat tmp.csv "A","B" 1,2 ,3 4,5これをRに読み込む。
> read.csv("tmp.csv")
A B
1 1 2
2 NA 3
3 4 5
もし解決したいなら、質問したい現象が100%再現できる最小のサンプルと実際の手順を示すべきです(最小のサンプルを準備している段階で9割がた自己解決するでしょう)。さもなければ、空欄になるNAになると水掛け論になり、不毛です。初心者 (2014-03-12 (水) 09:24:31)
初心者です。お願い致します。
"0","1","2","3","A","B"の5種類の要素からなるベクトルがあります。(以下のような例)c <- c("A","A","A","A","A","A","A","A","0","1","1","2","3","B",・・・)そこで初めて"A"以外の要素が出てくる要素の番号を求めたいです。
イメージとしては、「match("0"または"1"または"2"または"3"または"B",c)」みたいな感じなのですが、どのようにコーディングしたらよろしいでしょうか?
どなかご回答お願いします。
takeshik (2014-03-03 (月) 18:08:33)
Rstudio でプレゼンテーションを作成作成としているのですが、途中までは問題なくPreViewできて、途中追加でbarplotをしたところで、ずっと進捗マークがグルグルしている状態となってしまってほぼハングってるようになります。Previewできずそこから進められません。他のplotやグラフでないものは出力できるのですが、なんとも切り分けに行き詰まっています・・・。何か原因が考えられますでしょうか?
グラフとしては下記のようなものを3つ繰り返しています。(2つまではokで3つめを追加するとハングしたようになってしまう)```{r,fig.width=20,fig.height=10,echo=FALSE} barplot(prop.table(table(an$Q1)),ylim=c(0,1.0), main="あああ",cex.main=3,cex.names=2.2,cex.axis=2.5) ```
kepper (2014-02-28 (金) 10:25:17)
お世話になります。Win XP, R 3.02を使用しています。
以下のようにpatから要素を取り出してdatの何番目にマッチするか調べようとしています。しかし、i=3でdatの"aaa"のみにマッチしてほしいのですが、3つすべてにマッチしてしまいます。pat <- c("baaa","aaab","aaa") dat <- c("aaab","aaa","baaa") for (i in 1:3) print(grep(pat[i], dat))以下のようにしてもエラーになってしまいました。どのように対応すればよいでしょうか。
for (i in 1:3) print(grep(^pat[i]$, dat))宜しくお願いします。
for (i in 1:3) print(which((pat[i]==dat)==TRUE))で良いような気がしてきました。 -- kepper 2014-02-28 (金) 11:29:12
> sapply(pat, function(x){which(dat == x)})
baaa aaab aaa
3 1 2SK (2014-02-25 (火) 19:16:44)
水準が3つ(A,B,C,D)ありまして反復数がそれぞれ4,4,4,3であり、データはそれぞれA=392,440,376,317、B=317,368,254,309、C=268,310,290,280、D=236,223,339です。反復数が異なる場合なので、最小二乗平均値を求めないといけないと思うのですが、そのやり方がまず分かりません。初級Q&A(10)のところで,LSMEANについて記述されていましたが、中沢先生のホームページにリンクされず、やり方を理解することができませんでした。また、最小二乗平均値が分かったら、一元配置分散分析をしたいのですがDの反復数3はそのまま欠損値でやっていいのでしょうか。Rを使い始めてなれませんので、Rコマンダーでできるならありがたいです。どうぞよろしくお願いします。
カンゲツ (2014-02-25 (火) 17:09:35)
300万台の6ケタの数値6万件からなるデータをCSVからとりこみ変数oriに代入しました。
これをベクトル化するために変数dataに代入したところ値が変わってしまいます。ori<-read.table("CSVのファイル名") data<-c(ori[,1])dataを10レコード確認したところ
ori[1:10] [1] 11189 11190 11191 11192 (以下略)と11189からひとつずつ増える値になっています。(元データ) とは全く違う値です。
Rのバージョンは3.0.2です。
カンゲツ (2014-02-25 (火) 10:48:59)
お世話になっております。
ここに生年月日を6ケタの数値で管理しているデータがあります。
年の情報が元号でして100万の位が
1:明治
2:大正
3:昭和
4:平成
で3280306(この場合は昭和28年3月6日生まれ)などと表記されます。
このデータから各サンプルの年齢を計算し年齢別にまとめようとおもっています。
まず平成の人のみを抽出(ベクトルhs)したあとfor(i in length(hs)) { hs[i]-4000000 a<-hs[i]-4000000 b<-floor(a/10000) }で年まではなんとか切り分けることができましたが月と日がうまくいきません。
どのようにすればよいかご教授願います。
getAge <- function(num) {
nengo <- num %/% 1000000
num <- num %% 1000000
nen <- num %/% 10000 + c(1867, 1911, 1925, 1988)[nengo]
num <- num %% 10000
tuki <- num %/% 100
hi <- num %% 100
nen <- nen
age <- yy-nen
if (tuki > mm | (tuki == mm && hi > dd)) {
age <- age-1
}
return(age)
}
実行例
> test.dat <- c(1251023, 2090305, 3280306, 4230718) > yy <- 2014 # 年齢計算の基準点 > mm <- 2 > dd <- 25 > sapply(test.dat, getAge) [1] 121 93 60 2
wavelet (2014-02-23 (日) 22:00:04)
おかげ様で,0)生データから移動平均を中点にプロットするところまでできました.
1)次に,生データ−移動平均 を点でプロットします.
2)さらに,その値の移動平均を求めてプロットします.
以下のようにプログラムしましたが作図されません.
0,1,2の図は,重ねないで別々に作ります.
問題点を教えていただけるとうれしいです.
よろしくお願いします.AO1900.csvのデータも送ります.library("TTR") rw<-scan("AO1900.csv") ts<-rw sx=SMA(ts, 60) plot(ts,col=4,pch=1,cex=0.5) lines(sx[-(1:30)], type="b",cex=0.3) tss=ts-sx[-(1:30)] lines(tss, col=2,type="b",cex=0.3) sxx=SMA(tss,60) lines(sxx[-(1:30)], col=3, type="b",cex=0.3)
wavelet (2014-02-21 (金) 22:50:21)
移動平均をTTRを用いて求めています.点をプロットさせると移動平均は,最終月にプロットされて位相がずれてしまいます.中日にプロットする方法を教えてください.
以下のプログラムでは,月毎のデータを使って,100ヶ月の移動平均を求めています.
用いたデータも添付します.よろしくお願いします.library("TTR") rw <- scan("NAO1910.csv") ts <- rw sx <- SMA(ts, 100) plot(ts) par(new = TRUE) plot(sx, type = "b")
library("TTR")
par(mar=c(4, 4, 1, 4), mgp=c(1.8, 0.8, 0))
rw <- scan("nao54.csv")
ts <- rw
sx <- SMA(ts, 11)
plot(ts)
par(new = TRUE)
plot(c(sx[-(1:5)], rep(NA, 5)), type = "b", col="red", pch=19, xaxt="n", yaxt="n", ylab="")
mtext("moving average", 4, line=2)
axis(4)
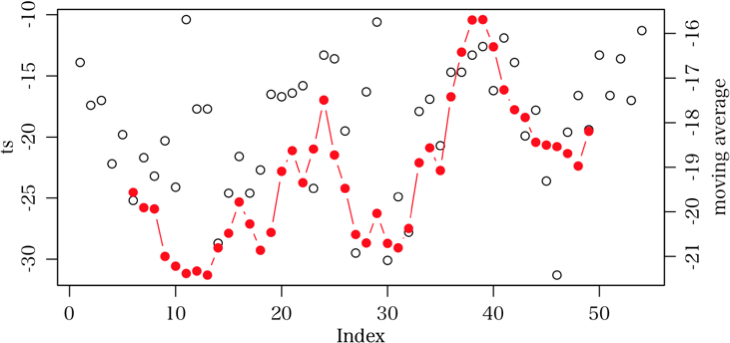
skgmsnb (2014-02-20 (木) 17:06:20)
複数のファイルを読み込んで、それぞれの標準偏差を求めようとしています。
ところが、fnames にファイル名を読み込んだ上で、apply(fnames, 1, calc.sd)
とすると "Error in apply(fnames, 1, calc.sd) : dim(X) は正の長さを持たねばなりません" というエラーが出てしまいます。
ここで、calc.sd というのは引数として与えられた文字列と同じファイル名を持ったデータをカレントディレクトリから読み込んで標準偏差を求める自作関数です。dim(fnames)
とすると "NULL" が返ってくることから、これは apply 関数が文字列を引数として関数に渡すことに対応していないからだと考えています。
このような場合、apply ではない他の関数を使うべきなのでしょうか?
ご教授よろしくお願い致します。
calc.sd <- function(fn) { sd(scan(fn, ""))}
sapply(c("1.txt", "2.txt"), calc.sd)
無名関数を書く方がスマートだけどね。scan はデータがどういう風に入っているかに依存するけどね。
sapply(c("1.txt", "2.txt"), function(fn) sd(scan(fn, "")))山田 (2014-02-19 (水) 18:45:06)
win8.1でRstudioはインストールできますか?
実行ファイルが見当たらないのですが。。。。
松田 (2014-02-19 (水) 18:40:14)
Mac(OSX10.9)でRをインストール後、画像を読み込みしました。
readImage()で読み込みdisplay()で表しましたが、Safariが立ち上がって画像が表示されます。windowsではないのですが、何か設定が間違っていますか?
JA (2014-02-17 (月) 20:13:53)
お世話になります。以下のように因子ごとに値をプロットしているのですが、
1.y軸のclassを数値ではなく元のA,B,Cとして認識させる
2.x軸にvalue、y軸にclassを表示する
をするためにはどのようにすればよいでしょうか。
宜しくお願いします。dat <- as.data.frame(matrix(0,100,2)) colnames(dat) <- c("value","class") dat[,1] <- runif(100) dat[,2] <- as.factor(rep(c("B","A","C","C","A"),20)) plot(dat)
dat <- as.data.frame(matrix(0, 100, 2))
colnames(dat) <- c("value", "class")
dat[, 1] <- runif(100)
dat[, 2] <- as.factor(rep(c("B", "A", "C", "C", "A"), 20))
plot(dat, yaxt="n")
axis(2, at=seq_along(levels(dat[, 2])), labels=levels(dat[, 2]))
なお,データフレームは以下のように作るだけで十分ですよ。
dat <- data.frame(value=runif(100),
class=rep(c("B", "A", "C", "C", "A"), 20))(2014-02-17 (月) 11:35:01)
個々の値が小さく、要素数が多いデータを円グラフにしてclockwise=Tを適用してラベルを表示すると、終端の辺りでラベルが重なってしまい読めません。ラベルの重複を防ぐ方法はありますでしょうか
pie(1:25, labels = c(rep(NA, 3), 4:25), clockwise = TRUE)
text((a <- locator(3)), as.character(1:3), pos = 3, offset = .1)
for (i in 1:3){
z <- atan(a$y[i]/a$x[i])
segments(a$x[i], a$y[i], 0.8 * cos(z), 0.8 * sin(z))
}
pie2 <- function(x, labels = names(x), edges = 200, radius = 0.8, clockwise = FALSE,
init.angle = if (clockwise)
90
else 0, density = NULL, angle = 45, col = NULL, border = NULL, lty = NULL,
main = NULL, special = NULL, ...) {
if (!is.numeric(x) || any(is.na(x) | x < 0))
stop("'x' values must be positive.")
if (is.null(labels))
labels <- as.character(seq_along(x))
else labels <- as.graphicsAnnot(labels)
x <- c(0, cumsum(x)/sum(x))
dx <- diff(x)
nx <- length(dx)
plot.new()
pin <- par("pin")
xlim <- ylim <- c(-1, 1)
if (pin[1L] > pin[2L])
xlim <- (pin[1L]/pin[2L]) * xlim
else ylim <- (pin[2L]/pin[1L]) * ylim
dev.hold()
on.exit(dev.flush())
plot.window(xlim, ylim, "", asp = 1)
if (is.null(col))
col <- if (is.null(density))
c("white", "lightblue", "mistyrose", "lightcyan", "lavender",
"cornsilk")
else par("fg")
if (!is.null(col))
col <- rep_len(col, nx)
if (!is.null(border))
border <- rep_len(border, nx)
if (!is.null(lty))
lty <- rep_len(lty, nx)
angle <- rep(angle, nx)
if (!is.null(density))
density <- rep_len(density, nx)
twopi <- if (clockwise)
-2 * pi
else 2 * pi
t2xy <- function(t) {
t2p <- twopi * t + init.angle * pi/180
list(x = radius * cos(t2p), y = radius * sin(t2p))
}
P.xy <- matrix(0, nx, 2)
for (i in 1L:nx) {
n <- max(2, floor(edges * dx[i]))
P <- t2xy(seq.int(x[i], x[i + 1], length.out = n))
polygon(c(P$x, 0), c(P$y, 0), density = density[i], angle = angle[i],
border = border[i], col = col[i], lty = lty[i])
P <- t2xy(mean(x[i + 0:1]))
P.xy[i, 1] <- P$x
P.xy[i, 2] <- P$y
lab <- as.character(labels[i])
if (!is.na(lab) && nzchar(lab) && !(i %in% 1L:special)) {
lines(c(1, 1.05) * P$x, c(1, 1.05) * P$y)
text(1.1 * P$x, 1.1 * P$y, labels[i], xpd = TRUE, adj = ifelse(P$x <
0, 1, 0), ...)
}
}
sh <- strheight("H")
for (i in 1L:special) {
temp <- P.xy[special, 2] + (special - i + 1) * sh * 1.2
lines(rep(P.xy[i, 1], 2), c(P.xy[i, 2], temp), xpd = TRUE)
text(P.xy[i, 1] - strwidth("H"), temp + 0.5 * sh, labels[i], xpd = TRUE,
adj = ifelse(P$x < 0, 1, 0), pos = 4, ...)
}
title(main = main, ...)
invisible(NULL)
}
ans <- pie2(1:25, radius = 0.6, labels = paste("L", 1:25, sep = "-"),
clockwise = TRUE, special = 8)
実行結果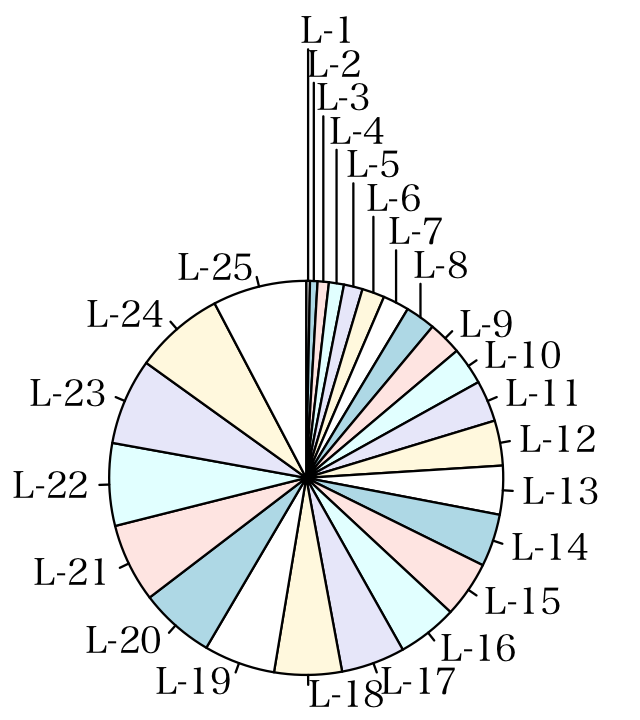
if (!is.na(lab) && nzchar(lab) && !(i %in% special:25L)) {
lines(c(1, 1.05) * P$x, c(1, 1.05) * P$y)
text(1.1 * P$x, 1.1 * P$y, labels[i], xpd = TRUE, adj = ifelse(P$x <
0, 1, 0), ...)
}
}
sh <- strheight("H")
for (i in special:25L) {
temp <- P.xy[special, 2] - (special - i-0.5 ) * sh * 1.2
lines(rep(P.xy[i, 1], 2), c(P.xy[i, 2], temp), xpd = TRUE)
text(P.xy[i, 1] + 0.25*strwidth("H"), temp + 0.5 * sh, labels[i], xpd = TRUE,
adj = ifelse(P$x < 0, 1, 0), pos = 2, ...)
}
変更後,
ans <- pie2(25:1, radius = 0.6, labels = paste("L", 25:1, sep = "-"),
clockwise = TRUE, special = 18)
で,動作確認。auto (2014-02-15 (土) 19:32:57)
お世話になります。
パッケージforecastのauto.arimaを使用して、次のようにSARIMAモデルの次数の自動選択を実行してみました。> library(forecast) > sarima1 <- auto.arima(log(AirPassengers), trace=T) ... ARIMA(0,1,1)(0,1,1)[12] : -401.9607 ... ARIMA(0,1,1)(2,1,2)[12] : -408.3862 Best model: ARIMA(0,1,1)(2,1,2)[12]その要約を出力すると次のようになります。
> summary(sarima1) Series: log(AirPassengers) ARIMA(0,1,1)(2,1,2)[12] Coefficients: ma1 sar1 sar2 sma1 sma2 -0.4291 -1.0022 -0.0196 0.4343 -0.4915 s.e. 0.0897 0.2632 0.1783 0.3023 0.1761 sigma^2 estimated as 0.001312: log likelihood=245.46 AIC=-478.92 AICc=-478.24 BIC=-461.67AICc=の値がモデル選択の過程における値と一致していません。
次に、Box and JenkinsのARIMA(0,1,1)(0,1,1)[12]モデルを当てはめてみます。> sarima2 <- Arima(log(AirPassengers), c(0, 1, 1), seasonal = list(order = c(0, 1, 1), period = 12)) > summary(sarima2) Series: log(AirPassengers) ARIMA(0,1,1)(0,1,1)[12] Coefficients: ma1 sma1 -0.4018 -0.5569 s.e. 0.0896 0.0731 sigma^2 estimated as 0.001348: log likelihood=244.7 AIC=-483.4 AICc=-483.21 BIC=-474.77Box and Jenkinsモデルの方がAICcが小さく、より優れたモデルであることが示唆されています。auto.arimaのトレースを見る限り、Box and Jenkinsモデルの方が劣っているのにです。この不一致の理由が理解できなくて困っています。どなたか、その原因を御教示いただければ幸いです。
ドベシイ (2014-02-12 (水) 22:07:54)
以下のようにして,時系列をウエーブレット係数で分解した時,ブロックのような表示になります.これを曲線で表示できないでしょうか.よろしくお願いします.
時系列のファイルも添付します.library(wavelets) rw <- scan("198311-200206.csv") ts <- rw # discrete wavelet wt <- dwt(ts, filter="d4", n.levels=5, boundary="periodic", fast=FALSE) plot(wt)
カンゲツ (2014-02-12 (水) 10:58:33)
はじめまして。円グラフを作成していてどうしても解決できなかった点がありまして質問をさせてください。
pie(1:10) を実行した際、時計で言う3時の位置に1番目の要素が出ます。
これを12時の位置に動かす方法はありますでしょうか。
ヒョウ エンフン (2014-02-08 (土) 10:29:36)
sem packageを使うとき、私は下のように入力しました。> library(sem) > coopd<- readMoments(names=c( + "y1","y2","y3","y4","y5","y6","y7","y8","y9","y10","y11","y12")) 1: 1.0 2: .160 1.0 4: .302 .341 1.0 7: .461 .400 .372 1.0 11: .299 .404 .552 .302 1.0 16: .152 .320 .476 .225 .708 1.0 22: .134 .403 .467 .256 .623 .324 1.0 29: .182 .374 .572 .255 .776 .769 .724 1.0 37: .251 .285 .316 .164 .361 .295 .260 .284 1.0 46: .372 .100 .408 .236 .294 .206 .071 .142 .295 1.0 56: .157 .291 .393 .229 .472 .351 .204 .320 .290 .468 1.0 67: .206 -0.014 .369 .224 .342 .202 .152 .189 .418 .351 .385 1.0 79: Read 78 items > model.coop <- specifyModel() 1: mother -> y1, b11, NA 2: mother-> y2, b21, NA 3: mother-> y3, b31, NA 4: mother-> y4, b41, NA 5: interaction-> y5, NA, 1 6: interaction-> y6, b62, NA 7: interaction-> y7, b72, NA 8: interaction-> y8, b82, NA 9: cooperative -> y9, NA, 1 10: cooperative -> y10, b103, NA 11: cooperative -> y11, b113, NA 12: cooperative -> y12, b123, NA 13: mother-> interaction, g21, NA 14: mother-> cooperative, g31, NA 15: y1 <-> y1, e1, NA 16: y2 <-> y2, e2, NA 17: y3 <-> y3, e3, NA 18: y4 <-> y4, e4, NA 19: y5 <-> y5, e5, NA 20: y6 <-> y6, e6, NA 21: y7 <-> y7, e7, NA 22: y8 <-> y8, e8, NA 23: y9 <-> y9, e9, NA 24: y10 <-> y10, e10, NA 25: y11 <-> y11, e11, NA 26: y12 <-> y12, e12, NA 27: mother<-> mother, NA, 1 28: interaction<-> interaction, delta2, NA 29: cooperative <-> cooperative, delta3, NA 30: Read 29 records > sem.coop <- sem(model.coop, coopd, N=50) 以下にエラー seq_len(ngroups) : 引数は非負の整数に変換できなければなりません 追加情報: 警告メッセージ: 1: In max(FLAT$group) : max の引数に有限な値がありません: -Inf を返します 2: In max(partable$group) : max の引数に有限な値がありません: -Inf を返します友達のコンピューターでやってみて、うまく結果ができましたが、自分のコンピューターではerrorが出ました。どこかに問題がありますか?
使うデータは全部ネットでdownloadしたデータです。間違えてはいないと思います。直す方法を教えてもらえませんか?
とまと (2014-02-05 (水) 22:30:40)
まだRを始めて間もない初心者です。
よろしくお願いいたします。> x<-c("a","a","a","a","b","b","b","a","b","b","a","a","b","a") > x > [1] "a" "a" "a" "a" "b" "b" "b" "a" "b" "b" "a" "a"のような"a"と"b"の2種類からなるベクトルがあります。 これを同じ文字が連続して存在する場合、連続する要素を結合して、
> x<-c("aaaa","bbb","a","bb","aa","b","a") > x > [1] "aaaa" "bbb" "a" "bb" "aa" "b" "a"という形に最終的にはしたいです。 一つ目のかたまりは
> R<- x[1:match("b",x)-1] > list<-paste(R, collapse = "") > x<-c(list,x[match("b",x):length(x)]) > x > [1] "aaaa" "b" "b" "b" "a" "b" "b" "a" "a" "b" > [11] "a"としましたが、ここからどのようにして繰り返しのループを組み立てていけばいいのかわかりません。 ベクトルの要素や長さはその時々によって変わるので、("a"と"b"の2種類からなるのは不変です。)変数のまま扱いたいです。 お教示お願いいたします。
> x <- c("a","a","a","a","b","b","b","a","b","b","a","a","b","a")
> ans <- rle(x)
> mapply(function(x, y) paste(rep(x, y), collapse=""), ans$values, ans$lengths)
a b a b a b a
"aaaa" "bbb" "a" "bb" "aa" "b" "a"> "aaaa" "bbb" "a" "bb" "aa" "b"となったベクトルの"a"を含む要素のうち
> Number<-grep("[a]",x2)
> Number
> [1] 1 3 5 7
「その要素の中の文字数が2以下ならば、その要素をベクトルから消去。2より大きいならば、その要素をベクトルの中にそのまま残しておく。」という条件で割り振りたいです。
(今回の例では、結果として"aaaa" "bbb" "bb" "b"というベクトルを得たいです。)
Number[1]に関しては以下のような操作を作ったのですが、ここからどのようにしてNumber内の全ての要素にループをかければいいのかが分かりません。
(説明上の問題で、以下の文では条件の文字数が5になっています。)
> f(nchar(x2[Number[1]])<5){x3<-x2[-Number[1]]} else {x3<-x2}
> x3
> b a b a b a
> "bbb" "a" "bb" "aa" "b" "a"
ご教示お願いします。(前回と同じ様なループの質問になってしまいすみません。)-- とまと 2014-02-06 (木) 11:48:30test.dat <- replicate(10, sample(letters[1:2], 10, replace = TRUE),
simplify = FALSE)
sapply(test.dat, function(x) {
ans <- rle(x)
cond <- ans$values == "b" |
(ans$values == "a" & ans$lengths >= 2) # 付け加える
ans$lengths <- ans$lengths[cond] # 付け加える
ans$values <- ans$values[cond] # 付け加える
mapply(function(x, y) paste(rep(x, y), collapse = ""),
ans$values, ans$lengths)
})ジェイ (2014-02-04 (火) 17:52:28)
お世話になります。R 3.02を使用しています。
for文を実行したところ、以下のようなWarning messagesが大量に出力されました。Warning messages: 1: In FUN(newX[, i], ...) : no non-missing arguments to min; returning Inf 2: In FUN(newX[, i], ...) : no non-missing arguments to min; returning Inf ・・・Warningが生じた箇所を特定するために、for文の最初の変数i=1に固定してfor文を実行したのですがWarningは生じませんでした。どうやらiが特定の値をとるときに問題が生じているようです。そのiの値を知りたいので、例えばWarningが生じた場合にfor文を抜けるということは可能でしょうか。
お手数ですが、ご回答宜しくお願いします。
> x <- rbind(1:5, rep(NA, 5), 11:15) # 例示のためのデータ(行列)
> options(warn=2) # Warning を Error とみなさせる
> for (i in 1:nrow(x)) {
+ print(i) # ループの制御変数 i の値を書く
+ print(x[i,]) # 処理に関連するデータを書く
+ min(x[i,], na.rm=TRUE) # Warning が起こる可能性のある処理
+ }
[1] 1 i の値
[1] 1 2 3 4 5 x[i,] の値
[1] 2 i が 2
[1] NA NA NA NA NA x[2,] は全部 NA なので,min がない!
以下にエラー min(x[i, ], na.rm = TRUE) : i が 2 のとき,Warning が起きた
(警告から変換されました) min の引数に有限な値がありません: Inf を返しますwavelet (2014-02-01 (土) 12:53:16)
以下のようにフーリエ解析をしたいのですが動きません.
何かパッケージをインストールしなくてはいけないのでしょうか.
x <- 100 f <- 1:x hz <- 1:( x/2 ) t<-seq(0.01,1.00, 1/length(f))# 4Hzの正弦波 data_waveA <- sin( 2 * pi * t * 4 )
# 18Hzの余弦波 data_waveB <- cos( 2 * pi * t * 18 )
# 上記2つの合成波 data_waveC <- data_waveA + data_waveB
# 波を描きます。 par( "mfrow" = c( 3, 1 ) ) plot( t, data_waveA, type="l" ) plot( t, data_waveB, type="l" ) plot( t, data_waveC, type="l" )
# 高速フーリエ変換を行ないます。 data_freqA <- fft( data_waveA - mean( data_waveA ) ) data_freqB <- fft( data_waveB - mean( data_waveB ) ) data_freqC <- fft( data_waveC - mean( data_waveC ) )
# 変換結果を表示します。 # 合成波の周波数も表示されています。 par( "mfrow" = c( 3, 1 ) ) plot( hz, abs( data_freqA[ hz ] ), type="l" ) plot( hz, abs( data_freqB[ hz ] ), type="l" ) plot( hz, abs( data_freqC[ hz ] ), type="l" )
x <- 100 f <- 1:x hz <- 1:(x/2) t <- seq(0.01, 1, 1/length(f)) # 4Hzの正弦波 data_waveA <- sin(2 * pi * t * 4) # 18Hzの余弦波 data_waveB <- cos(2 * pi * t * 18) # 上記2つの合成波 data_waveC <- data_waveA + data_waveB # 波を描きます。 par(mfrow = c(3, 1)) plot(t, data_waveA, type = "l") plot(t, data_waveB, type = "l") plot(t, data_waveC, type = "l") # 高速フーリエ変換を行ないます。 data_freqA <- fft(data_waveA - mean(data_waveA)) data_freqB <- fft(data_waveB - mean(data_waveB)) data_freqC <- fft(data_waveC - mean(data_waveC)) # 変換結果を表示します。 # 合成波の周波数も表示されています。 par(mfrow = c(3, 1)) plot(hz, abs(data_freqA[hz]), type = "l") plot(hz, abs(data_freqB[hz]), type = "l") plot(hz, abs(data_freqC[hz]), type = "l")投稿方法もお作法に従っていないし,お行儀の悪い子にはバチが当たりますよね(^_^) -- 河童の屁は,河童にあらず,屁である。 2014-02-01 (土) 21:54:13
ひろし (2014-01-31 (金) 16:12:54)
質問1
起動時に表示されるメッセージがR言語が日本語表示でRStudioのConsoleは英語表示の設定になっています。RStudioこの設定で最良でしょうか?
RStudioを日本語で快適に使用するための設定がもしあればアドバイス願います。
質問2
RStudioのConsoleの表示フォントをMS ゴシック(10pt)に設定していますが、ノートパソコンの画面では文字が薄く感じます。より輪郭が明瞭なぉすすめフォントをご存知ですか?あるいは太字指定ができますか?
実行環境
OS:Windows7 SP1 x86 日本語版
R言語:R 3.0.2
Rコマンダー:2.0-3
RStudio 0.98.501
宜しくお願いします。
稲田 (2014-01-30 (木) 23:58:43)
頻度ポリゴンを描く関数 hist2dを使用する際に、x軸とy軸で階級幅を変更するにはどうすればよいのでしょか。
hist関数では、breaksで指定できることがわかりました。
(下記を参照しました
http://www.econ.hokudai.ac.jp/~kakizawa/R/R_4.html)
2次元の場合の指定方法がわかりません。
よろしくお願いします。
panda (2014-01-30 (木) 14:36:30)
ある日付が記されているのデータが下までずっと続いてあるのですが、このdayという列のデータの右から10番目まで、すなわち月日時間を出力したいのです。labe day 323169 20101020092210 323169 20011020094231 323169 20131020152247 323169 20121020154501 723151 20121020071333 723151 20111020072611このような出力です。
labe day 323169 1020092210 323169 1020094231 323169 1020152247 323169 1020154501 723151 1020071333 723151 1020072611よろしくお願いします。
> a <- 20101020092210 > substr(a, 5, 14) # 文字列にしてから抽出が行われる [1] "1020092210" > as.integer(substr(a, 5, 14)) # 数値に戻しておきたいとき [1] 1020092210のようなことです。 -- 河童の屁は,河童にあらず,屁である。 2014-01-30 (木) 14:44:17
freshman (2014-01-29 (水) 15:28:38)
10000000を超える数値を扱っています。プロット関数で図を描きたいのですが、y軸ラベルを指数表記(scientific notation)にするにはどうしたらよいですか?アドバイスお願いします!
x <- 2:5 * 10^5 y <- c(1, 2, 3, 100) options(scipen=2) plot(x, y)リンク先みたいでなくて x.xxe+yy という形式でよければ,以下のようにでも。
x <- 2:5 * 10^5
y <- c(1, 2, 3, 100)
plot(x, y, axes = FALSE)
axis(1, at = 2:5 * 10^5, labels = sprintf("%.2e", 2:5 * 10^5))
axis(2, at = c(0, 100), labels = sprintf("%.2e", c(0, 100)))> sprintf("%.0e %.0f", 2*10^5, 2*10^5)
[1] "2e+05 200000"
この例だと 5 文字 - 6 文字で -1 なので,scipen=-2 にすればよいようで。panda (2014-01-28 (火) 16:33:24)
どなたかご説明お願いします。
以下のような3×3の行列が複数個ありまして、それぞれの対応する要素の平均値を求めたいです。
行列の数は以下には2つしか示しておりませんが、具体的な数を指定せずにn個など変数で行いたいです。
よろしくお願いします。A B C A B C 1 3 4 6 1 4 5 0 2 5 6 5 2 6 8 9 3 5 7 7 3 6 8 7
> x <- matrix(c(3, 4, 6, 5, 6, 5, 5, 7, 7), ncol=3, byrow=TRUE)
> y <- matrix(c(4, 5, 0, 6, 8, 9, 6, 8, 7), ncol=3, byrow=TRUE)
> z <- matrix(c(1, 4, 7, 7, 8, 6, 6, 4, 2), ncol=3, byrow=TRUE)
> w <- matrix(c(1, 2, 4, 4, 2, 6, 6, 3, 9), ncol=3, byrow=TRUE)
> a <- array(c(x, y, z, w), dim=c(3, 3, 4))
> a
, , 1
[,1] [,2] [,3]
[1,] 3 4 6
[2,] 5 6 5
[3,] 5 7 7
, , 2
[,1] [,2] [,3]
[1,] 4 5 0
[2,] 6 8 9
[3,] 6 8 7
, , 3
[,1] [,2] [,3]
[1,] 1 4 7
[2,] 7 8 6
[3,] 6 4 2
, , 4
[,1] [,2] [,3]
[1,] 1 2 4
[2,] 4 2 6
[3,] 6 3 9
> apply(a, 1:2, mean)
[,1] [,2] [,3]
[1,] 2.25 3.75 4.25
[2,] 5.50 6.00 6.50
[3,] 5.75 5.50 6.25
しかし,これでは,array 関数で配列を作るのがダサダサなので,テキストファイル foo.dat に 3×3 行列の要素を 1 行に 9 個ずつ含むように作り(列優先で配置),それを読み込んで array 関数を適用する。foo.dat は以下のようにもなるだろう。
3 5 5 4 6 7 6 5 7 4 6 6 5 8 8 0 9 7 1 7 6 4 8 4 7 6 2 1 4 6 2 2 3 4 6 9で,それを読み込んで,以下のようにする。
mat <- scan("foo.dat")
apply(array(mat, dim=c(3, 3, length(mat)/9)), 1:2, mean)
[,1] [,2] [,3]
[1,] 2.25 3.75 4.25
[2,] 5.50 6.00 6.50
[3,] 5.75 5.50 6.25
もし,foo.dat を用意したとすれば,以下のようにしても解は得られる。
> matrix(apply(matrix(mat, ncol=9, byrow=TRUE), 2, mean), 3)
[,1] [,2] [,3]
[1,] 2.25 3.75 4.25
[2,] 5.50 6.00 6.50
[3,] 5.75 5.50 6.25
そんなところかな。USB (2014-01-28 (火) 07:22:31)
x,y,z,a 1A5,584,127,65 681,862,98C,378 1A5,647,34,816 3489,65,261,936上記のように並んでいるデータをx列のデータを第一優先に、そして、xに同数字があった場合には、zの列のデータを第二優先に並べ替えたいです。
x,y,z,a 1A5,647,34,816 1A5,584,127,65 681,862,98C,378 3489,65,261,936結果的にはこのような形です。
Rを扱うのがまだ慣れていないためか、数字だけではないので、難しい気がしてならないです。この点で困っていたところ、この掲示板を見つけたので投稿させていただきました。どなたか回答をお願いいたします。
> d
x y z a
1 1A5 584 127 65
2 681 862 98C 378
3 1A5 647 34 816
4 3489 65 261 936
> (d <- d[order(sprintf("%20s", d$x), sprintf("%20s", d$z)),])
x y z a
3 1A5 647 34 816
1 1A5 584 127 65
2 681 862 98C 378
4 3489 65 261 936R初心者 (2014-01-27 (月) 11:02:51)
R初心者です。初めての投稿になります。
以前も同じような質問が出ていましたらすみません。
W,R,1,2,3,4の6種類の文字数字の列のデータから
(これらの文字数字はある現象のステージを示すものです。)
「W→1」「W→2」「W→3」「W→4」「W→R」
「1→2」「1→3」「1→4」「1→R」
「2→3」「2→4」「2→R」
「3→4」「3→R」
「4→R」
の文字の遷移の確率を出したいのですが、
(矢印の逆方向の場合も同様に必要です。)
連続する2数をどのように取り上げればいいのか分かりません。
あと、今回のように文字と数字が混合している場合の扱い方も分かりません。
どうかご教示、よろしくお願い致します。
> y<- with(rle(x), rep(values, ifelse( lengths, 1)))を用いて省略させるところまではわかりました。よろしくお願い致します。 -- R初心者 2014-01-27 (月) 11:26:15
> (a <- c("W", "R", 1:4))
[1] "W" "R" "1" "2" "3" "4"
連続する要素は,a[1:5] と a[2:6] のような2つのベクトルの要素を対比すればよいですね。
> a[-6] # a[1:5] と同じ [1] "W" "R" "1" "2" "3" > a[-1] # a[2:6] と同じ [1] "R" "1" "2" "3" "4"で,遷移行列は,table 関数を使えばよいですね。
> x <- "WWWWWW11111122222233333334444444443333322222RRRRRR3333222111WWWWW"
> y <- unlist(strsplit(x, ""))
> n <- length(y)
> (z <- table(y[-n], y[-1]))
1 2 3 4 R W
1 7 1 0 0 0 1
2 1 11 1 0 1 0
3 0 2 13 1 0 0
4 0 0 1 8 0 0
R 0 0 1 0 5 0
W 1 0 0 0 0 9
で,遷移確率が行方向の確率が1になるようにというものなら,prop.table 関数が使えます。小数点以下3桁まで出したいなら,round 関数を使って以下の通り。
> round(prop.table(z, 1), 3)
1 2 3 4 R W
1 0.778 0.111 0.000 0.000 0.000 0.111
2 0.071 0.786 0.071 0.000 0.071 0.000
3 0.000 0.125 0.812 0.062 0.000 0.000
4 0.000 0.000 0.111 0.889 0.000 0.000
R 0.000 0.000 0.167 0.000 0.833 0.000
W 0.100 0.000 0.000 0.000 0.000 0.900
投稿方法について,再確認して,汚くなった掲示板をキレイにしてくださいね。 -- 河童の屁は,河童にあらず,屁である。 2014-01-27 (月) 11:44:01> x<-read.table("Book1.txt",sep="\t",header=T) # タブ区切り,ヘッダあり
x
A B
1 1 WWWW11122233334443333222RRRRRR333222111WWW
2 2 WWWW11122233334443333222RRRRRR333222112WWW
3 3 WWWW11122233334443333222RRRRRR333222113WWW
4 4 WWWW11122233334443333222RRRRRR333222114WWW
5 5 WWWW11122233334443333222RRRRRR333222115WWW
> a<-"x[1,2]"
> y <- unlist(strsplit(a, ""))
> n <- length(y)
> (z <- table(y[-n], y[-1]))
, [ ] 1 2
, 0 0 0 0 1
[ 0 0 0 1 0
1 1 0 0 0 0
2 0 0 1 0 0
x 0 1 0 0 0
> round(prop.table(z, 1), 3)
, [ ] 1 2
, 0 0 0 0 1
[ 0 0 0 1 0
1 1 0 0 0 0
2 0 0 1 0 0
x 0 1 0 0 0x <- read.table("Book1.txt", sep="\t", header=TRUE, as.is=TRUE)
のように,as.is=TRUE を指定しないと。なお,TRUE は T のように省略しないようにした方がよいですね。a <- "x[1,2]"はだめです。x の1行目の2列目を取り出そうと思ったら,
a <- x[1, 2]とするのです。x に文字列が入っているからダブルクオートでくくろうと思ったのかもしれませんが,冷静に考えてください。"x[1,2]" は 6 文字からなる文字列ですよ。a <- "abcdef" である場合を考えれば分かるでしょう。 -- 河童の屁は,河童にあらず,屁である。 2014-01-28 (火) 12:03:32
> #データファイルをRに取り込む。
> x<-read.table("Book1.txt",sep="\t",header=TRUE,as.is=TRUE) # タブ区切り,ヘッダあり
> x
A B
1 1 WWWW11122233334443333222RRRRRR333222111WWW
2 2 WWWW11222233334443333222RRRRRR333222111WWW
3 3 WWWW12222233334443333222RRRRRR333222111WWW
4 4 WWWW22222233334443333222RRRRRR333222111WWW
5 5 WWWW22222233334443333222RRRRRR333222211WWW
> #tableを示す操作をまとめて定義(=too関数と名付けた。)
> too<-function(q){
> + a<-x[q,2]
> + y <- unlist(strsplit(a, ""))
> + n <- length(y)
> + z <- table(y[-n], y[-1])
> + round(prop.table(z, 1), 3)}
> #tableを行の数だけ表示する。(too関数を繰り返す。)
> lapply(1:nrow(x),too)
その後、表示されたテーブル同士の対応する数字同士の演算を一括で行い、同じような形式の表で示したいのですが、どのようにしたらよいでしょうか??-- R初心者 2014-01-29 (水) 12:04:33> too <- function(q) {
+ a <- x[q, 2]
+ y <- unlist(strsplit(a, ""))
+ n <- length(y)
+ z <- table(y[-n], y[-1])
+ # round(prop.table(z, 1), 3) # これを計算するのは,まだ時期尚早
+ return(z) # 集計結果(度数)の方を返す
+ }
> z <- lapply(1:nrow(x), too)
> z # 結果を見てみる
[[1]]
1 2 3 4 R W
1 4 1 0 0 0 1
2 1 6 1 0 1 0
3 0 2 8 1 0 0
4 0 0 1 2 0 0
R 0 0 1 0 5 0
W 1 0 0 0 0 5
[[2]]
1 2 3 4 R W
1 3 1 0 0 0 1
2 1 7 1 0 1 0
途中省略
[[5]]
1 2 3 4 R W
1 1 0 0 0 0 1
2 1 10 1 0 1 0
3 0 2 8 1 0 0
4 0 0 1 2 0 0
R 0 0 1 0 5 0
W 0 1 0 0 0 5
> (Sum <- Reduce("+", z)) # 足し算
1 2 3 4 R W
1 12 3 0 0 0 5
2 5 40 5 0 5 0
3 0 10 40 5 0 0
4 0 0 5 10 0 0
R 0 0 5 0 25 0
W 3 2 0 0 0 25
> round(prop.table(Sum, 1), 3) # 割合(確率)を計算
1 2 3 4 R W
1 0.600 0.150 0.000 0.000 0.000 0.250
2 0.091 0.727 0.091 0.000 0.091 0.000
3 0.000 0.182 0.727 0.091 0.000 0.000
4 0.000 0.000 0.333 0.667 0.000 0.000
R 0.000 0.000 0.167 0.000 0.833 0.000
W 0.100 0.067 0.000 0.000 0.000 0.833
こんなところでしょうか。 -- 河童の屁は,河童にあらず,屁である。 2014-01-29 (水) 12:28:24> apply(z,1:2,sum) 以下にエラー apply(z, 1:2, sum) : dim(X) は正の長さを持たねばなりません
> z2 <- array(0, dim=c(6, 6, 5)) # メモリ確保
> for (i in 1:5) {
+ z2[, , i] <- z[[i]] # 行列単位で代入
+ }
> apply(z2, 1:2, sum)
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 12 3 0 0 0 5
[2,] 5 40 5 0 5 0
[3,] 0 10 40 5 0 0
[4,] 0 0 5 10 0 0
[5,] 0 0 5 0 25 0
[6,] 3 2 0 0 0 25
のようにするのも一法。 -- 河童の屁は,河童にあらず,屁である。 2014-01-29 (水) 17:40:13> x
A B
1 1 WWWW11122233334443333222RRRRRR333222111WWW
2 2 WWWW11222233334443333222RRRRRR333222111WWW
3 3 WWWW12222233334443333222RRRRRR333222111WWW
4 4 WWWW22222233334443333222RRRRRR333222111WWW
5 5 WWWW22222233334443333222RRRRRR333222211WWW
行によって含む要素の数が違う場合
(例えば2,3行目が「1,2,W」の3要素しか含んでいない。)
はどのようにしたらよいでしょうか??
そのまま処理したら、2行目と3行目は3×3行列になってしまい、lapply関数後の処理がエラーになってしまいました。
> x
A B
1 1 WWWW11122233334443333222RRRRRR333222111WWW
2 2 WWW22211111111122222222221111111112222111W
3 3 WWWW11111112222222222222221111112222211WWW
4 4 WWWW22222233334443333222RRRRRR333222111WWW
5 5 WWWW22222233334443333222RRRRRR333222211WWW
含まれていない要素の値は0で示し、今までと同じように6×6行列のtableで表したくて、
データのそれぞれの行の遷移確率同士を演算したいです。
ご教示お願いいたします。-- R初心者 2014-01-31 (金) 09:33:50z2 <- lapply(z, function(x) {
names <- c(1:4, "R", "W")
y <- matrix(0, 6, 6, dimnames=list(names, names))
for (i in 1:nrow(x)) {
for (j in 1:ncol(x)) {
y[rownames(x)[i], colnames(x)[j]] <- x[i, j]
}
}
return(y)
})
これを,旨くやろうという魂胆で書き直すと,以下のようになるでしょうが,上のアルゴリズムが分かっていないと,よく分からないプログラムに見えるでしょうね。
z3 <- lapply(z, function(x) { # x が 6×6 行列になる場合を特別扱いしても良いけどめんどくせ!
names <- c(1:4, "R", "W")
y <- matrix(0, 6, 6, dimnames=list(names, names))
y[names %in% rownames(x), names %in% colnames(x)] <- x
return(y)
})
ともかくも,どちらのプログラムも同じ結果になるはずで,
> z[[2]]
1 2 W
1 18 2 1
2 3 14 0
W 0 1 2
> z2[[2]]
1 2 3 4 R W
1 18 2 0 0 0 1
2 3 14 0 0 0 0
3 0 0 0 0 0 0
4 0 0 0 0 0 0
R 0 0 0 0 0 0
W 0 1 0 0 0 2
> z[[3]]
1 2 W
1 12 2 1
2 2 18 0
W 1 0 5
> z3[[3]]
1 2 3 4 R W
1 12 2 0 0 0 1
2 2 18 0 0 0 0
3 0 0 0 0 0 0
4 0 0 0 0 0 0
R 0 0 0 0 0 0
W 1 0 0 0 0 5JJ (2014-01-22 (水) 08:36:01)
お世話になります。
bioconductorのメーリングリストに投稿し、回答を得たのですが、回答者への返信の仕方が分かりません・・・。単に回答者のメールアドレスにメールを送信すればよいのでしょうか?しかしそうするとスレッドに表示されないような気がしています。どなたかやり方をご存知でしたら教えて頂けませんか。
https://stat.ethz.ch/pipermail/bioconductor/2014-January/thread.html
宜しくお願いします。
TT (2014-01-17 (金) 21:09:02)
項目の困難度、識別力の算出はできたのですが、それらの標準誤差の算出方法をご存知の方がいらっしゃいましたら、ご教示お願いいたします。
fractal (2014-01-16 (木) 20:01:40)
ホームページに以下のように示されている、データの平均値の出しかたを教えてください。データ数は240個です。
データは、ホームページから、エクセルにコピーしてあります。
19 9 11 7 5 5 2 5 2 2 2 2 2 2 15 7 2 2 2 2 5 5 5 2 2 2 2 2 5 5 2 7
11 11 7 5 5 2 7 22 11 15 9 7 22 7 35 7 5 8 8 7 5 7 7 11 5 8 16 8 5 7 11 11
9 5 8 8 8 5 7 8 5 2 5 2 2 2 2 2 5 2 5 16 11 7 19 19 5 5 5 11 11 7 19 19
11 7 5 8 5 5 22 42 11 5 5 5 11 7 2 2 2 5 15 7 5 7 5 8 5 7 5 11 34 22 30 15
19 16 5 5 5 2 11 22 11 22 15 11 11 5 5 31 2 5 5 5 8 5 5 2 5 5 5 5 5 11 7 35
5 15 16 15 7 5 7 15 11 15 11 11 15 22 7 2 2 15 7 8 30 11 42 62 42 22 30 22 22 30 35 42
22 15 18 34 22 42 35 11 9 22 7 11 15 7 11 11 11 11 11 8 15 30 11 31 19 15 7 7 8 22 7 11
9 2 8 2 7 15 11 9 9 5 5 2 2 2 2 16 5 9 2 5 2 2 5 11
このような計算を1000する必要があるので、効率的な方法を知りたいです。
よろしくお願い致します。
getMean <- function(url) {
a <- scan(url)
stopifnot(length(a) == 3012)
a <- matrix(a, 251)[-(1:3),]
apply(a, 2, function(x) mean(x[x != -1]))
}
> getMean("http://isgi.latmos.ipsl.fr/source/indices/aa/aa1868.ima")
Read 3012 items
[1] 10.55645 15.96552 19.72177 20.97917 16.39919 17.94583
[7] 21.45565 19.37097 24.03750 25.94355 13.32083 13.68952
引数 url には,Web 上のページをそのまま読むなら "http://isgi.latmos.ipsl.fr/source/indices/aa/aa1868.ima" を指定,事前に作業ディレクトリに保存してあればそのファイル名("aa1868.ima" など)を指定します。getMean2 <- function(fileName) {
x <- scan(fileName)
mean(x[x != -1])
}
getMean2("aa11868.txt")
このレベルのプログラムを求めるというのでは,たくさんあるファイルに対してこの関数を適用するという部分を自分でプログラム化するのはとても,無理と言うことでしょうか?ファイル名の規則性はどのようになっていますか? -- 河童の屁は,河童にあらず,屁である。 2014-01-18 (土) 09:24:30files <- c("aa11868.txt", "aa11869.txt", "aa11870.txt", ...)
sapply(files, getMean)
とすればよいでしょう(プログラムの各部分がどのように機能しているのかはっきり分からないまま,適当にいじくっても動きませんよ。動いたように見えても,正しい答えが得られないことが多いでしょう。)。getMean3 <- function(url) {
a <- scan(url)
stopifnot(length(a) == 3012)
a <- matrix(a, 251)[-(1:3),]
mean(a[a != -1])
}
でよいわけですね。失礼しました。 -- 河童の屁は,河童にあらず,屁である。 2014-01-18 (土) 20:17:41よる (2014-01-16 (木) 15:29:06)
以前に似たような質問をしており恐縮なのですが、複数のバイナリセーブしたデータフレーム形式のオブジェクトを読み込み結合したいと思っています。ファイル名は「Data」に続いて日付と拡張子(.RData)のついたファイルが複数格納されているときに(例えば、Data20121101.RData, Data20121102.RData, ... Data20151231.RData)、特定の日付けのファイル(例えば、100日ぶん)をRに読み込んで一つのデータフレームにまとめるにはどうすればいいでしょうか?
それぞれのファイルは以下のようになっていて形式は同じです。> Data20121001 a b c 1 1 0.7420626 6.166458 2 2 -0.2008887 6.347304 3 3 1.4550494 6.860432ファイルがテキスト(アスキー)形式のときは「複数ファイルを、ファイル名を指定して読み込み、一つのデータフレームにまとめる方法」にて質問させていただき解決したのですが、バイナリセーブされた.RDataでは読み込む前にオブジェクト名が割りあてられているために応用できずにいます。
file <- "Data20121001.RData" # 起点となるファイル名
n <- 3 # 連結したいファイル数(日数)
date <- as.Date(paste(substr(file, 5, 8), substr(file, 9, 10), substr(file,
11, 12), sep = "-"))
file.Name <- as.character(date + 0:(n - 1))
result <- NULL # 結果
for (i in file.Name) {
string <- as.character(i)
object <- paste("Data", gsub("-", "", string), sep = "")
load(paste(object, "RData", sep = "."))
result <- eval(parse(text = sprintf("rbind(result, %s)", object)))
# 読み込んだオブジェクトを消去しておきたいとき
eval(parse(text = sprintf("rm(%s)", object)))
}
resultbuynnnmmm1 (2014-01-15 (水) 19:15:11)
wavelet処理できるRのパッケージはかなりあります。パッケージによって、用意されてるwaveletが異なると思います。私はまだwmtsaのwavCWTしか試してないのですが、周波数精度を比較的高目(時間精度はある程度犠牲にして)、処理したい場合、お勧めのwaveletパッケージや、それに適したwaveletのタイプについて教えてください。
(wmtsaの場合、ちょっと試した感じでは、wavelet="morlet", shiftをデフォルトよりちょっと大きめにすると、周波数精度を比較的高めの結果がでるのですが、高い周波数にあるはずのないピークがあらわれやすくなります。)
よろしくお願い致します。
(2014-01-12 (日) 23:19:08)
最近の質問者は,ありがともいえないのな...
松浦 (2014-01-12 (日) 11:25:36)
install.packagesによってcar他のパッケージをインストールしようとしていますが、Would you like to use a personal library instead? というメッセージが出て、Noと回答するとインストールできず、Yesと回答するとファイルは保存されるもののlibraryで呼び出すことができません。
パソコン(windows8です)のセキュリティの問題かもしれませんが、対応策をご存じの方がいらっしゃればご教示いただきたくよろしくお願いします。
R初心者 (2014-01-12 (日) 09:05:52)
R初心者です。
初めての投稿になりますよろしくおねがいいたします。
質問内容ですが、以下のような二つのcolumを作成した場合、> x<-c("a","b","c") > colum1<-matrix(x) [,1] [1,] "a" [2,] "b" [3,] "c" > y<-c("a","b","c","d","e") > colum2<-matrix(y) [,1] [1,] "a" [2,] "b" [3,] "c" [4,] "d" [5,] "e"colum2からcolum1の要素を削除するにはどのようにしたらよいでしょうか?
以下の様に2つのベクトルを結合し、unique()を用いましたが、> unique(rbind(colum1,colum2)) [,1] [1,] "a" [2,] "b" [3,] "c" [4,] "d" [5,] "e"重複した”a”,"b","c"が残ってしまいます。
非常に基礎的質問だとおもわれますが、ご回答頂ければ幸いです。
尚、動作環境は以下の様に成ります。R version 3.0.1 (2013-05-16) Platform: x86_64-apple-darwin10.8.0 (64-bit)
> setdiff(colum2, colum1) [1] "d" "e"が参考になるかな? -- 河童の屁は,河童にあらず,屁である。 2014-01-12 (日) 09:07:55
buynnnmmm1 (2014-01-11 (土) 21:31:22)
https://box.raksul.com/dl/bdbc45c2b62ff6a3ec489de2cf300f94
元データファイルと、処理したRソースを添付してます。
テスト用の音声ファイルは 1秒ごとに、880Hz,1760Hz,3520Hzのsin波の音になってます。
それを tuneRというパッケージの readWaveファイルで読み込んで、もともとステレオじゃない音ファイルのleftのデータを処理して時間と周波数の処理をしてます。
waveletまったく勉強しようとしてないわけじゃなく、図書館で本はかりて読んだっこはありますが、理解できてません。いろいろな種類の波形で処理ですっていうのと、連続と不連続の処理のしかたがある程度わかってます。
周波数の精度をあげると、時間の精度がさがると考えてて、(間違ってるかもしれませんが、)今のデフォルトのwavelet処理(wmstsaの wavCWTと wavDWT )はデフォルトだと、周波数分解能が悪いと思ってます。周波数方向(縦軸方向)でかなりぼやけてるので。
いっぽう、seewaveっていうパッケージもみつけてまして、こちらは、ちゃんと確認してませんが、マドをつくって、短時間のフーリエ変換して、周波数成分の時間変化をspectroで可視化できます。こちらは、窓の大きさを調節すればかなり周波数の精度をあげれました。デフォルトでもかなり縦方向(y軸方向)に狭くなってて、わかりやすいと思ってます。
waveletにしようと思ったのが、もっと周波数精度があがると思いました。数学的に、これいじょう細かく分解できない、周波数と時間に分解してくれるイメージがあったので(これもちゃんと理解できてないので、間違ってるのかもしれません。)
ということで、やりたいことは、時間の精度をもっと下げて、周波数の精度を上げたデータ処理をwaveletをつかってやりたいのですが、やり方がわかりません。
wavCWTとかに、周波数の分割数を変更するパラメータがあったのですが、それではほとんど変化ありませんでした。
本をかりたのはかなり前なのですが、その本では理解できなかったので、わかりやすい資料も探してます。これ読んで理解できたという資料があれば、それも紹介お願い致します。
よろしくお願い致します。
buynnnmmm1 (2014-01-11 (土) 19:19:03)
テスト用のオーディオファイルを作成して、それをwavelet処理し、比較的周波数分解能の良い処理を行ないたいと思ってます。しかしどのパラメーターを変更したら周波数分解能をあげて、時間分解能を下げれるのか良くわかりません。
seewaveという短時間フーリエ変換で時間と周波数の表示をするものがあって、そちらの方が周波数分解能がデフォルトで高いように思います。
こちらは窓を広くすると時間分解能がおちて周波数分解能があがるのですが。
http://note.chiebukuro.yahoo.co.jp/detail/n242822
の「wmstsaの場合」 の 「処理例」のところに1秒ごとに、880Hz,1760Hz,3520Hzのsin波の音を作って、それを処理して可視化したものをはりました。
処理はぜんぶデフォルトの条件で処理して、パラメーターは変更してません。
tuneRパッケージのwaveReadで音声ファイルを読み込んで、left要素のみをtsで時系列に変換してから、wavCWT と wavDWT にかけて、その出力をplot関数にかけただけです。
一番下のwaveseeの出力はspectro関数にかけて出力しました。
wavelet解析について、ほとんどなにも知りません。もし良い入門サイトや資料があれば、それも教えていただければ幸いです。
よろしくお願い致します。
いんせ (2014-01-09 (木) 12:26:29)
コメントありがとうございます. 冒頭部分の> data(…) > prcomp(…)の括弧内にエクセルのデータを入力しようとしました.
ファイル名を入れたり,実際の因子の名前などを入れて,D<-read.csv("R bact3.csv",header=T) D$A系-1<-factor(D$A系-1) D$A系-2<-factor(D$A系-2) head(D) summary(D) data(D) prcomp(D)としても,
警告メッセージ: In data(D) : データセット ‘D’ がありません > prcomp(D) 以下にエラー colMeans(x, na.rm = TRUE) : 'x' は数値でなければなりませんとでます.
どうすればよいのでしょうか.
いんせ (2014-01-09 (木) 00:35:30)
初めまして.Rを使い始めたばかりの初心者ですが,主成分分析をしなければなりません.データ元はエクセルで管理しているので,エクセルの拡張子をcsv形式にしてから,データをRに取り込んで作業しています.
他の簡単な操作では,エクセルから呼び出すことができるのですが,主成分分析では,エラーが出てしまい,作業が進みません.
つまり,ほぼ最初からうまくいっておりません.
自分でもいろいろ調べたりしているのですが,やはりわかりません.
勉強不足でありながら,誰かに聞くのは甘えでしかありませんがどなたか,具体的な手順など教えていただければ幸いです.
よろしくお願いいたします.
しょう (2014-01-03 (金) 14:50:11)
コメントありがとうございました。
なんとか計算ができました。
2つの時系列があった場合、それらの変化の仕方、上がったり下がったりの様子の類似度を見る相関係数のようなものはRにあるのでしょうか?
いわゆる相関係数が小さくても、時系列はよく似た変化をするものがあります。
どれくらい似ているかを数値化したいのですが。
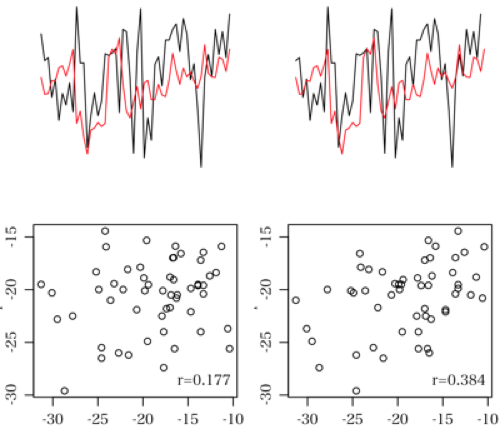
> n <- lowess(seq_along(nao$V1), nao$V1, f = 1/3) > s <- lowess(seq_along(sun$V1) - 4, sun$V1, f = 1/3) > cor(s$y[5:54], n$y[1:50]) [1] 0.9167944
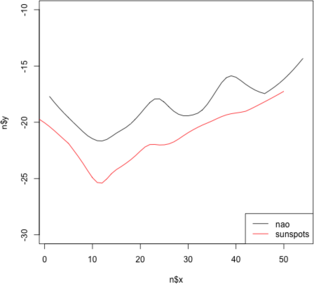
しょう (2014-01-02 (木) 20:05:12)
2つのcsv ファイルにある時系列の数字を読み込んで、2つの相関係数を求めることはできるでしょうか。
プログラムをを教えていただけるとうれしいです。
シップスクラーク (2013-12-30 (月) 23:19:13)
2つの別ファイルから共通した項だけを抽出したいです。
あるdata1というファイルとdata2というファイルがあるとします。例えば
data1xy,yz 9530,087361 9690,186479data2
xz,zy 9690,547895 5121,168415とデータだとすると、この2つのデータの左側の値が同じだった場合にその行の値を出力したいです。そのような方法はありますでしょうか?このデータは下に続いていると考えてください。Rを使い始めてまだ日が浅いので、別々のファイルでのデータの扱い方などがあまり理解出来ていないので、よろしくお願いします。
> data1 <- read.csv("data1")
> data2 <- read.csv("data2")
> merge(data1, data2, by.x="xy", by.y="xz")
xy yz zy
1 9690 186479 547895しょう (2013-12-27 (金) 18:04:33)
警告メッセージ: 1: In eval(expr, envir, enclos) : 複素数の虚部は、コネクションで捨てられました 2: In eval(expr, envir, enclos) :~ 複素数の虚部は、コネクションで捨てられました 3: In readLines("NAO1910.txt") : 'NAO1910.txt' で不完全な最終行が見つかりました 4: In eval(expr, envier, enclos) : 複素数の虚部は、コネクションで捨てられました 5: In plot.xy(xy, type, ...) : plot type 'line' will be truncated to first characterノイズをカットするプログラムです
x <- c(1, 1, 1, 1, 0, 0, 0, 0) y <- fft(x) z <- fft(fft(x), inverse = TRUE)/length(x) round(z, digit = 4) as.double(z) # cut high frequency from mid-lag # i.e, the center part mid <- (length(y) + 2)/2 lag <- 1 yy <- y for (i in (mid - lag):(mid + lag)) { yy[i] <- 0 } w <- fft(yy, inverse = TRUE)/length(yy) q <- as.double(w) plot(q) ### NAO ### # source data IOD_ENSO_NAO/NAO-monthly-Jim-Hurrell-1870-2000-1d.txt # xsource<- readLines("NAO50-08.txt") xsource <- readLines("NAO1910.txt") xorg <- as.double(xsource) # take part (e.g, 600 month =50 years) # x<-xorg[(length(xorg)-708+1):length(xorg)] x <- xorg[(length(xorg) - 1236 + 1):length(xorg)] # standardize(normalize) sx <- (x - mean(x))/sd(x) ## fft len <- length(sx) y <- fft(sx) # cut high frequency mid <- (length(y) + 2)/2 lag <- 250 - 1 yy <- y for (i in (mid - lag):(mid + lag)) { yy[i] <- 0 } # inverse w <- fft(yy, inverse = TRUE)/length(yy) q <- as.double(w) plot(q, type = "line") # plot(sx, type = "line")
z <- fft(fft(x), inverse = TRUE)/length(x) round(z, digit = 4) as.double(z)しかし,もう一度同じことをやっている以下のところで,エラーが出るのも同じですよ。複素数を as.double している。
w <- fft(yy, inverse = TRUE)/length(yy) q <- as.double(w)「'NAO1910.txt' で不完全な最終行が見つかりました」は,読み込んだファイルの最後が改行文字でないと言うこと。
しょう (2013-12-27 (金) 17:33:19)
as.double()にしたら動きました。ありがとうございました。
ただ、次のエラーが出てしまいました。助けてください。1: In eval(expr, envir, enclos) : 複素数の虚部は、コネクションで捨てられました 2: In eval(expr, envir, enclos) : 複素数の虚部は、コネクションで捨てられました 3: In file(con, "r") : ファイル 'NAO1910.txt' を開くことができません: No such file or directory
しょう (2013-12-27 (金) 15:41:52)
下のエラーが出てしまい困っています。
アドバイスください。以下にエラー eval(expr, envir, enclos) : 関数 "as.real" を見つけることができませんでした
みかん (2013-12-26 (木) 14:38:02)
投稿失礼します。
TSAパッケージのgarch.simに入っているrndというオプションを利用して、自由度を指定したt分布のシュミレーションを行いたいのですが、自由度を指定したところうまく動作しませんでした。
このパッケージではt分布の使用ができないのでしょうか?また、t分布を用いたgarchモデルの推定とシュミレーションができるパッケージがありましたら、お教え頂ければ幸いです。
よる (2013-12-25 (水) 22:56:31)
ディレクトリに「t」に続いて日付と拡張子(.txt)のついたファイルが複数格納されているときに(例えば、t20121101.txt, t20121102.txt, ... t20151231.txt,)、特定の日付けのファイル(例えば、100日ぶん)をRに読み込んで一つのデータフレームにまとめるにはどうすればいいでしょうか?
それぞれのファイルは以下のようになっていて形式は同じです。> t20121001 x y z 1 -0.9139121 0.9233681 1.34668386 2 -0.4414702 1.7249676 -0.03165230 : 9 0.6055080 -0.8037918 -3.16458153 10 -0.5272891 -1.2061249 1.42032382ディレクトリにあるすべての.txtファイルを読み込む際は、
library(plyr); d=ldply(dir(getwd(), "*.txt"), function(x) read.table(x, h=T))として読み込めたのですが、日付を特定しようとするとうまくいきません。
例えば、以下のようにしてファイル名のベクトルをつくって試してみたのですが、dateval = format(seq("2010/11/01", by="1 days", length=100, "%Y%m%d") filename = paste("t", dateval, ".txt", sep="") d=ldply(dir(getwd(), dateval), function(x) read.table(x, h=T))dir(getwd(), dateval)の部分で、datevalが最初の値(dateval[1])しか使われていないようで、うまくいきませんでした。
また、少し蛇足になってしまうかもしれませんが、11月10日から11月30日までのデータを呼ぼうとして以下を実行すると、11月すべてのデータが呼ばれてしまいました。dir(getwd(), "t201211*") [1] "t20121101.txt" "t20121102.txt" "t20121103.txt"... [8] "t20121108.txt" "t20121109.txt" "t20121110.txt"... [15] "t20121115.txt" "t20121116.txt" "t20121117.txt"... [22] "t20121122.txt" "t20121123.txt" "t20121124.txt"... [29] "t20121129.txt" "t20121130.txt"なぜ、"t20121101.txt"等が呼ばれてしまうのでしょうか?
上記の問題解決作をご教授いただければ幸いです。
x <- sprintf("t201211%02i.txt", 10:30)
# x <- c("t20121112.txt", "t20121115.txt", ..., "t20121124.txt") などなんでも
result <- NULL
for (i in x) {
d <- read.table(i, header=TRUE)
result <- rbind(result, d)
}
result
どうでしょ? -- 河童の屁は,河童にあらず,屁である。 2013-12-26 (木) 00:14:15rpt (2013-12-17 (火) 10:26:36)
お世話になります。
ライブラリrpartのCP(Complexity Parameters)について、理解できないで困っています。
例えばirisデータで次のように入力しますと> library(rpart) > iris.rpt <- rpart(Species ~ ., data=iris) > printcp(iris.rpt) Classification tree: rpart(formula = Species ~ ., data = iris) Variables actually used in tree construction: [1] Petal.Length Petal.Width Root node error: 100/150 = 0.66667 n= 150 CP nsplit rel error xerror xstd 1 0.50 0 1.00 1.23 0.047053 2 0.44 1 0.50 0.67 0.060888 3 0.01 2 0.06 0.08 0.027520のようにCP表が出力されます。
これについて大滝他「応用2進木解析法」(日科技連)によりますと、CPはrel errorに基づいて次のように計算するものとされ、> 0.6667*(1-0.5) [1] 0.33335 > 0.6667*(0.5-0.06) [1] 0.293348上記の出力結果のCPと一致しません。
回帰ツリーの場合は、rel errorがRSS比のことだということはよくわかるのですが、分類ツリーの場合のrel errorそのものについて、私が理解していないからかもしれません。
どなたか、この不一致の理由がわかる方、御教示いただければ幸いです。
> iris.rpt2 <- rpart(Species ~ ., data=iris,
+ control = rpart.control(minsplit=4, cp=0.0001))
> iris.rpt2
n= 150
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 150 100 setosa (0.33333333 0.33333333 0.33333333)
2) Petal.Length< 2.45 50 0 setosa (1.00000000 0.00000000 0.00000000) *
3) Petal.Length>=2.45 100 50 versicolor (0.00000000 0.50000000 0.50000000)
6) Petal.Width< 1.75 54 5 versicolor (0.00000000 0.90740741 0.09259259)
12) Petal.Length< 4.95 48 1 versicolor (0.00000000 0.97916667 0.02083333)
24) Petal.Width< 1.65 47 0 versicolor (0.00000000 1.00000000 0.00000000) *
25) Petal.Width>=1.65 1 0 virginica (0.00000000 0.00000000 1.00000000) *
13) Petal.Length>=4.95 6 2 virginica (0.00000000 0.33333333 0.66666667)
26) Petal.Width>=1.55 3 1 versicolor (0.00000000 0.66666667 0.33333333) *
27) Petal.Width< 1.55 3 0 virginica (0.00000000 0.00000000 1.00000000) *
7) Petal.Width>=1.75 46 1 virginica (0.00000000 0.02173913 0.97826087) *
> printcp(iris.rpt2)
...
Root node error: 100/150 = 0.66667
n= 150
CP nsplit rel error xerror xstd
1 0.5000 0 1.00 1.16 0.051277
2 0.4400 1 0.50 0.59 0.059828
3 0.0200 2 0.06 0.09 0.029086
4 0.0100 3 0.04 0.09 0.029086
5 0.0001 5 0.02 0.07 0.025833
のようになりますが、rel errorを各ステップにおける誤分類数と理解すると
> (100/150)/(100/150) [1] 1 > (50/150)/(100/150) [1] 0.5 > (6/150)/(100/150) [1] 0.06 > (3/150)/(100/150) [1] 0.03となり、4行目以降の数値が合いません。 また、次のようなモデルのCPテーブルを出力しますと、
> Country2 <- factor(60, levels=c("Japan", "Others")) #初期化
> Country2[car.test.frame$Country=="Japan"] <- "Japan"
> Country2[car.test.frame$Country!="Japan"] <- "Others"
> car.rpt <- rpart(Country2 ~ Price + Mileage + Type + Weight + Disp. + HP,
+ data=car.test.frame, control=rpart.control(cp=0.001, minsplit=5))
> printcp(car.rpt)
...
Root node error: 19/60 = 0.31667
n= 60
CP nsplit rel error xerror xstd
1 0.157895 0 1.00000 1.0000 0.18964
2 0.105263 1 0.84211 1.2632 0.19972
3 0.035088 6 0.31579 1.2105 0.19821
4 0.001000 9 0.21053 1.0526 0.19218
のように、一部の分岐のみしか表示されず、CPテーブルの表示ルールがわかりません。
重ねての質問で、誠に恐縮ですが、御教示いただければ幸いです。-- rpt 2013-12-18 (水) 15:18:30koheizm (2013-12-13 (金) 19:58:51)
データフレームに一定数の列を指定して追加する方法を考えております。
下の方に列を追加していく関数のQAがありましたが、それとは少し違って単純に列を追加したくおもいます。
rodiaさんのソースを拝借dat <- cbind(rep(x=c("male", "female"), times=10, each=2), rep(x=c("A", "AB", "B", "O"), times=10, each=2)) colname(dat) <- c("sex", "blood")このようなデータフレームがあるとしてsexとbloodの列の隣に複数行追加したいです。
入れる値は0でもNULLでも構いません。
現在は列数の違うデータフレームを統合する必要が生じて、ダミー列を作りたいなと思って詰まってしまいました。ご教授お願いします。
add.rows <- function(d, n, m) { # d: データフレーム,n, m: 追加列数
nr <- nrow(d)
x <- data.frame(matrix(0, nr, n+m))
return(cbind(d[1], x[1:n], d[2], x[-(1:n)]))
}
# 使用例
dat <- data.frame(sex=rep(x=c("male", "female"), times=10, each=2),
blood=rep(x=c("A", "AB", "B", "O"), times=10, each=2))
x <- add.rows(dat, 2, 4)
head(x)
しかし,あなたが本当にやりたいのがダミー行列の追加であるなら,枠だけ作っておいて後で値を埋めるなんてことしなくても,もっと効率的なプログラムをかけるでしょう。元の変数の隣にダミー行列を付加するなんてこともその後の利用を考えるとかえって使いにくいのでは? -- 河童の屁は,河童にあらず,屁である。 2013-12-13 (金) 23:34:04add.rows2 <- function(d, n) { # d: データフレーム,n: 追加列数
nr <- nrow(d)
x <- data.frame(matrix(0, nr, n))
return(cbind(d, x))
}
# 使用例
dat <- data.frame(sex=rep(x=c("male", "female"), times=10, each=2),
blood=rep(x=c("A", "AB", "B", "O"), times=10, each=2))
x <- add.rows2(dat, 4)
head(x)よる (2013-12-12 (木) 14:09:07)
以下のコードで生成したf1, f2, f3, ... というオブジェクトを、save関数を使ってプログラム中で、それぞれのオブジェクトを.RDataで保存するにはどうすればいいでしょうか?x <- 1:10 for (i in 1:10) { assign(paste("f", i, sep=""), x[i]) }実際のiは1:100000だったり、c("test1", "test2", ...) だったりします。
> x <- 1:10
> for (i in 1:10) {
+ name <- paste("f", i, sep="")
+ assign(name, x[i])
+ eval(parse(text=sprintf("save(%s ,file=\"%s\")", name, name)))
+ }レジェ (2013-12-10 (火) 19:53:22)
お世話になります。Windows XP, R3.0.1を使用しています。
以下のようなグラフの凡例の少し左側に、別の凡例を追加したいと考えています。そのために引数"topright"の座標の情報を取り出したいのですが、どのようにすればよいでしょうか?もしくはもっと簡単な方法があればご教示願います。
お手数ですが、ご回答宜しくお願いします。plot(rnorm(50), rnorm(50), xlim=c(-3,6), ylim=c(-3,3)) legend("topright", "sin", col="red", pch=3)
みかん (2013-12-10 (火) 15:35:26)
投稿失礼します。R初心者です。
EGARCHモデルを推定しようとして、以前あったegarchパッケージを探したのですが、パッケージのダウンロードサイトでは見つかりませんでした。このパッケージの入手方法をご存知の方がいましたら、お教え頂ければ幸いです。また、egarchパッケージを使用しない推定方法がありましたら、お教え頂ければ幸いです。
R初心者 (2013-12-08 (日) 17:41:10)
投稿失礼します。
「統計学 Rを用いた入門書」で初めてRを勉強しはじめた者です。
ですが、数ページにして詰まってしまいました。。
excelを持っていないため、openofficeを使用しcsv形式にしたら> worms <- read.csv("略/worms.csv",header=T,row.names=1) Warning message: In scan(file, what, nmax, sep, dec, quote, skip, nlines, na.strings, : EOF within quoted stringと出てきました。
odsのままにしてみたり、RopenofficeなるものをPCに入れようともしたのですが、開けないか開いてもバグった表が出てきました。
どなたかアドバイスいただけると嬉しいです!
よろしくお願い致します。
0.444 (2013-12-08 (日) 17:36:36)
psychはダウンロード出来ているようなのですが、パッケージとして存在していない旨のメッセージが出てきて使用できません。> library(psych) Error in library(psych) : there is no package called ‘psych’なおパッケージのインストールは既にRcmdrでも行っており、こちらと同じ手順でダウンロードしたのですが。
よろしく
rhodia (2013-12-05 (木) 18:07:48)
お世話になります。OS:Win7 64bit R3.0.2
以下のように男性女性の性別のカラムと血液型のデータを用意しました。
sexカラムがmaleであれば列を一行足して1,femaleならば0という関数を作りたく思います。dat <- cbind(rep(x=c("male", "female"), times=10, each=2), rep(x=c("A", "AB", "B", "O"), times=10, each=2)) colname(dat) <- c("sex", "blood") tmp_fnc <- function(x) { if (x == "male") x$m_flg = 1 else x$m_flg = 0 } tmp_fnc(dat)その結果このようにメッセージが出てきます。
Warning messages: 1: In if (x == "male") x$m_flg = 1 else x$m_flg = 0 : the condition has length > 1 and only the first element will be used 2: In x$m_flg = 1 : Coercing LHS to a listゴールとしてはsexカラムの中身がmaleならばm_flgが1になり、それ以外なら0とダミー変数をすべて0,1でつくりたいと考えております。
ご指導頂きたく思います。
dat <- data.frame(sex=rep(x=c("male", "female"), times=10, each=2),
blood=rep(x=c("A", "AB", "B", "O"), times=10, each=2))
dat$m_flg <- (dat$sex == "male")+0 # この一行でできます
dat
なお,データフレームの場合,文字型で入力したデータは Factor になるので,(dat$sex == "male")+0 の部分は 2-as.integer(dat$sex) やその他のやり方もあります。また,transform や with 関数を使うやり方もあります。![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべし。
」 を読むべし。
整形済みテキスト
行頭が半角空白で始まる行は整形済みテキストとなります。行の自動折り返しは行なわれません。
整形済みテキストは、他のブロック要素の子要素になることができます。
整形済みテキストは、他のブロック要素を子要素にすることができません。
整形済みテキストは、すべての子要素を文字列として扱います。
ということです。あなたの記事は,私が修正しておきました。-- 河童の屁は,河童にあらず,屁である。? 2013-12-05 (木) 19:15:10じゅん (2013-12-04 (水) 14:56:33)
お世話になります。Windows XP, R 3.0.1を使用しています。
以下のようにbarplotを実行しました。bmp(filename = "bar.bmp", width=1400, height=600) par(mar=c(5,5,4,2)) barplot(round(runif(100)*2000,0), names.arg=1:100, las=3, ylim=c(1,2000), ylab="value") dev.off()この結果を以下のように変更したいと思っています。どのようにすればよいかご教示頂けますか。
・y軸の0〜500を表示したい
・y軸と最初の棒グラフの間隔が空きすぎなので狭くしたい
・x軸の項目名が棒グラフの右寄りにずれているので中心に寄せたい
お手数ですが、宜しくお願いします。
barplot(round(runif(100)*2000,0), names.arg=1:100, las=3,
ylim=c(0,2000), ylab="value", xaxs="i")
KKNN (2013-12-02 (月) 16:30:27)
初めて質問させて頂きます。
各項目の最大値のみを抽出する方法を教えて頂きたいのです。
例えば、下記のようなデータフレームがあるとします。大統領名 在任期間 知名度 A 1 43 A 2 51 A 3 65 B 1 45 B 2 55上記の様なデータテーブルにおいて、各大統領において、在任期間が最大値の時の知名度を抽出したいのです。具体的に言うと、上記のテーブルから、 下記のようなサブセットを抽出するにはどのようにすれば良いでしょうか。宜しく御願い致します。
大統領名 在任期間 知名度 A 3 65 B 2 55
> (a <- data.frame(大統領名 = factor(c("A", "A", "A", "B", "B")),
在任期間 = c(1:3, 1:2),
知名度 = c(43, 51, 65, 45, 55)))
大統領名 在任期間 知名度
1 A 1 43
2 A 2 51
3 A 3 65
4 B 1 45
5 B 2 55
> by(a, a$大統領名, function(x) x[which.max(x$在任期間),])
a$大統領名: A
大統領名 在任期間 知名度
3 A 3 65
--------------------------------------------------------------
a$大統領名: B
大統領名 在任期間 知名度
5 B 2 55
> Reduce("rbind", by(a, a$大統領名, function(x) x[which.max(x$在任期間),]))
大統領名 在任期間 知名度
3 A 3 65
5 B 2 55galois (2013-11-30 (土) 13:27:24)
初めて質問させていただきます。
nlsで求めた式の確からしさはどのように評価したらいいのでしょうか?summaryで示されるt値やp値を使って自分で計算するのでしょうか?または赤池情報量などを用いるのでしょうか?ご存知の方、よろしくお願い致します。
TA (2013-11-25 (月) 17:37:31)
初めて質問させて頂きます。WindowsでRに取組んで1年弱です。
波形画像(jpg)をrimageでRに読み込み、locator()で波形の情報を読取ろうと考えています。しかし、rimageはコンソールから「パッケージのインストール」で呼び出す日本の3つのダウンロードサイトのリストにありません。そこでネットで調べて、http://cran.r-project.org からダウンロードを試みました。しかし、tarファイルであり、これだと、「ローカルにあるzipファイルからインストールする」ではエラーになります。
どのようにすれば良いのか、途方に暮れています。windowsでのrimageのインストール方法を御存知の方があれば、御教え下さい。
また、波形画像から、波形データをRに取り込む、もっと簡便な方法を御存知の方がいらしゃれば、その方法を御教え頂けると嬉しいです。
掃除好き (2013-11-22 (金) 12:37:04)
CAD,WER,ASD,XDR 1262041F20124,800,182139,195001 1262041F20124,900,181347,195003 12621B6F23168,810,153436,195081 12621B6F23168,910,154315,195089 1262236A23148,810,12251,195137 1262236A23148,910,12417,195141 1262236A23148,810,1431535,195005 1262236A23148,910,1502527,195137 1262239B23166,810,155554,195005 1262239B23166,910,123231,195015 126223AE23135,810,0750073,195001 126223AE23135,910,0842509,195013ここからCADの値が2つ続けて同じで、かつXDRの値が195001の次に195003となっている列の和を知りたいです。
そして、もしその195001の次に195003となっているのを計算し終えたら今度は195001の次に195005となっているものを、そして、XDRの値は195161まであるのでそれを全部出し終えたら、今度は195002の次が195003のものをというような感じで、それぞれの出力された結果がいくつあるかを知りたいです。==== 195001 --> 195003 ===== 1262041F20124 195001 182139 1262041F20124 195003 181347 count = 2 ==== 195001 --> 195013 ===== 126223AE23135 195001 0750073 126223AE23135 195013 0842509 count = 2というような出力にしたいです。
このプログラムのどこが間違っていますでしょうか?データ<-read.csv("データ/data.csv",header=TRUE) foo <- function(m, k) { count <- 0 ### 追加 件数をカウントする n <- nrow(データ) i <- 1 repeat { if (データ$CAD[i] == データ$CAD[i+1] && データ$XDR[i] == m && データ$XDR[i+1] == k) { if (count == 0) { ### 追加 該当があるときのみ先頭に出力 cat(sprintf("==== %i --> %i =====\n", m, k)) ### 追加 } ### 追加 cat(データ$CAD[i], データ$XDR[i], データ$ASD[i] "\n") cat(データ$CAD[i+1], データ$XDR[i+1], データ$ASD[i+1] "\n") count <- count+2 ### 追加 i <- i+1 } i <- i+1 if (i >= n) break } if (count != 0) { ### 追加 カウント数を出力 cat(sprintf("count = %i\n", count)) ### 追加 } ### 追加 } データ <- read.csv("データ/data.csv", as.is=TRUE) for (m in 195001:195161) { for (k in (m-1):1950162) { foo(m, k) # 関数を呼び出す } }
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべし。
「読んだけどわかんね〜よ」ということでしょうか。
」 を読むべし。
「読んだけどわかんね〜よ」ということでしょうか。
整形済みテキスト
行頭が半角空白で始まる行は整形済みテキストとなります。行の自動折り返しは行なわれません。
整形済みテキストは、他のブロック要素の子要素になることができます。
整形済みテキストは、他のブロック要素を子要素にすることができません。
整形済みテキストは、すべての子要素を文字列として扱います。
というところですよ。あなたの記事は私が修正しているので,投稿時とどこが違うかよく見てください。+ cat(データ$CAD[i], データ$XDR[i], データ$ASD[i] "\n")
エラー: 予想外の 文字列定数 です in:
" } ### 追加
cat(データ$CAD[i], データ$XDR[i], データ$ASD[i] "\n""
というのは,データ$ASD[i] "\n" のところにエラーがある。つまり,データ$ASD[i] の次にいきなり文字列 "\n" が出てきたよ!ということなので,カンマで区切りなさいということですよ。この次の行も。
cat(データ$CAD[i], データ$XDR[i], データ$ASD[i], "\n")それをなおせば,動くでしょう? -- 河童の屁は,河童にあらず,屁である。 2013-11-22 (金) 13:05:18
KK (2013-11-22 (金) 11:41:08)
はじめまして、macでRを使い始めて四苦八苦してます
ロジスティック解析を行う際、glm関数を使うとオッズ比まで出すことができるのですが、各説明変数の95%C.Iまで出すには、lrm関数を使うとよいと聞きました。しかしデフォルトのmac版Rでは、lrmがインストールされておらず、Designパッケージをインストールしたら良いとのところまで辿り着いたのですが、Designパッケージが見つからず困っております。大変申し訳ありませんがインストール方法を教えて下さい。
またロジスティック解析にて、各説明変数の95%C.Iを出すことが目的なので、他の方法で解決できるのであれば教えてください。お願いします。
> m.s <- summary(m) > exp(m.s$coefficients[2, 1] + c(-1, 1) * 1.96 * m.s$coefficients[2, 2])2つめは、
> exp(m.s$coefficients[3, 1] + c(-1, 1) * 1.96 * m.s$coefficients[3, 2])です。もちろん、confint()を使ってもよいです。
YH (2013-11-19 (火) 01:08:49)
はじめまして。表題の件がうまくいかなくてこちらで質問させてください。
「windows環境」で文字をja_JP.UTF-8にすることは可能でしょうか。
Sys.setlocale("LC_CTYPE", "ja_JP.UTF-8")で設定しようとすると下記のエラーがでてしまいます。> Sys.setlocale("LC_CTYPE", "ja_JP.UTF-8.") [1] "" 警告メッセージ: In Sys.setlocale("LC_CTYPE", "ja_JP.UTF-8.") : ロケールを "ja_JP.UTF-8." に設定せよとの OS のレポート要求は受け入れられません関連する情報を探していると、三年ほど前の記事や、一年ほど前の書き込みで
http://m884.hateblo.jp/entry/20100920/1284950844
http://like2ch.com/ag/uni/math/1294561909/500
が見つかりましたが、うまくいってないようです。
また、windowsの標準の文字など他の一部の文字コードでの設定ではうまくいくようです。((Sys.setlocale("LC_CTYPE","Japanese_Japan.20932")あるいは、Sys.setlocale("LC_CTYPE", "Japanese_Japan.932")など)
しかし、"ja_JP.UTF-8"を指定するとうまくいきませんでした。
なにか、解決策がありましたらご教示頂けると幸いです。
よろしくおねがいします。> sessionInfo() R version 3.0.2 (2013-09-25) Platform: i386-w64-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932
Montecarlo (2013-11-18 (月) 01:08:24)
いつもお世話になっております。
今回題名の通り Package(parallel) においてクラスタの生成に失敗したときをご教授いただきたいと思っています。
なお、 sessionInfo は以下の通りです。> sessionInfo() R version 3.0.2 (2013-09-25) Platform: x86_64-w64-mingw32/x64 (64-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base本題に入りますが、Package(parallel)において[makePSOCKcluster]を実行した際に、オブジェクトに格納し忘れると、その後並列関数に代入する[cluster object]が取得できず、再度[makePSOCKcluster]を実行すると、以下のエラーが出ますが、特段問題ないのでしょうか。
> library(parallel) > makePSOCKcluster(4) socket cluster with 4 nodes on host ‘localhost’ > cl <- makePSOCKcluster(4) .... 警告メッセージ: 1: 使われていないコネクション 6 (<-hogehoge) を閉じます 2: 使われていないコネクション 5 (<-hogehoge) を閉じます 3: 使われていないコネクション 4 (<-hogehoge) を閉じます 4: 使われていないコネクション 3 (<-hogehoge) を閉じます使われていないコネクションが警告によって閉じられているように見られますので、問題ないとは考えていますが、実際の所どうなのでしょうか。
詳しい方のお知恵を拝借したいと思っています。
どうぞよろしくお願い致します。
谷川 (2013-11-14 (木) 16:04:31)
R初心者です。よろしくお願いします。
VB.NETからStatconnDCOMを使用して、決定木のアルゴリズムを使用したいと考えています。
以下のようなコードを組んでおります。Dim lConnector As IStatConnector lConnector = New STATCONNECTORSRVLib.StatConnector lConnector.Init("R") lConnector.EvaluateNoReturn("library(mvpart);") lConnector.EvaluateNoReturn("Tdata <- read.csv(""C:\\T-DATA.csv"", header=T);") lConnector.EvaluateNoReturn("tree <- rpart(生死~等級+大人子ども+性別, data=Tdata, method=""class"");") lConnector.Evaluate("print(tree);") ・・・ (A)上記の(A)の行の出力をVB.NETで加工したいと考えておりますが、以下のメッセージで実行時エラーとなります。
『この接続 ID に対する接続がありません (HRESULT からの例外: 0x80040004 (OLE_E_NOCONNECTION))』
当方の学習不足なのですが、Evaluateメソッドの戻り値を取得する方法を教えていただきたく思います。
なお、環境は以下の通りです。VB.NET:2010 R:3.02(使用ライブラリ mvpart statconnDCOM:3.5-1B2以上よろしくお願いいたします。
RStudio初心者 (2013-11-12 (火) 09:48:09)
RStudioにてMessage Translationsが反映されません。当方の環境2種で試してみました。Windows Vista R version 3.0.1, R version 3.0.2 (両方で試しました) RStudio 0.97.551 - Windows XP/Vista/7/8 (2013-05-11) Windows 7 R version 3.0.1, R version 3.0.2 (両方で試しました) RStudio 0.97.551 - Windows XP/Vista/7/8 (2013-05-11)まず、R version 3.0.1をインストール、その際Message Translationsにチェック。起動時のConsoleは以下です。
R version 3.0.1 (2013-05-16) -- "Good Sport" Copyright (C) 2013 The R Foundation for Statistical Computing Platform: i386-w64-mingw32/i386 (32-bit) R は、自由なソフトウェアであり、「完全に無保証」です。 一定の条件に従えば、自由にこれを再配布することができます。 配布条件の詳細に関しては、'license()' あるいは 'licence()' と入力してください。 R は多くの貢献者による共同プロジェクトです。 詳しくは 'contributors()' と入力してください。 また、R や R のパッケージを出版物で引用する際の形式については 'citation()' と入力してください。 'demo()' と入力すればデモをみることができます。 'help()' とすればオンラインヘルプが出ます。 'help.start()' で HTML ブラウザによるヘルプがみられます。 'q()' と入力すれば R を終了します。次に、RStudio 0.97.551をインストール。起動時のConsoleは以下です。RStudio上ではエラーメッセージ等も日本語化されていません。
R version 3.0.1 (2013-05-16) -- "Frisbee Sailing" Copyright (C) 2013 The R Foundation for Statistical Computing Platform: i386-w64-mingw32/i386 (32-bit) R is free software and comes with ABSOLUTELY NO WARRANTY. You are welcome to redistribute it under certain conditions. Type 'license()' or 'licence()' for distribution details. R is a collaborative project with many contributors. Type 'contributors()' for more information and 'citation()' on how to cite R or R packages in publications. Type 'demo()' for some demos, 'help()' for on-line help, or 'help.start()' for an HTML browser interface to help. Type 'q()' to quit R.R version 3.0.2で試しても上手くいかず、OSを Windows7に変えて上記を試しても上手くいきません。
RStudio上でMessage Translationsを反映する方法をご存知の方は、どうかご教示いただけますと助かります。どうかよろしくお願い致します。
Montecarlo (2013-11-11 (月) 22:16:38)
立て続けに投稿して申し訳ないのですが、以前「全てのオブジェクトとその値を表示する関数」で質問させていただいたMonteCalroと申します。
何度も試したのですが、原因がつかめないので皆様のお知恵を拝借したく投稿します。
セッションインフォは以下の通りです。> sessionInfo() R version 2.15.2 (2012-10-26) Platform: x86_64-w64-mingw32/x64 (64-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base以前ヒントを頂いたので、以下の関数を新たに組んでみました。
ls.val <- function() { ls.all <- .Internal(ls(as.environment(-1L), FALSE)) ls.print <- ls.all[ls.all != "ls.val"] sapply(ls.print, function(temp) get(temp)) }グローバル環境では問題なく意図とした動作をします。
> a <- c(1:20); b <- c(5:8); c <- letters[3:12] > ls.val() $a [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 $b [1] 5 6 7 8 $c [1] "c" "d" "e" "f" "g" "h" "i" "j" "k" "l"この関数を、関数内でデバッグに使いたいと思いましたが、エラーが出て困っています。
f <- function() { #テスト用関数 x <- c(10:5) y <- c(3:9) z <- LETTERS[25:15] browser() } > f() Called from: f() Browse[1]> ls.val() 以下にエラー get(temp) : オブジェクト 'x' がありません Browse[1]> ls() [1] "x" "y" "z" Browse[1]> ls.str() x : int [1:6] 10 9 8 7 6 5 y : int [1:7] 3 4 5 6 7 8 9 z : chr [1:11] "Y" "X" "W" "V" "U" "T" "S" "R" "Q" "P" "O" Browse[1]> sapply(ls(), function(temp) get(temp)) $x [1] 10 9 8 7 6 5 $y [1] 3 4 5 6 7 8 9 $z [1] "Y" "X" "W" "V" "U" "T" "S" "R" "Q" "P" "O" Browse[1]> c関数「f」の中には、確かにx, y, z の3個のオブジェクトがあります。
かつ、[ls()] [ls.str()] は問題なく動作します。
そして以前教えていただいた [sapply(ls(), function(temp) get(temp)]も問題なく動作します。
なぜ、[ls.val()] は動作せず、エラーが出るのかを教えていただきたいと思います。
どうぞよろしくお願いいたします。
ls.val <- function() {
pe <- as.environment(-1)
ls.all <- ls(envir=pe)
ls.print <- ls.all[ls.all != "ls.val"]
sapply(ls.print, function(temp) get(temp, envir=pe))
}suho (2013-11-05 (火) 20:54:18)
潜在クラスモデルでクラスタ分析を行いたいデータがあります。
poLCA、Flexmix packageのマニュアルを読んだのですが、わからなかったのでお教えいただけないでしょうか。
なお、環境は以下の通りです。> sessionInfo() R version 3.0.2 (2013-09-25) Platform: i386-w64-mingw32/i386 (32-bit)扱いたいデータは、3000人にアンケートを行い、ある商品の選択理由を、20項目中から5項目を回答してもらったデータです。
ただし、回答してもらった5項目は、「最も強い選択理由(1番目)」〜「5番目の選択理由」というように、順番があります。
生データの状態は以下です(サンプルですが)[ID] [1st] [2nd] [3rd] [4th] [5th] 0001 5 8 4 19 10 0002 13 2 15 3 19 ・・・このような順序カテゴリーデータで、潜在クラスモデルを行えるRパッケージはあるのでしょうか?
poLCA、Flexmixのマニュアルで見つけられず。。。。(もし読み落としていたら申し訳ありません)
ご教示いただけますと大変ありがたいです。よろしくお願い致します。
Montecarlo (2013-11-03 (日) 18:26:50)
今回このWikiを検索しても分からなかったので、皆さんのお知恵をお貸し頂きたいと思い投稿します。
セッションインフォは以下の通りです。> sessionInfo() R version 2.15.2 (2012-10-26) Platform: x86_64-w64-mingw32/x64 (64-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods baseさて、質問内容ですが、現在の環境下のすべてのオブジェクトを表示する関数を書くことはできますか?
(グローバル環境で実行すると、その環境内にあるオブジェクト。関数のデバッグ中ではその関数内のオブジェクト)
具体例では、オブジェクトを以下のように代入します。val.a <- c(1, 2, 3); val.b <- c(4, 5, 6); val.c <- c(7, 8, 9) chr.x <- c("x", "y", "z"); chr.y <- c("Taro", "Hanako", "Jiro")その際に、ある関数hogeを実行すると以下のような答えが返ってきてほしいと思っています。
(list <- list(val.a = val.a, val.b = val.b, val.c =val.c, chr.x = chr.x, chr.y = chr.y)) $val.a [1] 1 2 3 $val.b [1] 4 5 6 $val.c [1] 7 8 9 $chr.x [1] "x" "y" "z" $chr.y [1] "Taro" "Hanako" "Jiro"どうぞよろしくお願いいたします。
> val.a <- c(1, 2, 3); val.b <- c(4, 5, 6); val.c <- c(7, 8, 9)
> chr.x <- c("x", "y", "z"); chr.y <- c("Taro", "Hanako", "Jiro")
> sapply(ls(), function(x) eval(parse(text=x)))
chr.x chr.y val.a val.b val.c
[1,] "x" "Taro" "1" "4" "7"
[2,] "y" "Hanako" "2" "5" "8"
[3,] "z" "Jiro" "3" "6" "9"
> val.a <- 1:5; val.b <- 10:19; val.c <- 100:103
> chr.x <- letters[1:7]; chr.y <- c("Taro", "Hanako", "Jiro")
> sapply(ls(), function(x) eval(parse(text=x)))
$chr.x
[1] "a" "b" "c" "d" "e" "f" "g"
$chr.y
[1] "Taro" "Hanako" "Jiro"
$val.a
[1] 1 2 3 4 5
$val.b
[1] 10 11 12 13 14 15 16 17 18 19
$val.c
[1] 100 101 102 103> ls.str() chr.x : chr [1:3] "x" "y" "z" chr.y : chr [1:3] "Taro" "Hanako" "Jiro" val.a : num [1:3] 1 2 3 val.b : num [1:3] 4 5 6 val.c : num [1:3] 7 8 9
hoge <- function(x) {
sapply(x, function(temp) get(temp))
}
hoge(ls())掃除好き (2013-11-01 (金) 15:25:53)
先ほども質問させてもらったのですが、また少し困ったことが起こりました。先ほどのプログラム自体は上手くまわったのですが、データ量が多いため、出力結果が非常に見難いため、求めようとしている結果が見つけるのにかなり時間を要することが分かりました。
そこで質問なのですが、先ほどのプログラムを使用して、195001→195002から195001→195003へと変わる際で区切りなどを入れてそれぞれがどの程度あるのか分かりやすくしたいです。区切りを入れるまたは195001→195002、195001→195003などそれぞれ出てきた結果を列としてとらえてそれらの列がいくつあるかを和として出力する方法はありますでしょうか?0E85051620170,195001 0E85051620170,195003 [1] 2 09690FD520157,195001 09690FD520157,195005 09692DF920168,195001 09692DF920168,195005 [2] 4 0CD30E8120165,195001 0CD30E8120165,195008 [3] 2 10491ADC20156,195001 10491ADC20156,195009 0E85080B20150,195001 0E85080B29150,195009 [4] 4といったような出力にしたいです。
そのようなやり方があるのであれば、教えていただけないでしょうか?
foo <- function(m, k) {
count <- 0 ### 追加 件数をカウントする
n <- nrow(ファイルデータ)
i <- 1
repeat {
if (ファイルデータ$CAR[i] == ファイルデータ$CAR[i+1] &&
ファイルデータ$SHA[i] == m &&
ファイルデータ$SHA[i+1] == k) {
if (count == 0) { ### 追加 該当があるときのみ先頭に出力
cat(sprintf("==== %i --> %i =====\n", m, k)) ### 追加
} ### 追加
cat(ファイルデータ$CAR[i], ファイルデータ$SHA[i], "\n")
cat(ファイルデータ$CAR[i+1], ファイルデータ$SHA[i+1], "\n")
count <- count+2 ### 追加
i <- i+1
}
i <- i+1
if (i >= n) break
}
if (count != 0) { ### 追加 カウント数を出力
cat(sprintf("count = %i\n", count)) ### 追加
} ### 追加
}
プログラムやデータなどを提示する方法について,「 ![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべし。ということについても,努力していないようですね。 -- 河童の屁は,河童にあらず,屁である。 2013-11-01 (金) 17:29:44
」 を読むべし。ということについても,努力していないようですね。 -- 河童の屁は,河童にあらず,屁である。 2013-11-01 (金) 17:29:44鬼の目にも涙 (2013-11-01 (金) 11:00:12)
出力結果をまた別のファイルに保存したいです。x y 200 300 300 400というような結果を別の新たなファイルに保存する方法はありますでしょうか?プログラムを組めばよいのでしょうか?
sink("ファイル名")
:
sink()
のようにすれば,sink("ファイル名") 以降 sink() までに,コンソールに出力されるであろうものが「ファイル名」とう名前を持つファイルに書き出されます。sink() を入れるまではファイルに出力され続けるので注意。![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべし。 -- 河童の屁は,河童にあらず,屁である。 2013-11-01 (金) 11:02:06
」 を読むべし。 -- 河童の屁は,河童にあらず,屁である。 2013-11-01 (金) 11:02:06河童の屁は,河童にあらず,屁である。 (2013-10-31 (木) 18:32:41)
# 危険!実行するな!無限ループ foo <- function() { i <- 1 repeat { i <- i+1 if (i >= 10) return } } foo() # () を付ければ問題ない foo2 <- function() { i <- 1 repeat { i <- i+1 if (i >= 10) return() } } foo2() # for なら問題ない foo3 <- function() { i <- 0 for (j in 1:100) { if (i >= 10) return } } foo3() # 不当なエラーメッセージが出る状況 a <- 1:20 foo4 <- function() { i <- 1 repeat { i <- i+1 if (a[i] >= 10) return # () を付けると無問題 } } foo4()のようなことに気づいた。
これは,Version 0.49 Beta (April 23, 1997) から(!!)今まで,ず〜〜っと同じである。ので,バグなどではない,のである
のざわ (2013-10-30 (水) 13:01:56)
http://cran.md.tsukuba.ac.jp/bin/windows/base/
が昨日から見えません。対応をお願いします。
掃除好き (2013-10-30 (水) 08:02:28)
何度もすみません。CAR,REC,TRW,DTM,SHA,KENSYU,ZIP,BIT 1262021A20150,0,800,20091019063645,195001,111,7600020,1950 0E85051620170,0,800,20091019063800,195001,112,, 0E85036820151,0,800,20091019063939,195003,111,7110931,1965 0969118E20153,0,800,20091019063942,195005,111,7630071,1958 09690FD520157,0,800,20091019063943,195001,111,7620006,1951 09692DF920168,0,800,20091019064203,195007,111,7618021,1955 1049204A20168,0,800,20091019064217,195001,111,7618013,1945 104920F920166,1,800,20091019064832,195005,111,7600001,1976 0CD30E8120165,1,800,20091019070210,195001,111,, 0CD305AE20156,0,800,20091019070223,195003,111,7620021,1949 10491ADC20156,0,800,20091019070537,195001,112,7690210,1993 0CD3028120168,0,800,20091019070716,195007,111,7600013,1947 0E85080B20150,0,800,20091019070830,195001,111,, 1049209920170,0,800,20091019071029,195009,111,7620046,1965 0E850FCF20170,0,800,20091019071125,195001,112,7618004,1991 10490C5F20148,0,800,20091019071135,195004,111,7000965,1976 0AFD0A5620166,0,800,20091019071523,195001,111,7600080,1944 1049214720169,0,800,20091019071906,195005,111,7618026,1980 10491BEC20152,0,800,20091019071935,195001,111,, 10490FE120148,0,800,20091019072112,195007,111,7630091,1962 ・ ・過去に質問した内容でプログラムを組んでみたのですが、上手くまわりません。このプログラムはどこが間違っていますでしょうか?教えていただければ幸いです。
foo <- function(m, k) for (m in 195001:195059) { for (k in (m+1):195060) { function(m, k) # 関数を呼び出す } n <- nrow(ファイルデータ) i <- 1 repeat { if (ファイルデータ$CAR[i] == ファイルデータ$CAR[i+1] && ファイルデータ$SHA[i] == m && ファイルデータ$SHA[i+1] == k) { cat(ファイルデータ$CAR[i], ファイルデータ$CAR[i], "\n") cat(ファイルデータ$CAR[i+1], ファイルデータ$CAR[i+1], "\n") i <- i+1 } i <- i+1 if (i >= n) return }
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべし。 -- 河童の屁は,河童にあらず,屁である。 2013-10-30 (水) 09:28:07
」 を読むべし。 -- 河童の屁は,河童にあらず,屁である。 2013-10-30 (水) 09:28:07
foo <- function(m, k) {
n <- nrow(ファイルデータ)
i <- 1
repeat {
if (ファイルデータ$CAR[i] == ファイルデータ$CAR[i+1] &&
ファイルデータ$SHA[i] == m &&
ファイルデータ$SHA[i+1] == k) {
cat(ファイルデータ$CAR[i], ファイルデータ$CAR[i], "\n")
cat(ファイルデータ$CAR[i+1], ファイルデータ$CAR[i+1], "\n")
i <- i+1
}
i <- i+1
if (i >= n) return
}
}
ファイルデータ <- read.csv("data.csv", as.is=TRUE)
for (m in 195001:195059) {
for (k in (m+1):195060) {
foo(m, k) # 関数を呼び出す
}
}スケルトン (2013-10-28 (月) 19:08:26)
Rのソフトをダウンロードしたのですが、内容全てが英語です。日本語のソフトか切替方法などはありませんでしょうか?
naca (2013-10-25 (金) 22:01:34)
下のような文字列xがあります。
xから"<>"とそれに囲まれた任意の文字列(ここでは<aaa>, <bb>, <cc 123 %>)を除いた文字列(ここでは「成功」)を抽出したいのですが、どのようなxでも対応できるような方法があればご教授いただけないでしょうか。
なお、<xxxx>のパターンは複数個あり、任意の文字列です。
よろしくお願いします。x <- "<aaa><bb>成功<cc 123 %>" gsub("<aaa>", "", gsub("<bb>", "", gsub("<cc 123 %>", "", x))) #さまざまなxに対応させたい。
func <- function(str) {
str <- unlist(strsplit(str, ""))
out <- TRUE
select <- logical(length(str))
for (i in seq_along(str)) {
if (str[i] == "<") {
out <- FALSE
select[i] <- FALSE
} else if (str[i] == ">") {
out <- TRUE
select[i] <- FALSE
} else {
select[i] <- out
}
}
paste(str[select], collapse="")
}
> x <- "<aaa><bb>成功<cc 123 %>"
> func(x)
[1] "成功"
> func("<html><head><title>タイトル</title></head><body>本体<br>文章</body></html>")
[1] "タイトル本体文章"func <- function(x) {
matched <- gregexpr("<[^>]*>", x)[[1]]
if (matched[1] == -1) {
return(x)
}
l <- attributes(matched)$match.length
# ↓手抜き。matchedの長さだけsubstrで切ったほうが良いと思います。
n <- unlist(mapply(FUN=seq, matched, matched + l - 1))
paste(strsplit(x, "")[[1]][-n], collapse="")
}掃除好き (2013-10-23 (水) 10:04:40)
CAR,REC,TRW,DTM,SHA,KENSYU,ZIP,BIT 1262021A20150,0,800,20091019063645,195001,111,7600020,1950 0E85051620170,0,800,20091019063800,195001,112,, 0E85036820151,0,800,20091019063939,195003,111,7110931,1965 0969118E20153,0,800,20091019063942,195005,111,7630071,1958 09690FD520157,0,800,20091019063943,195001,111,7620006,1951 09692DF920168,0,800,20091019064203,195007,111,7618021,1955 1049204A20168,0,800,20091019064217,195001,111,7618013,1945 104920F920166,1,800,20091019064832,195005,111,7600001,1976 0CD30E8120165,1,800,20091019070210,195001,111,, 0CD305AE20156,0,800,20091019070223,195003,111,7620021,1949 10491ADC20156,0,800,20091019070537,195001,112,7690210,1993 0CD3028120168,0,800,20091019070716,195007,111,7600013,1947 0E85080B20150,0,800,20091019070830,195001,111,, 1049209920170,0,800,20091019071029,195009,111,7620046,1965 0E850FCF20170,0,800,20091019071125,195001,112,7618004,1991 10490C5F20148,0,800,20091019071135,195004,111,7000965,1976 0AFD0A5620166,0,800,20091019071523,195001,111,7600080,1944 1049214720169,0,800,20091019071906,195005,111,7618026,1980 10491BEC20152,0,800,20091019071935,195001,111,, 10490FE120148,0,800,20091019072112,195007,111,7630091,1962 ・ ・上記のようなデータを読み込んだのですが、ここからCARの値が2つ続けて同じで、かつSHAの値が195001の次に195003となっている列の和を知りたいです。
そして、もしその195001の次に195002となっているのを計算し終えたら今度は195001の次に195005となっているものを、そして、SHAの値は195060まであるのでそれを全部出し終えたら、今度は195002の次が195003のものをというような感じで、それぞれの出力された結果がいくつあるかを知りたいです。
そのようなプログラムはありますでしょうか?
if文などを使えば良いような気がしますが、私自身プログラムに疎いのでRの練習がてらに少し難しいプログラムを学んでみたいので、分かります方は回答をお願いします。
for (i in 195001:195059) {
for (j in (i+1):195060) {
func(i, j) # 関数を呼び出す
}
}
しかしまあ,似たような質問があるもんだなあ。投稿方法について,「投稿における注意事項」と「 ![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべきであるというところまでそっくりだ。同じグループの質問者なんだろうか。 -- 河童の屁は,河童にあらず,屁である。 2013-10-23 (水) 10:24:35
」 を読むべきであるというところまでそっくりだ。同じグループの質問者なんだろうか。 -- 河童の屁は,河童にあらず,屁である。 2013-10-23 (水) 10:24:35yataro (2013-10-22 (火) 16:32:55)
Linux(Ubuntu)でRstudioを使ってRを使用しています。
使用環境は以下の通りです。sessionInfo() R version 3.0.2 (2013-09-25) Platform: x86_64-pc-linux-gnu (64-bit) locale: [1] LC_CTYPE=ja_JP.UTF-8 LC_NUMERIC=C LC_TIME=ja_JP.UTF-8 [4] LC_COLLATE=ja_JP.UTF-8 LC_MONETARY=ja_JP.UTF-8 LC_MESSAGES=ja_JP.UTF-8 [7] LC_PAPER=ja_JP.UTF-8 LC_NAME=C LC_ADDRESS=C [10] LC_TELEPHONE=C LC_MEASUREMENT=ja_JP.UTF-8 LC_IDENTIFICATION=C attached base packages: [1] stats graphics grDevices utils datasets methods base other attached packages: [1] fishmethods_1.5-0 MASS_7.3-29 foreign_0.8-56 boot_1.3-9 loaded via a namespace (and not attached): [1] tools_3.0.2ここで、以下のエラーが出てパッケージをインストールできません。
install.packages("truncnorm") Installing package into ‘/home/seishiro/R/x86_64-pc-linux-gnu-library/3.0’ (as ‘lib’ is unspecified) URL 'http://cran.rstudio.com/src/contrib/truncnorm_1.0-6.tar.gz' を試しています Content type 'application/x-gzip' length 9686 bytes 開かれた URL ================================================== downloaded 9686 bytes 以下にエラー setwd(od) : 作業ディレクトリを変更できません 以下にエラー setwd(startdir) : 作業ディレクトリを変更できません 実行が停止されました Warning in install.packages : installation of package ‘truncnorm’ had non-zero exit status The downloaded source packages are in ‘/tmp/Rtmpj8OYYl/downloaded_packages’ちなみにこのエラーは、端末から何らかの.rファイルを rstudio test.r として開いたときに起こり、rstudio のみを入力して新規に開いた場合にはこのエラーが起こることなくパッケージをインストール出来ます
先に新規で開いて、そのあとに.rファイルを開けば問題はないのですが、Projectを作成したときは毎度、別に新規で開くのは効率が悪いので、解決案をご教授いただければ幸いです。
> install.packages("truncnorm")
Installing package into ‘/home/xxxx/R/x86_64-pc-linux-gnu-library/3.0’
(as ‘lib’ is unspecified)
[snip]
gcc -std=gnu99 -I/usr/share/R/include -DNDEBUG
-fpic -O3 -pipe -g -c rtruncnorm.c -o rtruncnorm.o
[snip]
* DONE (truncnorm)Saito (2013-10-19 (土) 15:49:18)
いつもお世話になっております。
ネットや過去ログを調べましたが、見つからなかったので質問させてください。
座標と座標ごとに値が得られている場合に、それを3次元棒グラフで表示することは可能でしょうか。
さらに、その3次元棒グラフを近似するなんらかの関数を当てはめて、3次元棒グラフに重ね書きし、なめらかな曲面として表示することは可能でしょうか。
どなたか分かる方がいましたら、ご教授ください。
3次元棒グラフの書き方だけでも助かります。
どうぞよろしくお願い致します。
RSiteSearch("3d barplot")
で調べたら、vrmlgenパッケージのbar3d()とかscatterplot3dパッケージの例などがヒットしますよ。重ね書きは座標を計算してpoints()やtext()で書き込めば良いです。関数の当てはめと滑らかな曲面ももちろん可能ですが、私の知る限りは自分で手順を書いていく必要があります。スプライン関数を使うにしろ、Krigingで平滑化するにしろ、空間統計学の知識とRのコーディング知識が必要です。3次元を2次元に投影する変換関数の知識も必要です。詳しくは、間瀬茂先生の地球統計学とクリギング法RとgeoRによるデータ解析など、専門書を当たって下さい。 -- 2013-10-20 (日) 13:38:15データサイエンス (2013-10-17 (木) 14:08:14)
何度もすみません。今扱っているデータが膨大なので、プログラムが回せても結果がほとんど無視されてしまいます。無視された場合には、その結果を別のファイルに保存や、そのまま下に続けて別のプログラムに起用することは出来ないのでしょうか?
データサイエンス (2013-10-17 (木) 10:32:48)
もし、英語と数字が混ざっているような値をif文にかけるにはどうすればよろしいのでしょうか?
ZAW3
DE6A
IK9U
S1S3
というような英語と数字が混ざっているような値が下にずっと続いているのですが、例えばIK9Uだけを出力するといったような方法はあるのでしょうか?if文でやるのかなと思っているのですが、数字だけとは違うのでi+1といったことでは出来ないので困っています。
> a [1] "ZAW3" "DE6A" "IK9U" "S1S3" > a[a == "IK9U"] [1] "IK9U"「i+1といったことでは出来ない」とはどういうことでしょうね? -- 河童の屁は,河童にあらず,屁である。 2013-10-17 (木) 11:56:16
> grep("IK9U", a, value = TRUE)
[1] "IK9U"サッポロポテト (2013-10-17 (木) 01:44:42)
aa ab ac 126 800 19507 4AD 900 19501 4AD 800 19358 J89 900 19356 J89 901 19358 MK5 900 19538 SWK 911 19333 256 800 15689 ・ ・前回と同じような質問なのですが、データをファイルから出力したあと、このデータのabの列の901と911の行とその上の行以外を下記のように出力したいです。
126 800 19507 4AD 900 19501 4AD 800 19358 256 800 15689これはこのようなプログラムで本当に合っていますでしょうか?
b <- a$ab == 901&911 a[b | c(b[-1], TRUE),]
b <- a$ab == 901 | a$ab == 911 a[!(b | c(b[-1], FALSE)),]なぜそうしなくてはならないのかは,R の教科書などを参考にして考えてみてください。 -- 河童の屁は,河童にあらず,屁である。 2013-10-17 (木) 11:48:45
変体観測 (2013-10-14 (月) 14:11:53)
初めての投稿でまだ分からない部分もありますが質問させていただきます。「投稿における注意事項」等を読んで、自分で考えてはみたのですが、全くRについての知識がなく、どれも回すことが出来ませんでした。それでも回答していただけますならお願いします。x y 5555 358 5555 191 2951 358 2895 191 3515 358 3515 191 3515 193このようなデータをcsvテキストから読み込んだのですが、このデータのxの値が2つ続けて同じで、かつyの値が358の次に191となっている部分だけを出力したいです。
5555 358 5555 191 3515 358 3515 191このような形で出力するのはどうすればよろしいのでしょうか?
f1 <- function(a) {
n <- nrow(a)
b2 <- a$y == 191
b1 <- a$y == 358 & c(b2[-1], FALSE)
b1 <- b1 | c(FALSE, b1[-n])
c1 <- a$x == c(a$x[-1], FALSE)
c1 <- c1 | c(FALSE, c1[-n])
return(a[b1 & c1,])
}
しかしまあ,おまじないみたいになってしまうので,以下のように素直に書く方がよいでしょうね。
f2 <- function(a) {
n <- nrow(a)
i <- 1
repeat {
if (a$x[i] == a$x[i+1] && a$y[i] == 358 && a$y[i+1] == 191) {
cat(a$x[i], a$y[i], "\n")
cat(a$x[i+1], a$y[i+1], "\n")
i <- i+1
}
i <- i+1
if (i >= n) return
}
}
なお,もとのデータの記述で2列目の変数名が z になっていたのは y の単純な間違いと思うので,それを前提としてプログラムしました。 -- 河童の屁は,河童にあらず,屁である。 2013-10-14 (月) 15:02:55f2.2 <- function(a) {
n <- nrow(a)
ans <- logical(n)
i <- 1
repeat {
if (a$x[i] == a$x[i+1] && a$y[i] == 358 && a$y[i+1] == 191) {
ans[i] <- ans[i+1] <- TRUE
i <- i+1
}
i <- i+1
if (i >= n) return(a[ans,])
}
}
遅いのはいたしかたない。-- 2013-10-15 (火) 16:14:19f1.2 <- function(a) {
n <- nrow(a)
s1 <- 1:(n-1)
s2 <- 2:n
flag <- a$x[s1] == a$x[s2] & a$y[s1] == 358 & a$y[s2] == 191
return(a[c(flag, FALSE) | c(FALSE, flag),])
}
ほんのわずかだけど。f1.3 <- function(a) {
n <- nrow(a)
flag <- (a$x[1:(n-1)] == a$x[2:n]) * (a$y[1:(n-1)] == 358) * (a$y[2:n] == 191)
return(a[c(flag, FALSE) | c(FALSE, flag),])
}
& を * でやるなど,こそくな手段を使う。微々たる違いだが。 -- 2013-10-15 (火) 17:10:47> j <- (1:(nrow(a)-1))[diff(a$x) == 0] # j=c(1,5,6)
> k <- j[a$y[j]==358 & a$y[j+1]==191] # k=c(1,5)
> a[sort(c(k,k+1)),] # sort(c(k,k+1))=c(1,2,5,6)
x y
1 5555 358
2 5555 191
5 3515 358
6 3515 191まりも (2013-10-12 (土) 00:01:58)
残差分析(カイ2乗検定をした上で、さらに個別の項目のどこに差があるのかを探索するための)の手法と図示についてご教示ください。
残差には、標準化残差と、調整済み標準化残差とがあり、Chisq.test関数が返す $residuals と $stdres がそれぞれ対応していることを理解しました。
ネットを検索すると、調整済み標準化残差の値を標準正規分布のzスコアとしてp値を求める方法を紹介しているサイトが多く見つかります。
http://note.chiebukuro.yahoo.co.jp/detail/n71838
一方、観測値が期待値に対して有意差がある項目をわかりやすく図示する方法として、mosaicplot(data,shade=TRUE) や library(vcd) の mosaic(data, shade=TRUE) が頻繁に紹介されていますが、ここでは(調整済みでない)標準化残差が用いられています。
どちらをzスコアに用いるかは、あまり問題にならないのでしょうか。もし問題になるとすれば、調整済み標準化残差に基づいてモザイク図を塗ることはできるのでしょうか。
サッポロポテト (2013-10-11 (金) 15:32:53)
aa ab ac 126 800 19507 4AD 800 19501 4AD 800 19358 J89 800 19356 J89 900 19358 MK5 900 19538 SWK 900 19333 ・ ・このようなデータをファイルから出力したのですが、このデータのacの列の19358の行とその上の行だけを下記のように出力したいです。
4AD 800 19501 4AD 800 19358 J89 800 19356 J89 900 19358そのようなプログラムを組みたいのですが、初心者なので全く分かりません。
誰か教えていただきますでしょうか?
あとこれがいくつも下に続いていくので、その19358とその上の行がデータ内にいくつあるのかが分かるプログラムも教えていただければ光栄です。
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべきです。
」 を読むべきです。> b <- a$ac == 19358 > a[b | c(b[-1], FALSE),] aa ab ac 2 4AD 800 19501 3 4AD 800 19358 4 J89 800 19356 5 J89 900 19358このような行の組が何組あるかは,
> sum(b) [1] 2でよいのではないですか? -- 河童の屁は,河童にあらず,屁である。 2013-10-11 (金) 16:02:37
棒グラフ (2013-10-06 (日) 09:39:18)
エクセルのように縦棒の積み重ね棒グラフ自身に名前を埋め込むことはできるのですが,横棒の縦棒の積み重ね棒グラフにしたときにそれができません。ご教示いただけないでしょうか?
> fruits <- c(20, 30, 50)
> par(las=1)
> par(mgp=c(2, 0.8, 0))
> t = barplot(cbind(fruits), names.arg=c("fruits"))
> text(t[1],
+ c(fruits[1]/2, fruits[1]+fruits[2]/2,
+ fruits[1]+fruits[2]+fruits[3]/2),
+ c("lemon", "-orange", "apple"),
+ col=c("white", "black", "black"))
> axis(2, labels="%", at=100, padj=-1, las=1)![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべきです。
」 を読むべきです。text(c(fruits[1]/2, fruits[1]+fruits[2]/2, fruits[1]+fruits[2]+fruits[3]/2), t[1],
c("lemon", "-orange", "apple"), col=c("white", "black", "black"))
ちなみに,今の場合はラベルは 3 つしかないので示されたように座標を計算してもよいけど,たくさんある場合などには,
c(0, cumsum(fruits)[-length(fruits)])+fruits/2とするのがスマートでしょう。
はにわ (2013-09-30 (月) 15:57:33)
xlsxライブラリが使いたく,install.packages("xlsx") library(xlsx)とすると、以下のようなメッセージがでます。
> library(xlsx) 要求されたパッケージ xlsxjars をロード中です 要求されたパッケージ rJava をロード中です Error : .onLoadはloadNamespace()(xlsxjarsに対する) の中で失敗しました、詳細は: call: .jinit() error: Cannot create Java virtual machine (-4) 追加情報: 警告メッセージ: 1: パッケージ '‘xlsx’' はバージョン 2.14.2 の R の下で造られました 2: パッケージ '‘xlsxjars’' はバージョン 2.14.2 の R の下で造られました 3: パッケージ '‘rJava’' はバージョン 2.14.2 の R の下で造られました エラー: パッケージ '‘xlsxjars’' をロードできませんでしたWindows XP, R version 2.14.1です。
3か月前には同じコマンドで使えていたのでとても不思議です。
javaをインストールし直したりPATHを設定し直しましたがダメです。
何が原因か分かりますでしょうか。
平田光一 (2013-09-30 (月) 11:33:01)
RStudio0.97.551、R3.0.1を使用していますが、RStudioでは、動作しますが、RIDEでは、関数の呼び出し時に、ハングアップしてしまいます。
Win7 64bits、desktop PC、関数呼び出し後、直ぐに、print("abc")を挿入して見ましたが、表示する前に、ハングアップします。プログラムは、500行ぐらいで、変数は、大きいです。何か手がかりは無いでしょうか?
y (2013-09-26 (木) 13:53:02)
Mac OS Xユーザーです。
R for Mac OSXの最新版をインストールして、いくつかデータを打ち込んで練習していたところ、突然固まってしまったのでRを強制終了しました。
その後Rを何度クリックして起動しようとしても、うまく起動できません(ずっと虹色のくるくるが回ったままで、アプリケーションが応答しない)。
Rのアプリケーションをゴミ箱に入れて、再びインストールしてみたのですが、やはり起動できません。アプリケーション自体をインストールするところまではできます。
Rの関連ファイルがMac内に残っているのが悪いのかなと思い、いろいろと調べてみたのですが、どれがRの関連ファイルなのか、見つけられません。
おそらく、Rのアンインストールが不完全なのだと思います。
ですので、Rの関連ファイルとして考えられるものを、どの場所にあるかも含めて、教えていただけないでしょうか?
また、他にもなにかいい解決方法がありましたら、それもご教授いただきたいです。
よろしく願いいたします。
wataya (2013-09-26 (木) 06:09:48)
たびたびすみません。
Windows7 64bitでREXCELを使用しています。Rは32bit版で3.0.1。REXCELは3.2.13。EXCELは2007です。
VBAで花の名前の文字の入っている文字列配列のstr_moji(10)と、同名のデータセットをRに作成したいのですがどうすればいいのかわかりません。
Rinterface.PutDataframe "str_moji(0)", str_moji(0)
このようにすると、str_moji(0)でデータセットが作られてしまいます。
Rinterface.PutDataframe str_moji(0), str_moji(0)
このように""を抜いてしまうとエラーが出てとまります。
VBAから変数でデータセットを作る方法は無いんでしょうか。ご教授お願い致します。
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべきです。 -- 河童の屁は,河童にあらず,屁である。 2013-09-26 (木) 20:47:39
」 を読むべきです。 -- 河童の屁は,河童にあらず,屁である。 2013-09-26 (木) 20:47:39wataya (2013-09-25 (水) 21:30:38)
Windows7でRの32bit版を使用しています。Excelは2007です。
Rinterface.RRun "setwd (""C:/R/toukei/"") "
このようにフルパスで記述すれば、VBAで作業フォルダを指定できるのですが、
Dim strPath As String
strPath = ActiveWorkbook.Path
VBAで取得した文字列変数strPathのパスを
Rinterface.RRun "setwd ()"に書く方法がわかりません。
どのように書けばよいのか教えてください。宜しくお願い致します。
T (2013-09-25 (水) 13:22:27)
tapply関数でcsvファイルから要素別の平均値を出しているのですが, 要素の順序がアルファベット順になってしまいます.
mydataというcsvファイルの1行目に観測値が, 2行目に要素名が入っています.
要素.M <- tapply(mydata2$観測値, mydata2$要素, mean)
これを解消する方法は何かありませんでしょうか?
出力される要素の順序をこちらで指定できると有難いのですが..
宜しくお願いします
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) 」 を読むべきです。 -- 河童の屁は,河童にあらず,屁である。 2013-09-25 (水) 18:18:47
」 を読むべきです。 -- 河童の屁は,河童にあらず,屁である。 2013-09-25 (水) 18:18:47ndesse (2013-09-19 (木) 09:39:08)
R使いにも慣れて来たのですが、ループに困る事態が生じました。
原因が分からず、解決出来ません。お助け下さい。
処理したいデータは以下のようなものです。
コンマきり。V1=染色体番号、V2=位置、V3=遺伝子の長さ、V4=遺伝子名、V5=遺伝子相対位置、V6=計算したい値です。V1, V2, V3, V4, V5, V6, V7 chr01, 137.16, 1480, geneA, -1.50, 1.039, 1.351 chr01, 137.16, 1480, geneA, -1.50, 1.039, 1.351 chr01, 137.15, 1480, geneA, -1.49, 1.032, 1.353 chr01, 137.14, 1480, geneA, -1.48, 1.039, 1.355 chr01, 137.13, 1480, geneA, -1.47, 1.020, 1.349 chr01, 137.12, 1480, geneA, -1.46, 1.025, 1.344 chr01, 137.11, 1480, geneA, -1.45, 1.043, 1.337 chr01, 137.10, 1480, geneA, -1.44, 1.067, 1.316 chr01, 137.09, 1480, geneA, -1.43, 1.064, 1.305 chr01, 137.08, 1480, geneA, -1.42, 1.055, 1.311 chr01, 137.07, 1480, geneA, -1.41, 1.035, 1.315 chr01, 137.06, 1480, geneA, -1.40, 1.040, 1.297 chr01, 137.05, 1480, geneA, -1.39, 1.027, 1.294 chr01, 137.04, 1480, geneA, -1.38, 1.014, 1.321 chr01, 137.03, 1480, geneA, -1.37, 0.990, 1.333 chr02, 137.16, 1480, geneB, -1.50, 3.039, 1.351 chr02, 137.15, 1480, geneB, -1.49, 3.032, 1.353 chr02, 137.14, 1480, geneB, -1.48, 3.039, 1.355 chr02, 137.13, 1480, geneB, -1.47, 3.020, 1.349 chr02, 137.12, 1480, geneB, -1.46, 3.025, 1.344 chr02, 137.11, 1480, geneB, -1.45, 3.043, 1.337 chr02, 137.10, 1480, geneB, -1.44, 3.067, 1.316 chr02, 137.09, 1480, geneB, -1.43, 3.064, 1.305 chr02, 137.08, 1480, geneB, -1.42, 3.055, 1.311 chr02, 137.07, 1480, geneB, -1.41, 3.035, 1.315 chr02, 137.06, 1480, geneB, -1.40, 3.040, 1.297 chr02, 137.05, 1480, geneB, -1.39, 3.027, 1.294 chr02, 137.04, 1480, geneB, -1.38, 3.014, 1.321 chr02, 137.03, 1480, geneB, -1.37, 3.990, 1.333このファイルは、実際は相対位置が-1.5~3.5まで0.01づつ増加、遺伝子数は300ほどあります。
このデータに関して、相対位置 (V5) が同じ値のもののV6の平均を出したいと思っています。
私のScriptでは、ループ13回以降が動きません。この例のデータだと13番目で終わっており、実際の150回ループ(-1.5~3.5の0.01刻み)では、13回以降は「i NaN」という結果がループ分プリントされます。ループの始まりをずらして(例えば i=-1.37、14行目)スタートすると、また13番目で終わってしまいます。どこが間違っているのでしょうか?
書いたループはp <- read.table("test.txt", fill=TRUE) i=-1.50 while (i < -1.37) { p.sub <- subset(p, p$V5 == i, c(V6)) p.sub2 <- na.omit(p.sub) m <- mean(p.sub2$V6) sink("result.txt",append = TRUE) cat(i) cat("\t") cat(m) cat("\n") sink() i=i+0.01 }です。よろしくお願いします。
v5 <- sort( unique(p$V5) )
x <- matrix(0, nrow=length(v5), ncol=2)
for(i in 1:length(v5)){
x[i,] <- c(v5[i], mean(p$V6[p$V5==v5[i]]) )
}
> x
[,1] [,2]
[1,] -1.50 2.039
[2,] -1.49 2.032
[3,] -1.48 2.039
[4,] -1.47 2.020
[5,] -1.46 2.025
[6,] -1.45 2.043
[7,] -1.44 2.067
[8,] -1.43 2.064
[9,] -1.42 2.055
[10,] -1.41 2.035
[11,] -1.40 2.040
[12,] -1.39 2.027
[13,] -1.38 2.014
[14,] -1.37 2.490> s <- 0
> i <- 0
> while (i <= 1) {
+ s <- s+i
+ i <- i+0.01
+ }
> print(s) # 表示される値は 49.5 であるが,
[1] 49.5
> print(s == 49.5) # s の値は,正確な 49.5 ではない(真の値は 50.5 であるべき)
[1] FALSE
> print(sprintf("s = %a\n", s)) # 最下位にゴミがついている
[1] "s = 0x1.8c00000000004p+5\n"
> print(sprintf("s = %a\n", 49.5)) # なお,49.5 は 2 進数でも正確に表すことができる
[1] "s = 0x1.8cp+5\n"
次のプログラムと比較するとよい(これもあまりよいプログラムではないが)
> s <- 0
> i <- 0
> while (i <= 100) {
+ s <- s+i/100
+ i <- i+1
+ }
> print(s) # 正解
[1] 50.5
> print(s == 50.5)
[1] TRUE
> print(sprintf("s = %a\n", s))
[1] "s = 0x1.94p+5\n"
> print(sprintf("s = %a\n", 49.5))
[1] "s = 0x1.8cp+5\n"原 (2013-09-14 (土) 16:25:38)
R初心者です。初めて投稿いたします。お力をお貸しください。
生存率に影響を及ぼす因子を検討するため、Coxハザード回帰を使い検討しました。具体的には年齢、性別、LOHという因子を共変量として検討しました。しかし、以下のようにエラーが出ます。エラーのためハザード比もおかしなことになっています。
いろいろ検討してみましたが、どうしてもわからず質問させていただきました。どうかご教授ください。> d.cox<-coxph(Surv(duration,censor)~sex+age_56+LOH, method="exact", data=d) fitter(X, Y, strats, offset, init, control, weights = weights, 中で警告がありました: Loglik converged before variable 3 ; beta may be infinite. > summary(d.cox) Call: coxph(formula = Surv(duration, censor) ~ sex + age_56 + LOH, data = d, method = "exact") n= 53, number of events= 34 (15 observations deleted due to missingness) coef exp(coef) se(coef) z Pr(>|z|) sex 2.677e-01 1.307e+00 3.734e-01 0.717 0.4733 age_56 8.340e-01 2.303e+00 3.884e-01 2.147 0.0318 * LOH -1.958e+01 3.125e-09 5.875e+03 -0.003 0.9973 --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 exp(coef) exp(-coef) lower .95 upper .95 sex 1.307e+00 7.651e-01 0.6287 2.717 age_56 2.303e+00 4.343e-01 1.0754 4.930 LOH 3.125e-09 3.200e+08 0.0000 Inf Rsquare= 0.349 (max possible= 0.983 ) Likelihood ratio test= 22.78 on 3 df, p=4.487e-05 Wald test = 5.99 on 3 df, p=0.1121 Score (logrank) test = 16.42 on 3 df, p=0.0009295エラーメッセージの意味や対処方法など、ご教授頂ければ幸いです。
基本的なことが分かってないかもしれませんが、よろしくお願いいたします。
yuki (2013-09-14 (土) 16:17:17)
quantmodやTTRパッケージを使い株価チャートやテクニカル指標の表示をしたいです。
データの取得にgetSymbols()を使った場合、うまくいくのですが(下記コード参照)日本の株価を取得するためにRFinanceYJパッケージのquoteStockXtsData()を使い、同様に実行すると次のエラーが表示されうまく行きません。以下にエラー BBands(xx, n = n, maType = maType, sd = sd) : Price series must be either High-Low-Close, or Close/univariate.そこで対象のデータを、toyota[, c(2:4)]やtoyota[, c(4)]のようにもしましたがうまく行きませんでした。
以下にエラー runSum(x, n) : Invalid 'n'解決方法をご教授いただけませんでしょうか。よろしくお願い致します。
library(RFinanceYJ) library(quantmod) getSymbols("GS") str(GS) chartSeries(GS) addBBands() addCCI() #日本の株価データで同上のことを行う toyota <- quoteStockXtsData("7203.t", since="2013-08-23", date.end="2013-09-13", time.interval="daily") str(toyota) chartSeries(toyota) addBBands() # error addCCI() #error chartSeries(toyota[, c(2:4)]) chartSeries(toyota[, c(4)]) addBBands() # error addCCI() #error
toyota <- quoteStockXtsData("7203.t", since="2013-07-23",
date.end="2013-09-13", time.interval="daily")
toyota2 <- toyota[,1:4]
str(toyota2)
chartSeries(toyota2)
addBBands()
addCCI()> debug(addBBands)
> addBBands()
debugging in: addBBands()
debug: {
stopifnot("package:TTR" %in% search() ||
require("TTR", quietly = TRUE))
draw.options <- c("bands", "percent", "width")
draw <- draw.options[pmatch(draw, draw.options)]
lchob <- get.current.chob()
x <- as.matrix(lchob@xdata)
chobTA <- new("chobTA")
if (draw == "bands") {
chobTA@new <- FALSE
}
else {
chobTA@new <- TRUE
on <- NULL
}
xx <- if (is.OHLC(x)) {
cbind(Hi(x), Lo(x), Cl(x))
}
else x
bb <- BBands(xx, n = n, maType = maType, sd = sd)
chobTA@TA.values <- bb[lchob@xsubset, ]
chobTA@name <- "chartBBands"
chobTA@call <- match.call()
chobTA@on <- on
chobTA@params <- list(xrange = lchob@xrange, colors = lchob@colors,
color.vol = lchob@color.vol, multi.col = lchob@multi.col,
spacing = lchob@spacing, width = lchob@width, bp = lchob@bp,
x.labels = lchob@x.labels, time.scale = lchob@time.scale,
n = n, ma = maType, sd = sd, draw = draw)
return(chobTA)
}masa (2013-09-08 (日) 18:39:33)
リストのデータをwriteで出力すると以下の様なエラーがでます.
リストの出力はどのようにすればよいのでしょうか?以下にエラー cat(list(...), file, sep, fill, labels, append) : 引数 1 (タイプ 'list') は 'cat' で取り扱えません
set.seed(1234567)
a <- list(c(3L, 5L, 8L), x=1.23, a=rnorm(10),
b=letters[1:7], c=data.frame(a2=1:5, b2=factor(1:5),
c2=month.abb[1:5]), d=matrix(1:12, 3),
e=list(a3=1, b3="abc"), f=c(TRUE, FALSE),
g=factor(c("male", "female")))
dput(a, file="list.dat")
b <- source("list.dat")[[1]] # [[1]] を忘れずに
for (i in seq_along(b)) { # 同じであることを確認
cat(names(b)[i], all.equal(a[[i]], b[[i]]), "\n")
}
# dump を使う場合は,
# dump("a", "foo") の後,souce("foo") をすると a ができる
# ファイル内容を見れば dput と dump の違いが分かるはいよー (2013-09-06 (金) 14:42:37)
tcltkでリストボックスを2個表示してそれぞれの要素を選択した後、OKボタンでそれらの要素名を取得するための下記のようなスクリプトにおいて他方のリストボックスにフォーカスを移すと選択が解除されてしまい、結局どちらか一方の要素しか選択できません。tkpackにしたり、孫フレームで両者を分けたりしてみましたがダメでした。Rコマンダーの似たようなフォームでは選択が解除されないかと思いますがどのような手直しが必要でしょうか?よろしくお願いいたします。(R3.0.1、R2.15.2)
require(tcltk) wintop<-tktoplevel() OnOK <- function() { fru1 <- fruits1[as.numeric(tkcurselection(listbox1))+1] fru2 <- fruits2[as.numeric(tkcurselection(listbox2))+1] tkdestroy(wintop) msg <- paste(fru1,"and",fru2, "are selected.",sep=" ") tkmessageBox(message=msg) } listbox1 <-tklistbox(wintop, selectmode = 'single') fruits1 <- c("Apple","Orange","Banana","Pear") for (i in (1:4)){tkinsert(listbox1,"end",fruits1[i])} tkgrid(listbox1) listbox2 <-tklistbox(wintop, selectmode = 'single') fruits2 <- c("Cherry","Pear","Mango","Pear") for (i in (1:4)){tkinsert(listbox2,"end",fruits2[i])} tkgrid(listbox2) OK.but <-tkbutton(wintop,text=" OK ",command=OnOK) tkgrid(OK.but) tkfocus(wintop)
$ grep -l tklistbox Rcmdr/R/*.R Rcmdr/R/data-menu.R Rcmdr/R/statistics-models-menu.R Rcmdr/R/utilities.Rということなので、どれでもいいですが、例えば、utilities.Rの中を見ます。すると、
listbox <- tklistbox(frame, height=min(listHeight, length(variableList)),
selectmode=selectmode, background=bg, exportselection=export,
width=min(max(minmax[1], nchar(variableList)), minmax[2]))
と書いてあります。exportselectionが怪しいとあたりをつけて、検索すると複数のリストボックスを使用する場合などの解説が見つかります。かけだし (2013-09-04 (水) 17:24:48)
Rを始めたばかりのものです。R3.0.1 64ビット Windowsを使用しています。
8桁の数字データ(ex. 20130904)を日付型に変換する方法について教えてください。
日付型に変換するには、as.Dateを使えばよいと考えて、データを読み込んだ後、まずas.characterで文字型に変換しました。
modeで型変換ができていることは確認しました。
しかし、それをas.Date関数に入れても、エラーとなり文字型にできません。
実際にはデータフレームの1列を使ってやっているのですが、1例として示しますと、下記のような感じです。> as.Date("20130904") 以下にエラー charToDate(x) : 文字列は標準的な曖昧さのない書式にはなっていません > as.Date("2013/09/04")→スラッシュが入っているとうまくいくようです。
この場合、Rに読み込んだ後にどのような操作をすればよいのでしょうか。
ご教示いただけましたら幸いです。よろしくお願いいたします。
> a <- "20130904"
> as.Date(sprintf("%s/%s/%s", substr(a, 1, 4), substr(a, 5, 6), substr(a, 7, 8)))
[1] "2013-09-04"> as.Date("20130904", format = "%Y%m%d")
[1] "2013-09-04"
> as.Date("2013年9月4日", format = "%Y年%m月%d日")
[1] "2013-09-04"tamn (2013-09-03 (火) 01:44:54)
a.jpg, b.jpg, c.jpg(以下、a, b, c)という画像ファイルがあります。
データフレームの情報をもとに、aとbを横に並べた1つの画像(ab_yoko.jpg)や、bとcを縦に並べた1つの画像(bc_tate.jpg)を生成するような処理は可能でしょうか?
ライブラリなどあれば教えていただけないでしょうか。
難しければ、他の言語やソフトでということになるのですが、Rでできればと思ってます。
よろしくお願いします。
library(jpeg)
# read a sample file (R logo)
img <- readJPEG(system.file("img", "Rlogo.jpg", package="jpeg"))
# read it also in native format
img.n <- readJPEG(system.file("img", "Rlogo.jpg", package="jpeg"), TRUE)
# 画像データの結合
img2 <- rbind(img, img.n)
# img2 <- rbind(img, img)
# 保存
path <- paste(getwd(), "/img2.jpg", sep="")
writeJPEG(img2, target = path, bg = "white")> # read a sample file (R logo)
> img <- readJPEG(system.file("img", "Rlogo.jpg", package="jpeg"))
>
> # read it also in native format
> img.n <- readJPEG(system.file("img", "Rlogo.jpg", package="jpeg"), TRUE)
>
> # if your R supports it, we'll plot it
> jpeg("test.jpeg")
> if (exists("rasterImage")) { # can plot only in R 2.11.0 and higher
+ plot(1:2, type='n')
+
+ rasterImage(img, 1.2, 1.27, 1.8, 1.73)
+ rasterImage(img.n, 1.5, 1.5, 1.9, 1.8)
+ }
> dev.off()

> img <- readJPEG(system.file("img", "Rlogo.jpg", package="jpeg"))
> R <- img[,,1]
> G <- img[,,2]
> B <- img[,,3]
> str(img)
num [1:76, 1:100, 1:3] 1 1 1 1 1 1 1 1 1 1 ...
> img2 <- array(NA, c(76 * 2, 100, 3))
> img2[,,1] <- rbind(R, R)
> img2[,,2] <- rbind(G, G)
> img2[,,3] <- rbind(B, B)
> writeJPEG(img2, target = "/tmp/tmp.jpg")

mashi (2013-08-26 (月) 22:38:17)
長いベクトルを前から順番に,指定した長さで,分割する効率的な方法にはどのようなものがあるのでしょうか。
(例えば,10個の数字を,1,3,2,4こずつに分割するなど)
データ量が多いのでループを使うと処理しきれません。
よろしくお願いします。
func1 <- function(vec, len) {
n <- length(len)
end <- cumsum(len)
begin <- c(1, (end+1)[-n])
mapply(function(b, e) vec[b:e], begin, end)
}
func2 <- function(vec, len) {
n <- length(len)
ans <- vector("list", n)
end <- cumsum(len)
begin <- c(1, (end+1)[-n])
for (i in seq_along(begin)) {
ans[[i]] <- vec[begin[i]:end[i]]
}
return(ans)
}
ベンチマーク
> library(rbenchmark)
> vec <- 1:15000
> len <- rep(c(1, 3, 2, 4, 5), 1000)
> benchmark(func1(vec, len), func2(vec, len))
test replications elapsed relative user.self sys.self
1 func1(vec, len) 100 2.043 1.000 2.042 0.019
2 func2(vec, len) 100 2.378 1.164 2.387 0.011> x <- c(0:9,0:9,0:9) # 長い数値ベクトルのつもり
> X <- matrix(x,3,10,byrow=TRUE); X
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
[1,] 0 1 2 3 4 5 6 7 8 9
[2,] 0 1 2 3 4 5 6 7 8 9
[3,] 0 1 2 3 4 5 6 7 8 9
> X1 <- X[,1,drop=FALSE]; X1 # オプションdrop=FALSEに注意
[,1]
[1,] 0
[2,] 0
[3,] 0
> X2 <- X[,2:4,drop=FALSE]; X2
[,1] [,2] [,3]
[1,] 1 2 3
[2,] 1 2 3
[3,] 1 2 3
> X3 <- X[,5:6,drop=FALSE]; X3
[,1] [,2]
[1,] 4 5
[2,] 4 5
[3,] 4 5
> X4 <- X[,7:10,drop=FALSE]; X4
[,1] [,2] [,3] [,4]
[1,] 6 7 8 9
[2,] 6 7 8 9
[3,] 6 7 8 9> set.seed(123) > x <- round(runif(10000)) #長いベクトル > head(x, 20) [1] 0 1 0 1 1 0 1 1 1 0 1 0 1 1 0 1 0 0 0 1 > g <- abs(round(rnorm(1000))) + 1 #長さ指定のベクトル > head(g, 20) [1] 2 1 2 3 2 1 2 4 2 3 2 1 1 1 1 1 1 3 1 1gが指定する長さで、長いベクトルxを区切りたい。さて、どうしますか?河童さんの方法なら、一発で解決ですが、あなたの方法なら、2000個の添え字をいちいち計算しながら、入力しなくてはいけません。全く簡便ではないし、1000回も手で計算しているうちにミスも混入します。 ここはWikiシステムなので、ご自分の書き込みが不適切を思われれば、ご自分の書き込みとその反応(上の人の突っ込みと、私の書き込み、そして下のsample()を提案している人の書き込み)も含めてさくっと削除してもOKだと思います。
マルボロ川越 (2013-08-23 (金) 15:31:43)
選択結果(0,1)を被説明変数とした入れ子型ロジットモデルでの分析方法はわかりました。しかし、被説明変数をマーケットシェアとして分析した場合、t値が非常に小さくなりうまくいきません。
パッケージはlogit modelの対数尤度関数の定義であるfunction()やoptim()を用いています。
単純に選択結果をマーケットシェアに変更しただけではよろしくないのでしょうか?
(2013-08-17 (土) 09:25:35)
NAを含むデータで,青木先生のfactal2関数で因子得点を利用したいと思ったのですが,NAを含む行を削除しているために,既存のデータセットに加えることができません。以下のエラーが発生しました。以下にエラー data.frame(..., check.names = FALSE) : 引数は異なった列数を意味します: 5256, 5001, 4883, 487そこで,削除された行を挿入して,NAにしたいと思ったのですが,どのように行ったらよいのでしょうか。
set.seed(4649) d <- data.frame(matrix(rnorm(300), 50)) d[6, 3] <- d[24, 2] <- d[48, 5] <- NA A <- factanal2(d, factors=2, scores="regression", verbose=FALSE) B <- merge(d, A$scores, by="row.names", all=TRUE, sort=FALSE) C <- B[order(as.numeric(B[,1])),] rownames(C) <- C[,1] C <- C[,-1]
たく (2013-08-15 (木) 17:26:34)
例えば以下の例のようにtcltkでGUIで値を入れたりすることができますが、この例だと関数の中に変数が入ってしまうために、入力した値を変数として保存できません。
入力値を変数に残してその後使えるようにする良い方法をご存じでしたらどなたかお教えいただければ幸いです。
(環境はWin7 R3.01 64bitで、macではXのインストールが必要だと思います。)require(tcltk) tt<-tktoplevel() Name <- tclVar("Anonymous") entry.Name <-tkentry(tt,width="20",textvariable=Name) tkgrid(tklabel(tt,text="Please enter your first name.")) tkgrid(entry.Name) OnOK <- function() { NameVal <- tclvalue(Name) tkdestroy(tt) msg <- paste("You have a nice name,",NameVal) tkmessageBox(message=msg) return(NameVal) } OK.but <-tkbutton(tt,text=" OK ",command=OnOK) tkbind(entry.Name, "<Return>",OnOK) tkgrid(OK.but) doneval1 <- NameVal #関数の中なので変数として保存できない doneval2 <- tclvalue(Name) #関数の中なので変数として保存できない tkfocus(tt)
yaito (2013-08-11 (日) 17:32:25)
R初心者、というか今日から始めたばかりです。
EZRというコマンダーを使ってROC曲線を書いてみたのですが、いくつかのROCを重ねたFigureが作りたいのですが、作れません。2つまでならソフトにあるのですが、3つ以上重ねるにはどのようなスクリプトが必要なのでしょうか。何かおすすめの本などもありましたら御教授お願いします。
chaemon (2013-08-07 (水) 22:31:06)
初投稿です。
結合法則が成り立つ2項演算子☆が定義されているとして、ベクトルv=(v_1, v_2, ..., v_n)に対して(v_1☆v_2☆v_3☆・・・☆v_n)を高速に計算してくれるような関数はありますか。
+に対するsumのようなイメージです。
ベクトルによる並列処理などをしてくれて、for文で回すよりも早いものがよいです。
ないとすれば、作成したいのですが、その場合どちらのページを参考にするのがよいですか。
よろしくお願いします。
f1 <- function(x){
if(length(x) <= 1) return(x)
else {
i <- length(x)
y <- sapply(1:floor(i/2),
function(m){
x[2 * m - 1] + x[2 * m]
})
if(i %% 2 != 0) y <- c(y, x[i])
return(Recall(y))
}
}
> system.time(f1(1:10000))
ユーザ システム 経過
0.032 0.000 0.029
> system.time(sum(1:10000))
ユーザ システム 経過
0 0 0
従って、C言語のリファレンスを参照してださい。マニュアルのWriting R Extensionsには、C言語とRの連携について解説されています。library(inline)
src <- '
Rcpp::NumericVector x(X);
double ans = x(0);
for (int i = 1; i < x.size(); i++) {
ans += x(i);
}
return wrap(ans);
'
f2 <- cxxfunction(signature(X="numeric"), src, plugin="Rcpp")
f3 <- function(x){
ans <- 0
for (y in x) ans <- ans+y
return(ans)
}
実行結果
> n <- as.numeric(1:1e6)
> library(rbenchmark)
> benchmark(sum(n), f1(n), f2(n), f3(n), Reduce("+", n),
+ replications=10)
test replications elapsed relative user.self sys.self
2 f1(n) 10 32.789 964.382 32.750 0.244
3 f2(n) 10 0.063 1.853 0.064 0.000
4 f3(n) 10 3.144 92.471 3.144 0.014
5 Reduce("+", n) 10 3.544 104.235 3.522 0.037
1 sum(n) 10 0.034 1.000 0.034 0.000イズミ (2013-08-03 (土) 08:04:11)
初めてこちらに投稿します。
よろしくお願いします。
多分岐(C5.0)でツリー分析のモデルを作成しようとしたところ、以下のようなエラーメッセージが表示されました。
以下にエラー paste(apply (x,1,paste,collapse = ","),collapse = "\n"):
結果が2^31-1バイトを超えました
モデル作成は、以下のデータで行いました。
・データのファイル容量:約2.5GB
・データのレコード件数:約300万件
・説明変数とした項目数:約50項目
処理で使用したPCの物理メモリは32GBで、データ量を約40万件に減らしたところ、上記のエラーメッセージは表示されず、モデルは作成できました。
また、C5.0ControlでminCaseを10000とか20000にして、ノード内の最小データ件数を増やすことで、作成されるノード数を減らそうとしたのですが、同じメッセージが表示されます。
なお、上記データにて、tree関数で二分岐のツリーモデルを作成することができたので、データの中身は問題ないと考えています。
Google等で調べてもわからず、こちらにやってきました。
恐れ入りますが、解決策のご教示のほど、よろしくお願いします。
> a <- rep(1, 2^31) 以下にエラー rep(1, 2^31) : 'times' 引数が不正です 追加情報: 警告メッセージ: 強制変換により NA が生成されました > b <- rep(1, 2^31-1) # エラーなし > length(b) [1] 2147483647 > 2^31-1 [1] 2147483647ということなので,paste も「結果が2^31-1バイトを超えました」というからには,案外な限界を持っているということなのでしょう。
77 (2013-08-01 (木) 23:03:13)
chrome os でRはインストールできますか?
かものはし (2013-08-01 (木) 15:24:29)
下記のようなデータがあって、例えば13:00〜15:00のデータだけ抽出する場合、どのようにすれば良いでしょうか。また、数日間にわたって採取したデータの中から昼間のデータ(6:00〜18:00)だけとか、夜間のデータ(18:00〜翌朝6:00)だけを抽出することも可能でしょうか。Googleで調べてもそれらしい方法に出会えなかったのでここにやってきました。よろしくお願いします。x y 1 2010-08-01 12:14:00 230 2 2010-08-01 13:14:00 260 3 2010-08-01 14:14:00 300 4 2010-08-01 15:14:00 260 5 2010-08-01 16:13:00 240
hm <- as.integer(paste(substr(d$x, 12, 13), + substr(d$x, 15, 16), sep="")) d[1300 <= hm & hm <= 1500,] # 13:00 〜 15:00 d[0600 <= hm & hm <= 1800,] # 06:00 〜 18:00 d[!(0600 < hm & hm < 1800),] # 06:01 〜 17:59 でないもの秒まで考慮する必要があるならそれなりに。 -- 河童の屁は,河童にあらず,屁である。 2013-08-01 (木) 16:03:51
> a <- data.frame(x = as.POSIXct("2010-08-01 12:14") +
+ cumsum(c(0, rep(60 * 60, 47))),
+ y = round(runif(48, 200, 300)))
> head(a, 3)
x y
1 2010-08-01 12:14:00 241
2 2010-08-01 13:14:00 280
3 2010-08-01 14:14:00 239
というサンプルデータをあなたのデータに似せて作りました。
> a$hours <- format(a$x, "%H")時間を新しいカラムに入れます。13時から15時を取り出します。
> a[with(a, hours >= 13 & hours <= 15), ]
x y hours
2 2010-08-01 13:14:00 280 13
3 2010-08-01 14:14:00 239 14
4 2010-08-01 15:14:00 266 15
26 2010-08-02 13:14:00 237 13
27 2010-08-02 14:14:00 300 14
28 2010-08-02 15:14:00 270 15
目で見て分かりやすいようにhoursをデータフレームに追加しましたが、本当はそうする必要はないです。念のため。もし、秒まで考えたいなら0時0分0秒からの経過秒を考えればよいと思います。分かりやすいベタな方法はformat()とas.numeric()を使う方法です。
as.numeric(format(a$x, "%H")) * 60 * 60 +
as.numeric(format(a$x, "%M")) * 60 +
as.numeric(format(a$x, "%S"))
私は計算式文字列を作成してからeval()で計算する方が好みです。
sapply(a$x, function(i){eval(parse(text=format(i, "%H * 60 * 60 + %M * 60 + %S")))})さぼりん (2013-07-25 (木) 16:22:51)
さぼりんです。またお世話になります。
トレーニングデータをもとにモデルを構築し、テストデータを用いてモデルの当てはまりの良さを確認したいです。
トレーニングデータ"130725_test31_train.csv"は、X(入力)が7行x6列で、Y(出力)が7行x1列です。また、テストデータ"130725_test31_test.csv"は、Xが3行x6列で、Yが3行x1列です。(CSVファイルは別途アップロードします。)
下記のコマンドを実行したところ、モデルからの予測値(test.pred)が7行表示されました。テストデータに対する予測を行うのだから、値は3行表示されるべきだと考えています。コマンドの出力結果は正しいのでしょうか。
恐れ入りますが、ご教授をお願い致します。test31.train <- read.csv ("130725_test31_train.csv") Y <- as.matrix(test31.train[ ,7]) X <- as.matrix(test31.train[ ,1:6]) Y <- scale(Y) X <- scale(X) test31.pls <- plsr(OUTPUT ~ X, 2, data = test31.train, validation = "LOO") test31.test <- read.csv ("130725_test31_test.csv") Y <- as.matrix(test31.test[ ,7]) X <- as.matrix(test31.test[ ,1:6]) Y <- scale(Y) X <- scale(X) test.pred <- predict(test31.pls, ncomp = , test31.test$X) test.pred , , 1 comps OUTPUT 1 437321.1 2 437871.6 3 436570.5 4 439711.1 5 438163.7 6 440080.2 7 437147.9 , , 2 comps OUTPUT 1 439055.4 2 439095.0 3 436742.8 4 440570.5 5 436976.8 6 439369.7 7 435055.7
test31.train <- read.csv ("130725_test31_train.csv")
# 7 行 7 列,最終列の名前が OUTPUT
test31.pls <- plsr(OUTPUT ~ ., 2, data = test31.train,
validation = "LOO")
test31.test <- read.csv ("130725_test31_test.csv")
# 3 行 7 列,最終列の名前が OUTPUT
test.pred <- predict(test31.pls, test31.test[,1:6])
# OUTPUT を含まない場合は [,1:6] は不要
test.pred
# そもそも predict に OUTPUT は不要koheizm (2013-07-25 (木) 12:12:44)
少し統計よりの質問なので恐縮なのですが、下記のような期待値を算出したい時の関数やライブラリをご存知でしたらご教授願います。
===
コイントスで三枚同時に投げる
表の確率1/2 裏の確率1/2
表が出た回数をxとする
===
この時のE(x)とV(x)を導きたいのです。
n数が少ない場合は順を追って計算し算出出来るのですが~、もっと上手く算出できるのではないかなと思って質問させていただきます。
二項分布のdbnoimや一様分布のrunif、コインパッケージなどを調べてて行き詰まってしまいました。
func <- function(n) {
if (n <= 20) {
require(e1071)
f <- table(rowSums(bincombinations(n)))
x <- as.numeric(names(f))
mean <- sum(f*x)/2^n
variance <- sum(f*(x-mean)^2)/2^n
return(c(mean=mean, variance=variance))
}
}
使用例
> n <- 20
> p <- 1/2
> func(n)
mean variance
10 5
> c(n*p, n*p*(1-p)) # 理論値
[1] 10 5foo (2013-07-22 (月) 16:58:39)
質問事項
PLSに関するデータ構造の制限(仕様)を教えて下さい。 (例:0は処理できない、次元数はxxまで等)
以下、テスト内容。
1. PC環境
Windows 7 Enterprise Service Pack 1
Intel(R) Core(TM) i5 CPU 2.67 GHz
RAM 2.00GB
32bit
2. R環境
R-3.0.1
PLS 2.3.0
3. データ構造
3-1. Xが入力、Yが出力
X 19列x15行
Y 1列x15行
3-2. Xが入力、Yが出力
X 719列x23行
Y 1列x23行
4. PLS検証結果
4-1. 上記3-1、3-2のデータにおいて、
0が含まれると5.のエラーが出力され計算できない。
4-2. 上記3-1のデータで5.のエラーが出力され計算できない。
上記3-2のデータについては一度計算できた。<-- 再現性に疑問。
5. エラー
Invalid number of components, ncomp
> set.seed(37564) > x <- matrix(rnorm(23*719), 23) > y <- matrix(rnorm(23), 23) > x[1, 1] <- y[2, 1] <- 0 > a <- plsr(y~x, ncomp=2) # pcr にしても,エラーにはならない > summary(a) Data: X dimension: 23 719 Y dimension: 23 1 Fit method: kernelpls Number of components considered: 2 TRAINING: % variance explained 1 comps 2 comps X 4.406 8.622 y 95.943 99.886あなたの使い方が悪いのでは?関数をどのように使ったかわからないのでどうともいえないけど。
future21 <- read.csv ("xxx.csv")
Y <- as.matrix(future21[ ,720])
X <- as.matrix(future21[ ,1:719])
Y <- scale(Y)
X <- scale(X)
future21.pls <- plsr(BCD ~ X, 10, data = future21, validation = "LOO")
summary(future21.pls) library(pls)
future21 <- read.csv("pls.csv")
n <- 20
Y <- as.matrix(future21[,n])
X <- as.matrix(future21[ ,1:(n-1)])
# 1000 列+1列に水増し
n <- 1000
future21times50 <- cbind(data.frame(matrix(sample(X,
nrow(future21)*n, replace=TRUE), ncol=n)), Y)
Y <- as.matrix(future21times50[ ,n])
X <- as.matrix(future21times50[ ,1:(n-1)])
Y <- scale(Y)
X <- scale(X)
future21times50.pls <- plsr(Y ~ X, 10,
data = future21times50, validation = "LOO")
summary(future21times50.pls)tetsuro (2013-07-20 (土) 20:43:08)
こんにちは。
windows7 64bit で R 2.15.1 を使っています。
豊田氏のデータマイニング入門を読んでいるのですが、neuralパッケージがインストールできず困っています・・・。
install.packages(neural)としてもパッケージがありません。
この方の記事に書かれている通りに、下記リンク先からneural_1.4.2.zipをダウンロードしたところ、下記のようなメッセージがでました。
参照記事:
http://d.hatena.ne.jp/astrobot/20110628/1309278036
リンク先:
http://cran.ms.unimelb.edu.au/bin/windows/contrib/2.6/エラーメッセージ: utils:::menuInstallLocal() パッケージ ‘neural’ は無事に展開され、MD5 サムもチェックされました > library(neural) エラー: パッケージ '‘neural’' は R 2.10.0 以前に造らました。再インストールして下さい下記Q&Aの内容も参照したのですが、エラーメッセージが出てしまいました。
参照Q&A:
neuralパッケージのインストールについて
hiro? (2010-06-24 (木) 02:54:33)
http://cran.stat.nus.edu.sg/src/contrib/Archive/neural/にライブラリがありました。ここにアクセスし、neural_1.4.tar.gzをDL&解凍し、出てきたneuron/Rディレクトリをinstall.packages("出てきたRディレクトリ")で読み込んでやれば、インストール出来ました。お騒がせしてすみませんでした。 -- hiro? 2010-06-24 (木) 03:14:01エラーメッセージ: > install.packages("C:/Users/(ユーザー名)/Documents/R/win-library/ 2.15/neural/R") パッケージを ‘C:/Users/(ユーザー名)/Documents/R/win-library/2.15’ 中にインストールします (‘lib’ が指定されていないので) 警告メッセージ: package ‘C:/Users/(ユーザー名)/Documents/R/win-library/2.15/neural/R’ is not available (for R version 2.15.1)どなたかご教示いただけないでしょうか・・・。
よろしくお願いします。
かい (2013-07-10 (水) 13:31:52)
全ての変数の値を一括表示しようとしてfor (n1 in 1:length(ls())) { eval(parse(text = paste(ls()[n1],sep=""))) }をRで実行したのですが、何も表示されませんでした。試しに、簡略化した
for (n1 in 1:10) { ls() }を実行しても何も表示されませんでした。その場合でも ls() を単独で実行すると正常に変数の名前の一覧が表示されます。ls() を for で囲んでも機能するようにするためには、どうすれば良いでしょうか。
よろしくお願い致します。
> for (n1 in 1:length(ls())) {
+ print(eval(parse(text = paste(ls()[n1],sep=""))))
+ }
[1] 1
[1] 4