![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
初心者のための R および RjpWiki に関する質問コーナー
● スレッド
●● 目次 ●●
ひろ (2013-02-17 (日) 23:00:54)
kernlabパッケージのksvmを使用した下記コードを実行してplotすると確立分布図が現れますが,その図からサポートベクター(SV)(○・●,△・▲のプロット)を取り除く方法はありますでしょうか?(e1071パッケージでもoKです)
library(kernlab) # number of data points n <- 150 # dimension p <- 2 sigma <- 2 # variance of the distribution meanpos <- 0 # centre of the distribution of positive examples meanneg <- 3 # centre of the distribution of negative examples npos <- round(n/2) # number of positive examples nneg <- n-npos # number of negative examples # Generate the positive and negative examples xpos <- matrix(rnorm(npos*p,mean=meanpos,sd=sigma),npos,p) xneg <- matrix(rnorm(nneg*p,mean=meanneg,sd=sigma),npos,p) x <- rbind(xpos,xneg) # Generate the labels y <- matrix(c(rep(1,npos),rep(-1,nneg))) # Train a nonlinear SVM with Gaussian RBF kernel and default parameters svp <- ksvm(x,y,type="C-svc",kernel='rbf') plot(svp,data=x)
rkoma (2013-01-11 (金) 20:22:54)
R初心者です。よろしくお願い致します。
Rで作成する3次元散布図 (scatter3d)では、軸タイトルなどへのExpression関数への適用や、軸の最大・最小値の設定や、軸を対数目盛にすることは可能でしょうか?
Rcmdr の「3次元散布図」で作ったグラフをひな型にして、スクリプトウィンドウに追加して修正を行っています。具体的には、Rコマンダーから3次元散布図を作成して、scatter3d(x軸の値, y軸の値, z軸の値, surface=FALSE, residuals=TRUE, bg="white", axis.scales=TRUE, grid=TRUE, ellipsoid=FALSE, xlab=" ", ylab=" ", zlab=" ")となったところに log="x", log="z", xlim=c(0.1,100), zlim=c(0.1,100) およびExpression関数によるx,y,zlabのテキストの修正(上付き、下付き文字の追加)を加えています。特にエラーメッセージは出ないのですが、できませんでした。
2次元散布図では可能です。また、plot3d()では、最大値、最小値の設定はできたのですが、対数軸への変更はできませんでした。
R初心者、プログラミング初心者なので、全く原因がわかりません。単純に機能がないというだけでしょうか?Help()やRjpWikiの過去の質問なども含め、できるだけ調べてみたのですが、よくわかりませんでした。
また、scatter3d()やplot3d()に限らず、「最大値・最小値を設定できて軸を対数目盛りにできる3次元散布図」を作成する方法がありましたら、教えていただけると助かります。
OSはWindows2003、Rはver.2.15.2、Rcmdrはver.1.9-2を使用しています。
ちなみに、類似の質問をYahoo知恵袋にも投稿しています。そちらで解決しましたら、その旨ご報告します。
恐縮ですがご回答よろしくお願い申し上げます。
taipapa (2013-01-11 (金) 18:21:30)
某大学のパソコン(マック mountain lion)にR 2.15.2をインストールしました.その後にパッケージを色々入れようとすると,どのパッケージでも,以下の様なメッセージでうまくいきません.URL 'http://essrc.hyogo-u.ac.jp/ 省略 /pls_2.3-0.tgz' を試しています Content type 'text/html' length 182 bytes 開かれた URL ================================================== downloaded 182 bytes エラー: ファイル ‘/var/folders/5j/省略/pls_2.3-0.tgz’ は OS X バイナリパッケージではありません 追加情報: 警告メッセージ: 'tar' がゼロでない終了コード 1 を返しました tar: Unrecognized archive format tar: Error exit delayed from previous errors.なにをしても182バイトしかダウンロードしてくれません.
大学のネットワーク管理者に確認してもプロキシは使用していないとのことですので,プロキシの問題ではないと思います.また,幾つかのcranのミラーサーバーを試してみましたが,全てダメです.Bioconductorのパッケージをインストールしようとしても同様の症状です.マックのユーザーインターフェースの一つであるRパッケージインストーラでは,パッケージの一覧の取得は可能です.ですから,サーバーにはつながって中身も見えています.なのに,選択してインストールしようとすると上記のエラーでことごとくダメです.自宅ではなんの問題もなくR packageのインストールができているだけに,ほとほと困っております.うちの大学側のネットワーク絡みの問題なのでしょうが,かなり調べても同様の報告に行き当たることができず,こちらに相談する次第です.どなたかアドバイスいただければありがたいです.
たく (2013-01-11 (金) 11:21:32)
エディタでコードを色づけしてくれるものはいろいろありますが、ハイライトされた状態のカラーのまま印刷できるエディタや方法をご存じでしたらどなたかお教えいただけないでしょうか。環境はWin7を常用しています。
Montecarlo (2013-01-07 (月) 03:15:01)
何度か試行錯誤を重ねてみましたが、うまくいかなかったため皆様のお知恵を拝借したく投稿します。
よろしくお願いいたします。
セッションインフォメーションは以下の通りです。> sessionInfo() R version 2.15.2 (2012-10-26) Platform: x86_64-w64-mingw32/x64 (64-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base今したいことは題名にもあります通り、年次別に違うプロットマーカーを使用したいと思っています。
現在は以下のプログラムを走らせています。year <- c(rep(2008, 10), rep(2009,10), rep(2010, 10), rep(2011,10)) x <- rnorm(40) y <- rnorm(40) data <- data.frame(year, x, y) plot(data$x, data$y, type = "n") temp <- subset(data, data$year == 2008); points(temp$x, temp$y, pch = 0) temp <- subset(data, data$year == 2009); points(temp$x, temp$y, pch = 1) temp <- subset(data, data$year == 2010); points(temp$x, temp$y, pch = 2) temp <- subset(data, data$year == 2011); points(temp$x, temp$y, pch = 5)しかし、最後の4行を関数化しようと思い以下の通り変更してみましたが、すべてのデータが全てのプロットマーカーで上書きされていくだけで思い通りにいきません。
point.add <- function (year) { temp <- subset(data, data$year == year) if (year == 2008) { pch = 0 } else if (year == 2009) { pch = 1 } else if (year == 2010) { pch = 2 } else if (year == 2011) { pch = 5 } points(temp$x, temp$y, pch =pch) } point.add(2008) point.add(2009) point.add(2010) point.add(2011)うまくいかない原因がわからないので、どなたかご教授いただけないでしょうか。
よろしくお願いいたします。
year <- c(rep(2008, 10), rep(2009,10), rep(2010, 10), rep(2011,10)) x <- rnorm(40) y <- rnorm(40) data <- data.frame(year, x, y) plot(data$x, data$y, pch = c(0, 1, 2, 5)[data$year-2007])余計なことも不要だし,目的通りのことが実現できます。 -- 河童の屁は,河童にあらず,屁である。 2013-01-07 (月) 10:12:11
HIROi (2013-01-04 (金) 02:48:26)
初めて質問させていただく初心者です。read.csvを使って、カラム数23,行数600万件程度のcsvファイルを読み込もうとしたら、memory sizeの限度らしく失敗しました。実行結果(一部省略)は以下の通りです。EQ <- read.csv("VeryLargeData.csv") エラー: サイズ 45.4 Mb のベクトルを割り当てることができません 追加情報: 14 件の警告がありました (警告を見るには warnings() を使って下さい) > warnings() 警告メッセージ: 1: In scan(file, what, nmax, sep, dec, quote, skip, nlines, ... : Reached total allocation of 1535Mb: see help(memory.size) # 中略・・・全く同じメッセージの繰り返し 9: In type.convert(data[[i]], as.is = as.is[i], dec = dec, ... : Reached total allocation of 1535Mb: see help(memory.size) # 後略・・・全く同じメッセージの繰り返し、14.まで。使用したいカラムは「キーとなるコード1カラム+倍精度小数2カラム=合計3カラム」のみなので、「列を間引いて」読み出せれば用は足ります。しかし、最も柔軟にファイルを読み込めると聞くscanのヘルプを見ても、「最初の数行を間引く」方法はあっても、「カラムのうちいくつかだけを読み出す」方法というのはどうも見当たらないように思われます。どなたかご助言をお願いいただけないでしょうか。何卒よろしくお願いします。
投稿における注意事項
con <- file("iris.csv")
open(con)
repeat {
a <- readLines(con, 1) # 1 行ずつ読んで,
b <- as.numeric(unlist(strsplit(a, ","))[1]) # 自分でパースする
if (length(b) == 0) break
print(b)
}
close(con)$ cut -f2,5,11 -d"," 元.csv > 新.csvとして、新.csvを作成してこれをRに読み込む。 PerlでText::CSVを使ってもOK。 cutを使ったときの処理時間は、下記の程度。
$ wc -l test.csv 6000001 test.csv $ time cut -f2,5,11 -d"," test.csv > test_new.csv real 0m8.175s user 0m7.460s sys 0m0.688sさらに、これをRの中で行うと、もう少し時間がかかる。
> system.time(system('cut -f2,5,11 -d"," test.csv > test_new.csv'))
ユーザ システム 経過
7.492 0.640 10.167 system("gawk 'BEGIN{FS=\",\"; OFS=\",\"};{print $2, $5, $6}' VeryLargeData.csv > extract.csv")read.columns.csv <- function(file, columns = NULL, ...){
if(cmd <- system2("which", args = "cut")) stop("'cut' is not found.")
if(!missing(columns)){
out <- tempfile()
cols <- paste(columns, collapse = ",")
args <- paste("-f", cols, "-d','", file, ">", out)
system2("cut", args = args)
on.exit(file.remove(out))
}else{
out <- file
}
return(read.csv(out, ...))
}
使用例:> head(read.columns.csv("test1.csv", columns = c(2, 5, 11)), 2)
/usr/bin/cut
x1 x4 x10
1 0.1703623 -0.5631763 -0.734945
2 -1.4075415 -0.5450647 -1.758273
カラムを無指定にするとread.csv()と同じ挙動(本番ではhead()はもちろん不要です)。
> head(read.columns.csv("test1.csv"), 2)
/usr/bin/cut
id x1 x2 x3 x4 x5 x6 x7
1 1 0.1703623 0.2198511 -1.1616084 -0.5631763 -0.7652493 1.0642631 1.0568743
2 2 -1.4075415 0.5162073 -0.7501079 -0.5450647 -1.5637246 0.8976396 0.6194357
x8 x9 x10 x11 x12 x13 x14
1 1.6138930 0.41030405 -0.734945 1.13511914 0.77816141 0.04144169 0.262109
2 0.3551278 -0.08129068 -1.758273 -0.05866285 -0.09313449 -1.39641638 1.064553
x15 x16 x17 x18 x19 x20 x21
1 1.1631931 -0.6981225 -0.4869349 0.2129322 0.04856178 -0.2846752 -0.9029536
2 -0.9422174 0.7154750 0.7995136 -1.6102600 -2.92610442 -1.0668136 -1.8331052
x22 x23
1 0.6818277 0.4078844
2 -1.1319291 -1.0523440
cutコマンドがない環境でも、SQLiteがある環境ならsqldfパッケージのread.csv.sql()を使える模様(驚くほど短時間で600万行を読み込めた)。なお、R-ForgeにはColByColというテキストファイルの一部だけ読み込む専用パッケージもあるみたい(未検証)。tomo (2013-01-02 (水) 13:00:50)
閲覧ありがとうございます。
私はR言語で学習をしている初心者です。
その中で、irisデータを用いてコマンドの学習をしているのですがエラーが出てしまい先に進めなくなってしまいました。
以下にコマンドを載せますものが私の用いたものです。library(rpart) library(adabag) data <- iris ndata <- nrow(data) #乱数指定 set.seed(101) #学習データ(data.learn)とテストデータ(data.test)に分ける ridx <- sample(ndata, ndata * 0.5) data.learn <- data[ridx,] data.test <- data[-ridx,] #3-fold crossvalidation、弱識別器の数を10とし、 #学習データに対しboostingを行ったものをdata.adaCvに代入 data.adaCv <- boosting.cv(Species ~ .,data = data.learn,v=3, mfinal = 10) #テストデータに学習データを照らし合わせる。 resultPredict <- predict(data.adaCv, newdata = data.test, type="class")というコマンドですが「predict」を行った際に
以下にエラー UseMethod("predict") : 'predict' をクラス "character" のオブジェクトに適用できるようなメソッドがありませんというエラーが出てしまい、ネットや参考文献を見てもわからず投稿させていただきました。
このエラーの解決方法がお分かりのかたがいらっしゃいましたらよろしくお願いします。
お分かりの方いらっしゃいましたら例文等を添えてコメントいただけたら幸いです。よろしくお願いします。
> debug(predict)
> resultPredict <- predict(data.adaCv, newdata = data.test, type="class")
debugging in: predict(data.adaCv, newdata = data.test, type = "class")
debug: UseMethod("predict")
Browse[2]>
debugging in: predict.list(data.adaCv, newdata = data.test, type = "class")
debug: {
out <- lapply(object, predict, ... = ...)
if (!is.null(names(object)))
names(out) <- names(object)
out
}
Browse[3]>
debug: out <- lapply(object, predict, ... = ...)
Browse[3]>
debugging in: FUN(X[[1L]], ...)
debug: UseMethod("predict")
Browse[4]>
以下にエラー UseMethod("predict") :
'predict' をクラス "character" のオブジェクトに適用できるようなメソッドがありません library(rpart) library(adabag) data <- iris ndata <- nrow(data) # 乱数指定~ set.seed(101) # 学習データ(data.learn)とテストデータ(data.test)に分ける ridx <- sample(ndata, ndata * 0.5) data.learn <- data[ridx,] data.test <- data[-ridx,] # 弱識別器の数を10、3-fold crossvalidation?学習データに対しboostingを行ったものをdata.adaCvに代入 data.adaCv <- boosting(Species ~ .,data = data.learn, mfinal = 10,control=rpart.control(xval=3)) # テストデータに学習データを照らし合わせる。 resultPredict <- predict(data.adaCv, newdata = data.test, type="class") resultPredict・実行結果
$confusion Observed Class
Predicted Class setosa versicolor virginica
setosa 26 0 0
versicolor 0 17 3
virginica 0 1 28
$error
[1] 0.05333333
しかし私は先に述べたように交差検定の数をxvalの引数を変えることで変更できると認識していたのですが、xvalの引数を例えば3、5、10と変更しても実行結果に変化がありませんでした。ですがrpartのパッケージのxvalの説明には「number of cross-validations.」と書いてありました。そこで私が質問させていただきたいことはS (2012-12-28 (金) 11:29:56)
質問を書き込んでいるうちに自己解決しましたので、質問というより、同じ失敗をする人がでないように報告します。> Sys.Date() [1] "2012-12-28"今日は12月28日です。1月27日まで何日あるのか計算しました。
> difftime("2013-01-27", Sys.Date()) Time difference of 29.625 days > difftime("2013-01-27", "2012-12-28") Time difference of 30 daysSys.Date()を使った方はきっちり30日になりません。
> str(Sys.Date()) Date[1:1], format: "2012-12-28"Sys.Date()の出力はDateクラスで"2012-12-28"を持っているだけです。
> difftime("2013-01-27", as.Date("2012-12-28")) Time difference of 29.625 days2012-12-28をDateクラスにすると再現します。units = "days"を付けても変化はありません。
> difftime("2013-01-27", as.Date("2012-12-28"), tz = "JST") Time difference of 30 daysタイムゾーンを指定すると30日に戻りました。バックエンドで時間差計算を行ってそのままモデルに放り込んでいるとなかなか気がつかないエラーだと思います(もしかすると、私だけ?)。
yoro (2012-12-27 (木) 09:03:34)
お世話になります。
Windows7 (32或いは64bit)にて R 2.15.1 を使いっています。
お聞きしたいのは、hogehoge.R というようなスクリプトファイルをダブルクリックすることで、Rを起動させスクリプトを実行させる事は可能でしょうか?(バッチモードとしてではなく)
宜しくお願いします。
こさく (2012-12-26 (水) 18:42:17)
いつもお世話になっていますが、初めて投稿します。統計も素人ですがご容赦ください。
2群(a、b群各30名)で、それぞれ直線回帰できるようなデータがあるとします。実際のデータでは平均値には有意な差はなく、a、b群での交互作用(傾きの差)が主な関心事です。
下記は作ったデータですが、yをxとgrpでモデル化しようとしています。下記のモデル1-4のうちモデル1を採用したいと思いましたが、残念ながらgrp:xの有意差が出ませんでした。
ただ結果を見るとgrp:xの自由度が2になっています。浅学ですが、下記モデル2に示されるようにx、grpの自由度がそれぞれ1で、grp:xの自由度も1*1=1でよいのではと思ってしまいます。
他の統計ソフト(JMP)で解析すると、モデル1におけるgrp:xの自由度は期待通り1になっていました。近くの統計の先生にもモデル1におけるgrp:xの自由度は1でいいのではないかと言われました。
都合のいい結果を採用してもよいのですが、もう少し掘り下げることができればと思い投稿させていただきました。
何かコメント等ご指摘いただければ幸いです。
よろしくお願いいたします。set.seed(1234) # grp "a" のデータ x1 <- runif(30) y1 <- runif(30)+x1*0.3 # grp "b" のデータ x2 <- runif(30) y2 <- runif(30)-x2*0.3 x <- c(x1, x2) y <- c(y1, y2) # grp (2群のラベル) grp <- factor(c(rep('a', 30), rep('b', 30)))library(car) # type3 を指定するのは、問題とは関係ないですが
# ここからが問題 # モデルの固定因子を変えると、交互作用項の自由度が変わる # モデル 1、grp:x の自由度 2 > Anova(lm(y ~ grp+grp:x), type=3) Anova Table (Type III tests) Response: y Sum Sq Df F value Pr(>F) (Intercept) 1.5509 1 20.7241 2.904e-05 *** grp 0.1585 1 2.1178 0.15118 grp:x 0.3955 2 2.6423 0.08006 . Residuals 4.1908 56 # モデル 2、grp:x の自由度 1 > Anova(lm(y ~ x+grp+grp:x), type=3) Anova Table (Type III tests) Response: y Sum Sq Df F value Pr(>F) (Intercept) 1.5509 1 20.7241 2.904e-05 *** x 0.3939 1 5.2640 0.02554 * grp 0.1585 1 2.1178 0.15118 x:grp 0.2338 1 3.1245 0.08257 . Residuals 4.1908 56 # モデル 3、grp:x の自由度 1 > Anova(lm(y ~ x+grp:x), type=3) Anova Table (Type III tests) Response: y Sum Sq Df F value Pr(>F) (Intercept) 2.0111 1 26.357 3.573e-06 *** x 1.2837 1 16.823 0.0001318 *** x:grp 2.1557 1 28.251 1.843e-06 *** Residuals 4.3493 57 # モデル 4、grp:x の自由度 2 > Anova(lm(y ~ grp:x), type=3) Anova Table (Type III tests) Response: y Sum Sq Df F value Pr(>F) (Intercept) 2.0111 1 26.357 3.573e-06 *** grp:x 2.4071 2 15.773 3.533e-06 *** Residuals 4.3493 57
- それぞれ,Anova(lm(y ~ ほげほげ), type=3) というのを summary(lm(y ~ ほげほげ)) というようにやってみれば,どのようなパラメータ,幾つのパラメータが推定されているかがわかり,その数と Df がどういう関係になっているかがわかるでしょう。 -- 河童の屁は,河童にあらず,屁である。 2012-12-26 (水) 19:33:36
- コメントありがとうございます。summaryで出てくるパラメータの数とDfが対応していることがわかりました。なぜモデルにより交互作用項のパラメータの数が変わるかが謎ですが、とっかかりができたのでもう少し勉強してみます。またわからなかったらすいませんがアドバイスお願いいたします。ありがとうございました。 -- こさく 2012-12-27 (木) 16:43:49
りすりす (2012-12-26 (水) 12:17:20)
お世話になっております。
windowsでRを使用しています。
Rで、最尤法による共分散構造分析ができるパッケージはどれなのでしょうか?
semパッケージは、デフォルトで最尤法なのでしょうか?
どなたかわかるかたがいらっしゃれば、ご教授をお願いいたしす。
Saito (2012-12-21 (金) 21:29:04)
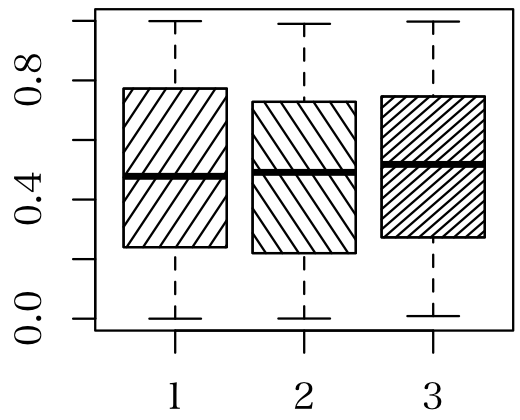
いつもお世話になっております。
boxplot()の図に斜線を入れたいのですが、どうすれば良いでしょうか。boxplot(runif(100), runif(200), runif(300))のそれぞれのグラフで、斜線の種類も変えられると嬉しいですが、とりあえず斜線が引きたいです。
どなたかご存知の方がいらっしゃいましたら、ご教授をお願い致します。
a <- boxplot(runif(100), runif(200), runif(300))
angle.p <- c(45, 135, 30)
density.p <- c(10, 10 , 20)
for (i in 1:3) {
rect(i-0.4, a$stats[2, i], i+0.4, a$stats[4, i],
angle=angle.p[i], density=density.p[i])
}
a <- boxplot(runif(100), runif(200), runif(300), ylim=c(-1, 3))
angle.p <- c(45, 135, 30)
density.p <- c(10, 10 , 20)
for (i in 1:3) {
rect(i-0.4, a$stats[2, i], i+0.4, a$stats[4, i],
angle=angle.p[i], density=density.p[i])
}
legend(1, 3, c("100", "200", "300"), lty=1)
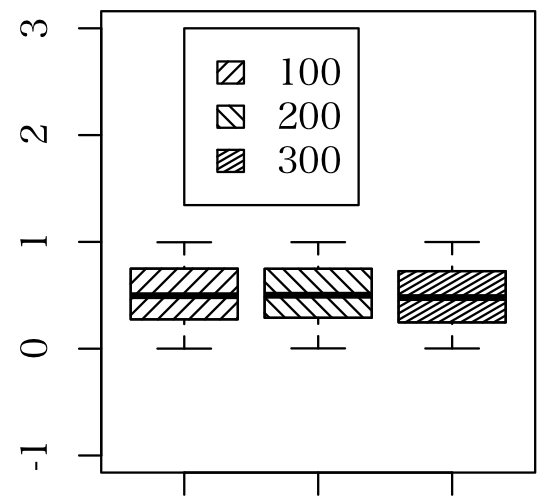
などとしたときに、legendの横にも同じ斜線の小さな四角、など表示出来るでしょうか?追加の形になってしまい申し訳ありません。どうぞよろしくお願い致します。 -- Saito 2012-12-21 (金) 22:11:50a <- boxplot(runif(100), runif(200), runif(300), ylim=c(-1, 3))
angle.p <- c(45, 135, 30)
density.p <- c(20, 20 , 40)
for (i in 1:3) {
rect(i-0.4, a$stats[2, i], i+0.4, a$stats[4, i],
angle=angle.p[i], density=density.p[i])
}
legend(1, 3, c("100", "200", "300"), angle=angle.p,
density=density.p)
BCD (2012-12-19 (水) 23:02:56)
いつもお世話になっております。
barplot関数を使ってグラフを作成した時にy軸に自動的に付加される値を取得したいです。
というのも小数点が多くつく場合、y軸が見づらいので10-3乗として表記をしたいからです。
barplotを使う前に元のデータに10の3乗をかけ算しても良いのですが、上記の方法は無いのか知りたいのです。
どうかご教示願います。
sori (2012-12-19 (水) 14:24:56)
データ解析の勉強を始めたのですが、なぜ警告メッセージが出るのかがわからないので困っています。どなたかよろしくお願いします。> x <- read.delim("clipboard") > x 学生名 性別 順位 国語 数学 英語 1 佐藤 1 5 64 48 78 2 鈴木 1 6 51 65 62 3 高橋 1 4 57 78 68 4 田中 1 14 38 62 42 5 渡辺 0 15 43 78 57 6 伊藤 0 13 52 73 53 7 山本 0 3 58 45 50 8 中村 0 1 72 36 71 9 小林 0 16 65 48 59 10 斉藤 1 17 53 58 35 11 加藤 0 12 30 55 53 12 吉田 0 18 56 72 49 13 山田 1 2 85 65 73 14 佐々木 1 19 69 62 66 15 山口 0 20 52 68 45 16 松本 0 11 55 52 63 17 井上 0 8 45 54 56 18 木村 1 7 61 32 88 19 林 1 10 43 57 43 20 清水 1 9 76 88 97 > attach(x) > 性別 [1] 1 1 1 1 0 0 0 0 0 1 0 0 1 1 0 0 0 1 1 1 > class(性別) [1] "numeric" > class <- factor(性別) > class(性別) [1] "numeric" > 性別 <- factor(性別) > class(性別) [1] "factor" > mean(x) 学生名 性別 順位 国語 数学 英語 NA 0.50 10.50 56.25 59.80 60.40 警告メッセージ: 1: mean(<data.frame>) is deprecated. Use colMeans() or sapply(*, mean) instead. 2: In mean.default(X[[1L]], ...) : 引数は数値でも論理値でもありません。NA 値を返します > mean(国語) [1] 56.25 > mean(x[,4:6]) 国語 数学 英語 56.25 59.80 60.40 警告メッセージ: mean(<data.frame>) is deprecated. Use colMeans() or sapply(*, mean) instead.
so (2012-12-19 (水) 13:50:56)
下記計算結果が導出される理由が分からず困っています。
ご教示いただけると幸いです。> floor( c( 230, 2.3*100, 2.3*10*10 ) ) [1] 230 229 230 > floor( c( 230000, 2.3*100000, 2.3*100*1000, 2.3*1000*100 ) ) [1] 230000 229999 229999 230000
> sprintf("%a", 2.3)
[1] "0x1.2666666666666p+1"
> sprintf("%a", 2.3*100)
[1] "0x1.cbfffffffffffp+7"
> sprintf("%a", 230)
[1] "0x1.ccp+7"
> sprintf("%a", 2.3*10)
[1] "0x1.7p+4"
> sprintf("%a", 23)
[1] "0x1.7p+4"
> sprintf("%a", 0.125)
[1] "0x1p-3"BCD (2012-12-17 (月) 12:44:46)
いつもお世話になっております。
グラフを描く時に、lasオプションを指定すればラベルの向きを変更することができますが、軸に対して斜め(45℃くらい角度をつける)にラベルを配置することはできないのでしょうか?
どうかご教示願います。
set.seed(1234) a <- barplot(runif(12)) text(a, -.01, month.name, srt = 45, adj = c(1, 1), xpd = TRUE)
a <- hist(rnorm(1000), xaxt="n", xlab="", main="") text(a$breaks+0.1, -2.5, a$breaks, srt=45, pos=1, xpd=TRUE)
library(plotrix)
set.seed(1234)
plot(runif(12), runif(12), xlab = "", xlim = c(0, 1),
ylim = c(0, 1), axes = FALSE)
ly <- round(c(0, (1:12)/12), 3)
lx <- ly[-13]
axis(1, at = lx, labels = rep("", 12), tck = -0.02)
staxlab(1, lx, month.name, srt = 45)
axis(2, at = ly, labels=rep("", 13))
staxlab(2, ly, ly, srt = 45)
box()yamaguchi (2012-12-16 (日) 17:27:44)
はじめまして。いつもお世話になっています。
初投稿という事で至らない点もあるかと思いますが、よろしくお願いします。
dealパッケージで多クラスのカテゴリデータでベイジアンネットワークを作成しようとしたところ、autosearch関数で下記のエラーが発生しました。>autosearch(dat.pre_net, dat.dummy, dat.prior) 以下にエラー apply(prior$jointalpha, didx, sum) : dim(X) must have a positive lengthどのように解決したらよいか、ご助言をいただけますか。
以下実行手順です。# サンプルとして成績データ作成 eng <- c("A", "B", "C", "C", "B", "D") math <- c("B", "B", "C", "A", "B", "D") sci <- c("A", "A", "B", "D", "B", "B") res <- c("A", "B", "C", "C", "B", "C") dat <- data.frame(eng, math, sci, res) dat eng math sci res 1 A B A A 2 B B A B 3 C C B C 4 C A D C 5 B B B B 6 D D B C青木氏のサイトのqt3, make.dummyを参考にして、カテゴリデータをダミーデータに変換。
上記サンプルではダミーデータに変換しなくてもネットワークを描画できたのですが、自分のデータではjointprior関数を実行する際に "too large than due to tequnical limitations in R"というエラーが起き、オブジェクトのサイズを減らすためにダミーデータに変換することにしました。dat.int <- dat dat.int[,1:ncol(dat)] <- lapply(dat, as.integer) # カテゴリを数値に変換 cat <- sapply(dat.int, max) # 各列が持つ水準の個数を保存 cname <- unlist(sapply(dat.int, levels)) # 水準の名称を保存 dat.colnames <- colnames(dat.int) dat.dummy <- make.dummy(dat.int) colnames(dat.dummy) <- paste(rep(dat.colnames, cat), cname, sep=".") # 列名.水準 というカラム名を設定 library('deal') dat.pre_net <- network(dat.dummy) dat.prior <- jointprior(dat.pre_net, 20) dat.dist <- learn(dat.pre_net, dat.dummy, dat.prior) # 有効辺ノードの設定 res_list <- grep("res.*", colnames(dat.dummy)) # このノードからは有効辺を引かない [1] 12 13 14 # res → res以外への有効辺を引かないようにする dat.col_num <- 1:length(colnames(dat.dummy)) # 各ノードを数字のベクトルで取得 dat.no_res <- length(dat.col_num) - length(res_list) # resがつかないカラム名の個数を取得 a <- sort(rep(res_list,length(colnames(dat.dummy)))) # res_listの各要素をカラム数分繰り返したベクトルを # ソート(有効辺が出てはいけないノード) b <- 1:length(colnames(dat.dummy)) # 有効辺が入るノード dat.ban <- cbind(a, b) # 12,12などの重複を削除 dat.ban_df <- as.data.frame(dat.ban) colnames(dat.ban_df) <- c("a", "b") dat.ban_df[dat.ban_df$a == dat.ban_df$b,] <- c(NA, NA) dat.ban_df <- na.omit(dat.ban_df) # 有効辺を引かないノードリストの設定を適用 dat.pre_net <- getnetwork(dat.dist) banlist(dat.pre_net) <- as.matrix(dat.ban_df) # モデル探索 dat.posterior <- autosearch(dat.pre_net, dat.dummy, dat.prior) 以下にエラー apply(prior$jointalpha, didx, sum) : dim(X) must have a positive lengthdebug関数でautosearch()の処理を追って見ましたが、私には原因がわかりませんでした。
読みづらい点もあるかと思いますが、対処策・アドバイスのほど、よろしくお願いします。
また、実行環境は下記のとおりです。>sessionInfo() R version 2.14.1 (2011-12-22) Platform: x86_64-pc-linux-gnu (64-bit) locale: [1] LC_CTYPE=ja_JP.UTF-8 LC_NUMERIC=C [3] LC_TIME=ja_JP.UTF-8 LC_COLLATE=ja_JP.UTF-8 [5] LC_MONETARY=ja_JP.UTF-8 LC_MESSAGES=ja_JP.UTF-8 [7] LC_PAPER=C LC_NAME=C [9] LC_ADDRESS=C LC_TELEPHONE=C [11] LC_MEASUREMENT=ja_JP.UTF-8 LC_IDENTIFICATION=C attached base packages: [1] stats graphics grDevices utils datasets methods base other attached packages: [1] deal_1.2-35
t.h (2012-12-13 (木) 17:28:14)
はじめまして。よろしくお願いします
題名の通りですが、package(e1071)におけるsvmについて質問があります。
私の最終的な目的は「1つのクラスに対して間違いを許さない2クラス判別を行うsvmをつくる」です。
(クラスAとBがあるとして、テストデータを入れたときにAがBと判定されることは許すが、BがAと判定することは許されないSVM、という意味です)
(1)このsvmに訓練データとしてデータを入れるとき、その順番は関係あるのでしょうか?
(e.g)「クラスA,クラスBという順番で入っている行列を訓練データと与えたとき」と、「クラスB,クラスAという順番で入っている行列を訓練データと与えたとき」に構成されるSVMは違うものになるのでしょうか?
(2)このsvmはデフォルトで実行すると、ソフトマージンsvm(最大マージンsvm)のようですが、ハードマージンsvmにするにはどのようにすればよいでしょうか?
(3)目的を達成するために、デフォルトのSVMからどのようにすればよいでしょうか?
(4*)デフォルトのsvmに3クラス以上のデータを訓練させた場合は1vs1式でのクラス分けなのか、1vsOtrhers式でのクラス分けなのかを教えていただきたいです
段々と抽象性の高い質問になっておりますが、((4)だけ別系統の質問です)最終的な目的を達成するにあたり、有用であればどの様な回答でも構いません。
見当違いの質問、読みづらい文章かもしれませんが、ご回答よろしくお願いします。
しまりす (2012-12-07 (金) 19:14:06)
お世話になります。
SEMパッケージの練習のため、下記URLの「食物とガン」の共分散分析を自分でやってみました。
http://wwwhum.meijo-u.ac.jp/labs/hh002/r/content/sem.html
共分散の結果は出せたのですが、最後のsummary()による適合度の出力ができませんでした。
summary(データ名)と入力しても、何も出力されず、エラーメッセージも出ません。
以下がプログラムと出力結果です。> # ガンデータのSEM # > > setwd("C:/Users/a2122062/work") > gandat <- read.table("gan.csv", sep=",", header = TRUE) > > # 相関行列を求める # > soukan <- cor(gandat) > soukan calory niku nyu sake daicho chokucho calory 1.0000000 0.7731109 0.7155250 0.6341241 0.7394903 0.8100869 niku 0.7731109 1.0000000 0.7227997 0.4515933 0.8526245 0.6718607 nyu 0.7155250 0.7227997 1.0000000 0.2321334 0.5787253 0.4785180 sake 0.6341241 0.4515933 0.2321334 1.0000000 0.5881328 0.6095649 daicho 0.7394903 0.8526245 0.5787253 0.5881328 1.0000000 0.7523879 chokucho 0.8100869 0.6718607 0.4785180 0.6095649 0.7523879 1.0000000 > > # 共分散する# > library(sem) 要求されたパッケージ MASS をロード中です 要求されたパッケージ matrixcalc をロード中です 警告メッセージ: 1: パッケージ '‘sem’' はバージョン 2.15.2 の R の下で造られました 2: パッケージ '‘matrixcalc’' はバージョン 2.15.2 の R の下で造られました > modelA.gan <- specify.model() 1: 洋食傾向 -> calory ,b11 ,NA 2: 洋食傾向 -> niku ,b21 ,NA 3: 洋食傾向 -> nyu ,b31 ,NA 4: 洋食傾向 -> sake ,b41 ,NA 5: 消化管ガン -> daicho ,NA ,1 6: 消化管ガン -> chokucho ,b62 ,NA 7: 洋食傾向 -> 消化管ガン,g21,NA 8: calory <-> calory ,e1 ,NA 9: niku <-> niku ,e2 ,NA 10: nyu <-> nyu ,e3 ,NA 11: sake <-> sake ,e4 ,NA 12: daicho <-> daicho ,e5 ,NA 13: chokucho <-> chokucho ,e6 ,NA 14: 洋食傾向 <-> 洋食傾向 , NA , 1 15: 消化管ガン <-> 消化管ガン , delta2,NA 16: Read 15 records 警 'specify.model' is deprecated. Use 'specifyModel' instead. See help("Deprecated") and help("sem-deprecated"). > > semgan <- sem(modelA.gan , soukan, N=47) > std.coef(semgan, digit=3) Std. Estimate 1 b11 0.90353083 calory <--- 洋食傾向 2 b21 0.88860193 niku <--- 洋食傾向 3 b31 0.72052466 nyu <--- 洋食傾向 4 b41 0.61993727 sake <--- 洋食傾向 5 0.89590512 daicho <--- 消化管ガン 6 b62 0.83980749 chokucho <--- 消化管ガン 7 g21 0.98052591 消化管ガン <--- 洋食傾向 8 e1 0.18363205 calory <--> calory 9 e2 0.21038661 niku <--> niku 10 e3 0.48084422 nyu <--> nyu 11 e4 0.61567779 sake <--> sake 12 e5 0.19735401 daicho <--> daicho 13 e6 0.29472337 chokucho <--> chokucho 14 1.00000000 洋食傾向 <--> 洋食傾向 15 delta2 0.03856894 消化管ガン <--> 消化管ガン 警告メッセージ: 'std.coef' is deprecated. Use 'stdCoef' instead. See help("Deprecated") and help("sem-deprecated"). > summary(semgan) >なぜなのでしょうか?
もしかしたらとても簡単なミスかもしれないのですが、どうぞよろしくお願いいたします。
さとし (2012-12-04 (火) 21:46:32)
お世話になります。Windows7のR 2.15.2を使っております。
lavaanパッケージで多母集団分析を行いました。lav.model.1 <- lavaan(model.1, data=d1, model.type="sem", group="group", int.ov.free=TRUE, auto.var=TRUE, auto.fix.first=TRUE, fixed.x=FALSE, auto.cov.lv.x=FALSE, auto.cov.y=FALSE) summary(lav.model.1, fit.measure=TRUE, standardized=TRUE)このようなオプションでsummaryを出力したところ,色々な適合度が表示されるのですが,GFIやAGFIが出力されません。
そこで質問なのですが,
1.GFIやAGFIの出力を可能にするオプションはあるのでしょうか?
2.GFIやAGFIの算出に必要な共分散行列を出力することはできるのでしょうか?
3.あるいは,出力された他の適合度指標からGFIやAGFIを式展開などで導出することは可能でしょうか?
よろしくお願いいたします。
moreFitIndices(lav.model.1)すると,nfi,ifi,gfi*,agfi*,ciac,ecvi,bic*,hqc,sicが出力されました。
れこ (2012-12-04 (火) 15:45:58)
お世話になります。WindowsのR 2.15.1を使っています。y <- rnorm(20) x <- factor(rep(c("AAAAA","BBBBB"),10)) stripchart(y ~ x, vertical=TRUE)このとき、x軸のラベルを反時計回りに90度回転させたグラフにしたいのですが、どのようにすればよいでしょうか?
宜しくお願いします。
y <- rnorm(20)
x <- factor(rep(c("AAAAA","BBBBB"),10))
stripchart(y ~ x, vertical=TRUE, las=2)Saito (2012-12-04 (火) 11:34:01)
いつもお世話になっております。
ある生物が、時間が経つに連れて大きくなり、一定時間が過ぎると子供を産んで(子の数は固定)次の新しい年級群が加入し、自然に死んだりたまに捕獲される、という生活史をシミュレーションしたいと思っています。ただ、プログラムに問題があり、大変時間がかかります。下記は私が作った例なのですが、新規に年級群が入るというイベントをif文で書いてしまっています。 実際には、時間も年級群の数ももっとべらぼうに多いですし、パラメタの値を色々変えて検討したいので、素早く計算出来るよう今よりも高速化をしたいと考えております。しかし、私の文法がへたくそなせいで、これ以上は高速化が出来ません。どなたかご教授頂けますと幸いです。Size <- seq(0.1, 30, by=0.5) #個体のサイズ分布 year <- 40 #時間 class <- 3 #生物の年級群の数 M <- catch <- matrix(0, ncol=year*class, nrow=length(Size)) ###Catchは既知のデータ### catch[, 5] <- rpois(length(Size), lambda=2) catch[, 10] <- rpois(length(Size), lambda=3) catch[, 56] <- rpois(length(Size), lambda=4) catch[, 77] <- rpois(length(Size), lambda=5) catch[, 105] <- rpois(length(Size), lambda=6) SS1 <- matrix(NA, nrow=length(Size), year*class) SS2 <- matrix(NA, nrow=length(Size), year*class) SS3 <- matrix(NA, nrow=length(Size), year*class) m1 <- runif(year*class, 0, 1) #平均成長の値(時間変化する) m2 <- runif(year*class, 5, 10) m3 <- runif(year*class, 15, 20) sd1 <- runif(year*class, 1, 5) #平均成長の分散(時間変化する) sd2 <- runif(year*class, 1, 3) sd3 <- runif(year*class, 1, 2) surv1 <- runif(year*class, 0.9, 0.99) #生存率(時間変化する) surv2 <- runif(year*class, 0.9, 0.99) surv3 <- runif(year*class, 0.9, 0.99) ###シミューション開始### M[1, 1] <- S2 <- S3 <- 40000 for(i in 2 : (year*class)){ if (i <= year) { A1 <- dnorm(Size, mean=m1[i], sd=sd1[i]) G1 <- sample(Size, sum(M[, i-1]), prob=A1/sum(A1), rep=T) M[, i] <- SS1[, i] <- rbinom(rep(1, length(Size)), table(cut(G1, breaks=c(Size, max(Size) + 0.5))), prob=surv1[i]) M[, i] <- M[, i] - catch[, i] M[which(M[, i] < 0), i] <- 0 S1 <- M[, i] } else { #次の年級群が加わってくる if (i > year & i <=(year*2+13)) { A1 <- dnorm(Size, mean=m1[i], sd=sd1[i]) A2 <- dnorm(Size, mean=m2[i], sd=sd2[i]) G1 <- sample(Size, sum(S1), prob=A1/sum(A1), rep=T) G2 <- sample(Size, sum(S2), prob=A2/sum(A2), rep=T) S1 <- SS1[, i] <- rbinom(rep(1, length(Size)), table(cut(G1, breaks=c(Size, max(Size) + 0.5))), prob=surv1[i]) S2 <- SS2[, i] <- rbinom(rep(1, length(Size)), table(cut(G2, breaks=c(Size, max(Size) + 0.5))), prob=surv2[i]) S1 <- S1 - round(catch[, i]*(SS1[, i]/(SS1[, i]+SS2[, i]))) #それぞれの年級群の数に比例した値で捕獲 S2 <- S2 - round(catch[, i]*(SS2[, i]/(SS1[, i]+SS2[, i]))) S1[which(is.na(S1))] <- 0 S2[which(is.na(S2))] <- 0 M[, i] <- S1 + S2 M[which(M[, i] < 0), i] <- 0 } else { #また次の年級群が加わってくるが、加わるタイミングが違う if (i > (year*2+13)) { A1 <- dnorm(Size, mean=m1[i], sd=sd1[i]) A2 <- dnorm(Size, mean=m2[i], sd=sd2[i]) A3 <- dnorm(Size, mean=m3[i], sd=sd3[i]) G1 <- sample(Size, sum(S1), prob=A1/sum(A1), rep=T) G2 <- sample(Size, sum(S2), prob=A2/sum(A2), rep=T) G3 <- sample(Size, sum(S3), prob=A3/sum(A3), rep=T) S1 <- SS1[, i] <- rbinom(rep(1, length(Size)), table(cut(G1, breaks=c(Size, max(Size) + 0.5))), prob=surv1[i]) S2 <- SS2[, i] <- rbinom(rep(1, length(Size)), table(cut(G2, breaks=c(Size, max(Size) + 0.5))), prob=surv2[i]) S3 <- SS3[, i] <- rbinom(rep(1, length(Size)), table(cut(G3, breaks=c(Size, max(Size) + 0.5))), prob=surv3[i]) S1 <- S1 - round(catch[, i]*(SS1[, i]/(SS1[, i]+SS2[, i]+SS3[, i]))) S2 <- S2 - round(catch[, i]*(SS2[, i]/(SS1[, i]+SS2[, i]+SS3[, i]))) S3 <- S3 - round(catch[, i]*(SS3[, i]/(SS1[, i]+SS2[, i]+SS3[, i]))) S1[which(is.na(S1))] <- 0 S2[which(is.na(S2))] <- 0 S3[which(is.na(S3))] <- 0 M[, i] <- S1 + S2 + S3 M[which(M[, i] < 0), i] <- 0 }}}} ###シミューション終わり### ###得たいもの1### SS1[is.na(SS1)] <- 0 SS2[is.na(SS2)] <- 0 SS3[is.na(SS3)] <- 0 SS <- SS1 + SS2 + SS3 SS[, c(5, 10, 56, 77, 105)] #(既知データである)捕獲があった時点での #捕獲前の推定個体数の総和 ###得たいもの2### M #捕獲を考慮して計算を続けたときの推定個体数
for (i in 2:year) {
最初の年級群
}
for (i in (year+1):(year * 2 + 13)) {
次の年級群
}
for (i in (year * 2 + 14):(year * class)) {
また次の年級群
}
もっとも,そのようにしても全然速くならない。当然だ,if-else 判定はたかだか 120 回しか行われないのだから,大勢に影響はない(結果が同じになることは確かめた)。もっと他の箇所を最適化しないと。 -- 河童の屁は,河童にあらず,屁である。 2012-12-04 (火) 13:11:09A1 <- dnorm(Size, mean=m1[i], sd=sd1[i]) A2 <- dnorm(Size, mean=m2[i], sd=sd2[i]) A3 <- dnorm(Size, mean=m3[i], sd=sd3[i])を
A123 <- mapply(function(m, s) dnorm(Size, m, s),
c(m1[i], m2[i], m3[i]), c(sd1[i], sd2[i], sd3[i]))
とするようなこと。平均値ベクトル,標準偏差ベクトルを c でつながなくてもよいように, m1, m2, m3 をひとまとめに行列で用意するなどというのも無意味でしょうね。結局,速度は全く影響を受けないでしょう。S1 <- S1 - round(catch[, i]*(S1/(S1+S2))) S2 <- S2 - round(catch[, i]*(S2/(S1+S2))) S3 <- S3 - round(catch[, i]*(S3/(S1+S2)))などは,S1, S2, S3 をベクトルではなく行列として使えばよいかも。おっと,S2 を計算するときには,直前に更新された S1 の結果を使うのですか(S3 も同じく?)... -- 河童の屁は,河童にあらず,屁である。 2012-12-04 (火) 13:57:56
S1 <- S1 - round(catch[, i]*(SS1[, i]/(SS1[, i]+SS2[, i]+SS3[, i])))のように、保存しておいた更新前のものを使ってcatchの影響をそれぞれの年級群に与えます。-- Saito 2012-12-04 (火) 14:17:49
catch[, 5] <- rpois(length(Size), lambda=2) catch[, 10] <- rpois(length(Size), lambda=3) catch[, 56] <- rpois(length(Size), lambda=4) catch[, 77] <- rpois(length(Size), lambda=5) catch[, 105] <- rpois(length(Size), lambda=6)は
catch[, c(5, 10, 56, 77, 105)] <- sapply(2:6,
function(lambda) rpois(length(Size), lambda=lambda))
# 2:6 の部分も実際は c(xxx, yyy, zzz,...) みたいなことだろうから
また,
m1 <- runif(year*class, 0, 1) #平均成長の値(時間変化する) m2 <- runif(year*class, 5, 10) m3 <- runif(year*class, 15, 20) sd1 <- runif(year*class, 1, 5) #平均成長の分散(時間変化する) sd2 <- runif(year*class, 1, 3) sd3 <- runif(year*class, 1, 2)は
m123 <- mapply(function(min, max) runif(year*class, min, max),
c(0, 5, 15), c(1, 10, 20))
sd123 <- mapply(function(min, max) runif(year*class, min, max),
c(1, 1, 1), c(5, 3, 2))
とか。そうすれば,前に書いた
A123 <- mapply(function(m, s) dnorm(Size, m, s),
c(m1[i], m2[i], m3[i]), c(sd1[i], sd2[i], sd3[i]))
は
A123 <- mapply(function(m, s) dnorm(Size, m, s), m123[i,], sd123[i,])のようになる。
a <- b <- c <- d <- matrix(NA, 10, 6)
for(i in 1 : 2) {
a[, i] <- rnorm(10, mean=0, sd=1)
d[, i] <- a[, i]
}
for(i in 3 : 4) {
a[, i] <- rnorm(10, mean=1, sd=2)
b[, i] <- rnorm(10, mean=0, sd=1)
d[, i] <- a[, i] + b[, i]
}
for(i in 5 : 6){
a[, i] <- rnorm(10, mean=2, sd=3)
b[, i] <- rnorm(10, mean=1, sd=2)
c[, i] <- rnorm(10, mean=0, sd=1)
d[, i] <- a[, i] + b[, i] + c[, i]
}
#このくらいしか思いつきません。for()消せないですかねぇ・・・↓
d <- matrix(NA, 10, 6)
for(i in 1 : 2) {
d[, i] <- rnorm(10, mean=0, sd=1)
}
for(i in 3 : 4) {
d[, i] <- apply(mapply(function(mean, sd)
rnorm(10, mean, sd), c(1, 0), c(2, 1)), 1, sum)
}
for(i in 5 : 6){
d[, i] <- apply(mapply(function(mean, sd)
rnorm(10, mean, sd), c(2, 1,0), c(3, 2,1)), 1, sum)
}set.seed(1234567)
Mean <- matrix(c(
# a b c
0, 0, 0, # i = 1
0, 0, 0, # i = 2
1, 0, 0, # i = 3
1, 0, 0, # i = 4
2, 1, 0, # i = 5
2, 1, 0), # i = 6
byrow=TRUE, ncol=3)
SD <- matrix(c(
1, 0, 0,
1, 0, 0,
2, 1, 0,
2, 1, 0,
3, 2, 1,
3, 2, 1),
byrow=TRUE, ncol=3)
シミュレーションデータは 3 次元の配列として用意する。n <- 10 i <- 6 # nrow(Mean) j <- 3 # ncol(Mean)n = 1:10 のそれぞれについて要素を定める。
i\j a b c 1 N(0, 1^2) 0 0 2 N(0, 1^2) 0 0 3 N(1, 2^2) N(0, 1^2) 0 4 N(1, 2^2) N(0, 1^2) 0 5 N(2, 3^2) N(1, 2^2) N(0, 1^2) 6 N(2, 3^2) N(2, 2^2) N(0, 1^2)以下のようにして「二次元配列」を作る。10行18列の「行列」。
m <- sapply(1:(i*j), function(ij) {
if (SD[ij] > 0)
rnorm(n, Mean[ij], SD[ij])
else
rep(0, n)
})
行列に配列の次元属性を与える(三次元配列)。配列要素の順序に注意。
m <- array(m, dim=c(n, i, j)) m3 次元配列の周辺和を求める。
apply(m, 1:2, sum) # これが d実行例
> m
, , 1
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0.156703769 1.1019974 -0.5878131 1.38177251 1.0866401 1.6239542
[2,] 1.373811191 1.5415915 2.1952178 0.22287438 2.5868151 -0.1643288
[3,] 0.730670244 -0.3782146 -0.9849293 -0.19496258 0.8157513 5.5951201
[4,] -1.350800927 -0.4985698 2.4799454 1.88217872 6.8272976 5.7091340
[5,] -0.008514961 -0.5412962 -0.1742176 -0.26483396 1.3814548 0.3559608
[6,] 0.320981863 0.5419221 0.7326528 -0.82759674 1.9827762 0.9208469
[7,] -1.778148409 -1.4575070 1.8153054 -3.16497120 -1.0243537 0.5033054
[8,] 0.909503835 0.1169604 1.4648845 0.08704023 2.4651632 0.9539357
[9,] -0.919404336 -0.1407206 -1.5649053 0.25347786 7.9319406 0.3369453
[10,] -0.157714831 0.0443457 2.0469119 0.47429132 4.6990698 6.6437705
, , 2
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0 0 -1.6481922 0.9301823 2.3629785 -0.07863734
[2,] 0 0 -0.5601030 0.2247429 1.7719864 2.32072697
[3,] 0 0 0.8362890 -1.3439788 1.0961511 0.03228667
[4,] 0 0 -2.1072330 0.6451209 1.5598118 1.45206790
[5,] 0 0 0.2892977 -1.9031158 3.4218537 -0.07945608
[6,] 0 0 -1.2472545 0.8929470 2.9554176 -3.03584486
[7,] 0 0 0.7074159 0.7091518 -0.8749767 1.79230210
[8,] 0 0 -1.6659138 -0.1559170 0.1048045 -0.82257526
[9,] 0 0 -1.4662284 2.1920211 -2.8208809 3.41325672
[10,] 0 0 0.2903617 -0.1537628 5.0290536 0.06835080
, , 3
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0 0 0 0 -0.1858555 0.1759547
[2,] 0 0 0 0 0.6234541 -1.2223501
[3,] 0 0 0 0 -0.8221098 -0.7782799
[4,] 0 0 0 0 1.6184734 1.7862177
[5,] 0 0 0 0 -0.2177257 -0.3615454
[6,] 0 0 0 0 -0.1005853 -1.6459583
[7,] 0 0 0 0 -0.6616456 -0.4109084
[8,] 0 0 0 0 0.4907506 -2.1850624
[9,] 0 0 0 0 -0.8438808 -0.3885813
[10,] 0 0 0 0 0.2083525 -0.9391815
> apply(m, 1:2, sum)
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0.156703769 1.1019974 -2.2360053 2.31195481 3.263763 1.72127163
[2,] 1.373811191 1.5415915 1.6351148 0.44761725 4.982256 0.93404801
[3,] 0.730670244 -0.3782146 -0.1486403 -1.53894141 1.089793 4.84912694
[4,] -1.350800927 -0.4985698 0.3727124 2.52729959 10.005583 8.94741966
[5,] -0.008514961 -0.5412962 0.1150801 -2.16794979 4.585583 -0.08504062
[6,] 0.320981863 0.5419221 -0.5146017 0.06535031 4.837608 -3.76095628
[7,] -1.778148409 -1.4575070 2.5227213 -2.45581941 -2.560976 1.88469909
[8,] 0.909503835 0.1169604 -0.2010293 -0.06887676 3.060718 -2.05370196
[9,] -0.919404336 -0.1407206 -3.0311337 2.44549896 4.267179 3.36162071
[10,] -0.157714831 0.0443457 2.3372736 0.32052849 9.936476 5.77293975
たぶん本番のプログラムは Mean,SD の定義法なども組み込まないといけないだろうが,大筋は変わらないであろう。set.seed(1234567) a <- b <- c <- d <- matrix(NA, 10, 6) for(i in 1 : 2) a[, i] <- rnorm(10, mean=0, sd=1) for(i in 3 : 4) a[, i] <- rnorm(10, mean=1, sd=2) for(i in 5 : 6) a[, i] <- rnorm(10, mean=2, sd=3) for(i in 3 : 4) b[, i] <- rnorm(10, mean=0, sd=1) for(i in 5 : 6) b[, i] <- rnorm(10, mean=1, sd=2) for(i in 5 : 6) c[, i] <- rnorm(10, mean=0, sd=1) for(i in 1 : 2) d[, i] <- a[, i] for(i in 3 : 4) d[, i] <- a[, i] + b[, i] for(i in 5 : 6) d[, i] <- a[, i] + b[, i] + c[, i] dは同じであり,このプログラムの答と私のプログラムの答は一致するので,私のプログラムも正しいと思います。 -- 河童の屁は,河童にあらず,屁である。 2012-12-05 (水) 16:32:03
set.seed(1)
Size <- seq(0.1, 30, by=0.5) #個体のサイズ分布
year <- 40 #時間
class <- 3 #生物の年級群の数
M <- catch <- matrix(0, ncol=year*class, nrow=length(Size))
Gs <- list(0, 0, 0) #classの数だけ
r_years <- c(year, year*2+13)
r_number <- c(40000, 40000)
###Catchは既知のデータ###
c_days <- c(5, 10, 56, 77, 105)
catch[, c_days] <- sapply(seq(2, 6),
function(x) {rpois(length(Size), lambda=x)}
)
SS <-SS2 <- As <- Probs <- matrix(NA, length(Size), class)
Mean <- m123 #mapply(function(min, max)
runif(year*class, min, max), c(0, 5, 15), c(1, 10, 20))
SD <- sd123 #mapply(function(min, max)
runif(year*class, min, max), c(1, 1, 1), c(5, 3, 2))
Surv <- surv123 #mapply(function(min, max)
runif(year*class, min, max), c(0.9, 0.9, 0.9), c(0.99, 0.99, 0.99))
k <- 1
SS2[1, 1] <- 40000
for(i in 1 : (year*class)){
As <- mapply(function(mean, sd)
dnorm(Size, mean, sd), Mean[i, ], SD[i, ])
Probs <- As/rep(apply(As, 2, sum), each=nrow(As))
Probs[which(is.na(Probs))] <- 0
for (j in 1 : ncol(Mean)) {
if(sum(Probs[, j]==0) < nrow(As))
Gs[[j]] <- sample(Size, sum(SS2[, j], na.rm=T),
prob=Probs[, j], rep=T)
}
for(j in 1 : ncol(As)){
SS[, j] <- rbinom(rep(1, length(Size)),
table(cut(Gs[[j]],
breaks=c(Size, max(Size) + 0.5))),
prob=Surv[i, j])
}
for(j in 1 : ncol(As)) {
SS2[, j] <- SS[, j] - round(catch[, i] *
SS[, j]/apply(SS, 1, sum))
SS2[which(SS2[, j] < 0), j] <- 0
}
SS2[which(is.na(SS2))] <- 0
M[, i] <- apply(SS2, 1, sum)
M[which(M[, i] < 0), i] <- 0
if(sum(i==r_years) > 0){
SS2[1, k+1] <- r_number[k]
k <- k + 1
}
}のぶ (2012-12-03 (月) 23:04:10)
お世話になります。
Rは始めたばかりの初心者です。
ご教示頂けると幸いです。
下記のようなデータフレームX、Yの、各A-A',B-B',C-C',D-D'間の、ペアT検定とWilcoxon符号順位検定を行い、各列の因子とp値一覧を出力したいと考えております。X <- data.frame(A = c(1,5,6,4,2), B = c(2,1,7,4,3) , C = c(1,NA,NA,NA,NA) , D = c(3,8,NA,4,1)) Y <- data.frame(A' = c(NA,3,2,8,1), B' = c(3,2,5,4,9) , C' = c(NA,2,8,3,10) , D' = c(1,3,NA,2,NA))検討したい因子がかなり多く、p値の一覧として出力した上で、BH法にてFDRを計算する予定です。
なので、検討できた数 p-value(paired-T.test) p-value(wilcoxon) A-A' 4 0.6892 0.7127 B-B' 5 0.4144 0.5807 C-C' 0 NA NA D-D' 3 0.09547 0.1736のように、要約された結果一覧を得たいです。
for (i in 1:4) print(t.test(X[,i],Y[,i],paired=TRUE)) for (i in 1:4) print(wilcox.test(X[,i],Y[,i],paired=TRUE))とするところまではたどり着きましたが、各検定結果において、何と何を比較したか分からない上にまとまっておらず、さらに全てNAの列があるとそこでスクリプトがとまってしまいます。
何か良い方法はありますでしょうか。
お手数をおかけいたしますが、よろしくお願いいたします。
X <- data.frame(A = c(1, 5, 6, 4, 2), B = c(2, 1, 7, 4, 3) ,
C = c(1, NA, NA, NA, NA) , D = c(3, 8, NA, 4, 1))
Y <- data.frame(A = c(NA, 3, 2, 8, 1), B = c(3, 2, 5, 4, 9) ,
C = c(NA, 2, 8, 3, 10) , D = c(1, 3, NA, 2, NA))
len <- length(X)
n <- pt <- wt<- rep(NA, len)
for (i in seq_len(len)) {
d <- na.omit(data.frame(x = X[, i], y = Y[, i]))
n[i] <- nrow(d)
if (n[i] >= 2) {
pt[i] <- t.test(d$x, d$y, paired = TRUE)$p.value
pw[i] <- wilcox.test(d$x, d$y, paired = TRUE)$p.value
}
}
data.frame(n, pt, pw, row.names=names(X))
実行結果(警告メッセージも出るが。不要なら出さないようにする)
n pt pw
A 4 0.68923296 0.7127019
B 5 0.41443008 0.5807122
C 0 NA NA
D 3 0.09546597 0.1735682悩めるポー (2012-12-01 (土) 18:40:47)
R version 2.13.0(2011-04-13)
mimR_2.6.2
gRbase_1.4.4の環境下で、mimRとMIM3.2.0.7のコネクションがうまくいかないのは何故でしょうか?
The mimR Package
January 31, 2006をなぞっても、、、。
asap (2012-11-30 (金) 21:30:28)
webページの内容はsrc <- readLines("http://www.jma.go.jp/jp/")のようにして取得できますが、そのページの更新日時を取得できる方法はありますか?
例えばfirefox(Win版)であれば、右クリック>ページの情報を表示 を選択すると
そのページの更新日時が表示されますが、それをRで取得したいと思っています
よろしくお願いします
> url <- "http://jasp.ism.ac.jp/~nakanoj/workshop12/2012Rmeeting.htm"
> grep("Last-Modified", system(paste("w3m -dump_head", url),
intern = TRUE), value = TRUE)
[1] "Last-Modified: Thu, 22 Nov 2012 05:14:24 GMT"
ただし、動的に作成されているページにはLast-Modifiedがないので上記は使えません。しかしその場合はアクセスした日時が更新日時ですからそもそも意味がないです。見た目が静的なページに見えても、実はSSIを使っている場合もあります。
なお、http://www.jma.go.jp/jp/は403 Forbiddenでした。url <- "http://jasp.ism.ac.jp/~nakanoj/workshop12/2012Rmeeting.htm"
grep("Last-Modified", system(paste("w3m -dump_head", url),
intern = TRUE), value = TRUE)
以下にエラー system(paste("w3m -dump_head", url), intern = TRUE) :
'w3m' not foundCOM (2012-11-26 (月) 10:02:04)
お世話になります。
stripchart() で因子別に一次元散布図を描き、対応するデータを線でつなぎたいと思っています。y <- rnorm(20) x <- factor(c(rep(1, 10), rep(2, 10))) stripchart(y~x, vertical=T)このとき、因子1の1番目と因子2の1番目、因子1の2番目と因子2の2番目・・・
というように点を線でつなぐにはどのようにすれ- d ばよいでしょうか?
宜しくお願いします。
やまだ (2012-11-24 (土) 06:24:36)
いつもお世話になっています。x [1] 2010/02/02 00:15:00 2010/02/02 01:15:00 2010/02/02 02:15:00 2010/02/02 03:15:00 [5] 2010/02/02 04:15:00 2010/02/02 05:15:00 2010/02/02 06:15:00 2010/02/02 07:15:00 [9] 2010/02/02 08:15:00 2010/02/02 09:15:00 2010/02/02 10:15:00 2010/02/02 11:15:00 [13] 2010/02/02 12:15:00 2010/02/02 13:15:00 2010/02/02 14:15:00 2010/02/02 15:15:00 [17] 2010/02/02 16:15:00 2010/02/02 17:15:00 2010/02/02 18:15:00 2010/02/02 19:15:00 20 Levels: 2010/02/02 00:15:00 2010/02/02 01:15:00 ... 2010/02/02 19:15:00 (y <- strptime(x, "%Y/%m/%d %H:%M:%S")) [1] "2010-02-02 00:15:00" "2010-02-02 01:15:00" "2010-02-02 02:15:00" "2010-02-02 03:15:00" [5] "2010-02-02 04:15:00" "2010-02-02 05:15:00" "2010-02-02 06:15:00" "2010-02-02 07:15:00" [9] "2010-02-02 08:15:00" "2010-02-02 09:15:00" "2010-02-02 10:15:00" "2010-02-02 11:15:00" [13] "2010-02-02 12:15:00" "2010-02-02 13:15:00" "2010-02-02 14:15:00" "2010-02-02 15:15:00" [17] "2010-02-02 16:15:00" "2010-02-02 17:15:00" "2010-02-02 18:15:00" "2010-02-02 19:15:00" length(x); length(y) [1] 20 [1] 9上記のようなコマンドを実行し、length(y)が9となるのはなぜでしょうか。どうぞご教授ください。
x <- factor(scan("", what=""))
"2010/02/02 00:15:00"
"2010/02/02 01:15:00"
"2010/02/02 02:15:00"
"2010/02/02 03:15:00"
"2010/02/02 04:15:00"
"2010/02/02 05:15:00"
"2010/02/02 06:15:00"
"2010/02/02 07:15:00"
"2010/02/02 08:15:00"
"2010/02/02 09:15:00"
"2010/02/02 10:15:00"
"2010/02/02 11:15:00"
"2010/02/02 12:15:00"
"2010/02/02 13:15:00"
"2010/02/02 14:15:00"
"2010/02/02 15:15:00"
"2010/02/02 16:15:00"
"2010/02/02 17:15:00"
"2010/02/02 18:15:00"
"2010/02/02 19:15:00"
x
(y <- strptime(x, "%Y/%m/%d %H:%M:%S"))
length(x); length(y)
実行結果は以下の通り
> x [1] 2010/02/02 00:15:00 2010/02/02 01:15:00 2010/02/02 02:15:00 2010/02/02 03:15:00 2010/02/02 04:15:00 [6] 2010/02/02 05:15:00 2010/02/02 06:15:00 2010/02/02 07:15:00 2010/02/02 08:15:00 2010/02/02 09:15:00 [11] 2010/02/02 10:15:00 2010/02/02 11:15:00 2010/02/02 12:15:00 2010/02/02 13:15:00 2010/02/02 14:15:00 [16] 2010/02/02 15:15:00 2010/02/02 16:15:00 2010/02/02 17:15:00 2010/02/02 18:15:00 2010/02/02 19:15:00 20 Levels: 2010/02/02 00:15:00 2010/02/02 01:15:00 2010/02/02 02:15:00 ... 2010/02/02 19:15:00 > (y <- strptime(x, "%Y/%m/%d %H:%M:%S")) [1] "2010-02-02 00:15:00" "2010-02-02 01:15:00" "2010-02-02 02:15:00" "2010-02-02 03:15:00" [5] "2010-02-02 04:15:00" "2010-02-02 05:15:00" "2010-02-02 06:15:00" "2010-02-02 07:15:00" [9] "2010-02-02 08:15:00" "2010-02-02 09:15:00" "2010-02-02 10:15:00" "2010-02-02 11:15:00" [13] "2010-02-02 12:15:00" "2010-02-02 13:15:00" "2010-02-02 14:15:00" "2010-02-02 15:15:00" [17] "2010-02-02 16:15:00" "2010-02-02 17:15:00" "2010-02-02 18:15:00" "2010-02-02 19:15:00" > length(x); length(y) [1] 20 [1] 20ちゃんと,両方とも長さ 20 ですけどねぇ。
> x <- factor(scan("", what=""))
1: "2010/02/02 00:15:00"
2: "2010/02/02 01:15:00"
3: "2010/02/02 02:15:00"
4: "2010/02/02 03:15:00"
5: "2010/02/02 04:15:00"
6: "2010/02/02 05:15:00"
7: "2010/02/02 06:15:00"
8: "2010/02/02 07:15:00"
9: "2010/02/02 08:15:00"
10: "2010/02/02 09:15:00"
11: "2010/02/02 10:15:00"
12: "2010/02/02 11:15:00"
13: "2010/02/02 12:15:00"
14: "2010/02/02 13:15:00"
15: "2010/02/02 14:15:00"
16: "2010/02/02 15:15:00"
17: "2010/02/02 16:15:00"
18: "2010/02/02 17:15:00"
19: "2010/02/02 18:15:00"
20: "2010/02/02 19:15:00"
21:
Read 20 items
> x
[1] 2010/02/02 00:15:00 2010/02/02 01:15:00 2010/02/02 02:15:00
[4] 2010/02/02 03:15:00 2010/02/02 04:15:00 2010/02/02 05:15:00
[7] 2010/02/02 06:15:00 2010/02/02 07:15:00 2010/02/02 08:15:00
[10] 2010/02/02 09:15:00 2010/02/02 10:15:00 2010/02/02 11:15:00
[13] 2010/02/02 12:15:00 2010/02/02 13:15:00 2010/02/02 14:15:00
[16] 2010/02/02 15:15:00 2010/02/02 16:15:00 2010/02/02 17:15:00
[19] 2010/02/02 18:15:00 2010/02/02 19:15:00
20 Levels: 2010/02/02 00:15:00 ... 2010/02/02 19:15:00
> (y <- strptime(x, "%Y/%m/%d %H:%M:%S"))
[1] "2010-02-02 00:15:00" "2010-02-02 01:15:00" "2010-02-02 02:15:00"
[4] "2010-02-02 03:15:00" "2010-02-02 04:15:00" "2010-02-02 05:15:00"
[7] "2010-02-02 06:15:00" "2010-02-02 07:15:00" "2010-02-02 08:15:00"
[10] "2010-02-02 09:15:00" "2010-02-02 10:15:00" "2010-02-02 11:15:00"
[13] "2010-02-02 12:15:00" "2010-02-02 13:15:00" "2010-02-02 14:15:00"
[16] "2010-02-02 15:15:00" "2010-02-02 16:15:00" "2010-02-02 17:15:00"
[19] "2010-02-02 18:15:00" "2010-02-02 19:15:00"
> length(x); length(y)
[1] 20
[1] 9
その一方で、以下に問題はありません。
> y[1]; y[9] [1] "2010-02-02 00:15:00" [1] "2010-02-02 08:15:00" > y[10] [1] "2010-02-02 09:15:00" > y[15]; y[20] [1] "2010-02-02 14:15:00" [1] "2010-02-02 19:15:00"問題となるのは以下のような場合です。
> m <- data.frame(V1=1:20, V2=1:20) > m[,1] <- x > m[,2] <- y Warning message: In `[<-.data.frame`(`*tmp*`, , 2, value = list(sec = c(0, 0, 0, : provided 9 variables to replace 1 variablesただ、原因の一つは旧バージョンのRを使っていることに原因の一つがあるようです。上記のエラーは以下の環境のときにおこるエラーです。
> sessionInfo() R version 2.8.1 (2008-12-22) i386-pc-mingw32 locale: LC_COLLATE=Japanese_Japan.932;LC_CTYPE=Japanese_Japan.932; LC_MONETARY=Japanese_Japan.932;LC_NUMERIC=C;LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base以下の環境だと問題なくうまくいきます。
> sessionInfo() R version 2.15.1 (2012-06-22) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods baseただ、R 2.8.1 でしか動作しないpackageを使用したいという事情があります。この場合、そのpackage使用の時のみでR 2.8.1を使用し、それ以外は最新バージョンを使用するという方法しかないでしょうか。なければ、そのように対応いたしますが、他に原因があるのであれば、ご教授いただければ幸いです。
Microsoft Windows XP
Professional
Version 2002
Service Pack 3
Intel(R) Core(TM)2 Duo CPU
E6850 @ 3.00GHz
2.99 GHz、1.95 GB RAM -- [[やまだ]] &new{2012-11-25 (日) 00:49:29};atr (2012-11-20 (火) 19:25:14)
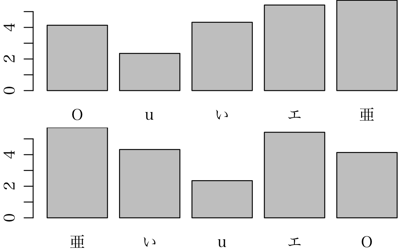
お世話になります。lab <- sample(rep(c("亜","い","u","エ","O"), length.out=10), 10) #サンプル sp <- runif(10, min=0, max=7) DF1 <- data.frame(lab, sp) MEAN1 <- tapply(DF1$sp, DF1$lab, mean) barplot(MEAN1)にて描画される棒グラフのバーの並び順を例えば左から「"亜","い","u","エ","O"」とする方法はないのでしょうか。
低い検索スキルのせいかRjpwiki内では見つかりませんでした。ご教授頂けると幸いです。
layout(matrix(1:2, 2))
par(mgp=c(1.6, 0.8, 0), mar=c(2, 2, 0, 0))
lab <- sample(rep(c("亜","い","u","エ","O"), length.out=10), 10) #サンプル
sp <- runif(10, min=0, max=7)
DF1 <- data.frame(lab, sp)
MEAN1 <- tapply(DF1$sp, DF1$lab, mean)
barplot(MEAN1)
DF1$lab<-factor(lab, c("亜","い","u","エ","O")) # ここがキモ!ですね
MEAN1 <- tapply(DF1$sp, DF1$lab, mean)
barplot(MEAN1)
layout(1)
P (2012-11-13 (火) 16:00:14)
はじめまして。よろしくお願いします。
今、問題になっているコードは以下の部分です。library(e1071) (eeg.svm <- svm(NAME~.,data=zz,cross=7))#svm summary(eeg.svm) pred2 <- predict(eeg.svm, zz) table(pred2,zz[,1])zzは、1列目NAMEに人物名(イニシャル)が入っており、2〜1201列目に各個人の波形が入っている、50×1201のデータフレームです。
このデータをsvmにかけて、個人の判別を行おうとしています。
そこで、summary(eeg.svm)を実行し結果を見るとCall: svm(formula = NAME ~ ., data = zz, cross = 7) Parameters: SVM-Type: C-classification SVM-Kernel: radial cost: 1 gamma: 0.0008333333 Number of Support Vectors: 50 ( 10 10 10 10 10 ) Number of Classes: 5 Levels: m N o u y 7-fold cross-validation on training data: Total Accuracy: 62 Single Accuracies: 42.85714 85.71429 42.85714 57.14286 85.71429 42.85714 75と出てきて、Accuracyが62%だとわかります。
では、その内訳がどうなっているのか、と予想からクロス表を作るためpred2 <- predict(eeg.svm, zz) table(pred2,zz[,1])を実行してみると、
pred2 m N o u y m 8 0 0 0 0 N 2 10 1 0 0 o 0 0 9 0 0 u 0 0 0 10 0 y 0 0 0 0 10と出てきます。このクロス表によればAccuracyは94%です。
なぜ、このように違った結果が出てしまうのでしょうか?
見辛い文章になっていたら申し訳ありません。(←編集し直しましたよ)
ご回答よろしくお願いします。
set.seed(12345)
zz <- data.frame(NAME=rep(c("m", "N", "o", "u", "y"), 10),
matrix(rnorm(50*1200), 50))
library(e1071)
eeg.svm <- svm(NAME~., data=zz, cross=7) # svm
summary(eeg.svm)
pred2 <- predict(eeg.svm, zz)
table(pred2, zz[,1])
やはり,データ数より変数の数が多いのでそういうことになるのでは?むしろ,あなたのデータで,pred2 の正解率が 100% でないほうがおかしいのかな? -- 河童の屁は,河童にあらず,屁である。 2012-11-13 (火) 17:31:58佐藤 (2012-11-07 (水) 22:38:03)
初めまして。今回、初めて投稿させて頂きます。
現在とあるシステムのエラー原因が分からず困っております。
ご指南を宜しくお願いします。
<状況>
4年前からあるシステムを運用していて、Rのソースを一切変えず、これまで運用をしてきたのですが、先日下記エラーが発生しました。想定しないデータが連携された為、以下のエラーが発生するようになったと思われるのですが、エラー内容をみたところ、何が原因か分かりません。
下記のエラー内容を見て、原因が分かれば教えて頂けないでしょうか?
<ログファイルからのエラー直後の箇所を抜粋>if (nrow(tDatStdModelPros) > 0) { res <- by(tDatStdModelPros, factorList, function(x) analyticalPerspectiveByStdModelFunc( x, stdPredictPros[(stdPredictPros[,"COLUMN_3"] == x[1,"COLUMN_3"]),], indexInfo[x[1,"COLUMN_3"],"COLUMN_11"], stdModelProsList[x[1,"COLUMN_2"],], startYM, 0)) # 結果が List なので matrix に変換 colName <- c("COLUMN_1", "COLUMN_2", "COLUMN_3", "COLUMN_4", "COLUMN_5", "COLUMN_6", "COLUMN_7", "COLUMN_8", "COLUMN_9", "COLUMN_10") predictValProsStd <- matrix(unlist(res), ncol=10, byrow = TRUE, dimnames = list(NULL, colName)) predictValProsStd[,"COLUMN_9"] <- 3 predictValProsStd[,"COLUMN_6"] <- 2 rm( res) } 以下にエラーmatrix(unlist(res), ncol = 10, byrow = TRUE, dimnames = list(NULL, : NULL に対して属性を設定しようとしました 実行が停止されました~
> x <- list(a=1:3, b="abc") > print(x) $a [1] 1 2 3 $b [1] "abc" > cat(x$a) 1 2 3 > cat(x$b) abc
> a <- NULL > attributes(a) <- list(a="aaa") > a list() attr(,"a") [1] "aaa"なんてやっても,エラーも出ないし,然るべき結果になるわけです。
mura (2012-11-07 (水) 14:18:02)
はじめまして。R初心者の大学院生で、修士論文のデータ整理で困っております。ご指南を宜しくお願いします。
実は、Jaccard(ジャッカード)係数で約10個のデータの相互の結びつきの強さを計算し、横書きのデンドログラムで描画したいと考えています。
スケールは、ジャッカード係数なのでデータは最大値は1.0で最小値は0となり0.XXXで表現され、数値が大きい(1に近い)程結びつきが強いということで樹形図は結節されていきます。
今、10個のデータ間のジャッカード係数を計算してある縦横の表(対照行列)がJaccard.CSVという名称のCSVファイルがあるとき、Rでどのようにすれば横書きの樹形図を書くことができるでしょうか?
Rについては超初心者で、本を見ながら練習をはじめたばかりです。
大変お手数ですが関数とその他引数も含めてどのようにすれば良いかご指南願えれば幸いです。
どうぞ宜しくお願い致します。
> set.seed(1234)
> x <- round(cor(matrix(runif(25), 5)), 1)
> row.names(x) <- colnames(x) <- LETTERS[1:5]
> x
A B C D E
A 1.0 -0.3 -0.4 -0.9 -0.4
B -0.3 1.0 0.5 0.3 -0.4
C -0.4 0.5 1.0 0.2 -0.3
D -0.9 0.3 0.2 1.0 0.6
E -0.4 -0.4 -0.3 0.6 1.0
という対称行列があったとして、
> library(rioja) > plot(chclust(as.dist(x)), hang = -1, horiz = TRUE)
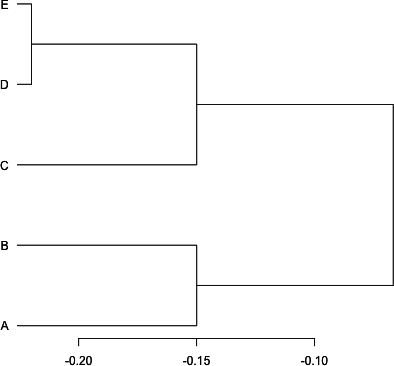
日頃、デンドログラムを描くことがないので、今までplot.hclust()ってhorizオプションがないことを知らなかった。何か理由があるのだろうか?
わnがねぇー (2012-10-27 (土) 11:23:59)
最近、CRAN より、インストールしたパッケージが、CRANサイトより消えてしまいました。
新しいPCに、Rをインストールしているのですが、このパッケージも利用する必要があります。
ンストール済みのパッケージをアーカイブ化(zip形式)して、新しいRの環境で利用する方法はあるのでしょうか?
satto (2012-10-26 (金) 02:26:34)
初めて投稿させて頂きます
以下のコードを実行した時に表示される要素番号とその要素が指す値が実際のものと一致しません。xn <- 2 yn <- 3 zn <- 4 a <- c(1:(xn*yn*zn)) dim(a) <- c(xn, yn, zn) f1 <- function(x, y, z) { cat("a[x,y,z] : ") cat( sprintf("[%d,%d,%d]=%2d,", x, y, z, a[x,y,z]) ) cat("\n") } f1(1:xn, 1:yn, 1:zn)おそらく、sprintf()の書式指定子に渡される順序と、配列の[]内でベクトルが展開される順序が一致していないのだと思いますが、このような表示を行うために何か良い方法はないでしょうか。
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください」というリンクがあります。ちゃんと読んで最初からちゃんとできる人もいます。
を読んでください」というリンクがあります。ちゃんと読んで最初からちゃんとできる人もいます。> for (k in 1:zn) {
+ for (j in 1:yn) {
+ for (i in 1:xn) {
+ f1(i, j, k)
+ }
+ }
+ }悩むぽー (2012-10-24 (水) 20:15:37)
お世話になります。
処理を繰り返すには、for ループを使えばいいんだろうな?と思うのです。
例えば、回帰分析の結果を変数の組み合わせで、数十回繰り返す場合、どんな処理をするのが、より効率的かと悩んでいます。文法をしっかりと理解しないまま使うことに課題はあるのですが、どなたかご教示ください。よろしくお願いします。
変数ABとvariable1の回帰分析をするとして、変数ABとn個の異なる変数の回帰分析を繰り返す処理です。下記のように、commandを繰り返し入力すれば出来ることですが、、、。RegModel.1 <- lm(AB~variable1, data=dataset) summary(RegModel.1) RegModel.2 <- lm(AB~variable2, data=dataset) summary(RegModel.2) RegModel.3 <- lm(AB~variable3, data=dataset) summary(RegModel.3)
AB variable1 variable2 variable3 variable4 variable5 variable6 variable7 variable8 variable9 1 60 47 51 67 50 46 58 42 55 58 2 50 57 51 51 70 54 31 46 44 61 3 38 44 34 48 33 67 51 37 40 56 4 38 66 46 46 38 48 50 55 49 44 :こんなふうにやる
n <- 9
for (i in 1:n) {
str <- sprintf("RegModel.%i <- lm(AB ~ variable%i, data=dataset); print(summary(RegModel.%i))",
i, i, i)
eval(parse(text=str))
}
別のやり方は,簡単。
ans <- lapply(dataset[,2:10], function(x) lm(dataset$AB~x)) lapply(ans, summary)ans はリストオブジェクトになります。
atr (2012-10-23 (火) 19:20:06)
お世話になります。
下記のように、'00〜'02年、'04年は測定実施、'03年測定せず、というデータに対し、年毎の平均値を棒グラフ化(または折れ線グラフ化)したい場合にyr <- sample(rep(c(2000:2002, 2004), length.out=10), 10) #サンプル sp <- runif(10, min=0, max=7) DF1 <- data.frame(yr, sp) MEAN1 <- tapply(DF1$sp, DF1$yr, mean) barplot(MEAN1)上記barplot(MEAN1)のように'02年の次が'04年とならず、下記barplot(MEAN2)のように、測定値のない'03年がゼロ等となるようにデータ自動補間またはプロット時に'03年が自動補間する方法はないでしょうか?
DF2 <- DF1 DF2[(nrow(DF2)+1), ] <- c(2003, 0) # '03年平均ゼロ化(便宜上) MEAN2 <- tapply(DF2$sp, DF2$yr, mean) barplot(MEAN2) #狙いの出力測定ナシの年のデータを手動追加で作成、はデータが膨大であり出来れば避けたいです。以上宜しくお願いします。
set.seed(123)
yr <- sample(rep(c(2000:2002, 2004), length.out=10), 10) #サンプル
sp <- runif(10, min=0, max=7)
DF1 <- data.frame(yr, sp)
MEAN1 <- tapply(DF1$sp, DF1$yr, mean)
func <- function(MEAN1) { # 飛び値のあるデータ
Names <- as.numeric(names(MEAN1))
MEAN2 <- numeric(diff(range(Names))+1)
MEAN2[Names-Names[1]+1] <- MEAN1
names(MEAN2) <- Names[1]:Names[length(Names)]
return(MEAN2) # 飛び値には 0 を補完したデータを返す
}
MEAN2 <- func(MEAN1)
barplot(MEAN2)
または,yr を,最大・最小を指定して factor にして以下のようにする
set.seed(123) yr <- sample(rep(c(2000:2002, 2004), length.out=10), 10) #サンプル sp <- runif(10, min=0, max=7) yr <- factor(yr, levels=min(yr):max(yr)) MEAN1 <- tapply(sp, yr, mean) barplot(MEAN1)お好きな方を
barplot(MEAN1[as.character(2000:2004)], names.arg=2000:2004)
vcd初心者 (2012-10-23 (火) 17:43:10)
お世話になります。
Windows 7上でR i386 2.15.1を使って、vcd_1.2-13を試しています。
パッケージvcdのモザイクプロットに関して、いくつか疑問があります。library(vcd) mosaic(Titanic)とすると、描画領域全体に広がるプロットが作成されますが、次のように仮想データを入力して描画すると、
tab <- matrix(c(10, 30, 40, 60, 70, 30), nrow=3, byrow=T) dimnames(tab) <- list(Size=c("S", "M", "L"), Gender=c("Male", "Female")) mosaic(tab, split_vertical=T)正方形の領域にしかプロットされません。
これを描画領域全体に広げるにはどうしたらよいのでしょうか。
また、度数を各セルに表示しようとして、次のように入力すると、mosaic(tab, split_vertical=T, labeling=labeling_cells(text=tab))度数は表示されるのですが、変数名と水準名のラベルが消えてしまいます。
最低、水準名は表示したいのですが、どうしたらよいのでしょうか。
さらに、ラベルを日本語で表示しようとして、次のように入力すると、> dimnames(tab) <- list(サイズ=c("S", "M", "L"), 性別=c("男", "女")) > mosaic(tab, split_vertical=T) 以下にエラー grid.Call.graphics(L_downviewport, name$name, strict) : Viewport 'cell:サイズ=S' was not foundとエラーになってしまいます。日本語が通らないようなのですが、何か対処法はありますでしょうか。
以上、長々と申し上げましたが、どなたか解決策を御教示いただければ幸いです。
ひろ (2012-10-23 (火) 12:02:25)
関数の引数(パラメータ)aの値がvalidか否かで処理をif分岐することを考えています.しかし,aの値の存在,validが正しく判断できずに困っています.
というのもexists("a")とするとTRUEが返りますが,他is.na,is.null,in.nan等で値の有無を調べると「wrapup 中にエラーが起こりました 'a'が見つかりません」とエラーが返ってきます.aは存在するのに,アクセス出来ない原因がわかりません.お知恵をお貸し頂けると助かります.
■ 追加ソースコードtest<-function(a,b){ browser() missing(a) # TRUE missing(b) # FALSE print(exists("a")) # TRUE print(exists("b")) # TRUE print(ls(a)) # [1] "a" "b" print(ls(b)) # 正常にオブジェクト一覧表示 print(search(a)) # wrapup 中にエラーが起こりました # 使われていない引数 (a) print(search(b)) # wrapup 中にエラーが起こりました # 使われていない引数 (b) is.na(a) # wrapup 中にエラーが起こりました # オブジェクト 'a' がありません is.na(b) # FALSE } test(b=1)
> test <- function(a, b) {
+ if (missing(a) || missing(b)) stop("Error 1")
+ else if(a < 0 || b < 10) stop("Error 2")
+ return(a + b)
+ }
> test(a=13)
以下にエラー test(a = 13) : Error 1
> test(b=1)
以下にエラー test(b = 1) : Error 1
> test(a=3, b=9)
以下にエラー test(a = 3, b = 9) : Error 2
> test(a=-10, b=20)
以下にエラー test(a = -10, b = 20) : Error 2
> test(a=50, b=100)
[1] 150
> test(10, 30)
[1] 40ひろ (2012-10-23 (火) 10:46:51)
添え字が許される範囲か否かを確かめる方法はありますでしょうか?
■ 試行内容:
SVMといった機械学習を用い,弁別予測・正解結果より精度を算出しようとしています.
ところが,結果の行列の構成が結果で異なります. 下記の様に,"正常","異常"の2つの行列が通常現れるpred 異常 正常 異常 100 0 正常 0 100ところが予測結果が悪いと次のように"異常"のみ現れ,"正常"行がないことがあります.結果res["正常",]とすると"wrapup 中にエラーが起こりました 添え字が許される範囲外です"とエラー.
pred 異常 正常 異常 100 100
kin (2012-10-23 (火) 07:59:59)
お世話になります。
hclustで樹形図を作成しています。
今回の質問は、各ラベルから延びている枝の高さを得たいのです。
→ 各ラベルに直接接続している枝の高さです。dat.hc <- hclust(dat, method="ward")とした場合、dat.hc$heightで各クラスターごとの枝の高さが得られ、dat.hc$merge でクラスター形成履歴?が判るので、そのラベル(マイナス値)と対応させるのかなと考えていますが、いまいちスマートなスクリプトが思い浮かびません。
アドバイスなど頂ければ幸いです。
宜しくお願いします。
> set.seed(4649)
> d <- data.frame(x=rnorm(10), y=rnorm(10), z=rnorm(10))
> a <- hclust(dist(d), method="ward")
> plot(a, hang=-1)
> cbind(
+ a$order,
+ sapply(a$order, function(x) {
+ a$height[which(a$merge == -x, arr.ind=TRUE)[1]]
+ })
+ )
[,1] [,2]
[1,] 9 4.161608
[2,] 7 1.761123
[3,] 1 1.093749
[4,] 3 1.093749
[5,] 6 1.310064
[6,] 10 1.310064
[7,] 4 3.018550
[8,] 2 1.690142
[9,] 5 1.290380
[10,] 8 1.290380Micheal (2012-10-23 (火) 05:09:56)
dataset <- 1:100 x <- matrix(dataset, 50, 2) w <- km <- kmeans(x, 2)の結果得られるClustering vectorを出力したくて
write(w, file="result.txt", ncolumns=1)としたのですが、うまく出力する事が出来ません。ご示唆を頂ければと思っています。よろしくお願い致します。
SKYLINE (2012-10-20 (土) 21:54:00)
http://mjin.doshisha.ac.jp/R/19.pdfを参考に mvpart 関数を用いて決定木を描画しました。library(mvpart) iris.rp <- mvpart(Species~., data=iris) plot(iris.rp, uniform=TRUE, branch=0.6, margin=0.05) text(iris.rp, all=T, use.n=TRUE)ところが、上記のサイトのような凡例 (色と品種の対応) が表示されません。
どうすれば凡例が表示されるのでしょうか?
ご教授お願い致します。
環境
MacOSX 10.8.2
R 2.15.1
RStudio 0.96.331
mvpart 1.6
アトム (2012-10-19 (金) 07:41:20)
100x250のマトリックス(dim(a)=100, 250)の中に1から9までの数字が入っています。この中から3だけを選んで10に変換したいと思い、b <- gsub(3, 10, a)を実行したのですが上手くいきません。ご示唆を頂ければと思っています。
> set.seed(888)
> a <- matrix(sample(9, 50, replace=TRUE), 10, 5)
> a
[,1] [,2] [,3] [,4] [,5]
[1,] 1 4 6 2 5
[2,] 4 1 9 2 2
[3,] 1 1 9 8 4
[4,] 7 4 7 1 5
[5,] 7 7 8 4 3
[6,] 1 9 6 3 8
[7,] 4 7 6 1 8
[8,] 3 3 2 9 3
[9,] 1 8 3 4 4
[10,] 9 7 8 4 1
> a[a==3] <- 10
> a
[,1] [,2] [,3] [,4] [,5]
[1,] 1 4 6 2 5
[2,] 4 1 9 2 2
[3,] 1 1 9 8 4
[4,] 7 4 7 1 5
[5,] 7 7 8 4 10
[6,] 1 9 6 10 8
[7,] 4 7 6 1 8
[8,] 10 10 2 9 10
[9,] 1 8 10 4 4
[10,] 9 7 8 4 1ひろ (2012-10-18 (木) 13:01:50)
何時も参考にさせて頂いています。R初心者です。x<-c(034, 0223, 098, 076, 054)というデータを読み込もうと思ったら0が表示されずに、>x 34, 223, 98, 76と読み込まれてしまいます。数字は数値ではありません。0も一緒に読み込む事は出来るのでしょうか?
> (x <- c("034", "0223", "098", "076", "054"))
[1] "034" "0223" "098" "076" "054" kin (2012-10-18 (木) 10:44:24)
お世話になります。
日本語のフレーズを指定文字数で改行したいと考えています。
しかし、ncharでは半角全角も1文字と判断するので半角と全角混在文字列では、指定文字数で改行を入れても1行の見た目の長さが異なってしまいます。
バイト単位で文字数を得るような関数はありませんか?
宜しくお願いします。
Micheal (2012-10-18 (木) 09:04:32)
R初心者です。ランダムな組み合わせの数字が入ったデータフレームを作成しました。y <- c(897, 34565, 32, 1,345, 324, 98765, 808, 897, 808, 808, 456, 3, 454, 3, …) length(y)=30000この中から同じ組み合わせの数字が幾つ出現しているかを調べ表示したいのですが、解決方法はありますでしょうか?得たい出力は、
(組み合わせの数字,出現回数)808, 2 1345, 35 32, 3 …です。よろしくお願い致します。
ひろ (2012-10-15 (月) 15:12:54)
いくつかの列は同じ列名で,幾らか異なる列名のdata.frameを2つ行連結したいのですが,列数が一致しないとエラーが返ってきて実現できません.解決方法がないでしょうか?
例えば,data.frame1とdata.frame2があるとします.2つの行連結すると,共通の列名が有るところは通常通り連結して,無い箇所は"NA"といった値で補完され,結果data.frame.Answerに成ることが希望です.
data.frame1:A C D 1 1 2 3 2 4 5 6data.frame2:
A B C D 1 7 8 9 10 2 11 12 13 14data.frame.Answer(<- data.frame1+data.frame2)
A B C D 1 1 NA 2 3 2 4 NA 5 6 3 7 8 9 10 4 11 12 13 14
> a A C D E F 1 1 2 3 1 4 2 4 5 6 4 6 > b A B C D X 1 7 8 9 10 4 2 11 12 13 14 9 > (n.a <- colnames(a)) # 列名を取り出す [1] "A" "C" "D" "E" "F" > (n.b <- colnames(b)) # 列名を取り出す [1] "A" "B" "C" "D" "X" > setdiff(n.a, n.b) # a にあって b にない列 [1] "E" "F" > a[setdiff(n.a, n.b)] # a から取り出す E F 1 1 4 2 4 6 > setdiff(n.b, n.a) # b にあって a にない列 [1] "B" "X" > b[setdiff(n.b, n.a)] # b から取り出す B X 1 8 4 2 12 9 > intersect(n.a, n.b) # 両方に共通する列 [1] "A" "C" "D" > a[intersect(n.a, n.b)] # a から取り出す A C D 1 1 2 3 2 4 5 6 > b[intersect(n.a, n.b)] # b から取り出す A C D 1 7 9 10 2 11 13 14
kin (2012-10-14 (日) 07:25:10)
時間がかかる処理の進行状況がわかるようにcatにてコンソールに出力しています。
しかし、コンソール画面の行数が26行とすると、そこまでの出力が溜まらないと画面に表示されないようです。
RGUI設定のbuffer console by deault?のチェックは外しています。
現在は、事前に改行(\n)を26個くらい出力することで対応していますが、他に良い方法はありますでしょうか?
cet (2012-10-14 (日) 01:46:07)
はじめまして。いつもこちらを参考にさせていただいています。
R初心者なのですが、どうしても研究でKrigingによる空間補間が必要になり、現在、R version 2.15.1(Win7/64bit)でgeoRのパッケージを利用しています。
緯度、経度、値の情報が入ったcsvを読み込みchl200708というデータフレームを作成しました。(緯度範囲:30-55N,経度範囲:140-180E データ行数:57690)
このような形式のデータを使用しております。> head(ttt200708) lon lat ttt 1 140.042 54.958 0.73995 2 140.083 54.958 0.74817 3 140.125 54.958 0.74817 4 140.167 54.958 0.77717 5 140.208 54.958 0.77717 6 140.250 54.958 0.78524(7~57690行目は省略いたします) このデータをパッケージgeoRコマンドを用いて
testgeo <- as.geodata(ttt200708)でgeodataの形式にすることはできました。
しかし、距離行列を計算しようとすると”負の長さのベクトルは許されません”というエラーが出てしまいます。
同様に、バリオグラムを作成しようとすると> vario1 <- variog(testgeo,max.dist=0.1) variog: computing omnidirectional variogram 以下にエラー dist(as.matrix(coords)) : 負の長さのベクトルは許されませんとなってしまいます。緯度・経度の並び順が悪いのかと思い、昇順・降順を入れ替えてみたりしましたが、それでも同様のエラーになってしまいます。
オンラインでもこの間違いの解決方法を探したのですが、みつかりませんでした。
勉強不足なのはわかっているのですが、どこかにこの解決方法が載っていますでしょうか?
もし、解決方法をご存知でしたら、教えてください。
どうぞよろしくお願いいたします。
> testgeo2 <- as.geodata(ttt200708_2)
> vario1 <- variog(testgeo2,max.dist=0.1)
variog: computing omnidirectional variogram
エラー:サイズ2.9Gbのベクトルを割り当てることができません
追加情報: 警告メッセージ: 1:In as.vector(dist(as.matrix(coords))) :
Reached total allocation of 8072Mb : see help (momory.size)
というエラーがでました。これを調べてみたところ、データが大きすぎて私のパソコンでは扱えないのだという理解に至りました。データが大きすぎるということは、小さな区画に分けて解析を行う以外の方法がないということでしょうか? -- cet 2012-10-14 (日) 13:00:15以下にエラー dist(as.matrix(coords)) : 負の長さのベクトルは許されませんMac の 64 bit 版の R ですが,この条件じゃあお手上げかな。
MY (2012-10-12 (金) 02:50:04)
いつもお世話になっております。
plot関数などの作図関数を使ってグラフのx軸、y軸の目盛りの線の長さを調節したいのですがどのようにすれば良いでしょうか?目盛りの太さを調節するにはlwd.ticksオプションをいじればよいことは分かったのですが...
どうぞよろしくお願い致します。
Micheal (2012-10-11 (木) 07:40:43)
R初心者の者です。
dataset=c(1,9,2,7,72,3,7,99,21,20,11,12,122,45,198,334,43,39,34,45…)
(要素数は100個)というデータセットにおいて、前から順に5個づつ合計していきたいのですが、計算方法が分かりません。イメージは(1+9+2+7+72, 3+7+99+21+20, 11+12+122+45+198, ….)です。どうぞ、宜しくお願い致します。
> dataset <- 1:100 # データ例
> x <- matrix(dataset, 5, 20) # 行列を作る
> x[,1:4]
[,1] [,2] [,3] [,4] 5 列目以降は略
[1,] 1 6 11 16
[2,] 2 7 12 17
[3,] 3 8 13 18
[4,] 4 9 14 19
[5,] 5 10 15 20
> colSums(x) # 列方向に(5 個ずつの要素の)和
[1] 15 40 65 90 115 140 165 190 215 240 265
[12] 290 315 340 365 390 415 440 465 490sheet (2012-10-09 (火) 23:11:19)
24行x500列の時系列データ(=data)で、1〜24行の各行が時間、1〜500列が人物の位置データになっています。1=家、2=オフィス、3=買い物…10=それ以外、で表しています。 (data[,1]=(10,10,10,10,10,10,10,10,4,4,2,2,2,2,2,2,2,2,2,2,3,4,4,10)、の様な)
これらのデータから各要素(1から10)の各時間毎の総計(rowSums(data==1), rowSums(data==2)….)を取り出し、更にその各時間毎(各行毎)に「新たにその行動を起こした人の数だけを数えたい」と思っています。例えばdata[,1]=(1,10,10,10,10,10,10,4,4,4,2,2,2,2,2,2,2,2,2,2,3,4,4,10) data[,2]=(10,10,10,10,10,10,10,10,4,4,4,4,2,2,2,2,2,2,2,2,3,4,4,10) data[,3]=(10,10,10,10,3,3,4,4,4,4,2,2,2,2,2,2,2,2,2,2,3,4,4,10)というデータセットにおいては、第一行のrowSums(data==10)=2, rowSums(data==1)=1になると思います。
ここでdata[,1]では第一行に1が初めて現れているので、3つのデータセット全てにおける第一行の1という要素の合計数は1(rowSums(data==1)=1)。反対に、data[,2]とdata[,3]においては10という要素が現れているので、第一行の10という要素の合計は2となります(rowSums(data==10)=1)。
しかし第二行を見てみると、data[,1]では10という要素が「新たに」現れているので、第二行の10という要素に関してはカウントするのですが、data[,2]とdata[,3]の第二行は10となっていて、第一行から連続しているので「新しく行動を起こした人」とは考えず、カウントしない事とします。この場合、第二行のrowSums(data==1)は1、第二行のrowSums(data==10)は2となる様に計算したいのですが、どの様にすれば良いのか検討が付きません。
何かご示唆を頂ければと考えております。よろしくおねがいします。
> d <- cbind(c(1,10,10,10,10,10,10,4,4,4,2,2,2,2,2,2,2,2,2,2,3,4,4,10),
+ c(10,10,10,10,10,10,10,10,4,4,4,4,2,2,2,2,2,2,2,2,3,4,4,10),
+ c(10,10,10,10,3,3,4,4,4,4,2,2,2,2,2,2,2,2,2,2,3,4,4,10))
> d2 <- apply(d, 2, function(x) {
+ y <- c(0, x[1:23])
+ ifelse(x==y, 0, x)
+ })
> d2
[,1] [,2] [,3]
[1,] 1 10 10
[2,] 10 0 0
[3,] 0 0 0
[4,] 0 0 0
[5,] 0 0 3
[6,] 0 0 0
以下略
> rowSums(d2==1)
[1] 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
> rowSums(d2==10)
[1] 2 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 3d <- cbind(c(0,0,0,10,10,10,0,0,0,0,0,10,10,10,10),
c(10,10,10,0,0,0,0,0,0,0,0,0,10,10,10),
c(0,10,10,10,10,10,10,0,0,0,0,0,0,0,0))
というデータにおいて、第一行においては10が3回と4回現れ、第二行においては10が3回と3回、第三行においては10が6回現れています。よってこれを
d <- cbind(c(0,0,0,3,3,3,0,0,0,0,0,4,4,4,4),
c(3,3,3,0,0,0,0,0,0,0,0,0,3,3,3),
c(0,6,6,6,6,6,6,0,0,0,0,0,0,0,0))
としたいのですが。よろしくおねがいします。 -- sheet 2012-10-12 (金) 08:29:24a <- sapply(apply(d, 2, rle), function(x) {
l <- x[[1]]
v <- x[[2]]
v <- ifelse(v, l, v)
rep(v, l)}
)
ad <- cbind(c(0,0,0,10,10,10,0,0,0,0,0,10,10,10,10),
c(10,10,10,0,0,0,0,0,0,0,0,0,10,10,10),
c(0,10,10,10,10,10,10,0,0,0,0,0,0,0,0))
というデータにおいて、同じ数(0は除く)が連続で出現した際に、その数が現れた回数の合計を数え、その計算結果を元々あった数字と入れ替えるという質問において下記の様な回答を得ました:
a <- sapply(apply(d, 2, rle), function(x) {
l <- x[[1]]
v <- x[[2]]
v <- ifelse(v, l, v)
rep(v, l)}
)
a
これを実行すると:
[,1] [,2] [,3] [1,] 0 3 0 [2,] 0 3 6 [3,] 0 3 6 以下略となり期待通りの結果が得られました。
Pen (2012-10-09 (火) 08:18:53)
時系列データを作っています。各列に個人のIDを振り、各行には毎分毎に、その個人から得られたデータを保存してあります。データは100人から得られたので、列の総計は100。行は1日を分毎に保存してあるので、24(h) x 60 (min) = 1440行となっていて、それを行列(=a)としてRに読み込みました。ここから時系列データにする為に、ts関数を使ってa <- ts(a, start=0, frequency=1440)としたら、
第一行:0,000000000 第二行:0,0006944444 第三行:0,0013888889 第四行:0,002083333 ....となってしまいました。
この数字は一体何なのでしょうか?
普通の時刻(00:00:00, 00:01:00, 00:02:00)で表示する事は可能なのでしょうか?
何かアドバイスを頂ければ幸いです。
> a <- ts(matrix(rnorm(1440*100), 1440, 100), start=0, frequency=1440)
> rownames(a) <- substring(as.POSIXct(0:1439*60, origin="2012-01-01"), 12, 19)
> a[1:10,1:5]
Series 1 Series 2 Series 3 Series 4 Series 5
00:00:00 0.3842177 -0.2572337 0.77637479 -1.4475976202 1.3714974
00:01:00 1.2847077 0.2472553 -0.33261748 1.8957044334 0.2877515
00:02:00 -1.8002028 -0.1887225 0.59084323 -0.6793383289 0.4185490
00:03:00 -0.8671542 0.6765212 0.34528150 -0.0985912968 0.2143888
00:04:00 0.9490067 -1.7888453 1.01909404 -0.4561435524 0.3423056
00:05:00 0.8383023 -0.2663956 1.14336704 0.7110252003 -0.2873150
以下略SP (2012-10-09 (火) 07:56:54)
PP.test の繰り返し計算で得られた結果を出力したいのですが、どうにも上手くいきません。for (i in 1:3) { b <- PP.test(a[, i]) print(b) }という計算式を作り、得られた結果は
Phillips-Perron Unit Root Test data: a[, i] Dickey-Fuller = -3.5585, Truncation lag parameter = 7, p-value = 0.03666 Phillips-Perron Unit Root Test data: a[, i] Dickey-Fuller = -3.8536, Truncation lag parameter = 7, p-value = 0.01631 Phillips-Perron Unit Root Test data: a[, i] Dickey-Fuller = -7.0615, Truncation lag parameter = 7, p-value = 0.01となりました。この P-value だけをまとめて出力したいと思っています。ファイル形式は何でも構いません。宜しくお願いします。
n <- 1000
a <- matrix(rnorm(500*n), ncol=n)
b <- numeric(n)
for (i in 1:n) {
b[i] <- PP.test(a[,i])$p.value
}
print(b)
のようにしてもよいし,下の「通りすがりの仮面ライダーさん」みたいにしてもよいし。 -- 河童の屁は,河童にあらず,屁である。 2012-10-09 (火) 10:17:21yy (2012-10-04 (木) 13:17:40)
対応分析や多次元尺度構成法などで散布図を作成しています。
データや解析手法により座標面の一部分に集中してプロットされることがしばしば発生しています。
この際に、作成されたプロットデータをスプリングモデルのような感じでノードの近いものは斥力が発生し、遠いものは引力が発生するみたいな感じで、ノードを分散させてみたいと考えていますが具体的な関数や手法の見当が全くついていません。
なにか、アドバイスをいただければ幸いです。
taked (2012-10-03 (水) 18:58:04)
関数の引数名を表示させたいのですが、可能でしょうか?
下はうまくいっていない例ですが、このような関数を実行したときに引数名(オブジェクトの変数名)である"y"をcat変数などで表示させたいです
yはベクトルです。よろしくお願いします。
y <- c(1.9, 0.8, 1.1, 0.1,-0.1,4.4,5.5,1.6,4.6,3.4) Data <- function(x){ # cat(names(x)) } Data(y)
> y <- c(1.9, 0.8, 1.1, 0.1,-0.1,4.4,5.5,1.6,4.6,3.4)
> Data <- function(x){
+ cat(deparse(substitute(x)), "\n")
+ }
> Data(y)
y
> long.name <- 1:10
> Data(long.name)
long.nameP (2012-10-03 (水) 15:42:52)
はじめまして。
線形判別分析を用いて、個人認証システムを実装しようと考えています。
元になる波形はcsv形式で保存されており、
30000行×10列で、1〜5列目が人物yの波形データ、残りが人物mの波形データです。eegMY <- matrix() #空の行列を用意 eegMY <- matrix(scan("csvファイルのパス",sep=',',n=30000),nrow=30000,ncol=10,byrow=T) #行列にデータを読み込む eegMYt <- t(eegMY) #転置する name <- matrix(, nrow=10, ncol=1) #個人を判別するための情報をy,mとして、行列を用意する name[1:5,1] <- "y" name[6:10,1] <- "m" x <- cbind(name,eegMYt) #データと個人識別行列を結合する z <- x[1:10,1:101] #※1 y <- as.data.frame(z) #ldaは行列形式は対応していないのでデータフレームに型変換する library(MASS) ldadata <- lda(V1~.,data = y) #実行できない(エラー:variables are collinear)※1 データが大きすぎるため実行できないようなので、とりあえず範囲を区切っています。
現在このようなコードになっています。
V1に用意した個人認証用の"y"か"m"が入っているので、それをもとにldaを実行したいのですが、In lda.default(x, grouping, ...) : variables are collinearという警告メッセージが表示されます。
どのようにすれば、このプログラムが完成に向かうでしょうか?
この様な、本格的なコードを書くのは初めてなので、見当違いの質問かもしれませんが、ご教授いただけたら幸いです。よろしくお願いします。
yds (2012-09-27 (木) 13:50:39)
hclustでデンドログラムを作成しpdfに出力しています。
pdf(family="Japan1GothicBBB", file="dendorogramTI.pdf", width=11.69*3, height=8.27)
td.hc <- hclust(td.d, method="ward")
plot(td.hc, cex=0.7, hang=-1, xaxs="i", yaxs="i")
dev.off()
良し悪しは別として、デンドログラムの項目(要素)が多くなり、例えばA4を横3枚に並べたようなサイズになってしまいます。
これをA4で3ページの1つのPDFとして出力することはできないでしょうか?
yy (2012-09-26 (水) 08:57:07)
windows7 32bit R i386 2.15.0 を使用しています。
Rのショートカットの作業フォルダは以下のようにしてデスクトップにRフォルダを作成しています。"C:/Users/hogehoge/Desktop/R"その配下にcsvフォルダと、scriptフォルダを作成して、各々csvファイルや拡張子rのスクリプトファイルを格納しています。
ユーザーは、ファイル → Rコードのソースを読み込み… でscriptフォルダに移動後に任意のrスクリプトファイルを読み込みます。
rのスクリプトファイルの最初には、下記のように記載されていてcsvファイル選択後、解析処理をさせています。library(svDialogs) dirBase <- getwd() setwd(paste(dirBase, "/csv" , sep="")) csvfile <- dlgOpen(title="対象のCSVファイルを指定してください。", filters=dlgFilters[c("All"), ])$res # 別のスクリプトを連続して実行する場合に備え、scriptフォルダに移動 setwd(paste(dirBase, "/script" , sep=""))その後、Rのコンソール画面で getwd() すると、
> getwd()~ [1] "C:/Users/hogehoge/Desktop/R/script"のように正しく変更されています。
◆ 今困っていることは、ファイル → Rコードのソースを読み込み…をクリックしても、最初の時はダイアログのフォルダが C:/Users/hogehoge/Desktop/R/csv となっています。
その後、ファイルを選択せずにダイアログを閉じ、もう一度、同じようにファイル → Rコードのソースを読み込み…を行うと、C:/Users/hogehoge/Desktop/R/script になっておりrスクリプトファイルを選択できるようになります。
◆ これを最初のRコードのソースを読み込み…で C:/Users/hogehoge/Desktop/R/script が選択できるようにしたいのですが、何か手立てはありますでしょうか?
Montecarlo (2012-09-25 (火) 23:10:06)
いつもお世話になっております。
新しいパソコンへRをインストールしたところ、以前のパソコンでは表示されなかった文言が表示されるようになってしまいました。[以前にセーブされたワークスペースを復帰します]この文言の意味するところが分からず、何か影響があるのでしょうか。 「ワークスペースを○○したい」のページは見つけましたが、あまりヒントになりませんでした。
ご存知の方がいらっしゃいましたら、なにとぞお教えいただけますようお願いいたします。 なお「sessionInfo()」は以下の通りです。> sessionInfo() R version 2.15.1 (2012-06-22) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base
- 前回 R を終了したときの状態を回復したと言うことで,気にする必要は全くありません。R を終了するときに定義した変数や関数など(要するにワークスペース)を保存し,次に R を立ち上げたときにそこから読み込んで,前の変数や関数を引き続き使えるようにすると言うことです。もし,終了時にそれ以前に定義した変数や関数などをチャラにしたいということなら,終了時に「ワークスペースのイメージファイルを保存しますか?(OS により文言は異なる)」に対して「保存しない」を選べばよいのです。 -- 河童の屁は,河童にあらず,屁である。 2012-09-26 (水) 06:44:50
- 河童の屁は、河童にあらず、屁である様 ご回答ありがとうございました。気にしなくていいということがわかり安心いたしました。 -- Montecarlo 2012-09-26 (水) 23:34:21
yy (2012-09-20 (木) 21:15:57)
お世話になります。
たとえばx <- "最新のコメントが付いた記事"とした場合、正規表現を "(最新|記事)" とした場合に「マッチした個数:この場合2個」を得たいのですが、regexprや grep ではできなさそうですが何か適当な関数はありますでしょうか?
よろしくお願いします。
# 重複があっても 1 回と数える
> x <- "最新のコメントが付いた記事の最初のコメント"
> sum(sapply(c("最新", "記事"), grep, x))
[1] 2
> sum(sapply(c("最新", "記事", "コメント"), grep, x))
[1] 3
# 重複はそれぞれを 1 回と数える
> sum(sapply((sapply(c("最新", "記事"), gregexpr, x)), length))
[1] 2
> sum(sapply((sapply(c("最新", "記事", "コメント"), gregexpr, x)), length))
[1] 4a.i (2012-09-19 (水) 04:32:46)
plot(x, y) text(x, y, label)での2次元分布図ではきちんと日本語表示されますが、
plot3d(x, y, z) text3d(x, y, z, label)でのrglパッケージで3次元分布図を作成すると日本語の部分だけが表示されないか文字化けしています。
きちんと日本語部分を表示する方法はあるでしょうか。
よろしくお願いします。
MY (2012-09-18 (火) 11:58:43)
Rを使って表を作成しpngなどのファイルに出力してグラフ化したいのですが、そういったことができる関数はありますか?
よろしくお願いします。
taked (2012-09-18 (火) 11:12:50)
小数点以下の表示をそろえるため、2.0や4.0を得たいのですが、調べても分からなかったので教えてください
よろしくお願い致します
下の例ではround関数を使っていますが、round関数でなくても、4.0(数値)の入力に対して、4.0を得られれば良いです> round(4.01,digit=0) [1] 4 > round(4.01,digit=1) [1] 4 > round(4.01,digit=2) [1] 4.01 > 4.0 [1] 4
> cat(sprintf("%.1f\n", 4.01))
4.0
> cat(sprintf("%.2f\n", 4.01))
4.01
> cat(sprintf("%.0f\n", pi))
3
> cat(sprintf("%.1f\n", pi))
3.1
> cat(sprintf("%.2f\n", pi))
3.14
> cat(sprintf("%.3f\n", pi))
3.142
> cat(sprintf("%.4f\n", pi))
3.1416
> cat(sprintf("%.5f\n", pi))
3.14159超初心者 (2012-09-14 (金) 15:52:22)
いつも掲示板を参考にしています。
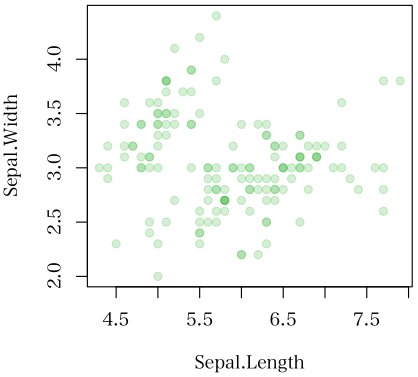
x<-matrix(c(2,2,3,6,2,0,6,7,9,1,4,1,4,2,1,3,5,6,3,2,6,4,5,7,5,1,1,7,3,2,0,5,0,8,1,8),9,4) rownames(x)<-c("みかん","いよかん","はっさく","ぽんかん","桃","すもも","白桃","20世紀梨","新高梨") colnames(x)<-c("あ町","い町","う町","え町") library(MASS) x.coa<-corresp(x,nf=4) biplot(x.coa)
上記のようなサンプルで出力されるbiplot図に対して、柑橘類(みかん、いよかん、はっさく、ぽんかん)はオレンジ色、(桃、すもも、白桃)はピンク色、(20世紀梨、新高梨)にはグリーンをあてたいのですが、どうやったらできるのでしょうか?
よろしくお願いします。
biplot.default <- edit(stats:::biplot.default)出てくるエディット画面で,51行目の
text(x, xlabs, cex = cex[1L], col = col[1L], ...)を
text(x, xlabs, cex = cex[1L], col = col, ...)に(col[1L] を col にする)
text(y, labels = ylabs, cex = cex[2L], col = col[2L], ...)を
text(y, labels = ylabs, cex = cex[2L], col = col[length(col)], ...)に(col[2L] を col[length(col)] にする)
(color <- rep(c("orange", "pink", "green", "red"), c(4, 3, 2, 1)))
"orange" が 4 回,"pink" が 3 回,"green" が 2 回。最後に "red" を 1 回ということ(描いてみれば分かるが,赤は,この例では町を表す文字列の色)。biplot(x.coa, col=color)その他の col[1L],col[2L] もしかるべき値に直した方がよいが,まあ決定的なものじゃないから省略。気になったらいじってみてください。 -- 河童の屁は,河童にあらず,屁である。 2012-09-14 (金) 18:54:52
(2012-09-11 (火) 15:22:23)
Mac OS X 10.8.1, R 2.15.1,Rstudio 0.96.330 を使っています。
R コンソールから pdf("file.png"...) のようにして書き出したファイル中の日本語は文字化けしませんが,Rstudio で,*.Rmd から *.html を生成すると,figure フォルダに作られる *.png ファイルの日本語は文字化け(トーフ)します。当然,できあがった html ファイル中の *.png 画像も文字化けしています。
Mac で作った *.Rmd を Windows に持って行って Rstudio で *.html にすると文字化けはしません。
Rstudio for Mac の場合には,フォントファミリーが日本語になっていないのだと思いますが,フォントファミリーを設定するオプションがわかりません。
R コンソールから knit2html から始めて debug でトレースしてみた限りでは,knit 関数の中にある process_file の中(あるいは更にその奧か)で *.png が作られるようですが,やはり,フォントファミリーの設定のタイミングが全くわかりませんでした。対処法はあるでしょうか。
画像に使われるフォントはpar(family="フォント名")で指定します.Macでヒラギノ角ゴシックを使う場合,
par(family="HiraKakuPro-W3")@
とします.
ウエモト (2012-09-10 (月) 16:15:33)
初めて質問させて頂きます。宜しくお願いします。
Rを使って線形判別法(lda)によってデータの分類・予測をしたいと思っております。library(MASS) group <- c("y","y","y","y","y","m","m","m","m","m")#人物の属性 wave <- c(y1,y2,y3,y4,y5,m1,m2,m3,m4,m5)#各要素は1列30000行の行列 traindata <- data.frame(GROUP=new{2012-09-17 (月) 21:48:32};
- group,WAVE=wave)#データフレームにする
(ldadata<-lda(GROUP~.,data = traindata))#ldaにかける plot(ldadata)#学習データの第一判別関数得点の分布 apply(ldadata$means%*%ldadata$scaling,2,mean)#判別得点 table(group,predict(ldadata)$class)#判別結果をクロス表にする←失敗このような状態です。実行すると最終行で、要素が一致せず、失敗してしまいます。
原因は、groupが要素数10なのに対し、predict(ldadata)$classが要素数30000だということに起因しています。
自分としては、データフレームtraindata中のwaveにおける、各要素である各行列がどちらのグループに属するのかを調べたいのですが、どうすればいいかで困っています。
google検索などを使ってみましたが、自分の力不足でどのようにすればいいのかわかりません。
Rの知識が少なく、低レベルな質問かとも思いますが、ご教授願います。
見辛い文面でしたら申し訳ありません。
> library(MASS)
> set.seed(123)
> ( d <- data.frame(group=rep(letters[1:2], each=5), x=rnorm(10), y=rnorm(10), z=rnorm(10)) )
group x y z
1 a -0.56047565 1.2240818 -1.0678237
2 a -0.23017749 0.3598138 -0.2179749
3 a 1.55870831 0.4007715 -1.0260044
4 a 0.07050839 0.1106827 -0.7288912
5 a 0.12928774 -0.5558411 -0.6250393
6 b 1.71506499 1.7869131 -1.6866933
7 b 0.46091621 0.4978505 0.8377870
8 b -1.26506123 -1.9666172 0.1533731
9 b -0.68685285 0.7013559 -1.1381369
10 b -0.44566197 -0.4727914 1.2538149
> ans <- lda(group~x+y+z, d)
> ans2 <- predict(ans)
> xtabs(~d$group+ans2$class)
ans2$class
d$group a b
a 4 1
b 3 2taka (2012-08-20 (月) 21:31:47)
お世話になります。
Rでcensored logit modelあるいはcensored probit modelを利用したのですが、ただのlogit model、probit model、またCensored regression modelのTobit modelについては、こちらのwikiの「Rでエコノメトリクス」のページなどを参照させていただき、それぞれglm(y ~ x1 + x2 + x3, family=binomial(link="logit")) glm(y ~ x1 + x2 + x3, family=binomial(linknew{2012-10-04 (木) 13:35:44}; ~="probit"))
vglm(y ~ x, tobit(Lower=0), trace=TRUE)などの関数を使うということまではわかったのですが、censored logit/probit modelではどのような関数を使えば良いのかがgoogleなどで調べられませんでした。
もしご教授いただけましたら幸いです。
当方、Rの基礎どころか統計の基礎も危うく、見当違いな質問でありましたら誠に申し訳ございません。
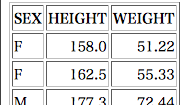
meet (2012-08-07 (火) 22:35:28)
xtableライブラリで、列ごとに小数点の桁数を指定する(2列目は小数点第一位まで、3列目は第二位まで)ことはできますか?また1列目に行番号をつけないようにできますか?ご教授お願いします。library("xtable") sex <- c("F", "F", "M", "M", "M") height <- c(158.0, 162.5, 177.3, 173.3, 166.3) weight <- c(51.22, 55.33, 72.44, 57.44, 64.33) ( x <- data.frame(SEX=sex, HEIGHT=height, WEIGHT=weight) ) mytable <- xtable(x) print(mytable, type="html")
mytable <- xtable(x, digits=c(0, 0, 1, 2)) print(mytable, type="html", include.rownames=FALSE)で,以下のようになるでしょう。
ランゲル・ハンス (2012-08-04 (土) 11:00:04)
いつも掲示板を参考にしています。
次のような行列Mがあります。m <- c(16, 15, 7, 14, 9, 11, 8, 4, 1, 16, 12, 17, 10, 9, 19, 6) M <- matrix(m, 4, 4) M行列Mの行をX、列をY、値をVとし、行列Mの左下が(X, Y)=(1, 1)になるように次のようなデータフレームを作りたいと思います。
X Y V 1 1 14 2 1 4 3 1 17 4 1 6 1 2 7 2 2 8 3 2 12 4 2 19 1 3 15 2 3 11 3 3 16 4 3 9 1 4 16 2 4 9 3 4 1 4 4 10方法をご教示いただけないでしょうか?
上の例は4行4列ですが、本当はn行m列でデータフレームを作りたいと思います。どうぞよろしくお願いします。
> N <- t(M[nrow(M):1,]) > data.frame(X=c(row(N)), Y=c(col(N)), V=c(N)) X Y V 1 1 1 14 2 2 1 4 3 3 1 17 4 4 1 6 5 1 2 7 SNIP 15 3 4 1 16 4 4 10
しまだ (2012-08-03 (金) 23:43:47)
お世話になります。
画像を与えた時、特定の色のピクセル数を得たいのです。そこで:library(biOps) data <- readJpeg("c:/hoge_20120101.jpg") x <- c(1:352) y <- c(1:288) blue <- c(0) grey <- c(0) white <- c(0) black <- c(0) yellow <- c(0) for ( i in x ){ for ( j in y ){ if (( data[i,j,1]==0 ) && ( data[i,j,2]==0 ) && ( data[i,j,3]==254 )) { blue <- ( blue + 1 ) } if (( data[i,j,1]==192 ) && ( data[i,j,2]==192 ) && ( data[i,j,3]==192 )) { grey <- ( grey + 1 ) } if (( data[i,j,1]==255 ) && ( data[i,j,2]==255 ) && ( data[i,j,3]==255 )) { white <- ( white + 1 ) } if (( data[i,j,1]==0 ) && ( data[i,j,2]==0 ) && ( data[i,j,3]==0 )) { black <- ( black + 1 ) } if (( data[i,j,1]==255 ) && ( data[i,j,2]==255 ) && ( data[i,j,3]==0 )) { yellow <- ( yellow + 1 ) } }}(1)処理を速くしたい
スクリプトを実行したところ、動いたのですが、処理が遅いように思います。 コーディングが素人で下手くそだと思うのですが、どこを修正すれば良いのでしょうか。
(2)png画像を扱いたい
biOpsは、png画像を読み込めないようです。オリジナルはpngのため、無理矢理jpegに変換しているのですが、pngが読み込めて同様の処理ができるパッケージはありませんでしょうか。
data <- readJpeg("c:/hoge_20120101.jpg")
d1 <- data[,,1]
d2 <- data[,,2]
d3 <- data[,,3]
blue <- sum(( d1==0 ) & ( d2==0 ) & ( d3==254 ))
grey <- sum(( d1==192 ) & ( d2==192 ) & ( d3==192 ))
white <- sum(( d1==255 ) & ( d2==255 ) & ( d3==255 ))
black <- sum((d1==0 ) & ( d2==0 ) & ( d3==0 ))
yellow <- sum(( d1==255 ) & ( d2==255 ) & ( d3==0 ))IonaZUNNNNNNN (2012-08-03 (金) 23:28:40)
マニュアルをちゃんと嫁
そもそも、そんなことをしたいというのは変態(そんなことする必要はない)
どうしてもしたければ、関数(プログラム)を書け
関数(プログラム)を書けないなら、あきらめよう
tt (2012-08-02 (木) 19:01:06)
n は、プラスの整数です。nが決まっていれば、以下でOKなのですが、異なった数字の時(実際にはせいぜい、5-10程度)には、プログラム中では対応できないですよね。mS <- data.frame(S1,S2, ---,Sn)一つの方法として、S1のみのデータフレームを作り、そこに1個ずつベクトルを追加していく方法はできるのですが、いかにも非効率ですよね。もう少しすっきりした方法は無いでしょうか?
よろしくお願いします。
# S1, S2, ..., Sn というベクトルがあって,それをデータフレームにする
> S1 <- rnorm(5)
> S2 <- rnorm(5)
> S3 <- rnorm(5)
> S4 <- rnorm(5)
> n <- 4
> mS <- eval(parse(text=paste("data.frame(", paste("S", 1:n, sep="", collapse=","), ")")))
> mS
S1 S2 S3 S4
1 -0.5025337 1.4369542 0.45664139 0.4367258
2 -0.5886718 -0.7321779 -0.08103579 1.3684718
3 -0.7793953 1.3541141 -0.98813613 -1.3115064
4 1.3410327 1.3565760 -1.19112426 0.3708082
5 0.5515542 0.3012129 0.61595356 -0.1799028data.frame(sapply(ls(pattern = "S[0-9]+"), get))
taked (2012-08-01 (水) 17:41:01)
Rstudio serverで、インストールできるパッケージとできないパッケージがあります。解決法があればご教授下さい。
CentOS 6.2、 rstudio-server-0.96.228-x86_64 です。
例えばggplot2はインストールできましたが、XMLなどいくつかのパッケージがうまくインストールできません。
パッケージがR自体のバージョン もしくは64bitに対応してないのでしょうか?> install.packages("XML") Installing package(s) into ‘/home/●○●○/R/library’ (as ‘lib’ is unspecified) trying URL 'http://cran.cnr.Berkeley.edu/src/contrib/XML_3.9-4.tar.gz' Content type 'application/x-gzip' length 923501 bytes (901 Kb) opened URL ================================================== downloaded 901 Kb * installing *source* package ‘XML’ ... ** package ‘XML’ successfully unpacked and MD5 sums checked checking for gcc... gcc checking for C compiler default output file name... a.out checking whether the C compiler works... yes checking whether we are cross compiling... no checking for suffix of executables... checking for suffix of object files... o checking whether we are using the GNU C compiler... yes checking whether gcc accepts -g... yes checking for gcc option to accept ISO C89... none needed checking how to run the C preprocessor... gcc -E No ability to remove finalizers on externalptr objects in this verison of R checking for sed... /bin/sed checking for pkg-config... /usr/bin/pkg-config checking for xml2-config... no Cannot find xml2-config ERROR: configuration failed for package ‘XML’ * removing ‘/home/●○●○/R/library/XML’ Warning in install.packages : installation of package ‘XML’ had non-zero exit status The downloaded source packages are in ‘/tmp/Rtmp5ZwZF3/downloaded_packages’
tadashi (2012-07-25 (水) 01:10:16)
こんな現象があります。
下記のようなsubset の使い方は間違いのようです。どこかにこの件について書いてあるでしょうか> subset(test,test[,"flag"]==flag)
> test<-data.frame(flag=sample(c("a","b","c"),rep=T,10)) > x<-"a" > subset(test,test[,"flag"]==x) flag 1 a 6 a 9 a > flag<-"a" > subset(test,test[,"flag"]==flag) flag 1 a 2 c 3 b 4 b 5 c 6 a 7 c 8 c 9 a 10 b
subset(test, test[, "flag"] == flag)は
attach(test) test[test[, "flag"] == flag, ] # ここのflagはデータフレーム内のflag列。 detach(test)といった処理が行われています。 -- Iona 2012-07-25 (水) 01:19:48
> set.seed(11119) > (a <- sample(c(TRUE, FALSE), 10, replace=TRUE)) [1] TRUE FALSE FALSE TRUE TRUE FALSE FALSE TRUE TRUE TRUE > (b <- sample(c(TRUE, FALSE), 10, replace=TRUE)) [1] FALSE FALSE TRUE TRUE FALSE TRUE TRUE FALSE FALSE TRUE > a | b [1] TRUE FALSE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE > a || b [1] TRUE > a & b [1] FALSE FALSE FALSE TRUE FALSE FALSE FALSE FALSE FALSE TRUE > a && b [1] FALSE
分散分析 (2012-07-20 (金) 11:26:29)
Rで二元配置分散分析をしたのですが、Residuals が表示されないことがあります。その理由を教えていただけると助かります。> a <- c(15.65, 14.40, 17.50, 15.90, 20.30, 19.45, 20.45, 19.05) > 要因1 <- factor(c("T1", "T1", "T2", "T2", "T3", "T3", "T4", "T4")) > 要因2 <- factor(c("Y1", "Y2", "Y1", "Y2", "Y1", "Y2", "Y1", "Y2")) > summary(aov(a ~ 要因1 * 要因2)) Df Sum Sq Mean Sq 要因1 3 34.026 11.3421 要因2 1 3.251 3.2513 要因1:要因2 3 0.151 0.0504よろしくお願いします。
ふしぎ (2012-07-19 (木) 17:53:11)
例えば、hoge.RdataをダブルクリックしてRを起動した後で、このファイル名"hoge.Rdata"を取得する関数や方法はありますでしょうか?
list.files() で作業フォルダ内のファイル名は取得できますが、他にも.Rdataの拡張子を持つファイルがある場合、hoge.Rdataを一意に決定できずに困っています。解決策やヒントなど、ご教示いただければ幸いです。
動作環境は、Win XP、R version 2.14.2です。
蓮井 (2012-07-15 (日) 06:45:20)
win-xp、R2.15.1、maptools0.8-16、sp0.9-99を使っています。
shapeファイルの各ポリゴンについて、
・面積を求める
・各ポリゴンがある座標値を含むかどうかを確認する
の2つの作業を実施したく、各ポリゴンの座標値を取り出し方についてご教示いただきたく、よろしくお願いします。
readShapeP最新 cat(sprintf(br;特別なことをするわけですから,なにもしないでいつでも好きなときに利用できるというわけにはいきません。でしょうね。 -- 河童の屁は,河童にあらず,屁である。 olyでポリゴンを読み、スロットpolygonsを表示させると、> map <- readShapePoly("sample.shp") > map1[1,] @ polygons > > An object of class "Polygons" > Slot "Polygons": > [[1]] > An object of class "Polygon" > Slot "labpt": > [1] -32959.94 -64662.22 > <中略> > Slot "coords": > [,1] [,2] > [1,] -32919.99 -64656.18 > [2,] -32920.01 -64659.99 > <中略> > [13,] -32919.99 -64656.18 > <後略>となりますが、このcoordsの値を取り出すためにはどうすればよろしいでしょうか。
「R の S4 クラス、メソッド入門」は読んでみたのですが、よくわからず…。
よろしくお願い申し上げます。
> map @polygons [[i]] @Polygons [[1]] @coordsで座標を取り出すことができました。 -- 蓮井 2012-07-15 (日) 18:11:58
kbys (2012-07-13 (金) 21:23:22)
sendmailRというパッケージでgmailを使ってメール送信する方法について教えてください。マニュアル(http://cran.r-project.org/web/packages/sendmailR/sendmailR.pdf)を参考にしたのが下のコードですがうまくいきません。よろしくお願いいたします。"oooooooooo@gmail.com"は仮のアドレスです。library(sendmailR) sendmail(from="oooooooooo@gmail.com", to="oooooooooo@gmail.com", subject="件名", msg="本文", control=list(smtpServer="smtp.gmail.com")) 以下にエラー socketConnection(host = server, port = port, blocking = TRUE) : コネクションを開くことができません 追加情報: 警告メッセージ: In socketConnection(host = server, port = port, blocking = TRUE) : smtp.gmail.com:25 cannot be opened
taked (2012-07-10 (火) 15:15:07)
お世話になります.rscriptでRスクリプトを実行して、グラフを保存したいのですがうまくできません.
以下のプログラムを、task.Rとしています.library(ggplot2) ratings <- qplot(rating, data=movies, geom="histogram") ggsave(ratings, file="fig1.png")
Rコンソールにコピペして実行するとグラフが保存できるのですが、コマンドプロンプトで
カレントディレクトリ>rscript task.R
として実行するとグラフが保存ができていません.コマンドプロンプトにはRコンソール実行時とほぼ同様のメッセージがでます.実行できているようですがグラフは保存されません.
Loading required package : methods
Saving 7 x 7 in image
stat_bin: binwidth defaulted to range/30. Use 'binwidth = x' to adjust this.
誤りなどあったらアドバイス頂けないでしょうか、よろしくお願いします.Win VISTA, R 2.15.1です
$ Rscript task.R 要求されたパッケージ methods をロード中です Saving 7 x 7 in image stat_bin: binwidth defaulted to range/30. Use 'binwidth = x' to adjust this. $ file fig1.png fig1.png: PNG image data, 2100 x 2100, 8-bit colormap, non-interlaced
Saito (2012-07-03 (火) 18:50:19)
いつもお世話になっております。
連続値を取る確率変数が複数種類あった場合に、その経験的な同時分布を求め、確率変数がある組み合わせをとる時の同時確率を求めたいです。
例えば、> library(np) > x <- rbeta(100, shape1=10, shape2=1) > y <- rgamma(100, shape=1) > z <- rnorm(100) > fit<-npudens(~x+y+z) > plot(fit)とすると、3変量の場合の同時確率分布が得られるようですが、その後に例えば、x=0.1, y=0.5, z=-0.1となるような確率を計算をしたいと考えています(出来ればベクトルの形で与えられるようにして、x=c(0.1, 0.2, 0.3), y=c(0.1, 0.2, 0.5), z=c(0.2, 0.4, -0.1)などと与えて、3つの同時確率が出てくると嬉しいです)。
また、上記の計算はおそらく連続値を丸める必要があると思われるのですが、そうではなく、x=0.9-1.1、y=0.4-0.6、z=-0.2-0.2といったように、特定の区間に入る確率も計算したいです。
ネットを調べていると、同じような質問はあったのですが、
https://stat.ethz.ch/pipermail/r-help/2010-November/260994.html
それに対する返答が私には理解できず、手づまりとなっています。
その他、色々と調べたのですが、二変量の場合や、正規分布に従う場合などはいくつか例があるようなのですが、特に分布形を仮定しない多変量となると資料が少なく途方に暮れております。
どなたか分かる方がいらっしゃいましたらご教授頂けると幸いです。
どうぞよろしくお願い致します。
KANA (2012-07-02 (月) 20:15:29)
Rにて下記のイメージように、ひとつのテーブルにある最頻出の読み方だけを抽出したいのですが、可能でしょうか。
AAAは、あああが3件・えーえーえーが2件のため「あああ」を抽出
UUUは、うううが2件・ゆーゆーゆーが1件のため「ううう」を抽出
■データ内容イメージ
テーブル名:table1
アルファベット,読み
AAA,あああ
UUU,ううう
AAA,あああ
EEE,えええ
AAA,えーえーえー
UUU,ゆーゆーゆー
EEE,いーいーいー
AAA,えーえーえー
EEE,いーいーいー
UUU,ううう
AAA,あああ
■抽出結果イメージ
AAA,あああ
UUU,ううう
EEE,いーいーいー
以上、よろしくお願いいたします。
> x
[1] "A" "U" "A" "E" "A" "U" "E" "A" "E" "U" "A"
> y
[1] "あ" "う" "あ" "え" "えー" "ゆー" "いー" "えー" "いー" "う"
[11] "あ"
> table(paste("(",x,",",y,")",sep=""))
(A,あ) (A,えー) (E,いー) (E,え) (U,う) (U,ゆー)
3 2 2 1 2 1> d <- structure(list(アルファベット = structure(c(1L, 3L, 1L,
+ 2L, 1L, 3L, 2L, 1L, 2L, 3L, 1L), .Label = c("AAA", "EEE", "UUU"
+ ), class = "factor"), 読み = structure(c(1L, 3L, 1L, 4L, 5L,
+ 6L, 2L, 5L, 2L, 3L, 1L), .Label = c("あああ", "いーいーいー",
+ "ううう", "えええ", "えーえーえー", "ゆーゆーゆー"
+ ), class = "factor")), .Names = c("アルファベット", "読み"
+ ), class = "data.frame", row.names = c(NA, -11L)) # ここまでテストデータ作成
> ans <- xtabs(~., data=d)
> cbind(apply(ans, 1, function(x) colnames(ans)[which.max(x)]))
[,1]
AAA "あああ"
EEE "いーいーいー"
UUU "ううう"
同順位の処理はなんとかする。 -- 河童の屁は,河童にあらず,屁である。# テストデータを作成する
gen <- function(n=10000, len=2) {
m <- sample(len, n, replace=TRUE)
sapply(m, function(k) paste(LETTERS[sample(5, k, replace=TRUE)], collapse=""))
}
set.seed(111111)
n <- 2000 # 2000 組(行)のデータ
d <- data.frame(item=gen(n), def=gen(n, len=4))
テストデータ(データフレーム)
item def
1 CD DCD # CD は DCD みたいなこと
2 BE D
3 AA D
4 B ADB
中略
1998 A C
1999 E CEEE
2000 CC BDA
中間集計
ans <- xtabs(~., data=d)分析結果
cbind(apply(ans, 1, function(x) colnames(ans)[which.max(x)])) [,1] A "C" AA "A" AB "B" AC "C" AD "B" 中略 ED "A" EE "A"(2) 今回提示する結果表示
col.name <- colnames(ans)
row.name <- rownames(ans)
for (i in 1:nrow(ans)) { # 集計表の各行について
x <- ans[i,]
max.val <- max(x) # 最大頻度
items <- x[x==max.val] # 最大頻度と同じ頻度を持つ要素
cat(row.name[i], ":", items[1], "-", names(items), "\n")
}
A : 13 - C # A に対して C が 13 回
AA : 5 - A
AB : 3 - B D # AB に対して B, D がそれぞれ 3 回
AC : 5 - C
AD : 4 - B C
中略
ED : 3 - A B C
EE : 2 - A B C CEE D Ed <- read.csv("table1.csv")
> 「どちらか一方表示できれば大丈夫」,そんなわけ無いのでは!ymbr (2012-06-30 (土) 16:35:57)
お世話になっております。
2e+05などの大きな値を実数表記する際にoptions(scipen=○○)を使って『200000』と表示のではなく、上付き文字の指数を用いることで『2×10^5(実際は上付き)』のような書式で表示することはできますでしょうか?
よろしくお願い致します。
x <- 2:5 * 10^5 y <- c(1, 2, 3, 100) plot(x, y, axes = FALSE) axis(1, at = 2:5 * 10^5, labels = parse(text = paste(2:5, "%*%", "10^5"))) axis(2, at = c(0, 100), labels = expression(0, 1 %*% 10^2))
taked (2012-06-29 (金) 17:46:33)
お世話になります.lm()で 線形モデルによる回帰を行いたいのですが、データフレームが大きいためかエラーとなります.> memory.size(max = FALSE) [1] 14.01 > df <- read.csv("data.csv", as.is=T) > memory.size(max = FALSE) [1] 471.71 # (中略) > memory.size(max = FALSE) [1] 747.44 > LM <- lm(df$y ~ . , data = df ) エラー: サイズ 268.0 Mb のベクトルを割り当てることができません > memory.size(max = FALSE) [1] 1210.4 >memory.limit(size = NA) [1] 2047 > object.size(df) 229790744 bytes(質問1) もともとの"data.csv"は約100MB(250列×20万行です)です.これを読込すると、object.sizeは約220MBとなりRが約450MBのメモリを使うのは正常でしょうか?
(質問2) lm()の実行前のメモリ使用量は約747MBですが、lm()などを実行するにはやはりメモリ(memory.limit=2047MB)が不足なのでしょうか?何か他の原因が考えられるでしょうか?
解決方法や回避策など(変数等を減らす以外で)アドバイスを頂けると幸いです.よろしくお願い致します.
R 2.14.1 , --max-mem-size=2047MB, Windows vista 32bit, メモリ 3.00 GB, 起動させているソフトはRとテキストエディタのみです.
esta (2012-06-29 (金) 05:07:05)
いつもお世話になっております。
2変量ガウスコピュラ関数について、確率密度関数をグラフで表示したいのですが、2変量ガウスコピュラ密度関数の中に標準正規分布の逆関数があります。この逆関数を表現できず困っています。標準正規分布の逆関数はどのように表現すればよいのでしょうか。
> qnorm(c(0.025, 0.5, 0.975, 0.7)) [1] -1.9599640 0.0000000 1.9599640 0.5244005そうでないとしたら,もっと正確な定義を述べるよう望む。 -- 河童の屁は,河童にあらず,屁である。 2012-06-29 (金) 06:43:46
hiro (2012-06-20 (水) 11:56:54)
Rを始めたばかりです。
現在、ロジスティック回帰分析にかけるために、総当り法でAICからモデル選択を行なっています。
変数が20個と多いので、all.logisticの結果はすべて表示されず、デフォルトだとdeviance順に20個しか表示されません。なので、print.all.logisticを使ってAICの小さい順にソートしたいのですが、エラーが出てしまいます。> print.all.logistic(obj, sort.by=c("AIC")) 以下にエラー data.frame(obj$deviance, obj$AIC, obj$formula) : オブジェクト 'obj' がありませんどのようにすればよいのでしょうか。
よろしくお願いいたします。
> lr <- read.table("lr.data", header=TRUE)
> obj <- all.logistic(lr)
> print(obj, sort.by="AIC", models=100)
のようにするようですけど,そのようにしていますか?上の3行を実行したらどうなります。同じようにエラーが起きますか?使用方法を理解せず,勝手な解釈で不適切な使用法でエラーがでても,自己責任ですよ。 -- 2012-06-20 (水) 12:15:12tadashi (2012-06-17 (日) 18:15:17)
実際のデータを読み込むときには、データクリーニングをする必要があります。
そういった作業をRだけできるだけやる方法を書いてあるページはないでしょうか?
Rクックブックには、そういったのはRは不得意だから、やらないほうがいいとかいてありますが、もしかしたらあるのではないかと思っての質問です。
とりあえず、scan で全部読み込んで、それからいろいろ処理しましょうといような内容になるかと予想しています。
しかしまあ,昔っから,データクリーニングといってきた。 -- 河童の屁は,河童にあらず,屁である。 2012-06-18 (月) 15:48:38データベースの中から誤りや重複を洗い出し、異質なデータを取り除いて整理すること。データベースの精度を高めることにより、経営やマーケティングに有用な相関関係やパターンを探り出すデータマイニングなどに役立てることができる。データクリーニング。
esta (2012-06-15 (金) 16:30:16)
はじめまして。R歴1週間の初心者です。f<-function(x)exp(-3.0346+0.0558*x-0.0003*x*x)/{1+exp(-3.0346+0.0558*x-0.0003*x*x)} plot(f,0,250)これを実行するとfのグラフが表示されます。mathematicaで最大値とそのときのxの値を求めたところ、x=93のときf(93)=0.391765が最大値として得られました。これをRで求めるにはどのようにしたらよいのでしょうか?
The R Tipsを一通り読んでみましたが分かりませんでした。
どうかよろしくお願いいたします。
> z <- plot(f,0,250) > max(z$y) [1] 0.3917469 > z$x[which.max(z$y)] [1] 92.5
> func <- function(x) {
+ -1 / (1 + exp(3.0346 - 0.0558 * x + 0.0003 * x^2))
+ }
> ans <- optimize(func, interval=c(0, 250))
> cat(ans$minimum,を取得する関数や方法はありますでしょうか?~
list.files()
で作業フォルダ内のファイル名は取得できますが、他にも.Rdataの拡張子を持つファイルがある場合、hoge.Rdataを一意に決定できずに困っています。解決策やヒントなど、ご教示いただければ幸いです。93 0.3917648とすればよい。 -- 河童の屁は,河童にあらず,屁である。 2012-06-15 (金) 23:23:12
BCD (2012-06-14 (木) 11:06:29)
いつもお世話になっております 。
Linux、R2.1.4環境下でlibrary(doMC)のregisterDoMC()を使ってforeach~%dopar%{で並列処理を行っているのですが、Rがクラッシュしてしまい処理を終えることができません。解決策をご存知の方がいらっしゃいましたら御教授願います。
library(doMC) registerDoMC() foreach(i = 1:3) %dopar% sqrt(i)このような簡単な処理ならクラッシュせずに処理を行えるのでしょうか。 -- Iona 2012-06-14 (木) 21:44:58
tadashi (2012-06-12 (火) 10:08:39)
前も聞いているのですが、できるんでしょうか? UTF-8で、CSVで保存すれば、なんとでもなるだろうというのはわかっているのですが、バイナリで共通してもてると便利だと思っています。それほどめんどうでなく、いい方法はないのでしょうか?
TK (2012-06-09 (土) 21:27:13)
Rコマンダーで折れ線グラフを作成した結果思うようなグラフができたのですが、X軸の目盛が0,50,100....と50刻みで表示されました。これを0,30,60,90,....360まで30刻みにしたいのですが、作成方法を詳しくお願いします。
R初心者ですのでプログラムの書き込み場所手順等、詳しくお願いします。
今は、Rコマンダーでデーターセットしてグラフ作成する事しかわかってませんので宜しくお願いします。
layout(matrix(1:2, 1)) plot(iris$Petal.Width, iris$Sepal.Length) plot(iris$Petal.Width, iris$Sepal.Length, xaxt="n", yaxt="n", xlim=c(0, 3)) axis(1, at=seq(0, 2.5, by=0.25)) axis(2, at=seq(4, 8, by=1)) layout(1)要するに,plot コマンドで xaxt="n", yaxt="n" 必要があれば xlim, ylim そして,axis(1, ...), axis(2, ...) を使う。高水準描画関数はデフォルトの描画をしてくれるが,それを抑制して,低水準描画関数で細かな指定をする。デフォルトで我慢できないのなら,詳細指定は自分で設定するしかない。 -- 河童の屁は,河童にあらず,屁である。 2012-06-09 (土) 22:09:39
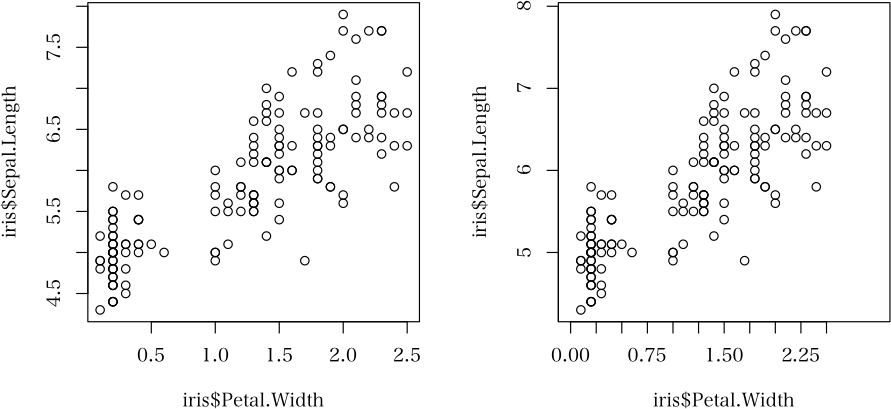
[[E&T]] (2012-06-06 (水) 21:34:40)
Efron and Tibshiraniの"An Introduction to the Bootstrap"でブートストラップの勉強をしています。同書のp.99-102の「8.6 移動ブロック・ブートストラップ」に関して、質問があります。
時系列データからランダムに3つの観測値からなるブロックを復元抽出して、新しい時系列を200系列作成します。それに対して1次の自己回帰モデルを当てはめ、回帰係数βのブートストラップ推計値を求めようというものです。
これを追試しようとして、library(boot)のtsbootを適用すべき問題であろうと推測し、以下のようなコードを試してみました。(対象とする時系列は、library(bootstrap)のlutenhorm$V4です。)library(bootstrap) library(boot) block.beta <- function(tsb){ ar.fit <- ar(tsb, order.max=1) as.vector(ar.fit$ar) } lut.block3 <- tsboot(lutenhorm$V4, statistic=block.beta, R=200, l=3, sim="fixed")すると、以下のようなエラーメッセージが出て、うまくいきません。
以下にエラー tsboot(lutenhorm$V4, statistic = block.beta, R = 200, l = 3, : replacement(置き換え)の長さが0です何か、根本的なところで私自身に誤解があるのかもしれませんが、原因がよく分かりません。どなたか解決策を御教示いただければ幸いです。
Rbeginer (2012-06-05 (火) 11:10:03)
Rで時系列解析を考えている初心者です。
AR(自己回帰モデル)を用いた時系列解析に関する質問です。
RでARを用いた伝達関数のゲインやコヒーレンス、位相を計算することを検討しています。
調べていてもどうもたどり着けません。
Rでそもそも可能でしょうか、またパッケージを導入すれば可能なのでしょうか?
ご教授いただければ幸いです。
sato (2012-06-05 (火) 10:39:04)
R初心者です.
Rで折れ線グラフや棒グラフなどの単純な作図を行なっています.
軸の目盛りですが,デフォルトでは外向きとなっていると思います.
これを内向きにしたいと思って,いろいろ調べたのですが,よくわかりません.
ご教授くださいますようお願いいたいします.
BCD (2012-06-02 (土) 21:07:42)
いつもお世話になっております。
グラフ内における座標について質問があります。
ベクトルがいくつかあり、それらをfor文で繰り返しを用いてそれぞれをplot関数で折れ線グラフを作成しています。各々のグラフにおいて作図領域の中の同じ位置にlines関数を用いて線を表示したいのです。それぞれのグラフでは座標位置が異なるのでlines関数に渡すxとy座標を統一することができません。for文を使って繰り返しの中で自動的に線を加えたいので各グラフにおける座標を取得することができれば良いと思うのですが方法が分からないのです。
何かご存知の方がいらっしゃれば御教授願います。
Rstudent (2012-05-31 (木) 09:29:52)
Rのグラフィックデバイスについて質問があります。
Mpeg等の動画像を手動で一定のフレーム数進め、その都度、動画像上のある点の座標を取得したいと考えております。
座標の取得はlocator()を利用すれば可能だと思いますが、以下について方法がありましたら、ご教示願います。
1.動画像を手動で進める方法
animationパッケージのani.start()を用いてHTML上で再生すると、コマ送りのボタンが表示されますが、このようなことをRのグラフィックデバイス上でできないものでしょうか。
2.ショートカットキーを用いてグラフィックデバイスにコマンドを与える方法
動画像を進める際にショートカットキーでコマ送りを行ったり、ショートカットキーで変数に値を入れたりすることができると非常に効率的になるのですが、そのような方法はあるのでしょうか。
以上、どうぞよろしくお願いいたします。
timesaver (2012-05-21 (月) 18:48:18)
コンマ秒以下の経過時間を計算したい場合、例えば
as.difftime(c("0:3:20", "0:3:20.5"),units="sec")
とすると結果は
Time differences in secs
[1] 200 200
attr(,"tzone")
となり秒以下は無視されます。これを解決する簡単な方法はどなたかご存じ
ありませんか?過去記事やパッケージはないかと探したのですが見当たりませんでした。
my.difftime <- function(time) { # units="secs" を仮定
unname(sapply(time, function(t) {
str <- unlist(strsplit(t, ":"))
if (length(str) != 3) return(NA)
return(sum(as.numeric(str)*c(3600, 60, 1)))
}))
}
> as.difftime(c("1:3:20", "2:3:20.5"), units="secs")
Time differences in secs
[1] 3800 7400
attr(,"tzone")
[1] ""
> my.difftime(c("1:3:20.123", "2:3:20.523"))
[1] 3800.123 7400.523> difftime("2012-05-22 00:34:56.789", "2012-05-22 00:12:34.567", units="secs")
Time difference of 1342.222 secs> difftime("2011-10-11 15:08:01.800 JST", "2011-10-11 15:08:02.900 JST", units="secs")
Time difference of -1.1 secs
> difftime("2011-10-11 15:08:01.800 JST", "2011-10-11 15:08:01.900 JST", units="secs")
Time difference of -0.1000001 secs> options(digits=20) # コンソールに表示する数値の桁数を20桁にする > time1 <- "2011-10-11 15:08:01.800 JST" # この日付は, > time1 <- as.POSIXct(time1) # コンピュータ上ではこのように格納される > unclass(time1) # 数値としては以下の通り [1] 1318313281.7999999523 # 10進数でのキレイな数になっていない attr(,"tzone") [1] "" > time2 <- "2011-10-11 15:08:01.900 JST" # もう一つの日付についても同じく > time2 <- as.POSIXct(time2) > unclass(time2) [1] 1318313281.9000000954 # 10進数でのキレイな数になっていない attr(,"tzone") [1] "" > unclass(time1)-unclass(time2) # 数値の差を取ると [1] -0.10000014305114746094 # -0.1 ではない,ということになった!!!! attr(,"tzone") [1] ""そもそも,"2011-10-11 15:08:01.800 JST" が 1318313281.7999999523 ということはどういうことか。それは,1970年1月1日を0として,経過日時を表す数値です。有効桁15桁では正確には表せない数値です。正確に表せない2つの数値の差も正確には表せないと言うことです。
> difftime("1970-01-01 15:08:01.800 JST", "1970-01-01 15:08:01.900 JST", units="secs")
Time difference of -0.10000000000218278728 secs # かなり,正確になったように見えるが我々の感覚では不正確
> options(digits=7) # 当然と言えば当然だが,表示桁数を通常の7桁にすれば,
> difftime("1970-01-01 15:08:01.800 JST", "1970-01-01 15:08:01.900 JST", units="secs")
Time difference of -0.1 secs # 一見正しい答えが表示される(が,コンピュータ上では -0.1 ではない)
ということですね。「コンピュータは,正確な答えを表示しているとは限らない」> my.difftime2 <- function(time1, time2) {
+ t <- function(time) {
+ sum(as.numeric(unlist(strsplit(time, ":"))) * c(3600, 60, 1))
+ }
+ t(time1)-t(time2)
+ }
> my.difftime2("15:08:01.800", "15:08:01.900")
[1] -0.1
プログラムが書けない?...がんばってもらうしかないなあ。optimal (2012-05-20 (日) 01:03:58)
期間10年、9カ国、説明変数10から成るパネルデータを分析しています。分析対象は、経済学でいうところのグラビティモデルです。
プーリング推計と固定効果推計については出力できたのですが、ランダム効果推計を行おうとすると、下記のようなエラーがでてしまいます。
パネルデータには欠損値はありません。どなたか、対処法をご教示くださいませんでしょうか?なお、期間10年対象国21カ国(上記9カ国含む)のパネルデータ、期間10年対象国12カ国のパネルデータについては推計が出来ました。> north.pl <- plm(form, data=north, model="pooling") series are constants and have been removed > north.wi <- plm(form, data=north, model="within") series are constants and have been removed > north.ra <- plm(form, data=north, model="random") series are constants and have been removed 以下にエラー if (sigma2$id < 0) stop(paste("the estimated variance of the", : TRUE/FALSE が必要new{2012-07-14 (土) 04:53:48};
- tadashiさん、Ionaさんありがとうございます。pythonのsmtplibをrJythonから利用する方法を調べてこのサイト(http://r.789695.n4.nabble.com/Email-out-of-R-code-td3530671.html)で、特にファイル添付できるnutterb 氏の投稿を参考にしています。送信はできたのですが本文に日本語を送信すると???となって受信されます。エンコードを明示的に指定しないといけないことが分かり修正を試みているのですがpythonも初心者なものでうまくいっておりません。大変お手数おかけしますが、nutterb 氏の投稿の中で修正箇所・コードについて教えて頂けないでしょうか? -- kbys なところが欠損値です 使用環境は以下の通りとなります。
R version 2.15.0 (2012-03-30)
Platform: x86_64-pc-mingw32/x64 (64-bit)
以上につき、ご教示お願い致します。
> north <-read.csv("north.csv", header=T)
> north <-plm.data(north, index=c("country","year"))
> north
country year ctt edu gdppc 以下略
1 Australia 2000 14.44600 2.276241 10.225650 以下略
2 Australia 2001 14.88955 2.276817 10.276530 以下略plm:::swar
function (formula, data, effect)
{
within <- plm.fit(formula, data, model = "within", effect = effect)
within$args <- list(effect = effect, random.method = "swar")
中略
if (balanced) {
sigma2$one <- arg.other * deviance(between)/df.residual(between)
sigma2$idios <- deviance(within)/df.residual(within)
sigma2$id <- (sigma2$one - sigma2$idios)/arg.other
if (sigma2$id < 0) # sigma2$id が NA であるため
stop(paste("the estimated variance of the", effect,
"effect is negative"))
以下略
sigma2$id が NA なのは,モデルに対して解が求まっていないから。解が求まらないのは,説明変数が多すぎるから。 -- 河童の屁は,河童にあらず,屁である。 2012-05-23 (水) 19:47:05taked (2012-05-18 (金) 17:40:50)
Rを使って、簡単なレポートを作成したいと思ってます。
pdf()関数で、グラフはpdfに出力できます。
例えば、任意のテキスト、データフレーム、グラフを並べてpdfに出力することは可能でしょうか?ご教授お願いします。df <- data.frame(x=c(1:5), y=sample(100,5)) pdf("c:/test.pdf") plot(df) dev.off()
> grDevices::pdf.options(useDingbats = FALSE); utils::Sweave('test1.Rnw')
Writing to file test1.tex
Processing code chunks with options ...
1 : keep.source term verbatim
2 : keep.source term tex
3 : keep.source term verbatim pdf
You can now run (pdf)latex on 'test1.tex'
�x�����b�Z�[�W�F #文字化け
1: 'test1.Rnw' has unknown encoding: assuming Latin-1
2: package 'xtable' was built under R version 2.14.2
>
Running texi2dvi on test1.tex...failed
Error running C:/w32tex/bin/pdflatex.exe (exit code 9) 1: 'test1.Rnw' has unknown encoding: assuming Latin-1 2: package 'xtable' was built under R version 2.14.2ということですが,Sweave 関数にエンコーディングの指示はしましたか?
tadashi (2012-05-17 (木) 14:28:27)
Apacheのcombined形式のログをRで読み込んで処理しようと思っていますが、読み込んだあとの処理(正規表現)で結構苦労しています。rubyでやれば超簡単なのですが、Rでは非常に難しいのでしょうか? やりたいことは、IPアドレスやエージェントなどの項目への切り分けです。一行のアクセスログを綺麗に分解したいのです。非常に面倒かどうかを知りたいのです。
Toy (2012-05-16 (水) 22:03:33)
Windows XPとWindows 7でR version 2.15.0を使用しています。
プロットのタイトルやラベルに日本語を使用して、これをepsファイルに保存すると、警告メッセージ: 1: 'mbcsToSbcs' 中の '繧ウ繝ウ繝励Λ繧、繧「繝ウ繧ケ' で変換に失敗: <e3>をドットで置き換えました (以下略)のようなワーニングが大量に出て、作成されたepsファイルを見ると、確かに日本語の部分がドットに置き換えられています。
これは、既知のバグなのでしょうか?
それとも、何か対処法があるのでしょうか?
tadashi (2012-05-12 (土) 14:18:50)
いろいろあるみたいです。たとえば、札幌R勉強会とかあるみたいですが、情報ご存知のかた、教えてください。
のざわ (2012-05-03 (木) 15:18:29)
soundと言うライブラリで、色々な音が作れる事が判りました。(だだしWindowsマシンだけ)。ここで質問なのですが、折角作ったサウンドファイルをファイルとして出力し保存したい場合のやりかたが判りません。ファイル出力の方法を教えて下さい。宜しくお願いします。
タカ (2012-05-03 (木) 06:37:33)
いつもお世話になっております。
グラフィカルモデリングを行うために、mimRをインストールしたのですが、説明書通りに打ち込んでもエラーになってしまいます。> data(carcass) > gmd.carc <- as.gmData(carcass) > m.main <- fit(mim(".", data=gmd.carc)) MIM returned NULL or NA, can not continue 以下にエラー strsplit(mimFormula, "/") : 文字列でない引数です原因を御存じの方がいらっしゃいましたら、ご指導いただけると助かります。
よろしくお願いいたします。
使用環境は、
R version 2.15.0 (2012-03-30)
Platform: i386-pc-mingw32/i386 (32-bit)
タカ (2012-04-29 (日) 21:24:21)
いつもお世話になっております。
データフレームを作成する際、変数名を他の変数で指定したいのですが、
> x #xはデータフレーム var 1 5 2 4 3 3 4 2 5 1 > y="var2" #変数名をvar2にしたい > z=c(1,1,1,1,1) > x=cbind(x,y=z) > x var y 1 5 1 2 4 1 3 3 1 4 2 1 5 1 1
上のように変数名をvar2として追加したいのですが、yになってしまいます。
変数で指定することはできないのでしょうか?
よろしくお願いいたします。
> x var 1 5 2 4 3 3 4 2 5 1 > z=c(1,1,1,1,1) > x=cbind(x,var2=z) > x var var2 1 5 1 2 4 1 3 3 1 4 2 1 5 1 1
> colnames(x) <- c("var",y)
> x
var var2
1 5 1
2 4 1
3 3 1
4 2 1
5 1 1
もっと簡単には
> colnames(x)[2] <- "var2"
tadashi (2012-04-27 (金) 16:33:04)
POSデータの詳細行のようなテストデータをつくろうと思っています。簡単にRでできることなのでしょうか?ある範囲の文字列(orange,lemon,apple) があってそれに何個というのをランダムにつけます。下記のようなデータです。orange 2 lemon 3 apple 5 lemon 2 apple 4のようなサンプルデータです。for などつかわずにできそうな気がするのですが、できるのでしょうか?
やりたいことは、文字列一覧からランダムに文字列を選んで、それにランダムに自然数をわりあてます。Rだとなんかさくっと出来る方法があるような気がする気がしています。
n <- 10
Max <- 20
Kudamono <- c("orange", "lemon", "apple")
d <- data.frame(kudamono = sample(Kudamono, n, replace = TRUE),
kosuu = sample(Max, n, replace = TRUE))
こんな風なデータフレームが出来ますけど?
kudamono kosuu
1 orange 18
2 orange 5
3 apple 7
4 orange 6
5 lemon 1
6 lemon 11
:> X <- as.data.frame(table(sample(month.abb[1:5],1000,rep=TRUE))) > X Var1 Freq 1 Apr 196 2 Feb 220 3 Jan 204 4 Mar 191 5 May 189
yamda (2012-04-22 (日) 11:51:20)
例えば,1月〜12月に対し,変数A(90,86,88,82,75,67,55,46,36,21,17,10)と変数B(72,61,52,49,39,36,30,27,23,20,16,12)が与えられているときに,AとBの回帰直線を同時に描くにはどのようにすればよいでしょうか?ご教示いただけますと,幸甚です。
A <- c(90,86,88,82,75,67,55,46,36,21,17,10) B <- c(72,61,52,49,39,36,30,27,23,20,16,12) x <- 1:12 plot(x, A) abline(lm(A~x)) points(x, B, col="red") abline(lm(B~x), col="red")でよいんじゃない? -- 河童の屁は,河童にあらず,屁である。 2012-04-22 (日) 13:03:46
hayashi (2012-04-19 (木) 20:51:21)
変数名y1,y2,y3,y4,y5,.....y100 があり,"y1 + y4","y1 + y2 + y4 +y5"のような文字列が与えられたときに,文字列に出現した変数名をカウントしたいのですが,どのようにすればよいか教えていただけないでしょうか
最終的に欲しい結果は,次のような結果ですy1 y2 y3 y4 y5....y100 1 1 0 0 1 0....0 2 1 1 0 1 1....0 df <- - data.frame(y1=0, y2=0, y3=0, y4=0, y5=0) r1 <- "y1 + y4" r2 <- "y1 + y2 + y4 +y5" df x1 <- c(1, 0, 0, 1, 0) x2 <- c(1, 1, 0, 1, 1) df2 <- rbind(df, x1) df2 <- rbind(df2, x2) df2
> set.seed(123456789)
> df <- as.data.frame(matrix(sample(0:1, 50, replace=TRUE), 10))
> colnames(df) <- paste("y", 1:5, sep="")
> df
y1 y2 y3 y4 y5
1 1 0 0 0 1
2 1 1 1 1 1
3 1 1 1 0 1
4 1 0 0 1 1
5 1 0 0 1 0
6 1 0 1 0 1
7 0 0 0 1 1
8 1 0 0 0 0
9 0 1 1 0 0
10 0 1 1 0 0
> r1 <- "y1 +y4"
> r2 <- "y1 + y2 + y4 +y5"
> func <- function(str) {
+ r1.e <- paste("rowSums(df[,",
+ paste("c(",
+ paste(
+ paste("'", unlist(strsplit(str, " *\\+ *")), "'", sep=""),
+ collapse=", "), ")", sep=""), "])", sep="")
+ return(eval(parse(text=r1.e)))
+ }
> df2 <- df
> df2$r1 <- func(r1)
> df2$r2 <- func(r2)
> df2
y1 y2 y3 y4 y5 r1 r2
1 1 0 0 0 1 1 2
2 1 1 1 1 1 2 4
3 1 1 1 0 1 1 3
4 1 0 0 1 1 2 3
5 1 0 0 1 0 2 2
6 1 0 1 0 1 1 2
7 0 0 0 1 1 1 2
8 1 0 0 0 0 1 1
9 0 1 1 0 0 0 1
10 0 1 1 0 0 0 1 r1 <- "y1 +y4",
r2 <- "y1 + y2 + y4 +y5"
y1 y2 y3 y4 y5
1 1 0 0 0 1
(略)
11(r1) 1 0 0 1 0
12(r2) 1 1 0 1 1
(以下続く)> ncol <- 5 # 後で,100 にしてね
> func <- function(str) {
+ result <- integer(ncol)
+ x <- as.integer(gsub("y", "", unlist(strsplit(str, " *\\+ *"))))
+ result[x] <- 1
+ return(result)
+ }
>
> r1 <- "y1 +y4"
> r2 <- "y1 + y2 + y4 +y5"
>
> df <- data.frame(matrix(func(r1), 1))
> colnames(df) <- paste("y", 1:ncol, sep="")
> df <- rbind(df, func(r2))
> df <- rbind(df, func("y1+y3+y5"))
> # あと,好きなだけ繰り返す
> df
y1 y2 y3 y4 y5
1 1 0 0 1 0
2 1 1 0 1 1
3 1 0 1 0 1
で,いいいですか?細かな仕様は自分で調整 -- 河童の屁は,河童にあらず,屁である。 2012-04-19 (木) 23:24:44BCD (2012-04-19 (木) 18:42:45)
非常に桁数の大きな計算をしています。例えば以下のような計算式です。> (1e+307)*(1e+307) [1] Infしかしながら、計算の桁数が大きいと計算結果が全てInfになってしまい実際の数値を知ることが出来ません。どうすれば計算結果をInfでなく数値として表示させることができるのでしょうか?よろしければ御教授願います。
# フィボナッチ数列の 10000 項目を求める--- 2090 桁の数値
library(gmp)
a <- b <- as.bigz(1) # この場合は as.bigq でも同じ
for (i in 3:10000) {
c <- a+b
a <- b
b <- c
}
print(c)> x <- lchoose(1000, 500) / log(10) # 1000から500を選ぶ二項係数の常用対数値 > x [1] 299.4318271518637288864 > 10 ^ (x - floor(x)) [1] 2.702882409454304912799 # つまり 2.7028...e+299
> library(help=gmp)
> chooseZ(1000, 500)
Big Integer ('bigz') :
[1] 27028824094543656951561469362597527549615200844654828700739287510
662542870552219389861248392450237016536260608502154610480220975005067
991754989421969951847542366548426375173335616246407973788734436457416
111949760457104498575628788051460099421942675236691585660313686260248
4428109296905863799821216320
なんて具合にね。さと (2012-04-18 (水) 09:58:44)
4月から新しい環境で仕事を始めたのですが、インターネットに接続してRに新しいパッケージをインストールしようとしたところ、> utils:::menuInstallPkgs()~ --- このセッションで使うために、CRANのミラーサイトを選んでください --- 以下にエラー m[, 1L] : 次元数が正しくありませんなるエラーメッセージが出てインストールができませんでした。以前の職場ではそのような不都合はなかったのですが、原因がわからず困っております。
新しい職場のシステム管理者はR使いではないので原因がわからなかったのですが、何か解決のヒントをご教授いただければ幸いです。
教師 (2012-04-15 (日) 23:05:22)
講義で一連のR命令を実行していく様子を,プロジェクターでスクリーンに映したいと考えています.命令を書いたソースファイルを読み込み,一行ずつ実行し,その都度解説をしたいと考えています.
何か適当な工夫,もしくはそれ用のパッケージをご存知なら教えてください.
例えば,par(ask=TRUE) でひき続く画像出力を段階的に出力するイメージです.
よろしくおねがいします.
> Demo <- function(x) {require(evaluate);invisible(lapply(evaluate(file(x)),
+ function(y) {replay(y); invisible(readLines(n=1))}))}
> Demo("L1.R") # L1.R はR命令を羅列したテキストファイル
+ # (以下CRを押すごとに命令とその結果がR端末に)
Time difference of -1.1 secs
表示される)
Loading required north.ra package: evaluate
> ### R のデータタイプ
> x <- 1 # 数値(倍精度実数)
> x
[1] 1
> (x <- 1) # 付値と結果の表示を同時に行う
[1] 1
> (xx = 2) # 付値のもうひとつの形式
[1] 2
(以下省略)> func <- function() {
+ n <- 10
+ s <- 0
+ for (i in 1:n) {
+ s <- s+i^2
+ }
+ print(s)
+ }
> debug(func)
> func()
debugging in: func()
#1 の debug: {
n <- 10
s <- 0
for (i in 1:n) {
s <- s + i^2
}
print(s)
}
Browse[2]>
#2 の debug: n <- 10
Browse[2]>
#3 の debug: s <- 0
Browse[2]>
#4 の debug: for (i in 1:n) {
s <- s + i^2
}
Browse[2]>
#4 の debug: i
Browse[2]> cat(i, s)
0
Browse[2]>
#5 の debug: s <- s + i^2
Browse[2]>
#4 の debug: i
Browse[2]> cat(i, s, n)
1 1 10
Browse[2]>
#5 の debug: s <- s + i^2
Browse[2]>
#4 の debug: i
Browse[2]>
#5 の debug: s <- s + i^2
Browse[2]>
#4 の debug: i
Browse[2]>
#5 の debug: s <- s + i^2
Browse[2]> cat(s)
14
Browse[2]> cat(i)
4
Browse[2]> c
#7 の debug: print(s)
Browse[2]> Q
これはこれで,なかなか大変。 -- 河童の屁は,河童にあらず,屁である。 2012-04-16 (月) 22:49:15北原 (2012-04-13 (金) 18:09:05)
Rとより一般的な統計の質問かもしれません。
AICはパラメトリックモデルの比較に使う指数だと知っていますが、ノンパラメトリックとパラメトリックモデルの比較にはどのようなものが使えるんでしょうか(Rで計算できる指数の中で)?
よろしくお願いいたします。
教師 (2012-04-09 (月) 19:40:44)
今年度からRを使った講義を受け持つことになった理工系大学の教員です.もっぱら Ubuntu Linux でしかRを使った経験がないのですが,おそらく学生は Windows を使っているものが多いのではと考えています.Windows 版 (日本語化)Rのインストールは今ではほぼ自動化されているのでしょうか.それとも多少の手動による設定が必要でしょうか.Windows のバージョンでかなり違うのでしょうか.
Windows 版 R のインストールについて現在最も参考になるサイト,もしくは本を教えてください.この Wiki で見つかるインストール法が最良でしょうか.
よろしくお願いします.
Hiro? - 2 (2012-04-09 (月) 12:22:04)
cmprsk_2.2-1 を利用して、競合リスクを考慮したevent のCumulative incidence を下記のようなプログラムで計算しました。しかし、これですと 95% CI が計算されません。どのようなコマンドを加えるとよいでしょうか。ご指摘いただいたように実際のデータをつけて再投稿いたします。
どうかお願いします。install.packages("cmprsk") library(cmprsk) MYDATA (症例は25例で、days:観察期間、censor:eventあり=1、eventなし=0、競合リスク=2) (下記データをexcel上に作成) days censor group 1 3 1 1 2 4 1 1 3 4 1 1 4 5 1 1 5 6 1 1 6 5 1 1 7 11 1 1 8 6 1 1 9 2 1 1 10 19 2 1 11 21 2 1 12 3 1 1 13 2 2 1 14 3 1 1 15 3 1 1 16 7 1 1 17 28 0 1 18 5 1 1 19 5 1 1 20 3 1 1 21 3 1 1 22 4 1 1 23 5 1 1 24 4 1 1 25 3 1 1 MYDATA <- read.delim(pipe("pbpaste"), header=TRUE) #(ここで上記excelデータをコピー) Event <- MYDATA Event$censor <- ifelse(MYDATA$censor == 1, 1, 0) Comp <- MYDATA Comp$censor <- ifelse(MYDATA$censor == 2, 1, 0) Mixed <- MYDATA Mixed$censor <- ifelse(MYDATA$censor == 1, 1, ifelse(MYDATA$censor == 2, 2, 0)) result <- cuminc(MYDATA$days, MYDATA$censor, MYDATA$group, cencode=0) result2 <- timepoints(result, Event$days) result2$"est" 2 3 4 5 6 7 11 19 21 28 (days) group 1 0.04 0.32 0.48 0.68 0.76 0.80 0.84 0.84 0.84 0.84(目的とする事象の累積%) group 2 0.04 0.04 0.04 0.04 0.04 0.04 0.04 0.08 0.12 0.12(競合リスク)
tadashi (2012-04-06 (金) 00:58:14)
windows 用のまずつくってみました。
http://www.e-stat.go.jp/SG1/estat/List.do?bid=000001034991&cycode=0
の1番目のです。
ダウンロードは下記からできます。解凍してから読み込んでください。
http://it.isogaya.co.jp/koku001.zip
文字コードがshift-jis ぽいので、Linux ,Mac では読めないと思います。共通して読めるようにするのはどうしたらいいのでしょうか?> load("koku001.RData") > names(koku001) [1] "地域コード" "地域識別コード" "境域年次.2010." "境域年次.2000." [5] "地域名称" "人口22" "人口17" "人口増減数" [9] "人口増減率" "面積" "人口密度"下記は、全国の人口を求めています。全国の集計を除いて、都道府県で人口を集計しています。当たり前ですが、全国の集計と同じ結果になります。
t <- subset(koku001, 地域識別コード == "a") t[1, 6] t <-t[2:48,] sum(as.numeric(t$人口22))
tadashi (2012-04-05 (木) 17:57:36)
アクセスログをread.table で読み込んでいますが、結構トラブルが発生して取り込めなくなっています。簡素化すると下記のようになります。"a" "b" "c" "aa"a" "bbb" "ccc"を
read.table(file = "test.tsv", sep = "\t", header = TRUE)で読み込むと、
In read.table(file = "test.tsv", sep = "\t", header = TRUE) : 'test.tsv' の readTableHeader で不完全な最終行が見つかりましたというメッセージがでます。データ作成をRuby でやっているので、そこで処理すればいいともいえるのですが、R の中ではダブルクオーテーションがはいっていて読み込みエラーがでるのは、どうやって処理するのが、よく行われているのでしょうか?
tadashi (2012-03-26 (月) 00:17:16)
公開されているデータから、いろいろ面白い分析ができると思っています。衆議院小選挙区と国勢調査を結びつけて分析するような動きは日本であるのでしょうか? 現在、Rで小選挙区と国勢調査の地域コードをリンクしたRのdataをつくりました。こういったことに興味ある方はいらっしゃるのでしょうか?
> (e <- load("e"))
[1] "shu45"
「テキストエディット」で読んでみたけど,
北原 (2012-03-21 (水) 21:39:27)
Rの初心者のものです。
Rでパワースペクトル解析(spectrum)を行い、plotで図を描いています。ところで、横軸(x軸)も対数をとった図にしたいんですが、どうすればいいんですか?
教えてください。
layout(matrix(1:2, 1)) spg.out <- spectrum(UKgas, method="ar") plot(spg.out$freq, spg.out$spec, log="xy", type="l") layout(1)これにより,以下の図が描かれます。 -- 河童の屁は,河童にあらず,屁である。 2012-03-22 (木) 09:54:12
順天太郎 (2012-03-21 (水) 09:30:32)
R 及び数学には初心者です。
質問させていただきます。
幾つかの点がxyz座標上にあります。
その点を P (1,2,3), Q (3,-2,4), R (-,2.-5, 8), 1:5, sep=, S(-2,4, 8), T (-2,-4, -3)とします。
あるxyz軸に対してか平面”(平面Aとする)を設定し、その平面がA1x + B1y + C1z + D1=0とした時、A1 = -0.01、B1 = 0.05, C1= -0.03, D1 =1 すなわち -0.01x + 0.05y – 0.03z + 1 = 0で表されるとします。
2. 座標(?)全体を回転し、平面Aが平面xyと重なり、かつP, Q, Rの三点の中心を通る様にしたいです。(平面A’)。
3. この回転運動を行ったとき、上記P,Q,R,S, Tの新しい位置はどの様になるでしょうか?またその式はどの様に求められるのでしょうか。
宜しくお願いたします。
順天太郎
パン屋 (2012-03-20 (火) 20:57:18)
こんばんは。
igraphを用いてグラフを書きました。c1 <- c("a", "a", "a", "b", "b", "c") c2 <- c("b"," c", "d", "c", "d", "d") c3 <- c(5, 10, 1, 0, 100, 20) tmp1 <- data.frame(c1 = c1, c2 = c2, c3 = c3) library(igraph) tmp2 <- graph.data.frame(tmp1[1:6,], directed = F) E(tmp2)$weight <- c3 print(tmp2, e = TRUE, v = TRUE) plot(tmp2, vertex.label = V(tmp2)$name, layout = layout.fruchterman.reingold, edge.label = E(tmp2)$weight)としたのですが、重み(c3)が結果に反映されてないようです。どのようにしたらよいかご教授いただけないでしょうか。よろしくお願いします。
R version 2.14.2 (2012-02-29) Platform: i386-pc-mingw32/i386 (32-bit) other attached packages: [1] igraph_0.5.5-4
サワボノリ (2012-03-16 (金) 23:49:52)
こんにちは。plot(0, xlab="") mtext(side=1, at=0.8, text="panda score=0", line=-4, adj=1) mtext(side=1, at=0.8, text="cat score=1+", line=-5, adj=1)とした時に 0 と 1 が揃うように表示したいのですが、やり方がわかりません。family="mono" であれば "panda score=0 " とするだけで良いのですが、 family="sans" の時はそうシンプルにはできません。
"+" と同じ幅の空白を挿入する方法がありましたら教えてください。
latex の \phantom{+} と同じようなものはないのでしょうか?
\begin{overpic}[
bb= 0 0 504 504, trim=30 30 30 50,
width=0.48\linewidth, height=0.48\linewidth,
clip]{abc.eps}
\put(65,35){\tiny
\begin{tabular}[t]{|lcr|} \hline
○:ABC\rule[0pt]{0pt}{5pt} &$=$ & 0 \\
△:DEF\rule[-2pt]{0pt}{5pt} &$=$ & +1 \\\hline
\end{tabular}}
\end{overpic}mtext2 <- function(string, at, ...) {
eq <- regexpr("=", string)
len <- strwidth(substring(string, eq+1))
mtext(text=string, at=at+len, adj=1, ...)
}
plot(0, xlab="")
mtext2("panda score=0", 0.8, side=1, line=-4)
mtext2("cat score=1+", 0.8, side=1, line=-5)
このままでは汎用性がありませんから(等号がふたつ以上あるとおかしくなるなど)、あとは必要と実際の使用環境に応じて例外処理などをつけたせばよいかと。 -- 2012-03-17 (土) 23:20:20Cent (2012-03-13 (火) 13:43:33)
こんにちは。Ceut OS 5.1, R 2.13.2を使用しています。
名前にスペースが含まれているディレクトリにsystem()でunixコマンドを実行したいのですが、スペースが邪魔でエラーになってしまいます。簡素化した例を挙げると#"aaa bbb"というディレクトリにUNIXコマンドを実行する dir_name <- "aaa bbb" dir.create(dir_name) path <- paste(getwd(),"/",dir_name, sep="") system(paste("ls ",path," > ",path,"/ls_res.txt",sep=""))この場合、以下のエラーメッセージが表示されます。
ls: bbb: そのようなファイルやディレクトリはありません ls: bbb/ls_res.txt: そのようなファイルやディレクトリはありませんgsub()でディレクトリ名の"aaa bbb"の半角スペースをエスケープしてからsystem()に渡せば良いと思ったのですが、
gsub("aaa bbb","aaa\ bbb",path)や
gsub("aaa bbb","aaa\\\\ bbb",path)としてみましたが、 "/root/aaa\ bbb"という文字列をつくり出すことができません。どのようにすればsystem()で処理できるでしょうか?
宜しくお願いします。
% ls aaa bbb ls: aaa: No such file or directory ls: bbb: No such file or directoryはだめだけど,以下の2つはちゃんと動く
% ls 'aaa bbb' xxx.txt test99.dat test99.txt test99.log % ls "aaa bbb" xxx.txt test99.dat test99.txt test99.logこれが理解できれば,R から system 関数を使って操作するのも同じこと。
dir_name <- "aaa bbb"
dir.create(dir_name)
path <- paste(getwd(),"/",dir_name, sep="")
system(paste("ls '",path,"' > '",path,"/ls_res.txt'",sep=""))
つまり,あなたのスクリプトで system に渡される文字列は
> paste("ls ",path," > ",path,"/ls_res.txt",sep="")
[1] "ls /foo/bar/aaa bbb > /foo/bar/aaa bbb/ls_res.txt"
なのですが, ' を挿入した文字列は以下のようになるのです。
> paste("ls '",path,"' > '",path,"/ls_res.txt'",sep="")
[1] "ls '/foo/bar/aaa bbb' > '/foo/bar/aaa bbb/ls_res.txt'"
で,こうやって ' でくくられていればパス名に空白を含んでいても構わない。 -- 河童の屁は,河童にあらず,屁である。 2012-03-13 (火) 17:15:14SAS ユーザ (2012-03-10 (土) 14:10:23)
Rで行単位にテキストファイル※を読み込み随時処理を行うことは可能でしょうか?
目的は一度にメモリに乗り切らないような大量データの処理です。
AWKやSASのデータステップをイメージしています。
readlines、及びscanは一度に全ファイルを読み込みに行くので目的に沿いません。
※可能であればデータフレームに対しても同様の処理を行いたいです。
tadashi (2012-03-09 (金) 17:36:51)
アクセスログをRで分析するために、データフレームの繰り返し処理をしたいのですが、ある程度、詳しく書いたページはないでしょうか?
for (適当な変数名 in データフレーム) { データフレーム名$変数 etc}
で使えるようなのですが、普通は使わないのでしょうか?
for (file.name in c("a.dat", "b.dat", "c.dat")) {
x <- read.table(file.name)
x を使った処理
}
としてもよいし,処理の部分を関数にして
処理 <- function(ファイル名) {
処理の実体
}
実際の実行はそれぞれのファイルに対してということで
sapply(c("a.dat", "b.dat", "c.dat"), 処理)
とやってもよいだろう。後者の場合は,試験的にというか,少数個のデータについて,
処理("foo.dat")
などということが出来るということだろう(前者でも出来るけど,for の中を書き換えなきゃならん)。> data.frame(t$a1,t$a2,paste(t$a1,t$a2)) t.a1 t.a2 paste.t.a1..t.a2. 1 a a a a 2 b c b c 3 c b c b
> data.frame(s21$seq,s21$ip,s21$ip1,(s21$ip != s21$ip1 )+0) 以下にエラー Ops.factor(s21$ip, s21$ip1) : 因子の水準セットが異なっていますできた。式の間に空白をいれてはいけないのですね。
> data.frame(t$a1,t$a2,result=(t$a1!=t$a2)+0)
t.a1 t.a2 result
1 a a 0
2 b c 1
3 c b 1
> t<-data.frame(a1=c('a','b','c'),a2=c('a','c','b'))
> data.frame(t$a1,t$a2,result=(t$a1!=t$a2)+0)
t.a1 t.a2 result
1 a a 0
2 b c 1
3 c b 1dashi]] &new{2012-03-10 (土) 21:07:14};fr='20111010.tsv'文字での読み込みにして、因子化を避けます。
log<-read.table( file=fr,header=FALSE,sep="\t",as.is=c(TRUE))通常のログの形式の先頭に、unix timestamp などをつけています。
names(log)<-c( "utime","yyyy","mm","dd","hour","minute","second", "ip","identd","user","datetime","request","code", "bytes","referer","agent")ip と時刻で並べ替え、
t<-order(log$ip,log$utime) log<-log[t,]ip,utimeを切り出します。この時点で、因子化されてしまいます。
s1<-data.frame(ip=log$ip,utime=log$utime) s2<-data.frame(ip=log$ip,utime=log$utime) t<-data.frame(ip="999.999.999.999",utime=0)ずらして並べます。
s2<-rbind(s2,t)
s2<-s2[-1,]
t<-nrow(s1)
s1<-cbind(seq=1:t,s1)
s2<-cbind(seq=1:t,s2)
names(s2)<-c('seq','ip1','utime1')
s12<-merge(s1,s2)
因子をcharacter に戻します。
s12$ip<-as.character(s12$ip) s12$ip1<-as.character(s12$ip1)ip アドレスが変わるところに 1 をたてます。
s12$test<-(s12$ip != s12$ip1)+0 head(s12) seq ip utime ip1 utime1 test 1 1 114.80.93.71 1318209476 114.80.93.71 1318209532 0 2 2 114.80.93.71 1318209532 114.80.93.71 1318209532 0 3 3 114.80.93.71 1318209532 119.235.237.16 1318186208 1 4 4 119.235.237.16 1318186208 119.235.237.16 1318207179 0 5 5 119.235.237.16 1318207179 119.235.237.19 1318187392 1 6 6 119.235.237.19 1318187392 119.235.237.19 1318257867 0
のす (2012-03-09 (金) 06:21:30)
よろしくお願いしますA B C D 1 赤 青 黄 白 2 赤 黄 白 白 3 青 白 白 黒 4 白 黄 赤 黒 5 黒 白 青 青 6 赤 赤 赤 赤このような表から
A B C D 赤 3 1 2 1 青 1 1 1 1 黄 0 2 1 0 白 1 2 2 2 黒 1 0 0 2このようなクロス集計表を得たいのですが、どのようにしたら出来るのでしょうか。
いままでtableを使って列ごとに処理した後、エクセルで成型していました。
環境は Win7 64, R2.14.1です。
> sapply(lapply(d, factor, levels=names(table(sapply(d, levels)))), table) A B C D 黄 0 2 1 0 黒 1 0 0 2 青 1 1 1 1 赤 3 1 2 1 白 1 2 2 2
> a <- data.frame(color = c(sapply(d, factor)),
+ group = rep(LETTERS[1:4], each=6),
+ count = rep(1, 24))
> xtabs(count ~ color + group, a)
group
color A B C D
黄 0 2 1 0
黒 1 0 0 2
青 1 1 1 1
赤 3 1 2 1
白 1 2 2 2以下にエラー table(sapply(d, levels)) : 全ての引数は同じ長さでなければなりませんというエラーが出力されるのですが、理由がわかるでしょうか。データに問題があるかと思い、いろいろと試していますが、規則性がよくわかりません。-- のす 2012-03-09 (金) 15:34:06
A B C D 1 黄 黄 赤 黄 2 黄 黄 白 青 3 白 黄 赤 白 4 赤 黄 黄 白 5 赤 黄 黒 白 6 黄 黄 白 黒エラーが起きる原因は,sapply(d, levels) の結果得られるリストの要素数が異なるということです。
> sapply(d, levels) $A [1] "黄" "赤" "白" $B [1] "黄" $C [1] "黄" "黒" "赤" "白" $D [1] "黄" "黒" "青" "白"つまり,この例だと A の要素数は3,B の要素数は 1,C,D の要素数はともに4というようになっているのがまずく,この結果に table 関数を作用させると,
> table(sapply(d, levels)) 以下にエラー table(sapply(d, levels)) : 全ての引数は同じ長さでなければなりませんというエラーが出てくるのですね。
> table(list(a=c("a", "b"), b=c("a", "c"), c=c("c", "x")))
どんな答えが出てくるか,プログラムを実行せずに予測してみてください。私は,出てくるのとは全く違った答えを予想していたのです。> sapply(lapply(d, factor, + levels=names(table(unlist(sapply(d, levels))))), + ~~~~~~~ ~ + table) A B C D 黄 3 6 1 1 黒 0 0 1 1 青 0 0 0 1 赤 2 0 2 0 白 1 0 2 3当然ながら,別法による答えと同じになることは確かめました。
> a <- data.frame(color = c(sapply(d, factor)),
+ group = rep(LETTERS[1:m], each=n),
+ count = rep(1, n*m))
> xtabs(count ~ color + group, a)
group
color A B C D
黄 3 6 1 1
黒 0 0 1 1
青 0 0 0 1
赤 2 0 2 0
白 1 0 2 3
思い込みとは恐ろしいものです。 -- 河童の屁は,河童にあらず,屁である。 2012-03-21 (水) 18:32:02kejuyan (2012-03-08 (木) 00:06:40)
Rでグラフなどの出力をMathematicaのノートブック風にできないでしょうか?
私の環境は、Windows7,R-2.12です。
色々調べたところTeXmacs + Rという組み合わせで実現できるような記述も見つけたのですが、設定の仕方が分かりませんでした。
TeXmacs + Rに限らず、Windows7でRの出力をMathematica風に出来る方法があれば、具体的な設定方法も含めてご教授宜しくお願いします。
tadashi (2012-03-05 (月) 15:26:37)
apache のアクセスログのデータフレームで、user-agent のところで、bot の文字がはいっているデータ(列)を抽出したいのですが、どうするのがいいのでしょうか、正規表現で、簡単にできると思うのですが、書き方がわかりません。
subset(データフレーム、条件(正規表現))のように書くと思われるのですが、可能ではないでしょうか?
> (d <- data.frame(
UserAgent=c("foo", "bar", "bot", "baz", "robots"),
x=LETTERS[1:5]))
UserAgent x
1 foo A
2 bar B
3 bot C
4 baz D
5 robots E
# データフレームの UserAgent 列に "bot" という文字列を含む行を取り出す
> d[grepl("bot", d$UserAgent),]
UserAgent x
3 bot C
5 robots Eユーザー (2012-03-05 (月) 11:11:10)
read.tableコマンドでCSVファイルを読み込もうとしましたがう下記のような警告がでてうまくいきません。どうすればよいでしょうか?
以下、コマンドと警告です。> Ar1TestData <- read.table("C:\Users\g-fujimoto\Dropbox\Ar1TestData.csv",header=TRUE) エラー: "C:\U"で始まる文字列の中で8進文字なしに'\U'が使われています
saito (2012-03-04 (日) 10:20:33)
OS Windows7 x64を使用しておりますがイベントID(E)63のエラー報告がされます。メッセージは次の様です。"C:program files\R\r-2.14.1\Tc\bin64\tk85.dll"のアクティブ化コンテキストの生成に失敗しました。マニフェストまたポリシーファイル"C:program files\R\r-2.14.1\Tc\bin64\tk85.dll"行9のエラーです。要素"assemblyIdentity"の属性"processerArchitecture"に無効な値"x64"が指定されています。
上記のようなエラーメッセージですが気に病むような問題なのか無視しても構わないのか判断に迷っております。何方か解決策をご教授願えないでしょうか?お忙しい中申し訳ありませんが宜しくお願い致します。
みのむし (2012-03-02 (金) 15:24:49)
Rを使って計量経済学の分析をしています。
最近ある研究者が自分の開発した手法をRで使うためのパッケージを開発して、学術目的に限り無料で配布しています。
私は彼の手法を少し改良したものを実際の分析で使いたいと考えています。
そこで、彼の作成したパッケージに手を加えたいのです。
ところが、ダウンロードしたパッケージを見ると、拡張子がrdbやrdxとなっており、もととなったコードを読むことができません。
Rのパッケージのソースコードを読むにはどうすれば良いのでしょうか?
どなたかご教示いただければ大変幸いです。
tsujimo (2012-03-01 (木) 00:41:25)
最近Rを触り始めました#用語がおかしかったらお知らせください,なるべく改めて行きたいと思います
データフレームからデータを抽出するのに以下を試みましたが,結果がどうも解せません
c(1,3)で抽出しようとしたものは何が抽出されたのでしょうか?
どういうデータが取りこぼされたのでしょうか?#結果のデータを見比べて見ても特に規則性は見えませんでした
> nrow(raw[which(raw["Q1"]==c(1), useName=TRUE, arr.ind=TRUE),]) [1] 8526 > nrow(raw[which(raw["Q1"]==c(3), useName=TRUE, arr.ind=TRUE),]) [1] 14750 > nrow(raw[which(raw["Q1"]==c(1,3), useName=TRUE, arr.ind=TRUE),]) [1] 11546ある行"Q1"を選択肢に複数の不連続な選択番号で抽出したかったのですがどうすれば良かったのでしょうか?
複数の不連続な選択番号(ここではc(1,3))を関数の引数として受け取りたいとも考えています
ループ版は作れたのですが悲しい位遅いです
以上よろしくお願いします
s2;
> set.seed(1)
> (raw <- data.frame(Q1=1:5, x=sample(LETTERS, 5))) # 簡単な例を作る
Q1 x
1 1 G
2 2 J
3 3 N
4 4 U
5 5 E
> raw[which(raw["Q1"]==c(1), useName=TRUE, arr.ind=TRUE),]
Q1 x
1 1 G
1.1 1 G
# arr.ind を指定するのがおかしい
> which(raw["Q1"]==c(1), useName=TRUE, arr.ind=TRUE)
row col
[1,] 1 1
# raw へ渡される引数(行番号)がおかしいことが分かる
> raw[which(raw["Q1"]==c(3), useName=TRUE, arr.ind=TRUE),]
Q1 x
3 3 N
1 1 G # 意図しないものも抽出されている なぜだろうかと考える
> which(raw["Q1"]==c(3), useName=TRUE, arr.ind=TRUE)
row col
[1,] 3 1
> raw[which(raw["Q1"]==c(1, 3), useName=TRUE, arr.ind=TRUE),]
Q1 x
1 1 G
1.1 1 G
> which(raw["Q1"]==c(1, 3), useName=TRUE, arr.ind=TRUE)
row col
[1,] 1 1
> raw[raw[,"Q1"] %in% c(1, 3),] # これが解
Q1 x
1 1 G
3 3 Nそとの (2012-02-28 (火) 10:15:35)
ネットでみつけたデータを使って回帰分析の勉強をしています。d <- read.table("http://personality-project.org/r/datasets/R.appendix2.data", header=T) lm1 <- lm(Alertness ~ Gender * Dosage, data=d)上の回帰分析で求められる切片と下の計算で得られる平均が同じになることはわかったのですが、両者のsdの違いが説明できません。
mean(d$Alertness[d$Gender=='f' & d$Dosage=='a'] ) summary(lm1)$coef[1,2] sd(d$Alertness[d$Gender=='f' & d$Dosage=='a'] )このふたつは同じになるものではないのですか?直接的なRの質問でありませんが、よろしくおねがしいます。
tadashi (2012-02-25 (土) 10:38:36)
http://pisa2009.acer.edu.au/downloads.php の生徒の回答(テキストで、非圧縮で1G程度)をRに読み込むと、150メガくらいになります。64bit のwindows で、メモリは、何Gぐらいあると快適に動くのでしょうか?
akira uegaki (2012-02-22 (水) 23:10:18)
Mac OS X (10.7.2)ですが、パッケージlmtestをインストール後、これをロードしようとすると、「エラー: パッケージ '‘zoo’' をロードできませんでした」となって、ロードできません。したがって、DWテストができません。Windowsで同じことをしたときは簡単に成功したのですが。いろいろネット上で探ってみたのですが、わかりません。どなたか、ご教示ください。
tadashi (2012-02-21 (火) 18:16:56)
dataset パッケージ library/datasets/html/00Index.html にいろいろデータがあるのですが、少し詳しく解説しているページがあったら教えてください。
ひろ (2012-02-20 (月) 23:09:37)
Rで開発したソフトを配布したいのですが,ソースコードが丸見えなため,どうにかできないかと悩んでおります.
1.Rの難読化か機械語にコンパイル方法ありますでしょうか?私が調べてみたところ存在せずでした.(RCC(R2C++)は入手できず,Pコンパイラやらはありましたが...)
2.compilerパッケージでコンパイルしてみましたが,ソースコードが添付されてしまいます.ソースコードやコメントを除去する方法はございますでしょうか?英語のマニュアル読んでみましたが,それらしきは見当たりませんでした.もしかしたら,英語苦手なので見落としている可能性はありますが.
ひろ (2012-02-20 (月) 15:10:24)
UTF8に依存した文字コードを含む文字列データをR言語を使って処理しようとしています.Linuxと違ってWinではUTF8の読み込める物の,エラーメッセージが文字化けしたりと困っておりまして,そこでお聞きしたいのが,
1. WindowsでもUTF8はShift-JISの同様に簡単に処理できますか?UTF8の文字列を扱う度iconvを使って変換が必要でしょうか?
2. Mac/Linuxへの移行を検討した方がいいでしょうか?
3. Windows版Rは近い未来にUTF8に対応するか否か?
おちあい (2012-02-20 (月) 04:47:08)
モデルに基づくクラスター分析の勉強のために、以下のスクリプトをあるウエブサイト(http://mjin.doshisha.ac.jp/R/29/29.html)から使用しています。> install.packages("mclust") > iris2<-iris[51:150, 1:4] > library(mclust) > plot(EMclust(iris2)) > mhc <- hc(modelName="EEE", data=iris2) # もとはEEEここまで来ると
以下にエラー hcEEE(data = iris2) : hc for EEE model is not currently supportedが表示されます。
他のモデル、EII、VII、VVVではこのエラーは表示されません。
EEEのモデルを採用し処理を続けるにはどうしたらよいのでしょうか。アドバイスを頂けたらと思います。
使用環境
R version 2.14.0 (2011-10-31)
Platform: x86_64-pc-mingw32/x64 (64-bit)
Windows 7
stop("hc for EEE model is not currently supported")
temp[[4]] <- temp[[4]][1:2]
temp[[5]] <- temp[[5]][1:2]
: 以下略ひらやま (2012-02-18 (土) 10:40:35)
分布モデル作成に有用なdismoパッケージを使っているのですが、先日から突然このパッケージが読み込めなくなってしまいました。読み込もうとすると「応答なし」となって固まってしまいます。その他のパッケージは問題なく読み込むことができ、dismoパッケージのみこの症状が出ます。
以下のことは試してみましたが解消されません。
・パッケージの更新
・パッケージの削除、再インストール
・旧バージョンのパッケージの使用
・Rをアンインストールし、再インストール
また、昨日まで問題なくdismoパッケージが読み込めていた別PCでも、今朝になると全く同じように読み込めなくなっていました。昨夜から今朝にかけて行っていた作業内容はパッケージdismo内のmaxent()というコマンドを使って、架空データで10000回の分布モデル作成でした。
どのように対処していいものか悩んでおります。ご助言いただけますと助かります。よろしくお願いします。
使用環境
OS: Windows 7
R version: 2.14.1
対象package: dismo
ただばやし (2012-02-04 (土) 14:51:06)
積み上げグラフを書いていて、バーを塗りつぶした上に模様を描きたいと考えています。(例えば群集組成を示す図で生物の属ごとに色を与え、種ごとに模様を変えたい)
barplotで図を書いているのですが、angleとcolで模様を変えたり模様に色を付けることはできるのですが、バー自体を塗りわけることができず困っております。
ご助言頂けますと助かります。
よろしくお願いします。
x <- matrix(sample(10:50, 20), 4)
dimnames(x) <- list(LETTERS[1:4], letters[1:5])
barplot(x, col = c("#aa000070", "#00aa0070",
"#0000aa70", "#aa660070"))
par(new = TRUE)
barplot(x, col = 1:4, angle = 1:4 * 45, density = 10)
mp <- barplot(x, col = seq_len(nrow(x)) + 1)
rect(mp - 0.5, 0, mp + 0.5, colSums(x), col = "black", density = 10,
angle = 45 * seq_len(ncol(x)))
のざわ (2012-02-04 (土) 08:36:34)
宜しくお願いします。
以下のようなプログラムで和分を実数と整数(1)にしてsummaryを見ているのですが相関係数もAICも全く変わりません。
何かバグでもあるのでしょうか?library(fracdiff) AP.d <- fdGPH(AirPassengers)$d AP.fra <- fracdiff(AirPassengers, nar = 3, dtol = AP.d, nma = 1) summary(AP.fra) AP.fra <- fracdiff(AirPassengers, nar = 3, dtol = 1.0, nma = 1) summary(AP.fra)
Yuki (2012-02-01 (水) 16:10:38)
多数の時系列データをARIMAモデルで予測したいと考えています。
try関数によりエラーとなった場合は次のデータに行くようなプログラムを作成しようとしていますが、try関数を使ってもうまく戻り値を捉えることができず困っています。対処方法をご存じの方がいらっしゃいましたらお教え頂けますか?
私が引っかかっている箇所は以下になります。
利用データ:(エラーとなった1データのみ記載しています。実際はループして処理をしています。)myts <-ts(c(92,85,77,178,174,161,93,81,120,136,84,117,92,108,87,97,82,99, 142,205,142,75,60,92,113,93,128,64,72,71,89,98,103,127,97,101, 86,81,85,57,79,86,83,99,89,132,136,89,85,107,50,22,97,148,102, 79,79,80,40,99,178,77,97,89,81,73,126,129,160,98,23,86,105,113, 120,106,105),freq=7, start=c(7,13))arimaで処理が正常に終わる場合、以下のように、
> (class(try(fit <- arima(myts,order=c(1,1,1),method="CSS-ML"),silent=true))) [1] "Arima"が返ってきますので、下記のようにifelse関数を用いてmyresに戻り値を格納し、myresの値を利用して、FALSEの場合、例外処理に行くようにしたいと考えました。
> (myres <-ifelse(class(try(fit <- arima(myts, order = c(1, 1, 1), method = "CSS-ML"), silent = true)) == "Arima", TRUE, FALSE) ) [1] TRUE正常にARIMAで予測できる場合は、期待通り「TRUE」が返ってきます。 しかし、エラーとなるmethod="ML"で実行すると、期待した「FALSE」が返ってきません。
> (myres <-ifelse(class(try(fit <- arima(myts, order = c(1, 1, 1), method = "ML"), silent = true)) == "Arima", TRUE, FALSE) ) 以下にエラー value[[3L]](cond) : オブジェクト 'true' がありません 追加情報: 警告メッセージ: In arima(myts, order = c(1, 1, 1), method = "ML") : possible convergence problem: optim gave code=1のようにエラーとなりますが、myresの値は更新されず、以下のように前の値のままです。
> myres [1] TRUE念のため、myresをrmしてから確認すると、以下のようにmyresに値が帰っていないことがわかりました。
> rm(myres) > (myres <-ifelse(class(try(fit <- arima(myts, order = c(1, 1, 1), method = "ML"), silent = true)) == "Arima", TRUE, FALSE) ) 以下にエラー value[[3L]](cond) : オブジェクト 'true' がありません 追加情報: 警告メッセージ: In arima(myts, order = c(1, 1, 1), method = "ML") : possible convergence problem: optim gave code=1 > myres エラー: オブジェクト 'myres' がありません利用環境は以下のとおりです。
Rのバージョン:2.14.1 OS:WindowsXP SP3 利用パッケージ: stats graphics grDevices utils datasets methods baseどうぞよろしくお願い致します。
> (myres <-ifelse(class(try(fit <- arima(myts, order = c(1, 1, 1), + method = "ML"), silent = TRUE)) == "Arima", TRUE, FALSE) ) [1] FALSE ←←← FALSE になりますよ 警告メッセージ: In arima(myts, order = c(1, 1, 1), method = "ML") : possible convergence problem: optim gave code=1それと,ifelse は不要。以下と同じ。
> (myres <-class(try(fit <- -data.frame(ip=arima(myts, order = c(1, 1, 1), + method = "ML"), silent = TRUE)) == "Arima" )
まあくん (2012-01-26 (木) 10:32:57)
expression()の使い方ですが、expression(paste(bgroup("(", atop(n, x), ")"), p^x, q^{n-x}))を
temp <- "q^{n-x}" expression(paste(bgroup("(", atop(n, x), ")"), p^x, temp))のように書き換えたいのですが、うまくいきません。どのようにすればうまくいくでしょうか?
temp <- "q^{n-x}"
eval(parse(text=paste('expression(paste(bgroup("(", atop(n, x), ")"), p^x, ',
temp, '))', sep='')))のす (2012-01-23 (月) 18:02:07)
投稿ルールも読まず、汚い投稿をしてしまい重ね重ね申し訳ありません。
お詫びします。
先ほどの、質問ですが、以下の様な処理を行った場合、「小学生…」のグラフと、「イチゴ…」のグラフが重ねて出力されます。
ところが、「Excelで学ぶコレスポンデンス分析」(高橋信 著)の123ページには「大学生、高校生…」のグラフと、「なし、いちご…」のグラフとを別々に描いてもかまわないと記述されています。
これをRで行い、2つのグラフが得られるようにしたいのですが、よくわかりません。
よろしくお願いします。
環境はwin7(64)
R ver 2.14.1です。library(MASS) y <- data.frame(小学生 = c(4, 3, 4, 7, 3), 中学生 = c(5, 9, 6, 7, 4), 高校生 = c(6, 2, 3, 7, 6), 大学生 = c(7, 1, 4, 6, 7)) rownames(y) <- c("イチゴ", "みかん", "ぶどう", "りんご", "なし") y.coa <- corresp(y, nf=2) biplot(y.coa, main="好きな果物調べ")
layout(matrix(1:2, 2)) old <- par(mar=c(3, 3, 1, 3), mgp=c(1.6, 0.6, 0)) plot(y.coa$rscore) text(y.coa$rscore, label=dimnames(y.coa$rscore)[[1]], pos=4, xpd=TRUE) plot(y.coa$cscore) text(y.coa$cscore, label=dimnames(y.coa$cscore)[[1]], pos=4, xpd=TRUE) par(old) layout(1)
るち (2012-01-23 (月) 15:11:05)
はじめまして。
今T^2管理図の管理限界楕円を図に出したいのですが、その前に陰関数を図に表せるかが知りたいと思っています。
円であれば半径の指定さえすれば表示できることはわかるのですが、楕円になる陰関数での図式化の方法があれば教えていただけたらと思います。
> i <- seq(from = 0, to = 2 * pi, length = 50) > plot(6 * cos(i), 2 * sin(i), type = "l", asp = 1)
ななみ (2012-01-21 (土) 21:55:03)
はじめまして。npパッケージでモデルの検定をしています。model <- lm(Y ~ X, y = TRUE, x = TRUE) X <- data.frame(X) npcmstest(model = model, xdat = X, ydat = Y, bws = 1)のようにバンド幅を指定すると
「仮引数 ”bws” が複数の実引数にマッチしました」というエラーがでてしまいます。
どなたか、これに対する解決方法をご存じでしたらお教え頂きますようお願い致します。
masa (2012-01-19 (木) 14:13:00)
お世話になります。
ヘルプやこのサイトを検索して見つけられず、非常に基本的な点ではと思うのですが、ご教授ください。
下記のような関数で階層化クラスタリングを行おうとしています。dataset の名称を文字列として、ところどころ(# here の行)で使いたいのですが、下記のままだとオブジェクトの内容そのものが入ってしまいます。myhc <- function(varset, dataset) { hc <- hclust(dist(model.matrix(varset, dataset)), method= "ward") plot(hc, main= paste("Cluster Dendrogram for Solution", dataset, sep=""), # here xlab= paste("Observation Number in", datasetname), sub="Method = Ward; Distance = Euclidian", hang=-1) rect.hclust(hc, k = 5, border = "red") dev.print(jpeg, filename = paste(dataset, ".jpg"), width = 800, height = 600) # here } sessionInfo()~ [#w973c77e] R version 2.14.1 (2011-12-22) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] splines graphics stats utils grDevices tcltk base other attached packages: [1] RODBC_1.3-4 Rcmdr_1.8-1 relimp_1.0-3 car_2.0-11 [5] survival_2.36-10 nnet_7.3-1 MASS_7.3-16 loaded via a namespace (and not attached): [1] tools_2.14.1
zou (2012-01-16 (月) 16:33:26)
merge()で、by= に複数列指定する方法があれば教えて下さい。(x <- data.frame( name1 = c("tanaka", "tanaka", "tanaka", "suzuki", "suzuki", "suzuki"), year1 = c("2000", "2001", "2005", "2000", "2005", "2008"), test1 = 1:6)) (y <- data.frame( name2 = c("tanaka", "tanaka", "suzuki", "tanaka", "suzuki", "suzuki"), year2 = c("2000", "2001", "2000", "2005", "2005", "2008"), test2 = c(11, 12, 14, 13, 15, 16))) # m1 <- merge(x, y, by.x = "name1", by.y = "name2", all = TRUE))ほしい結果は次のものです。よろしくお願いします。
なお、ソートして結合するやり方ではなく、merge()を使いたいです。tanaka 2000 1 11 tanaka 2001 2 12 tanaka 2005 3 13 suzuki 2000 4 14 suzuki 2005 5 15 suzuki 2008 6 16
> merge(x, y, by.x = c("name1", "year1"), by.y = c("name2", "year2"),
+ all = TRUE, sort = FALSE)
name1 year1 test1 test2
1 tanaka 2000 1 11
2 tanaka 2001 2 12
3 tanaka 2005 3 13
4 suzuki 2000 4 14
5 suzuki 20, 05 5 15
6 suzuki 2008 6 16mamiy (2012-01-16 (月) 11:52:48)
いつもお世話になっております。
R2.13.2を使用していましたが、最近、R2.14.1に変更しました。
R2.13をアンインストールしてから、R2.14.1をインストールしたのですが、Rcmdrにて、日本語を含むパスのファイルのデータインポートでエラーが出ます。[3] エラー: <86><e3><82>ケ繝育畑繝<87>繝シ繧ソ/test.csv", header=TRUE, sep=",", na.strings="NA", dec=".", strip.white=TRUE)に不正なマルチバイト文字がありますR2.13.1でもR2.13.2でもエラーはなく、日本語を含むファイルパスのものも、データインポートできていたのです。何か設定などしなければいけないのでしょうか。
データのインポートを実行した際にRコマンダーのスクリプトウィンドウに表示されるスクリプトを選択して、右クリックで「実行」を選択すると、エラーは出ません。アクティブデータセットには、セットされませんが。
ご存じの方、おられましたら、お教えください。よろしくお願いいたします。
sessionInfo()の結果はR version 2.14.1 (2011-12-22) Platform: i386-pc-mingw32/i386 (32-bit) locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 LC_MONETARY=Japanese_Japan.932 [4] LC_NUMERIC=C LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods baseよろしくお願いいたします。
宗二 (2012-01-15 (日) 16:02:34)
よろしくお願い致します。下のプログラムは、2列×10行の配列bを作り、要素が999の行で区切られたそれぞれの部分を線プロットしています。a1 <- seq(1, 10, by = 1) a2 <- seq(1, 5.5, by = 0.5) b <- cbind(a1, a2) b[1, ] <- b[4, ] <- b[10, ] <- 999 separator <- which(b[, 1] == 999) for (i in 1 : (length(separator) - 1)) { data <- b[(separator[i] + 1) : (separator[i + 1] - 1), ] if (i == 1) { plot(data, xlim = c(1, 10), ylim = c(1, 10), type = "l") } else { par(new = TRUE) plot(data, xlim = c(1, 10), ylim = c(1, 10), type = "l", axes = F, ann = F) } }このサンプルのように区切りの数が少なく、forループの数が少なければ問題ないのですが、区切りの数が莫大になると、作図するのにかなり時間がかかってしまいました。そこで、forループを使わないで高速化できないか考えたのですが、具体的な方法が浮かばず悩んでいます。何か高速化するいい方法、もしくはアイディアがあればご教授お願いします。
竹下 (2012-01-15 (日) 08:03:21)
こんにちは。
response variable が割合 (p) なのですが、 lm( log(p / (1-p)) ~ x ) とするのは統計学的に良いやり方なのでしょうか?
asap (2012-01-12 (木) 18:36:36)
今ある行列irisに、n列の場合分けに応じた新しい列を追加したいのですが、どのようにすればよいでしょうか
新しい列の変数には次のような値を入れたいのですがif (iris$Species[i] == "setosa") { iris$Sp[i] <- 1 } else { iris$Sp[i] <- 0 }
まち (2012-01-11 (水) 21:49:52)
geoRパッケージのvariogで標本バリオグラムの計算を行うと最大距離を持つポイントが省かれてしまいます。
同じデータでgstatパッケージvariogramを使うと省かれません。
パラメータの指定が悪いのでしょうか?
自己相関が無くなる距離なので推定には問題ないと思いますが気になります。
よろしくお願いします。library(geoR) library(gstat) # データの読み込み data(ca20) # geoR vcloud <- variog(ca20) (vcloud) plot(vcloud) # gstat ca20gstat <- cbind(ca20$coords[, "east"], ca20$coords[, "north"], ca20$data) colnames(ca20gstat) <- c("east", "north", "data") ca20gstat <- as.data.frame(ca20gstat) vfgstat <- variogram(data ~ 1, loc = ~east+north, data = ca20gstat, cutoff = vcloud$max.dist, width = vcloud$max.dist / 13) (vfgstat) plot(vfgstat)下記の 13 行目で geoR の場合 $n が 12 点でなく 11 点になってしまいます。
geoRの場合$n $u $v 1 543 43.77376 55.50552 2 1648 131.3213 78.09254 3 2364 218.8688 97.05838 4 2246 306.4163 117.1554 5 2531 393.9638 129.0646 6 2152 481.5114 146.4889 7 1708 569.0589 151.9713 8 1309 656.6064 153.6482 9 694 744.1539 144.7442 10 343 831.7014 146.8878 11 144 919.249 131.8958 12 59 1006.796 139.2288 13 11 1094.344 139.2727gstatの場合
np dist gamma 1 543 60.35719 55.50552 2 1648 132.58886 78.09254 3 2364 220.90992 97.05838 4 2246 305.65494 117.15539 5 2531 391.80321 129.06460 6 2152 482.02453 146.48885 7 1708 568.46999 151.97131 8 1309 652.62969 153.64820 9 694 739.59074 144.74424 10 343 826.14460 146.88776 11 144 913.61963 131.89583 12 59 995.10190 139.22881 13 12 1082.94416 165.16667使用環境
R version 2.12.1 (2010-12-16) Platform: i386-pc-mingw32/i386 (32-bit) gstat_1.0-8 geoR_1.6-35
hiro (2012-01-10 (火) 16:39:38)
R-2.14.1 for Windows(32bit)を使っています.
R-2.13.2までは、以下のサンプルでスライダーを動かすと、グラフはヒストグラムとカーネル密度共に滑らかに表示されます.
しかし、2.14以降、スライダーを動かしている間は一部のグラフが表示されなくなりました.この例ではカーネル密度が時々消えます.
グラフが複雑化すると、目立って見苦しくなります.
複数のWindowsPC(XP/Win7)で試しましたが、100%再現します.
もし解決策を御存知の方がいらっしゃれば、ご教示頂けないでしょうか.
宜しくお願いします.library(tcltk) draw.graph <- function(...) { upper <- as.double(tclvalue(Slider)) hist(x, freq = FALSE, ylim = c(0, upper), col = "cyan", border = "white") lines(den, lwd = 3) } x <- rnorm(200) den <- density(x) tt <- tktoplevel() Slider <- tclVar("0.5") slider <- tkscale(tt, from = 0.1, to = 1, resolution = 0.01, showvalue = TRUE, variable = Slider, orient = "horizontal", command = draw.graph) tkgrid(tklabel(tt, text = "upper"), sticky = "w") tkgrid(slider) tkfocus(tt)