![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
初心者のための R および RjpWiki に関する質問コーナー
以下の 4 つの「●項目」のどれかをクリック!!
● 新規投稿欄: --- その前に,「投稿における注意事項」を読んでから!
● このページの目次 --- 質問への回答・コメントの参照
● 最新のスレッド --- 最も最近に書き込まれた「親」記事(最新のコメントが付いた記事ではありません)
● Q&A (初級者コース) 過去の目次 --- 今までの 16 個の書庫の参照
参照は,個々の項目をクリック(質問への回答・コメントの参照も,個々の項目をクリック)
●● 新規投稿 ●●
まずは,「投稿における注意事項」を読んで,それに従ってください。
これに従わない場合には,回答が得られにくい場合があります。
univ (2019-12-16 (月) 23:52:06)
OECDによる国際調査PIAACを分析しているのですが、table作成時にエラーが出て困っています。「オブジェクト###がありません」というエラーへの対処を教えていただければ幸いです。よろしくお願いいたします。library("intsvy") data("piaac", package= "PIAAC") ptable <- piaac.table(variable = "AGEG10LFS", data = piaac)を実行すると、
data.frame(table(data[[variable]][drop = TRUE]), Percentage = round(as.numeric(tabtot), でエラー: オブジェクト 'tabtot' がありませんとエラーが出ます。
tabtotはintsvy.table関数内で定義されているようです。intsvy.table <- function (variable, by, data, config) { table.input <- function(variable, data, config) { if (sum(is.na((data[[variable]]))) == length(data[[variable]])) { result <- data.frame(NA, Freq = 0, Percentage = NA, Std.err. = NA) names(result)[1] <- variable return(result) } if (config$parameters$weights == "JK") { R.wt <- sapply(1:max(data[[config$variables$jackknifeZone]]), function(x) ifelse(data[[config$variables$jackknifeZone]] == x, 2 * data[[config$variables$weight]] * data[[config$variables$jackknifeRep]], data[[config$variables$weight]])) if (isTRUE(config$parameters$varpv1)) { tabrp <- as.matrix(sapply(1:ncol(R.wt), function(x) percent(as.factor(as.numeric(data[[variable]])), total = FALSE, weights = R.wt[, x]))) tabtot <- percent(as.factor(as.numeric(data[[variable]])), weights = data[[config$variables$weight]], na.rm = TRUE, total = FALSE) if (length(tabtot) != 1) { tabse <- apply((tabrp - tabtot)^2, 1, sum)^(1/2) } else { tabse <- 0 } } else { R.wt2 <- sapply(1:max(data[[config$variables$jackknifeZone]]), function(x) ifelse(data[[config$variables$jackknifeZone]] == x, 2 * data[[config$variables$weight]] * ifelse(data[[config$variables$jackknifeRep]] == 1, 0, 1), data[[config$variables$weight]])) R.wt <- cbind(R.wt, R.wt2) tabrp <- as.matrix(sapply(1:ncol(R.wt), function(x) percent(as.factor(as.numeric(data[[variable]])), total = FALSE, weights = R.wt[, x]))) tabtot <- percent(as.factor(as.numeric(data[[variable]])), weights = data[[config$variables$weight]], na.rm = TRUE, total = FALSE) if (length(tabtot) != 1) { tabse <- (apply((tabrp - tabtot)^2, 1, sum)/2)^(1/2) } else { tabse <- 0 } } } if (config$parameters$weights == "BRR") { tabrp <- as.matrix(sapply(1:config$parameters$BRRreps, function(i) percent(as.factor(as.numeric(data[[variable]])), total = FALSE, weights = data[[paste(config$variables$weightBRR, i, sep = "")]], na.rm = TRUE))) tabtot <- percent(as.factor(as.numeric(data[[variable]])), weights = data[[config$variables$weightFinal]], na.rm = TRUE, total = FALSE) if (length(tabtot) != 1) { tabse <- sqrt(rowSums((tabrp - tabtot)^2)/20) } else { tabse <- 0 } } result <- data.frame(table(data[[variable]][drop = TRUE]), Percentage = round(as.numeric(tabtot), 2), Std.err. = round(tabse, 2)) names(result)[1] <- variable return(result) } if (missing(by)) { output <- table.input(variable = variable, data = data, config = config) } else { for (i in by) { data[[c(i)]] <- as.character(data[[c(i)]]) } output <- ddply(data, by, function(x) table.input(data = x, variable = variable, config = config)) } class(output) <- c("intsvy.table", "data.frame") output }環境は
R version 3.6.2 (2019-12-12) Platform: x86_64-w64-mingw32/x64 (64-bit) Running under: Windows 10 x64 (build 17134) Matrix products: default locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] grid stats graphics grDevices utils datasets methods [8] base other attached packages: [1] ggplot2_3.2.1 svyPVpack_0.1-1 survey_3.36 survival_3.1-8 [5] Matrix_1.2-18 memisc_0.99.20.1 MASS_7.3-51.4 lattice_0.20-38 [9] sca_0.9-0 remotes_2.1.0 intsvy_2.4 loaded via a namespace (and not attached): [1] Rcpp_1.0.1 pillar_1.4.2 compiler_3.6.2 [4] RColorBrewer_1.1-2 plyr_1.8.5 base64enc_0.1-3 [7] tools_3.6.2 rpart_4.1-15 digest_0.6.23 [10] checkmate_1.9.4 htmlTable_1.13.3 jsonlite_1.6 [13] tibble_2.1.1 gtable_0.3.0 pkgconfig_2.0.3 [16] rlang_0.4.2 DBI_1.0.0 rstudioapi_0.10 [19] xfun_0.11 gridExtra_2.3 withr_2.1.2 [22] stringr_1.4.0 knitr_1.26 repr_1.0.1 [25] cluster_2.1.0 htmlwidgets_1.5.1 mitools_2.4 [28] nnet_7.3-12 data.table_1.12.8 reshape_0.8.8 [31] foreign_0.8-72 latticeExtra_0.6-28 Formula_1.2-3 [34] magrittr_1.5 backports_1.1.5 scales_1.0.0 [37] Hmisc_4.3-0 htmltools_0.4.0 splines_3.6.2 [40] colorspace_1.4-1 stringi_1.4.3 acepack_1.4.1 [43] lazyeval_0.2.2 munsell_0.5.0 crayon_1.3.4です。
OSはwindows10 64bitです。
if (config$parameters$weights == "JK") {
tabtot <- ...
}
if (config$parameters$weights == "BRR") {
tabtot <- ...
}
原因はわかりましたが,回避方法はないでしょう。だって,table.input を書き直さないといけないですが,どのように書き直すか知らないから。 -- 2019-12-17 (火) 13:49:00library("intsvy")
piaac.table2 = function (variable, by, data, export = FALSE, name = "output",
folder = getwd())
{
piaac_conf = list(
variables = list(
weightFinal = "W_FSTUWT",
weightBRR = "W_FSTURWT"),
parameters = list(
BRRreps = 80,
weights = "BRR"
)
)
output <- intsvy.table(variable, by, data, config = piaac_conf)
if (export) {
write.csv(output, file = file.path(folder, paste(name,
".csv", sep = "")))
}
return(output)
}
使用例は以下のように。
> piaac.table2(variable = "AGEG10LFS", data = piaac) AGEG10LFS Freq Percentage Std.err. 1 24 or less 29242 19.17 0 2 25-34 28779 18.87 0 3 35-44 30705 20.13 0 4 45-54 31338 20.55 0 5 55 plus 32450 21.28 0しかし,piaac.table2 はもともとこんなに遅いのか?
piaac.table3 = function(variable, digits = 2) {
n = table(piaac[, deparse(substitute(variable))])
total = sum(n)
percent = round(n / total * 100, digits)
df = data.frame(c(n), c(percent))
colnames(df) = c("n", "percent")
return(df)
}
これを使ってやってみるとね,以下のような結果になるよ。
> piaac.table3(AGEG10LFS)
n percent
24 or less 29242 19.17
25-34 28779 18.87
35-44 30705 20.13
45-54 31338 20.55
55 plus 32450 21.28
> piaac.table3(GENDER_R)
n percent
Male 72241 47.37
Female 80271 52.63
どうだかね???tkzhsymd (2019-12-16 (月) 15:13:48)
Rstdio1.2.5でベイズ推定汎用ソフトBiipsをインストールしました。https://biips.github.io/install/から所定のファイルをダウンロードしてinstall.packages()にかけたのですが、以下の警告が出ました。
package ‘Rbiips_0.10.0.zip’ is not available (for R version 3.5.3)
警告だけだから行けたのかと思ってlibrary(Rbiips)としたところ、
‘Rbiips’ was installed by an R version with different internals; it needs to be reinstalled for use with this R version
となり、RstdioでないR3.5.3にはインストールできます。このメッセージはRstudioでダメなのはR3.5.3にインストールされているからということでしょうか。両者は干渉しあうものでしょうか。
R3.5.3の方は問題なくインストール、library()と行けるのですが。
install.packages("Rbiips_0.10.0.zip",dependencies=TRUE)
パッケージを ‘C:/Users/xxx/Documents/R/win-library/3.6’ 中にインストールします
(‘lib’ が指定されていないため) 'pkgs'から 'repos = NULL' を推測
パッケージ ‘Rbiips’ は無事に展開され、MD5 サムもチェックされました
library(Rbiips)
エラー: package or namespace load failed for ‘Rbiips’:
package ‘Rbiips’ was installed by an R version with different internals;
it needs to be reinstalled for use with this R version
つぐ太郎 (2019-12-03 (火) 08:55:42)
pcaMethodsとglmnetのパッケージが見当たりません。代替パッケージ等教えていただけないでしょうか。よろしくお願いいたします
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("pcaMethods")
でインストールできるよと書いてあったりするし。yoshida931 (2019-11-20 (水) 11:13:17)
以下のようなフレームを
Group pre-post Outcome
1 "治療前" 23.5
1 "治療前" 21.17
1 "治療後" 15.5
1 "治療後" 20.5
2 "治療前" 21.17
2 "治療前" 11.17
2 "治療後" 19
2 "治療後" 20.5
次のような表記に変更することは可能でしょうか?
Group <- c(1,1,1,1,2,2,2,2)
pre-post <- c("治療前","治療前","治療後","治療後","治療前","治療前","治療後","治療後")
Outcome <- c(23.5, 21.17, 15.5, 20.5, 21.17, 11.17, 19, 20.5)
サンプルサイズが大きい場合でも一瞬でできたら助かります。
> Group <- df[,1] # 各列をベクトル変数に代入する
> pre.post <- df[,2]
> Outcome <- df[,3]
> dump("Group", file="Group.R") # 変数名をダブルクオートで囲むこと(つまり,文字列で指定する)
> dump("pre.post", file="pre.post.R") # 指定したファイルに書き出される
> dump("Outcome", file="Outcome.R")
データフレームの各列の代入先の変数名も file 名も自由にしてよい。> dput(df[,1]) c(1L, 1L, 1L, 1L, 2L, 2L, 2L, 2L) > dput(df[,1]+0) # L がつくのを嫌がるなら次のようにすればよい c(1, 1, 1, 1, 2, 2, 2, 2)「変数名 <- 」と入力してから,コピペする。 -- 2019-11-20 (水) 14:08:34
かもめのジョナ子 (2019-11-17 (日) 23:20:21)
read.csvで読み込んだデータ(列数150)を呼び出す際に,50未満の列数のものはx[3]やx["変数名"]で呼び出せますが,50以上のものですとx[55]と書くと「未定義の列が選ばれました」とエラーメッセージが出ます。x["変数名"]と書いた場合はデータを呼び出せます。50以上の列数のものもスムーズに呼び出せるようにするにはどのようにしたらよいのでしょうか。よろしくお願いいたします。(既出でしたら申し訳ありません。)
x = data.frame(matrix(rnorm(300*150), 300, 150)) # 300 行,150 列のデータフレーム
colnames(x) = paste0("d", 1:150) # d1, d2, ..., d150 の列名を付ける
dim(x) # 300, 150 が表示される
head(x) # 先頭の 6 行が表示される
x[150] # 150 番目の列の 300 個の要素が 1 行 に 1 個,300 行出力される
それでもやはりだめならば,読み込み元のデータをよく見てみる必要があるでしょう。 -- 2019-11-18 (月) 09:44:54[ トップ | Tips紹介 | 初級Q&A | R掲示板 | 日本語化掲示板 | リンク集 ] [ 編集 | 差分 | バックアップ | 添付 | リロード ] [ 新規 | 一覧 | 単語検索 | 最終更新 | ヘルプ ]ってのが,あるでしょ?その中の「添付」ってのをクリックしなさい。 -- 2019-11-18 (月) 13:57:29
ya (2019-11-12 (火) 16:31:37)
バッファを割り当てることができません
お世話になります。
検索エンジンや過去の質問で見つからなかったので質問させていただきます。既出でしたら申し訳ありません。
R consoleでパッケージをインストールしようとすると、以下の表示が出てしまいます。
BiocManager::install("IlluminaHumanMethylationEPICanno.ilm10b4.hg19")
Bioconductor version 3.10 (BiocManager 1.30.9), R 3.6.1 (2019-07-05)
Installing package(s) 'IlluminaHumanMethylationEPICanno.ilm10b4.hg19'
installing the source package ‘IlluminaHumanMethylationEPICanno.ilm10b4.hg19’
URL 'https://bioconductor.org/packages/3.10/data/annotation/src/contrib/IlluminaHumanMethylationEPICanno.ilm10b4.hg19_0.6.0.tar.gz' を試しています
Content type 'application/x-gzip' length 158431882 bytes (151.1 MB)
downloaded 151.1 MB
installing *source* package 'IlluminaHumanMethylationEPICanno.ilm10b4.hg19' ...
using staged installation
data
moving datasets to lazyload DB
Error in lazyLoadDBinsertVariable(vars[i], from, datafile, ascii, compress, :
バッファを割り当てることができません
ERROR: lazydata failed for package 'IlluminaHumanMethylationEPICanno.ilm10b4.hg19'
removing 'C:/Users/902722/Documents/R/win-library/3.6/IlluminaHumanMethylationEPICanno.ilm10b4.hg19'
設定環境は以下のようになります。
R version 3.6.1 (2019-07-05)
Platform: i386-w64-mingw32/i386 (32-bit)
Running under: Windows 7 (build 7601) Service Pack 1
Matrix products: default
locale:
[1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C LC_TIME=Japanese_Japan.932
attached base packages:
[1] stats graphics grDevices utils datasets methods base
loaded via a namespace (and not attached):
[1] BiocManager_1.30.9 compiler_3.6.1 tools_3.6.1
また、現在のメモリの最大割り当て数は1535MBです。
バッファ値の設定を変更すればインストールできるようになるのでしょうか。もしそうなら、どのようにバッファ値の設定を行えば良いのでしょうか。
どうぞよろしくお願いいたします。
BiocManager::install("IlluminaHumanMethylationEPICanno.ilm10b4.hg19")
を入力したら,なんだかんだで,ずいぶん長い間,なんだかんだインストールしたあげく
The downloaded source packages are in ‘/private/var/folders/s5/rt_bzvk53_d9w2gm5_w9gfmr0000gq/T/RtmpXxmpv8 /downloaded_packages’ 警告メッセージ: install.packages(update[instlib == l, "Package"], l, repos = repos, で: installation of package ‘hexbin’ had non-zero exit statusなんていう,ぶきみなエラーメッセージを出してとまりましたけどね
> BiocManager::install("IlluminaHumanMethylationEPICanno.ilm10b4.hg19")
Bioconductor version 3.10 (BiocManager 1.30.9), R 3.6.1 Patched (2019-09-23
r77210)
Installing package(s) 'IlluminaHumanMethylationEPICanno.ilm10b4.hg19'
installing the source package ‘IlluminaHumanMethylationEPICanno.ilm10b4.hg19’
URL 'https://bioconductor.org/packages/3.10/data/annotation/src/contrib/IlluminaHumanMethylationEPICanno.ilm10b4.hg19_0.6.0.tar.gz' を試しています
Content type 'application/x-gzip' length 158431882 bytes (151.1 MB)
==================================================
downloaded 151.1 MB
* installing *source* package ‘IlluminaHumanMethylationEPICanno.ilm10b4.hg19’ ...
** using staged installation
** data
*** moving datasets to lazyload DB
** inst
** help
*** installing help indices
** building package indices
** testing if installed package can be loaded from temporary location
** testing if installed package can be loaded from final location
** testing if installed package keeps a record of temporary installation path
* DONE (IlluminaHumanMethylationEPICanno.ilm10b4.hg19)
The downloaded source packages are in
‘/private/var/folders/s5/rt_bzvk53_d9w2gm5_w9gfmr0000gq/T/RtmpXxmpv8 /downloaded_packages’
どうしてくれます?(^_^;)-- 2019-11-12 (火) 21:53:32jj (2019-11-05 (火) 01:50:34)
Rstudioのヘルプペインにて、虫眼鏡アイコンの検索ボックスへ移動するショートカットキーは設定可能でしょうか。
「Ctrl+3」でヘルプへ移動後、「Ctrl+F」では、「Find in topic」の方しか選択できないようです。
キーボードショートカットのヘルプ、およびModifyについても目を通したものの、発見することができませんでした。
もしご存知の方がおられましたら、ご教示頂けると助かります。
環境
R version 3.5.3 (2019-03-11)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 18362)
zstk5 (2019-10-29 (火) 20:58:03)
回答を期待する記事は原則 質問コーナー Q&A にお願いします。
と書かれているその下に,質問するとはなんたる肝っ玉。
投稿場所,投稿書式共に,はなはだ不適切だったので,ここにこのように再投稿させました。これだけで,原投稿者にはプレッシャーとは思うが,それは仕方ない(規則だから)
以 下 が 投 稿 内 容。 生 暖 か い 目 で 見 て 答 え て や っ て ち ょ ! !
* * * * * * * * * * * * * * *
こんにちは。
現在、Rを使用してrandomforestを実行しているのですが下記コードを実行すると保存されるobjファイルのサイズが大きく変わってしまいます。
(実際はもう少しデータサイズが大きく複雑なのですが同様の事象は下記コードでも発症します)
違いは、randomforestを実行する部分を自分で作成した関数内においているかおいていないかの差になります。
そのため処理は変わらないと考えているのですが出来上がるobjファイルのサイズが関数化している方は119KB, 関数化していないほうは61KBとなります。
なぜ、関数化するとモデルのサイズが大きくなってしまうのでしょうか。
原因分かる方いましたらご教示お願いいたします。
以下サンプルコード###################### #パターン1 ###################### test <- function() { library(randomForest) set.seed(123) ## 実行 rf <- randomForest(Species ~ ., data = iris, proximity = TRUE, importance = TRUE) saveRDS(rf, file = "test.obj") } test() ###################### #パターン2 ###################### library(randomForest) set.seed(123) ## 実行 rf <- randomForest(Species ~ ., data = iris, proximity = TRUE, importance = TRUE) return(iris) saveRDS(rf, file = "test_.obj") ---環境情報--- OS:Windows Server2012R2(64bit) R:version 3.6.1 Rstudio:
.. ..- attr(*, ".Environment")=<environment: 0x7fc91ecd9438> 関数内で保存した場合 .. ..- attr(*, ".Environment")=<environment: R_GlobalEnv> トップレベルで保存した場合まあ,save したものを load するときの事を考えると,「どの環境に load するか」という情報が必要なんでしょうね。そして,環境内という情報はトップレベルにおける情報と量が違うとか?
tkzhsymd (2019-10-23 (水) 09:53:52)
現時点でのRstdioの最新バージョンはそれぞれ1.5xxだと思います。その状態でライブラリをインストールすると”3.6ではダメ”というメッセージになりました。Rstdioの中にRが入っているということでしょうか。
ところで、Rstdioがあれば別途インストールしておくRは不要ということになるのでしょうか。周囲にはRstdioがあればいらないということのようですが。
R version 3.6.1 Patched (2019-09-23 r77210) -- "Action of the Toes" Copyright (C) 2019 The R Foundation for Statistical Computing Platform: x86_64-apple-darwin15.6.0 (64-bit) 以下略というのが示されるでしょう?RStudio は R を使っているんです。
Tomo (2019-08-09 (金) 10:16:14)
こんにちは。
lpSolveやRglpkを使って線形計画法をトライしているのですが、結果が正しく出ません。
制約条件の設定が意図通りに効きません。以下のコードの何がおかしいでしょうか。
すみませんが分かる方教えてください。
やりたいこと:
・弁当、菓子パン、おにぎり、お菓子、ジュースを 与えられた制約条件の中で
“最も短い時間で作りたい”(目的関数)
・弁当、菓子パン、おにぎり、お菓子、ジュースは、
それぞれ機械A、B、Cの“いずれか1つを使う”(制約条件1)
・機械A、B、Cは 使える合計時間上限が決まっている(制約条件2)
・弁当、菓子パン、おにぎり、お菓子、ジュースは機械A、B、C
どれを使うかによって、作るのにかかる時間が違う(マトリックス)
以下が作るのにかかる時間(マトリックス)と制約条件2ですマトリックス 機械A 機械B 機械C 弁当 27 70 149 ← 機械AかBかCのどれか1つを使う 菓子パン 23 58 122 おにぎり 18 46 97 お菓子 25 36 76 ジュース 14 35 73 制約条件2→ 60 70 80 ←機械Aは合計で60時間まで使える > library(Rglpk) > # 目的関数の係数 > k.obj <- c(27, 23, 18, 25, 14, 70, 58, 46, 36, 35, 149, 122, 97, 76, 73) > # 制約式の左辺の係数 > k.mat <- matrix(c(1,1,1,1,1,0,0,0,0,0,0,0,0,0,0, + 0,0,0,0,0,1,1,1,1,1,0,0,0,0,0, + 0,0,0,0,0,0,0,0,0,0,1,1,1,1,1, + 1,0,0,0,0,1,0,0,0,0,1,0,0,0,0, + 0,1,0,0,0,0,1,0,0,0,0,1,0,0,0, + 0,0,1,0,0,0,0,1,0,0,0,0,1,0,0, + 0,0,0,1,0,0,0,0,1,0,0,0,0,1,0, + 0,0,0,0,1,0,0,0,0,1,0,0,0,0,1), ncol=15, nrow = 8, byrow=TRUE) > # 制約式の等号・不等号 > k.dir <- c("<=", "<=", "<=", "==", "==", "==", "==", "==") > # 制約式の左辺 > k.rhs <- c(60, 70, 80, 1, 1, 1, 1, 1) > Rglpk_solve_LP(k.obj, k.mat, k.dir, k.rhs, max=FALSE)上のコードを走らせると
$optimum [1] 107 $solution [1] 1 1 1 1 1 0 0 0 0 0 0 0 0 0 0 $status [1] 0 $solution_dual [1] 0 0 0 0 0 43 35 28 11 21 122 99 79 51 59 $auxiliary $auxiliary$primal [1] 5 0 0 1 1 1 1 1 $auxiliary$dual [1] 0 0 0 27 23 18 25 14 $sensitivity_report [1] NAと結果が出ますが、これだと全部機械Aで作れと言っていることになり、且つ、制約条件の60を超えてしまっています。すみませんが分かる方教えてください。
実際には食品数が200種類ほどあります。 -- Tomo 2019-08-09 (金) 16:50:10
κ(かっぱ) (2019-08-07 (水) 05:21:53)
こんにちは。> x <- c('織田信長', 'パンダ')という文字列があった時に、その要素と同じ名前のファイルが存在するかチェックしたいです。
> y <- list.files() > y [1] "織田信長" "パンダ"とすると、「織田信長」も「パンダ」も存在するのですが、
> x %in% y [1] TRUE FALSEとなりました。ファイル名から引っ張ってきた「パンダ」 (y[2]) を見てみると、濁音、半濁音が変なことになっています。
> substr(y[2], 1,1) [1] "ハ" > substr(y[2], 2,2) [1] "゚"ハと半濁音の丸が別々の文字となっているようです。濁音も同じです。直接入力した「パンダ」とlist.files() からの「パンダ」と同じと認識させるにはどうしたら良いものでしょうか?
ファイル名に日本語を使うのは好ましくないのですが、実際には人名のファイルが大量に存在しており、それがデータベースと一致しているか調べたいのであります。
x <- c('織田信長', 'パンダ')
x <- iconv(x, "UTF-8", "UTF-8-MAC")
y <- list.files()
x %in% y
> x %in% y
[1] TRUE TRUE
旨くいってもいかなくっても,お返事を。 -- 2019-08-07 (水) 08:33:58SABI (2019-08-06 (火) 09:27:02)
お世話になります。
下記のようにggraphパッケージでネットワーク図を作成しているのですが、右側の凡例a,b,cの〇を大きくするにはどうすればよいでしょうか。
library(ggraph) require(igraph) gr <- graph_from_data_frame(data.frame( from = c(1, 1, 2, 2, 3, 3, 4, 5), to = c(2, 1, 5, 4, 2, 2, 1, 1), class = sample(letters[1:3], 8, TRUE) )) V(gr)$class <- c('a', 'b', 'c', 'a', 'b')
gr %>% ggraph(layout = "auto") + geom_edge_link(colour = "black") + geom_node_point(aes(colour=class, size=10)) + geom_node_label(aes(label=name), repel=T)
内部的に使用しているigraphやggplot2のヘルプファイルも確認し、
theme(legend.key.size=grid::unit(30, units="points"))やscale_size_manual(values=~~)、などで対応できると推測したのですが変化がありません。
環境はigraph_1.2.4.1, ggraph_1.0.2, R version 3.5.0.です。
宜しくお願い致します。
がんば (2019-06-27 (木) 13:04:33)
お世話になります。周辺に誰も経験者がいなかったのでここを始めとしてネットで勉強してRstudioを使って測定結果から信頼度95%で上限下限の線が出せるようになった段階の者です。plotの後でlocator()の実行でマウスによりグラフ上の数値を取り出せるというとこまでは分かったですが特定のX値(又はY値)に対応するグラフ上の点の数値を返す関数はないのでしょうか(上限線、最尤値線、下限線のY値又はX値が返ってくるようなもの)?
使用環境:
R version 3.5.2 (2018-12-20)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows >= 8 x64 (build 9200)
Matrix products: default
locale:
[1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932
[3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C
[5] LC_TIME=Japanese_Japan.932
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] WeibullR_1.0.10
loaded via a namespace (and not attached):
[1] compiler_3.5.2 tools_3.5.2 Rcpp_1.0.0
d = data.frame(x = c(256, 198, 216, 236, 268, 416, 431, 473, 238, 269),
y = c(318, 63, 279, 304, 301, 305, 367, 370, 247, 444))
new = data.frame(x = seq(min(d$x), max(d$x), length=1000))
ans = lm(y ~ x, data=d)
pred.w.plim <- predict(ans, new, interval = "prediction")
pred.w.clim <- predict(ans, new, interval = "confidence")
matplot(new$x, cbind(pred.w.clim, pred.w.plim[,-1]),
lty = c(1,2,2,3,3), col=c(1,2,2,3,3),
type = "l", xlab = "x", ylab = "predicted y")
points(d)
ここで,locator を使って x, y 座標を取得し,その x 座標をデータフレーム new2 にして 予測限界,信頼限界を計算し,表示する
xy = locator(5) new2 = data.frame(x = xy$x) pred.w.plim <- predict(ans, new2, interval = "prediction") pred.w.clim <- predict(ans, new2, interval = "confidence") cbind(new2, pred.w.plim, pred.w.clim[,-1])分からない所は,またオンラインヘルプをひく。 -- 2019-06-27 (木) 13:39:21
初心者 (2019-06-15 (土) 23:51:19)
初心者です
エクセルファイル(単純なXY の表)で作成したマトリックスをRに導入したところ、 エラー: vector memory exhausted (limit reached?) が出ました。自身でも原因をgoogle で調べたところ恐らくexcelファイルが大きすぎることが原因であるかと思いました。一応対処方法を見つけて実践してみたのですが、うまくいきませんでした。理解不足なだけかもしれませんが。googleで調べた解決方法は以下を導入することかと思いました。usethis::edit_r_environ("project")
改善方法や他の解決方法を知っている方がいたら教えてください。
よろしくお願いします。
初心者 (2019-06-07 (金) 08:37:14)
分散分析の棄却域を求める関数qtukey(上側/下側確率, 平均値の数, 群内の自由度)とありますが、この平均値の数というのは具体的に何を指しているのでしょうか?
投稿先が間違えていたので,移動したよ。
あなたが記事を書いた枠のすぐ上に,「回答を期待する記事は原則 質問コーナー Q&A にお願いします。」って書いてあったでしょ。
> qtukey(0.01, 5, 10, lower.tail=FALSE) [1] 6.136093lower.tail というのは,下側確率を求めるよということで,lower.tail=FALSE は上側確率を求めるということだ(デフォルトでは lower.tail=TRUE になっているので,上側確率を求めるときには必ず lower.tail=FALSE を指定しなければならないよ)
初心者 (2019-05-31 (金) 06:53:53)
初心者です。
1)histやplot等で図を複数作り、重ねて表示したいです。この際に作図した後に一旦次のコードを入力しようと画面をクリックすると、開いていたウィンドウが閉じてしまい、重ねて作図することができません。どうしたら良いのでしょうか?
2)また、hist(x)等で図を表示して一度そのウィンドウを閉じると、以後全く同じコードを入力しても図が表示されません。このため再度同じ図を表示するときには、一度ソフトそのものを終了しなくてはなりません。これは正常なのでしょうか?
ハヤト (2019-05-08 (水) 09:01:15)
どうしたらいいのでしょうか?
(2019-05-06 (月) 12:07:33)
psych を使おうとすると,エラーメッセージが出ます。
R 3.6 のくせに,R 3.5 の libgfortran.3.dylib を読もうとして失敗しているようです。> library(psych) エラー: package or namespace load failed for ‘psych’: .onLoad は loadNamespace()('mnormt' に対する)の中で失敗しました、 詳細は: call: dyn.load(file, DLLpath = DLLpath, ...) error: 共有ライブラリ '/Library/Frameworks/R.framework/Versions/3.6/Resources/library/mnormt/libs/mnormt.so' を読み込めません: dlopen(/Library/Frameworks/R.framework/Versions/3.6/Resources/library/mnormt/libs/mnormt.so, 6): Library not loaded: /Library/Frameworks/R.framework/Versions/3.5/Resources/lib/libgfortran.3.dylib Referenced from: /Library/Frameworks/R.framework/Versions/3.6/Resources/library/mnormt/libs/mnormt.so Reason: image not foundpsysh は
Package: psych Version: 1.8.12 Date: 2018-12-31なので,R 3.6 に対応できていないのも仕方ないとは思いますが。
R のバージョン情報は> sessionInfo() R version 3.6.0 Patched (2019-05-02 r76458) Platform: x86_64-apple-darwin15.6.0 (64-bit) Running under: macOS Mojave 10.14.4
てりたま (2019-04-29 (月) 10:05:09)
お世話になります。
パッケージはインストールできますが、宣言すると下記エラーがでます。
ダウンロードされたパッケージは、以下にあります C:\Users\teric\AppData\Local\Temp\RtmpyiJ0Xk\downloaded_packageslibrary (Rcmdr)
要求されたパッケージ splines をロード中です 要求されたパッケージ RcmdrMisc をロード中です 要求されたパッケージ car をロード中です 要求されたパッケージ carData をロード中です 要求されたパッケージ sandwich をロード中です 要求されたパッケージ effects をロード中ですError: package or namespace load failed for ‘effects’ in loadNamespace(j <- i1L, c(lib.loc, .libPaths()), versionCheck = vIj):
‘survey’ という名前のパッケージはありません エラー: パッケージ ‘effects’ をロードできませんでした
あるべきものがないといっているので、メモリ不足?
空き領域190G インテルI5 256メモリ Win10使用です。
解決策を教えていただけますでしょうか。
YT (2019-04-12 (金) 19:01:08)
お世話になっております。
最近Rを使い始めた者です。
データの処理のときにnls関数を用いて、非線形最小二乗法でフィッティングを行っています。
その際正の値であるべきパラメータ(フィッティングして得られるパラメータ、start=で定めるパラメータ)が負の値になってしまいます。
これらのパラメータが負にならないように制限を付けることはできるのでしょうか?
どなたかわかる方がいらっしゃいましたら、教えていただけると幸いです。
宜しくお願い致します。
Sai (2019-04-10 (水) 16:27:33)
お世話になっております。
下記のプログラムで、map_masterというオブジェクトのカテゴリー数(例では、A, B, Cの3つのカテゴリーで、それぞれ3, 2, 1個の要素を持つ)とkを増減させた時に、mapも自動で変わるようなプログラムを作りたいと思っています。
今はカテゴリー数が3つの例でやっていますが、実際はもっといくつもあるので、手作業で入れるのは厳しい状況です。mapと同じ出力を得たいのですが、可能でしょうか。
kの数および、A, B, Cのカテゴリー数を手動で増減(D以降を付け足したり、Bだけ抜いたり)させたときに、mapが自動で変わるようにしたいです。
どなたかわかる方がいらっしゃいましたら、ご教授頂けますと幸いです。
どうぞよろしくお願いいたします。
#各カテゴリーの名前とカテゴリーに含まれる要素数(既知) nA <- c(3, 1, 4) nB <- c(2, 4, 5) nC <- c(1, 3, 5) k <- 1 map_master <- c("A=factor(rep(NA, nA[k]))", "B=factor(rep(NA, nB[k]))", "C=factor(rep(NA, nC[k]))") #カテゴリー数 LM <- length(map_master) #入れ物の用意 map <- NA K <- gamma(LM+1) for(i in 1 : K) { map <- c(map, list(NA)) } #各カテゴリーの組み合わせ(この部分を自動で得たい) map[[1]] <- list() map[[2]] <- list(A=factor(rep(NA, nA[k]))) map[[3]] <- list(B=factor(rep(NA, nB[k]))) map[[4]] <- list(C=factor(rep(NA, nC[k]))) map[[5]] <- list(A=factor(rep(NA, nA[k])), B=factor(rep(NA, nB[k]))) map[[6]] <- list(A=factor(rep(NA, nA[k])), C=factor(rep(NA, nC[k]))) map[[7]] <- list(B=factor(rep(NA, nB[k])), C=factor(rep(NA, nC[k])))
library(e1071)
b <- bincombinations(LM)
n <- nrow(b)
s <- character(n-1)
for (i in 1:(n-1)) {
s[i] <- sprintf("%2d ", sum(b[i,]))
sep <- ""
for (j in 1:LM) {
if (b[i, j] == 1) {
t <- sprintf("%s%s=factor(rep(NA, n%s[k]))", sep, LETTERS[j], LETTERS[j])
sep <- ", "
s[i] <- paste0(s[i], t)
}
}
}
s <- sort(s)
map <- vector("list", n-1)
for (i in 1:(n-1)) {
t <- sprintf("map[[%d]] <- list(%s)", i, substr(s[i], 4, nchar(s[i])))
eval(parse(text=t))
}
実行が終わって,map を表示すると,お望みのものが出来ているはず。 -- 2019-04-10 (水) 18:15:07nhoge <- c(3, 1, 4)
nB <- c(2, 4, 5)
nA1t <- c(1, 3, 5)
map_master <- c("hogehoge=factor(rep(NA, nhoge[k]))",
"B=factor(rep(NA, nB[k]))",
"A1t=factor(rep(NA, nA1t[k]))")
私の例が悪かったせいで、二度手間になってしまい大変申し訳ありません。map_master <- c("hogehoge=factor(rep(NA, nhoge[k]))",
"B=factor(rep(NA, nB[k]))",
"A1t=factor(rep(NA, nA1t[k]))")
があって,以下の 3 行で name1 にカテゴリー名, name2 に要素数が入りますよ。
a <- matrix(sapply(map_master, function(s) unlist(strsplit(s, "[= \\[]"))[c(1,3)]), 2) name1 <- a[1,] name2 <- a[2,]実行結果
> name1 [1] "hogehoge" "B" "A1t" > name2 [1] "nhoge" "nB" "nA1t"あとは,前の LETTERS のかわりに name1,後ろの LETTERS のかわりに name2 を書けばよいだけでしょ。
初心者k (2019-03-18 (月) 21:06:03)
最近Rを始めたものです。
以下のようなデータフレームXの変数名をデータフレームYのCATで置き換えて最終段のようなデータフレームを作成する方法をご教示いただきたいです。
大変初歩的な質問と思いますが、どうぞよろしくお願いいたします。データフレームX A B C D 1 F 153 50 2 F 161 53 3 M 180 74 4 M 169 58 データフレームY Name CAT A ID B SEX C H D W ID SEX H W 1 F 153 50 2 F 161 53 3 M 180 74 4 M 169 58
colnames(x) <- y[, "CAT"]または
colnames(x) <- y[, 2]注意すべきは,[ の後に , が必要ということくらいかな? -- 2019-03-19 (火) 08:48:30
ゆたたみ (2019-03-05 (火) 18:04:16)
お世話になります。
library(FactoMineR)
CA(データ名, graph = FALSE)#ggplot2ベースのバイプロットを描く
library(factoextra)
fviz_ca_biplot(データ名)
上記にてバイプロット図を描くと軸の横に%が表示されます
例(Dim1(26.3%))
この%は何を意味しているのでしょうか?
詳しい方教えていただけますと幸いです。
K.Tanaka (2019-02-26 (火) 20:19:08)
お世話になります。環境はWindows 10Pro 64bit + R version 3.5.2 (+ RStudio)です。> a <- c(1,3,3,4,6,6,7,8,8,8) > (flg <- c()) NULL~ > (flg <- c(rep(0,10))) [1] 0 0 0 0 0 0 0 0 0 0 > for(i in 1:10){ + if(a[i]==a[i+1]){ + flg[i] <- 1 + } + } Error in if (a[i] == a[i + 1]) { : missing value where TRUE/FALSE needed > (flg) [1] 0 1 0 0 1 0 0 1 1 0ベクトルaで、同じ数値が続く最初の部分にフラグ(flg=1)を立てたいと思っています。
しかし、スクリプトを実行すると、上記のエラーが出てしまいます。
また、この例ではきちんとフラグが立っていますが、実際に処理したい3,000行ほどのベクトルでは、ほとんどコードは同じなのですが、すべてのフラグに1が立ってしまいます。
エラーコードでググって見ましたが、 https://code.i-harness.com/ja-jp/q/703b33
http://mako-shark.hatenablog.com/entry/2017/11/14/140146
http://www.okadajp.org/RWiki/?%E5%88%9D%E7%B4%9A%EF%BC%B1%EF%BC%86%EF%BC%A1%20%E3%82%A2%E3%83%BC%E3%82%AB%E3%82%A4%E3%83%96%282%29#u8ec8994
などがヒットしたので見てみましたが、理解できませんでした。
やろうとしていることは非常に単純ですし、上で挙げた例では欠損値の影響も無さそうです。どなたか解決方法をご教示下さいますようお願い致します。
> a <- c(1,2,3,3,3,3,4,4,4,6,6,6,6,7,8,8,8,8,8,9,9,10,11) > b <- numeric(length(a)) > x <- c(0, rle(a)$lengths) > b[(cumsum(x[-length(x)])+1)[x[-1] != 1]] <- 1 > b [1] 0 0 1 0 0 0 1 0 0 1 0 0 0 0 1 0 0 0 0 1 0 0 0
徒弟 (2019-02-25 (月) 10:22:13)
お世話になっております。
3次元座標(x,y,z)にプロットした散布図があります。元のデータもあります。
この点を一本の曲線式で近似したいと考えております。
そのためにスプライン近似が使えるのではないかと考えておりますが、
Rで3次元座標に対するスプライン近似を行う方法が分かりません。
どのように行えば良いかご教示いただければ幸いです
jj (2019-02-01 (金) 07:33:32)
今までpatient at riskを含んだカプランマイヤー曲線を描くのにnpsurvとsurvplotを使用していたのですが、最近になって使用できなくなりました。rmsのパッケージに変更があったのでしょうか?ご存知の方がいらっしゃいましたらご教授願います。
> library(rms) エラー: package or namespace load failed for ‘rms’: オブジェクト 'plotp' を見つけることができませんでした (名前空間 'rms' のロード中に)って出るからね。
Maintainer: Frank E Harrell Jr
<f.harrell@vanderbilt.edu>
に連絡してみれば? -- 2019-02-01 (金) 18:18:43other attached packages: [1] Hmisc_4.2-0 ggplot2_3.1.0 Formula_1.2-3のようにバージョン番号が出力されている部分がある。(単に library(help=Hmisc) でもよいけど) -- 2019-02-27 (水) 11:19:36
r (2019-01-21 (月) 09:24:17)
初心者です。
Ri386 3.4.3をwindows7professionalで使用しています。
A B C D E F G H
1 71.18 3.16 76.90 27.73 55.74 5.27 11.07
1 68.86 9.30 71.44 28.83 60.54 6.20 13.04
1 71.41 2.74 72.77 28.44 56.49 4.28 10.77
1 NA 11.38 NA NA 62.20 4.05 11.07
1 67.13 8.71 74.36 25.11 63.07 2.34 10.45
1 71.64 4.93 75.14 28.89 56.52 5.41 10.80
途中省略
3 71.85 1.61 78.25 28.89 46.76 5.47 7.67
3 73.94 2.63 71.32 29.54 55.34 3.77 8.33
3 73.36 1.31 78.84 NA 55.05 4.94 8.53
3 72.42 3.80 82.98 27.27 52.07 6.25 9.89
3 71.53 2.66 81.99 26.39 55.57 5.76 10.02
途中省略
6 74.56 2.77 74.58 29.18 52.10 8.06 10.59data=read.table("clipboard",header=TRUE)
result3<-lm(data[,1]~data[,2]+data[,3]+data[,4]+data[,5]+data[,6]+data[,7]+data[,8])
result4<-step(result3)
Start: AIC=78.25
data[, 1] ~ data[, 2] + data[, 3] + data[, 4] + data[, 5] + data[,6] + data[, 7] + data[, 8]
Df Sum of Sq RSS AIC
- data[, 7] 1 0.0795 179.80 76.285
- data[, 5] 1 0.1613 179.88 76.326
- data[, 4] 1 1.8827 181.61 77.183
- data[, 2] 1 3.9465 183.67 78.200
none> 179.72 78.245
6] + data[, 8]
Df Sum of Sq RSS AIC
none> 179.80 76.285
- data[, 3] 1 10.2626 190.07 79.281
- data[, 8] 1 11.2881 191.09 79.765
- data[, 6] 1 13.8466 193.65 80.962
Error in step(result3) :
number of rows in use has changed: remove missing values?なぜ、エラーになるのか分かりません。
どうぞご教示下さい。
A B C D E F G H 1 NA 11.38 NA NA 62.20 4.05 11.07 3 73.36 1.31 78.84 NA 55.05 4.94 8.53 ★このデータ 1 71.18 3.16 76.90 27.73 55.74 5.27 11.07 1 68.86 9.30 71.44 28.83 60.54 6.20 13.04 : 以下略まず,重回帰分析に限らないが,NA を含む行は分析には用いられない。
> data2 = na.omit(data) # na.omit で欠損値を含む行をオミットする
> nrow(data2) # 残った行数が 90 になった
[1] 90
> nrow(data) # 元のデータは欠損値を含む 2 行があった
[1] 92
> result <- lm(A ~., data2) # 重回帰モデルの指定は,こんな風にする
> result2 <- step(result) # AIC を見る
Start: AIC=78.25
A ~ B + C + D + E + F + G + H
Df Sum of Sq RSS AIC
- G 1 0.0795 179.80 76.285
- E 1 0.1613 179.88 76.326
- D 1 1.8827 181.61 77.183
- B 1 3.9465 183.67 78.200
<none> 179.72 78.245
- H 1 7.4237 187.15 79.888
- F 1 9.3509 189.07 80.810
- C 1 10.3408 190.06 81.280
Step: AIC=76.28
A ~ B + C + D + E + F + H
Df Sum of Sq RSS AIC
- E 1 0.1715 179.97 74.371
- D 1 2.0560 181.86 75.308
- B 1 3.8690 183.67 76.201
<none> 179.80 76.285
- C 1 10.2626 190.07 79.281
- H 1 11.2881 191.09 79.765
- F 1 13.8466 193.65 80.962
Step: AIC=74.37 この段階で E は使わないということになるが
A ~ B + C + D + F + H 分析例数は変わらない(90行のまま)ので,エラーは起きない
Df Sum of Sq RSS AIC
- D 1 3.0666 183.04 73.891
<none> 179.97 74.371
- B 1 5.3420 185.32 75.003
- C 1 10.0913 190.07 77.281
- H 1 11.3027 191.28 77.853
- F 1 13.7468 193.72 78.995
Step: AIC=73.89
A ~ B + C + F + H
Df Sum of Sq RSS AIC
<none> 183.04 73.891
- B 1 4.5611 187.60 74.107
- H 1 9.6070 192.65 76.495
- F 1 12.0254 195.07 77.618
- C 1 13.0502 196.09 78.090
可能なら,投稿記事を編集したらよい -- 2019-01-21 (月) 13:58:47r (2019-01-11 (金) 10:35:26)
初心者です。グループ1 <-c(0.214868106,0.130905896)
グループ2 <-c(0.363994669,0.133351903)
グループ3 <-c(0.314337464,0.33376496)
グループ4 <-c(0.24814533,0.435896438)
グループ5 <-c(0.460955618,0.348227084)
グループ6 <-c(0.479455052,0.4087283)
全グループ<-c(グループ1, グループ2, グループ3, グループ4,グループ5,グループ6)
薬の種類 <-c(rep("A", 2), rep("B", 2), rep("C", 2), rep("D", 2), rep("E", 2), rep("F", 2))
薬の種類
[1] "A" "A" "B" "B" "C" "C" "D" "D" "E" "E" "F" "F"薬の種類2 <-factor(薬の種類)
薬の種類2
[1] A A B B C C D D E E F FLevels: A B C D E F
oneway.test(全グループ~薬の種類2, var.equal=TRUE)
Error in model.frame.default(formula = 全グループ ~ 薬の種類2) :variable lengths differ (found for '薬の種類2')以上のとおりエラーが出てしまうのはなぜでしょうか。
ご教授お願い致します。
> グループ1 <-c(0.214868106,0.130905896)
> グループ2 <-c(0.363994669,0.133351903)
> グループ3 <-c(0.314337464,0.33376496)
> グループ4 <-c(0.24814533,0.435896438)
> グループ5 <-c(0.460955618,0.348227084)
> グループ6 <-c(0.479455052,0.4087283)
> 全グループ<-c(グループ1, グループ2, グループ3, グループ4, グループ5, グループ6)
> 薬の種類 <-c(rep("A", 2), rep("B", 2), rep("C", 2), rep("D", 2), rep("E", 2), rep("F", 2))
> 薬の種類
[1] "A" "A" "B" "B" "C" "C" "D" "D"
[9] "E" "E" "F" "F"
> 薬の種類2 <-factor(薬の種類)
> 薬の種類2
[1] A A B B C C D D E E F F
Levels: A B C D E F
> oneway.test(全グループ~薬の種類2, var.equal=TRUE)
One-way analysis of means
data: 全グループ and 薬の種類2
F = 2.102, num df = 5, denom df
= 6, p-value = 0.1961
日本語なんて使うと,特に Windows の場合は入力は面倒くさいし,見た目が同じようなフォントのため今回のような発見の難しいバグを発生させたり,ろくな事ないですよ。今回は該当しなかったけど,全角空白があったりすると,七転八倒することもある。(エディタで検索すればどうってことないけど,初心者はドツボに嵌まるだろうか)> x <- c(0.214868106,0.130905896, + 0.363994669,0.133351903, + 0.314337464,0.33376496, + 0.24814533,0.435896438, + 0.460955618,0.348227084, + 0.479455052,0.4087283) > drug <-factor(rep(LETTERS[1:6], each=2)) > drug [1] A A B B C C D D E E F F Levels: A B C D E F > oneway.test(x~drug, var.equal=TRUE) One-way analysis of means data: x and drug F = 2.102, num df = 5, denom df = 6, p-value = 0.1961どれだけ簡単か。 -- 2019-01-11 (金) 18:45:13
tuji (2019-01-04 (金) 20:29:05)
お世話になります。
複数の要素を持つものを比較したときに、他のものそれぞれと比較していずれか要素で大きな値を示す、下位互換ではない(パレート最適である)ものを抽出したいです。
実際は遺伝に関するデータで100*100*100*200行程度、30程度の要素を比較したいと考えています。
例として1クラス40名にトランプを各スート毎に1枚ずつランダムに配って、スート毎に大きいほうが勝つというようなゲームを考え
クラス内の誰に対しても、すべてのスートで負けてしまうことはない生徒をピックアップすることをしました。
下記スクリプトにより問題なく計算はできました。しかし冗長に感じます。
もし有効な関数・パッケージ等ご存知でしたら、教えていただけませんでしょうか。pop <- 40 suit <- 4 mymat1 <- matrix(as.integer(runif(pop * suit,1,14)), pop,suit) #40名にスーツごとに(無限セットの)トランプから1枚を配る mymat2 <- cbind(1:pop, mymat1[order(-mymat1[,1],-mymat1[,2],-mymat1[,3],-mymat1[,4]),]) #最初のスーツの順で並べ替え、各名にIDを振る。スーツが増えた場合スクリプトを書きかえなければいけない compatible <- as.logical(numeric(pop)) #誰の下位互換でもない場合をT、そうでない場合をFとする用の論理値ベクトル myfunc <- function(x, y){ any(x > y) | setequal(x, y) } tm <- mymat2 #並べ替えているため、最初のIDは必ず誰の下位互換でもない for (u in 1:pop){ Limit <- apply(tm[,-1],1,myfunc,tm[1,-1]) #applyで行毎に判定するのとは別に、行が増えたとき用に #tm[,2] > tm[1,2] | tm[,3] > tm[1,3] | tm[,4] > tm[1,4] | tm[,5] > tm[1,5] | apply(tm[,-1],1,setequal,tm[1,-1]) #と書く方がよいでしょうか? compatible[tm[Limit,][1]] <- T #tmの1行目に来るIDの下位互換ではない次のIDは必ず誰の下位互換でもない。 #IDの1列目がcompatibleの要素にアクセスするために必須になっている。 tm <- tm[Limit,][-1,] #tmの1行目と1行目の下位互換のIDを省く print(u) if (length(tm) == suit + 1) { compatible[tm[1]] <- T break } } mymat2[compatible,] #確認用
> pop <- 100*100*100*200 > suit <- 30 > mymat1 <- matrix(as.integer(runif(pop * suit, 1, 14)), pop, suit) エラー: ベクトルのメモリを使い切りました (上限に達した?)
ポンタ (2018-12-27 (木) 22:07:12)
お世話になります。
初めて投稿します。
昨年(2017年)会社でRを入れてもらえるようになり、本業の傍らで、データの分析を始めたものです。
本日、会社のPC(windows7 32bit)でR(3.4です。RStudioは入れてません)を使ってdplyr::filterを動かしていたら、急にエラーが出始めました。(理由が良くわかりません。勝手にRを再インストールできる現場でもありません)
Error in filter_impl(.data, quo) :
Evaluation error: `as_dictionary()` is defunct as of rlang 0.3.0.
ネットで調べていたら、
https://github.com/tidyverse/dplyr/issues/3941
に同じエラーメッセージが出ていましたが、どのように対応したらよいのか良くわかりません。
rlangというのが悪さをしているようなのでこれの1つ前のバージョン(rlang_0.2.2.tar.gz)を入れなおし、dplyrとRcppも入れなおしましたが、エラーのままです。
どのようにすれば、エラーが出なくなるかご教示頂けないでしょうか?
R version 3.5.2 (2018-12-20) -- "Eggshell Igloo"
Copyright (C) 2018 The R Foundation for Statistical Computing
Platform: x86_64-w64-mingw32/x64 (64-bit)
R は、自由なソフトウェアであり、「完全に無保証」です。
一定の条件に従えば、自由にこれを再配布することができます。
配布条件の詳細に関しては、'license()' あるいは 'licence()' と入力してください。
R は多くの貢献者による共同プロジェクトです。
詳しくは 'contributors()' と入力してください。
また、R や R のパッケージを出版物で引用する際の形式については
'citation()' と入力してください。
'demo()' と入力すればデモをみることができます。
'help()' とすればオンラインヘルプが出ます。
'help.start()' で HTML ブラウザによるヘルプがみられます。
'q()' と入力すれば R を終了します。
> utils:::menuInstallPkgs()
--- このセッションで使うために、CRAN のミラーサイトを選んでください ---
URL 'https://cran.ism.ac.jp/bin/windows/contrib/3.5/dplyr_0.7.8.zip' を試しています
Content type 'application/zip' length 3069918 bytes (2.9 MB)
downloaded 2.9 MB
パッケージ ‘dplyr’ は無事に展開され、MD5 サムもチェックされました
ダウンロードされたパッケージは、以下にあります
C:\Users\user\AppData\Local\Temp\RtmpE9GCqc\downloaded_packages
> library(dplyr)
次のパッケージを付け加えます: ‘dplyr’
以下のオブジェクトは ‘package:stats’ からマスクされています:
filter, lag
以下のオブジェクトは ‘package:base’ からマスクされています:
intersect, setdiff, setequal, union
> data.frame(a=10) %>% filter(a>4)
a
1 10
> sessionInfo()
R version 3.5.2 (2018-12-20)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 17134)
Matrix products: default
locale:
[1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932
[3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C
[5] LC_TIME=Japanese_Japan.932
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] bindrcpp_0.2.2 dplyr_0.7.8
loaded via a namespace (and not attached):
[1] tidyselect_0.2.5 compiler_3.5.2 magrittr_1.5 assertthat_0.2.0
[5] R6_2.3.0 tools_3.5.2 pillar_1.3.1 glue_1.3.0
[9] tibble_1.4.2 crayon_1.3.4 Rcpp_1.0.0 pkgconfig_2.0.2
[13] rlang_0.3.0.1 purrr_0.2.5 bindr_0.1.1
> library(dplyr)
> data.frame(a=10) %>% select(a)
a
1 10
>jun (2018-12-16 (日) 18:49:05)
衛星画像の土地被覆分類をSVMを用いて行いたいと考えているのですが、R初心者のため手順が全くわかりません。
簡単で構いませんので手順を教えていただけると幸いです。
画像の赤、青、緑、近赤外の各バンドの画素値の抽出まではしているのでそこからどのような手順を踏めばいいのかわかっていない状況です。
pinoj (2018-12-06 (木) 20:53:46)
ハザード比を出す方法に関する質問をさせて頂きたく思います。
例えば、生存期間を決定する因子として、以下の3つの因子が見つかったとします。
性別、年齢、喫煙(すべて01因子とします)
群別する変数に「性別」を、調整に用いる変数に「年齢と喫煙」を設定した場合、
これらの因子で調整した性別に関する生存曲線は、下記のコマンドで作成できました。
coxmodel <- coxph(Surv(Days,Survival==1)~ age + smoking + strata(sex), data=dat, method="breslow")
cox <- survfit(coxmodel)
plot(cox)
上記で、2本のカーブを描くことができました。
しかしながら、この性別の2群間のハザード比(および95%信頼区間)を出す方法がわかりません。
print(coxmodel)
を入力すると、性別ではなく、その他の2因子(年齢と喫煙)に関するハザード比しか得られません。
恐れ入りますが、どうかご教示をお願い申し上げます。
hisapon (2018-12-01 (土) 16:02:58)
pequodよりlmresを使用して交互作用項を含む3要因の重回帰分析をしておりますが、その交互作用項が連続変数×ダミー変数の場合でも、以下のような式で大丈夫でしょうか。従属変数:dep 独立変数1(連続変数):ind1 独立変数2(連続変数):ind2 独立変数3(ダミー変数):ind3.dummy
lmres(dep~ind1*ind2*ind3.dummy,centered=c("ind1","ind2","ind3.dummy"),data=dat)ダミー変数である独立変数3を中心化させる必要があるのか、また、その後の単純効果分析ではsimpleSlope(・・・,pred="ind1",mod1 = "ind2",mod2="ind3.dummy")で分析を進めれば、-1SDと+1SDで高低群が決定されて効果分析が行われますが、そもそもind3.dummyが0,1の値しかとらないデータとなるとsimpleSlope(・・・,pred="ind1",mod1 = "ind2",mod2="ind3.dummy")で分析しても正確な結果は出てこないのではないかと考えております。
このあたりのことについて教えていただけると有難いです。あるいは、ダミー変数を含む複数要因の重回帰分析でlmres以外の手法でよい方法や手順があればご教示いただきたいです。よろしくお願いいたします。
noria (2018-11-28 (水) 00:49:28)
皆様お世話になります。
欠損値のあるデータの多重代入後の解析、統合方法についてお教え頂けないでしょうか。
現在、多重代入はAmeliaパッケージ、解析はsurv IDINRIパッケージを用いて2つのモデルのNRIとIDIを求めたいと思っています。下記のスクリプトを入力しましたところ、多重代入(n=5)後の5つの結果を算出できましたが、これをどのようにして統合して良いのか分かりません。お忙しいところ恐縮ですがご教授ください。
スクリプト
dat1 <- read.csv("/Users/###/Desktop/dat1.csv")
library(Amelia)
x = data.frame
m = 5
dat1 <- subset(dat1, select=c("time","status","age","copd","nn","dm","bb","ras"))
dat1.out <- amelia(x = dat1, m = 5)
library(survIDINRI)
for(i in 1:m){idinri <- dat1.out$imputations[[i]] D = subset(idinri, select=c("time","status","age","copd","nn","dm","bb","ras")) D$status = as.numeric(D$status==1) D=D[!is.na(apply(D, 1, mean)),]; dim(D) mydata = D t0 = 365 indata1 = mydata; indata0 = mydata[,c(-3)]; n=nrow(D); covs1 <- as.matrix(indata1[,c(-1,-2)]) covs0 <- as.matrix(indata0[,c(-1,-2)]) x <- IDI.INF(mydata[,1:2],covs0,covs1,t0,npert=200)
IDI.INF.OUT(x); }
結果
Est. Lower Upper p-value
M1 0.034 0.000 0.109 0.030
M2 0.203 -0.027 0.402 0.060
M3 0.001 -0.008 0.154 0.607
Est. Lower Upper p-value
M1 0.039 0.002 0.127 0.020
M2 0.203 0.019 0.377 0.030
M3 0.002 -0.010 0.162 0.517Est. Lower Upper p-valueM1 0.041 0.002 0.138 0.020
M2 0.203 -0.018 0.367 0.060
M3 0.001 -0.008 0.139 0.488Est. Lower Upper p-valueM1 0.033 -0.001 0.135 0.090
M2 0.130 -0.203 0.392 0.109
M3 0.002 -0.010 0.210 0.547
Est. Lower Upper p-value
M1 0.031 0.000 0.119 0.070
M2 0.203 0.000 0.368 0.050
M3 0.001 -0.007 0.187 0.498
-0.889 (2018-11-27 (火) 15:44:27)
初心者です。
Ri386 3.4.3をwindows7professionalで使用しています。
1列目を目的変数として2~8列目を説明変数とした重回帰分析を行おうと以下の入力を行ったところ、result1 <-lm(data[,1]~data[,2]+data[,3]+data[,4]+data[,5]+data[,6]+data[,7]+data[,8])
Error in data[, 1] : object of type 'closure' is not subsettableというエラーメッセージが表示されてしまいます。
解決法などご教示頂けると助かります。
-0.889 (2018-11-16 (金) 15:50:38)
初心者です。
Ri386 3.4.3をwindows7professionalで使用しています。
重回帰分析を行いたいため、エクセルのファイルデータをコピー&ペーストするとエラーがでます。
Error in scan(file = file, what = what, sep = sep, quote = quote, dec = dec, :line 59 did not have 5 elements4列までは、ペーストできるのですが、5列以上はエラーとなります。
コピー&ペースト方式には、ペーストできる量に制限があるのでしょうか。また、解決法などご教示いただけけると助かります。
よろしくお願い致します。
(2018-11-11 (日) 12:32:06)
以下の投稿は,「投稿における注意事項」に反する方法で掲載され,投稿フォームを破壊した上,ページの構成を乱しました。
「投稿における注意事項」および「投稿文書の書式」をよく読むよう,注意喚起しておきます。
httrパッケージにおけるPOST関数のURLエンコードについて。あるURLからデータを取得するため、httrパッケージのPOST関数を使用しています。その中で引数bodyに日本語をしているのですがPOST関数はUTF-8でURLエンコードを行っているようです。データ取得先のサーバーに合わせてSHIFT-JISでURLエンコードを行いたいのですが方法はありますでしょうか。
東京都下 (2018-10-15 (月) 17:00:39)
皆様始めまして
過去の業務の統計部分を引き継ぐことになり、いちから勉強しています。
2群の直線判別分析を行っているのですがRがpredict計算で出す判別結果と関連係数を組み合わせ手計算で出す結果の値が異なっていました。
結果を再度見てみると定数項が違うことに気づきました。
lda関数を利用した判別関数の定数項導出でもし留意点などございましたらご教示ください
変数は2つしかなくてたとえばa,bとして
result <- lda(y~a+b)
predict_result <- predict(result)
result$means
result$scalingLD1a ***
b ***
result$means%*%result$scaling
apply(result$means%*%result$scaling,2,mean)
★ここで定数項が出る
とやってみました。
aとb(係数)については問題ないのですが、定数項があいません。
「あわない」というのは同じデータを別のソフトで解析して出した結果と比較してみて分かったものです。predictで出す判別結果とそのソフトで出す結果の数値はぼぼ同じでした。
今扱おうとしているデータ25000件位有り、2つの整数数値列+1カテゴリーのデータです。具体データは出せないのでもうしわけないのですが、いくつかネット上のサンプル(10*2のデータ)で実施したところ問題はありませんでした。
> y = factor(rep(0:1, c(5, 7)))
> a = c(3,2,2,3,4,5,6,4,4,2,3,4)
> b = c(3,2,3,4,5,4,3,7,6,5,4,6)
> library(MASS)
> result <- lda(y~a+b)
> predict_result <- predict(result)
> result$means
a b
0 2.8 3.4
1 4.0 5.0
> result$scaling
LD1
a 0.5955855
b 0.5845561
> result$means%*%result$scaling
LD1
0 3.655130
1 5.305123
> apply(result$means%*%result$scaling,2,mean)
LD1
4.480126> predict_result$x
LD1
1 -1.0772009
2 -2.2573426
3 -1.6727864
4 -0.4926448
5 0.6874968
6 0.6985262
7 0.7095556
8 1.8566091
9 1.2720530
10 -0.5036742
11 -0.4926448
12 1.2720530
> mean(predict_result$x)
[1] -3.515345e-16> pred <- cbind(a, b) %*% result$scaling
> pred
LD1
[1,] 3.540425
[2,] 2.360283
[3,] 2.944839
[4,] 4.124981
[5,] 5.305123
[6,] 5.316152
[7,] 5.327181
[8,] 6.474235
[9,] 5.889679
[10,] 4.113952
[11,] 4.124981
[12,] 5.889679
> offset <- apply(result$means%*%result$scaling,2,mean)
> offset
LD1
4.480126
> pred2 <- pred-offset # offset を調整する!!
> mean(pred2[1:5])
[1] -0.8249962
> mean(pred2[6:12]) # これで,2 群の判別値の平均値の真ん中が 0 になる
[1] 0.8249962nkrit (2018-10-11 (木) 20:48:13)
初心者です。
lst <- list(A=c(1), B=c(2,3), C=c(3,4,5))
のようなリストがありまして、これを
A 1
B 2,3
C 3,4,5
のような2列の表形式に保存したいと考えましたが、
lapply(lst, function(x) write(x, "data", append=T))
これでは、A, B, Cなどの名前が入りませんし、どうしたら簡潔でスマートなのか、思いつかず、困っております(実際は、もっと要素の多いリストなので、手作業でやるのは、かなり骨が折れます)。お忙しいところを恐縮ですが、ご助言いただけましたら、幸いに存じます。
> junk = mapply(function(x, y) {cat(x, "\t", sep=""); cat(y, sep=","); cat("\n")}, names(lst), lst)
A 1
B 2,3
C 3,4,5lst <- list(A=c(1), B=c(2,3), C=c(3,4,5))
con <- file("out.dat", "w")
junk <- mapply(function(x, y) {cat(x, "\t", sep="", file=con, append=TRUE); cat(y, sep=",", file=con, append=TRUE); cat("\n", file=con, append=TRUE)}, names(lst), lst)
close(con)cat(paste(names(lst), lapply(lst, paste, collapse = ","), sep = "\t"), sep = "\n", file = "lst.txt")行名と以降の区切りや、値間の区切りはpasteのsepとcollapseで適宜変えてください。 -- 2018-10-12 (金) 10:29:54
nkrit (2018-10-04 (木) 02:49:41)
欠損値を含むデータフレームの各列から欠損値だけ抜いて、それぞれリストに格納して、次の解析に使いたいのですが、ほとんど手作業のようなコードしか書けずに悩んでいます。
NAのある行をそっくり抜くのではなく,A1なら2要素、A2なら1要素A3なら3要素とも残したいのですが。
どうすればRらしく、すっきりと書けるのでしょうか。何卒よろしくお願いいたします。
df <- data.frame(A1=c("a","b",NA),A2=c("c",NA,NA),A3= c("d","e","f"))
g1 <- df[,"A1"]
g1 <- g1[!is.na(g1)]
g2 <- df[,"A2"]
g2 <- g2[!is.na(g2)]
g3 <- df[,"A3"]
g3 <- g3[!is.na(g3)]
lst <- list(g1,g2,g3)
names(lst) <- c("A1","A2","A3")
うどん (2018-10-03 (水) 23:07:32)
初心者です。
R, Rstudioをmacで使っています。どちらも最新のものにアップデート済です。
no,city,facility_category,facility_name,site,lat,lon,sta,fin
No002,01室蘭,Harbor,崎守埠頭,3,42.365545,140.931244,0,24
No003,05登別,Station,JR登別,9,42.452047,141.180894,9,17
上記のようなcsvファイルを読み込むと、下記のように日本語の部分だけマルチバイト化して表示されてしまいます。
No002 01\216\272\227\226 Harbor \215\350\216\347\225u\223\252 3 42.36554 140.9312 0 24
No003 05\223o\225\312 Station JR\223o\225\312 9 42.45205 141.1809 9 17
一通り調べてみたのですが、エラーも出ず、文字化けでもないようなのでエンコードの問題でもなさそうです。
Rstudio上でfilesから該当ファイルをview fileで読み込むと正常に表示されるのですが、import datasetだとやはりマルチバイトのようになってしまいます。
何か解決策はないでしょうか。よろしくお願いします。
During startup - Warning messages: 1: Setting LC_CTYPE failed, using "C" 2: Setting LC_COLLATE failed, using "C" 3: Setting LC_TIME failed, using "C" 4: Setting LC_MESSAGES failed, using "C" 5: Setting LC_MONETARY failed, using "C"
pkopw2 (2018-09-24 (月) 12:27:12)
library(RMeCab)
res <- RMeCabC("すもももももももものうち")
2行目を入力したとたんに、落ちてしまいます。エラーメッセージもありません。
Rは64/32bitのいずれも試しましたが、同じ現象でした。
どこがにミステイクがあるのでしょうか。どうかよろしくお願いします。
install.packages("RMeCab", repos = "http://rmecab.jp/R") でダウンロードしました。
使用環境:windows10pro(64bit)、R3.4.1 です。
(2018-09-22 (土) 22:17:08)
下記の変換用データフレームと変換前データフレームがあった時、
変換後データフレームを生成するにはどのようなコードを書けばよいでしょうか。
変換用データフレームは値の上限と下限、変換する値の3項目を持ちます。
変換前データフレームの変数vの範囲を判定して、変換用データフレームの値に変換したいです。
なお実際はどのデータフレームも件数が数千件ほどあります。
変換用データフレーム
lower,upper,value
0,1,7
1,2,5
2,3,4
3,4,3
変換前データフレーム
v
1.5
2.5
変換後データフレーム
v,w
1.5,5
2.5,4
変換用 = data.frame(lower = 0:3, upper = 1:4, value = c(7, 5, 4, 3 ))
変換前 = data.frame(v = c(1.5, 2.5, 999, 3.2))
z = lapply(変換前$v, function(x) {
y = which(変換用$lower <= x & x < 変換用$upper)
ifelse(length(y) == 0, NA, 変換用$value[y])
})
変換後 = 変換前
変換後$w = unlist(z)
> 変換後
v w
1 1.5 5
2 2.5 4
3 999.0 NA
4 3.2 3nkrit (2018-09-04 (火) 10:21:20)
merge()の結合様式に関するパラメータを外部から入力することを考えているのですが、
前回のご指摘を踏まえて、本質的な部分だけの質問を心がけて、コンソールからの外からの入力に対応するスクリプト部分は省略します。
ポイントは、たとえば、whichに "L"が入力されたら、論理型で all.x=T, all.y=Fと返してくれて、"R"が入ったら、all.x=F, all.y=Tとなり、該当するローマ字を入れなければ、自動的にall.x=T, all.y=F (left merge) が選ばれるような仕組みを作りたいと思いました。
そこで、which <-"L" # may change to others xyTF <- list(c(T,F),"left") # default here --> all.x=T, all.y=F switch(which, "L" = xyTF <- list(c(T,F),"left"), "R" = xyTF <- list(c(F,T),"right"), "O" = xyTF <- list(c(T,T),"outer"), "I" = xyTF <- list(c(F,F),"inner"), "l" = xyTF <- list(c(T,F),"left"), "r" = xyTF <- list(c(F,T),"right"), "o" = xyTF <- list(c(T,T),"outer"), "i" = xyTF <- list(c(F,F),"inner"), cat("\n When other letters => 'left merge' is automatically selected.\n\n") ) cat("Mode: ", which,"=[" , xyTF[[2]], "]\n") all.x=xyTF[[1]][1] all.y=xyTF[[1]][2] # example: Mdata <- merge(Ldata, Rdata, by.x=Lkey, by.y=Rkey, all.x=xyTF[[1]][1], all.y=xyTF[[1]][2])としてみました。なにぶん独学で、周囲に批判的なコメントをくださる人がいないもので、ふつうは、どうすべきなのかよくわからず、もっと筋の良い(うまい)プログラミングってあるのかと思い、質問させていただきます。なにとぞアドバイスのほど、よろしくお願いいたします。答えは出るのだから、これでも構わないとかでも、ありがたいです。
![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください」もあるよ。
を読んでください」もあるよ。which <- toupper(which) # 小文字で指定されたら大文字にする
all.x <- TRUE; all.y <- FALSE # デフォルトの場合の定義
if (which == "R" || which == "I") all.x = FALSE # デフォルトでない場合の再定義
if (which == "R" || which == "O") all.y = TRUE # デフォルトでない場合の再定義
cat("all.x =", all.x, "all.y =", all.y, "\n")nkrit (2018-08-29 (水) 00:51:11)
ベタに下記のようなスクリプトを書いてみたのですが、とても時間がかかるので、Rのままで、大幅に時間を節約する方法はないかと思いました。
双方のファイルをソートしてから始めると少しは節約になるのかと思いましたが、それでもわかりやすいスクリプトが書けません。アイデアをいただけましたら幸いです。
args <- commandArgs(TRUE)
working.dir <- dirname(args[1])
q.file <- basename(args[1])
p.file <- args[2]
setwd(working.dir)
Qdata <- read.table(q.file, header = T, sep = "\t")# Qdata[,5] = query pvalue dataset (ex.5,000)
Pdata <- read.table(p.file, header = T, sep = "\t")# Pdata[,1] = compared pvalue dataset (ex.10,000)
Qdata$count <- 0
Sys.time()
for (i in 1:nrow(Qdata)){for (j in 1:nrow(Pdata)){ if (Qdata[i,5] >= Pdata[j,1]) Qdata$count[i] <- Qdata$count[i]+1 # when pvalue in the query dataset is larger than pvalue in the compared dataset, one count is added } # ex. nrow(Pdata) = 10,000}
Sys.time()
Qdata$p_value <- Qdata$count/nrow(Pdata)
pvalue_q.file <- paste0("pvalue_", q.file)
write.table(Qdata, pvalue_q.file, quote = F, sep = "\t", row.names = F)
x = c(2, 6)
y = c(3, 4, 1, 0, 9)
count = integer(length(x))
for (i in seq_along(x)) {
sum = 0
for (j in seq_along(y)) {
if (x[i] >= y[j]) {
sum = sum + 1
}
}
count[i] = sum
}
count初心者です (2018-08-11 (土) 08:05:21)
もう一点、こちらも初歩的な質問となり大変恐縮ですが、①に合致するものを②から選択したいです。選択された結果は③のようになります。
①
sample month
1 201701
1 201702
1 201703
1 201704
1 201705
1 201708
1 201709
2 201705
2 201706
2 201709
2 201712
②
sample sex age site month
1 Male 34 a 201701
1 Male 34 b 201701
1 Male 34 a 201702
1 Male 34 b 201702
1 Male 35 a 201703
1 Male 35 a 201704
1 Male 35 a 201705
1 Male 35 a 201706
1 Male 35 b 201708
1 Male 35 a 201709
1 Male 35 b 201709
1 Male 35 b 201711
2 Female 63 c 201701
2 Female 63 c 201705
2 Female 63 d 201705
2 Female 63 c 201706
2 Female 64 c 201707
2 Female 64 c 201709
2 Female 64 d 201709
2 Female 64 c 201710
2 Female 64 d 201712
③
sample sex age site month
1 Male 34 a 201701
1 Male 34 b 201701
1 Male 34 a 201702
1 Male 34 b 201702
1 Male 35 a 201703
1 Male 35 a 201704
1 Male 35 a 201705
1 Male 35 b 201708
1 Male 35 a 201709
1 Male 35 b 201709
2 Female 63 c 201705
2 Female 63 d 201705
2 Female 63 c 201706
2 Female 64 c 201709
2 Female 64 d 201709
2 Female 64 d 201712
merge(データフレーム①,データフレーム②,by.x=”sample”,by.y=”month”)とやってみましたが,column name ‘sample’ is duplicated in the resultというエラーがでます。
大変お手数をおかけしますが、ご教示いただけると幸いです。どうぞよろしくお願いいたします。
初心者です (2018-08-10 (金) 17:20:37)
R初心者です。初歩的な質問で申し訳ございません。
例えば以下のようにサンプルごとに複数の月がデータとしてある場合、重複せずにMonthのデータをピックアップするにはどのように書けばよいでしょうか?
sample Month
1 201701
1 201701
1 201702
1 201703
1 201703
1 201704
1 201704
1 201705
1 201708
1 201708
1 201709
2 201705
2 201706
2 201706
2 201709
2 201709
2 201712
結果は以下のようになります。
sample Month
1 201701
1 201702
1 201703
1 201704
1 201705
1 201708
1 201709
2 201705
2 201706
2 201709
2 201712
大変お手数をおかけしますが、どうぞよろしくお願いします。
むー (2018-08-09 (木) 22:17:57)
変数設定について質問です。英語での入力は問題ないのですが、日本語にするとエラーが出てしまいます。この場合、どの様に設定をすればよろしいでしょうか。因みにMacOSです。
誰か分かる方がおられましたらよろしくお願いします。
R version 3.5.1 ...... -- "Feather Spray" Copyright .... Platform: x86_64-apple-darwin15.6.0 (64-bit) R は、自由なソフトウェアであり、「完全に無保証」です。 一定の条件に従えば、自由にこれを再配布することができます。 配布条件の詳細に関しては、'license()' あるいは 'licence()' と入力してください。以下略
pkopw2 (2018-08-08 (水) 16:30:48)
作図したグラフをメタファイルとしてパワポに貼り付ける方法に関しての質問です。
右クリック>メタファイルにコピー>パワポにコピペ
とすると、グラフが左上に偏ってしまい、右側・下側に無駄な余白が生じてします。その結果、グラフが小さく貼り付けられてしまいます。
(R(3.5.1)、windows10 pro、powerpoint 2016)
この現象は、例えるなら、
par(mfrow=c(2,2))
のコマンドを入力した時に、左上の領域にグラフが描出されるようなイメージです。
前に使っていたパソコン(ver3.2.1、windows8)では、作図したグラフをそのままのサイズで張り付けることができました(そのままというのは、”ビットマップにコピー”として張り付けた時のイメージです)。
結局、パワポにコピペした後にグループ化を解除したりして一応何とかなるのですが、描出されたグラフをそのままのイメージで、メタファイルとして貼り付けたいのです。
作図領域とデバイス領域がそろっていないのかと思い、下記のコマンドを色々と試してみましたが、
fig = c(0, 1, 0, 1)
par(oma = c(0, 0, 0, 0))
plt = c(0, 1, 0, 1)
par(mfrow=c(1,1))
昨日からずっと解決できていません。
初歩的な質問で恐縮ですが、どうかよろしくお願いします。
win.metafile("test.wmf")
hist(rnorm(10000))
dev.off()
ここに示す画像のようになっておらず,大きな余白があるのか(赤枠は画像の範囲を示すためにこちらで追加した) -- 2018-08-09 (木) 16:05:15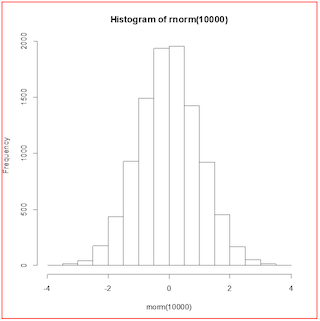
$fig [1] 0 1 0 1 $mai [1] 1.02 0.82 0.82 0.42 $mar [1] 5.1 4.1 4.1 2.1 $mfg [1] 1 1 1 1 $oma [1] 0 0 0 0 $omd [1] 0 1 0 1 $omi [1] 0 0 0 0 $plt [1] 0.1205513 0.9382542 0.1499541 0.8794487 $usr [1] 0 1 0 1それ以上は,わからない。 -- 2018-08-11 (土) 14:17:58
sk (2018-08-04 (土) 08:03:33)
複数のデータフレームを同じ操作で加工したいのですが、うまくできません。
ベクターでも一緒なので、やりたいことは下記のような形です。
a <- c(1,2)
b <- c(3,4)
a <- append(a, 9)
b <- append(b, 9)
これを
for (i in list(a,b) ){i <- append(i, 9) View(i)}
のように書くと、Viewでは見えるのですが、
それぞれのデータフレーム(ベクター)は変化していません。
lapply(dlist, function (x) {x <- append(x, 9); View(x)})
も考えたのですがこれも違いました。
for (s in c("a","b") ){eval(parse(text = paste0(s," <- append(", s, ", 9)"))) eval(parse(text = paste0("View(", s, ")")))}
これはできるのですが、操作が多いので、すべてを文字列で書くのが面倒ですし視認性も悪いです。本当はどうするのがよいのでしょうか。
func = function(d) {
d$x2 = d$x^2
d$y2 = 1/d$y
d
}
> set.seed(987)
> a = data.frame(x=1:5, y=rnorm(5))
> a
x y
1 1 -0.05701369
2 2 0.26999481
3 3 0.87447166
4 4 -0.19034659
5 5 1.10069779
> a = func(a)
> a
x y x2 y2
1 1 -0.05701369 1 -17.5396479
2 2 0.26999481 4 3.7037749
3 3 0.87447166 9 1.1435476
4 4 -0.19034659 16 -5.2535746
5 5 1.10069779 25 0.9085146
> b = data.frame(x=1:10, y=rnorm(10))
> b
x y
1 1 -0.893758950
2 2 -0.954967917
3 3 -0.855857687
4 4 -1.113020051
5 5 -1.288726486
6 6 0.757694061
7 7 -0.876224265
8 8 1.758459078
9 9 -0.009684665
10 10 -0.448383447
> b = func(b)
> b
x y x2 y2
1 1 -0.893758950 1 -1.1188699
2 2 -0.954967917 4 -1.0471556
3 3 -0.855857687 9 -1.1684186
4 4 -1.113020051 16 -0.8984564
5 5 -1.288726486 25 -0.7759598
6 6 0.757694061 36 1.3197939
7 7 -0.876224265 49 -1.1412603
8 8 1.758459078 64 0.5686797
9 9 -0.009684665 81 -103.2560250
10 10 -0.448383447 100 -2.2302340
> c = data.frame(x=c(2,4,8), y= c(0.2, 0.34, 0.87))
> c
x y
1 2 0.20
2 4 0.34
3 8 0.87
> result = lapply(list(a, b, c), func) # lapply でまとめて実行
> result
[[1]]
x y x2 y2
1 1 -0.05701369 1 -17.5396479
2 2 0.26999481 4 3.7037749
3 3 0.87447166 9 1.1435476
4 4 -0.19034659 16 -5.2535746
5 5 1.10069779 25 0.9085146
[[2]]
x y x2 y2
1 1 -0.893758950 1 -1.1188699
2 2 -0.954967917 4 -1.0471556
3 3 -0.855857687 9 -1.1684186
4 4 -1.113020051 16 -0.8984564
5 5 -1.288726486 25 -0.7759598
6 6 0.757694061 36 1.3197939
7 7 -0.876224265 49 -1.1412603
8 8 1.758459078 64 0.5686797
9 9 -0.009684665 81 -103.2560250
10 10 -0.448383447 100 -2.2302340
[[3]]
x y x2 y2
1 2 0.20 4 5.000000
2 4 0.34 16 2.941176
3 8 0.87 64 1.149425
> result_c = result[[3]]
> result_c
x y x2 y2
1 2 0.20 4 5.000000
2 4 0.34 16 2.941176
3 8 0.87 64 1.149425a <- c(1,2) b <- c(3,4)は代入だけど,こんなのを 100回書くわけはないが,ファイルからの入力にしても,たとえば
a <- read.table("file-a.dat")
なんてことになり,結局100回書かなくてはならないことに違いない。変更も 100回書かなきゃならないし,利用も同じ。変更が複雑なら,その複雑さを100回そのまま書くよりは,複雑な処理の記述を1回だけ関数で書いて,せめて100回書くときは文字数の少ないfunc(a)としようというのが先の回答。for (i in 1:100) {
f = sprintf("x%03i.dat", i)
x = read.table(f)
x = fnc(x)
write.table(x, f) # 変更を書き出す必要があれば
}
とすればよいだけ。file.name = c("foo.dat", "bar.dat", "nanntakakantaranagainamae.dat", "jugemujugemu.dat", ....) 全部で100個の定義
for (f in file.name) {
x = read.table(f)
x = fnc(x) # ここに処理内容を書けば,処理内容は1回だけ書けばよいので,関数にしたものを呼ぶ必要はないけど,読みやすいように書く。
write.table(x, f) # 変更を書き出す必要があれば
}
要するに,「繰り返しはループでする」,「ループで処理できるようにデータ構造およびデータ記述法を考える」ということ。 -- 2018-08-05 (日) 06:05:43sh (2018-07-26 (木) 17:33:16)
用語があっているか自信がないのですが・・・
Database(postgreSQL)接続してテーブルを参照し、そのカラムの1つを(重複削除して)リスト形式にすることで、selectizeInputの選択肢にしています。
■読み込んだテーブル
|ID|要素|
|1|赤|
|1|黄|
|1|青|
|2|赤|
|2|青|
|3|青|
|3|緑|
|4|赤|
|4|緑|
■selectizeInputの選択肢:テーブルの<要素>カラムをunique化したもの
[赤,黄,青,緑]
この選択肢に対して、「選択内容をすべて含むID」を表示したい場合はどうすれば良いのでしょうか。
或いは絞り込むために、表をもっと加工したほうが良いのでしょうか。。
※例
選択:[赤,青] ⇒ 表示ID:1,2
選択:[緑] ⇒ 表示ID:3,4
> data = data.frame(ID=c(1,1,1,2,2,3,3,4,4),
+ 要素=c("赤","黄","青","赤","青","青","緑","赤","緑"))
> f = function(選択) {
+ tbl = xtabs(~ID+要素, data)[, 選択, drop = FALSE]
+ as.integer(rownames(tbl)[rowSums(tbl)==length(選択)])
+ }
> f(c("赤", "青"))
[1] 1 2
> f("緑")
[1] 3 4nkrit (2018-07-20 (金) 22:01:39)
一般化混合線形モデルのパッケージglmmMLで、
result <- glmmML(status ~ x3, data = x, family = binomial, cluster = Columns)
のような形で結果を取得して、係数の推定値と係数の標準偏差は、
result$coefficients[2]
result$coef.sd[2]
のように取得できましたが、
z値やそのp値は、どのように取得すれば良いのでしょうか。
result自体は、coef se(coef) z Pr(>|z|)(Intercept) -0.1233 0.845 -0.1459 0.884
x3 14.4545 79.892 0.1809 0.856
のような形で表示されてくるので、取得の方法があるのではと思いました。
推定値/標準偏差を後で計算して、2*pnorm()によってP値を取得することもできるかと思いましたが、せっかくもともと表示されているのでそのまま取得したいと思いました。やり方をお教え頂けましたら、大変ありがたく思います。
どうぞ宜しくお願い致します。
念のためresultのstructureを添付させていただきます。str(result)
List of 18$ boot : num 0 $ converged : logi TRUE $ coefficients : Named num [1:2] -0.123 14.455 ..- attr(*, "names")= chr [1:2] "(Intercept)" "x[[3]]" $ coef.sd : num [1:2] 0.845 79.892 $ sigma : num 0.0194 $ sigma.sd : num 31.6 $ variance : num [1:3, 1:3] 7.14e-01 6.54e-02 5.89e-03 6.54e-02 6.38e+03 ... $ aic : num 28.1 $ bootP : logi NA $ deviance : num 22.1 $ df.residual : num 13 $ cluster.null.deviance: num 22.1 $ cluster.null.df : int 14 $ posterior.modes : num [1:16] -0.00018 -0.000178 -0.000178 -0.000203 -0.000182 ... $ prior : chr "gaussian" $ terms :Classes 'terms', 'formula' language status ~ x[[3]] .. ..- attr(*, "variables")= language list(status, x[[3]]) .. ..- attr(*, "factors")= int [1:2, 1] 0 1 .. .. ..- attr(*, "dimnames")=List of 2 .. .. .. ..$ : chr [1:2] "status" "x[[3]]" .. .. .. ..$ : chr "x[[3]]" .. ..- attr(*, "term.labels")= chr "x[[3]]" .. ..- attr(*, "order")= int 1 .. ..- attr(*, "intercept")= int 1 .. ..- attr(*, "response")= int 1 .. ..- attr(*, ".Environment")=<environment: R_GlobalEnv> .. ..- attr(*, "predvars")= language list(status, x[[3]]) .. ..- attr(*, "dataClasses")= Named chr [1:3] "numeric" "numeric" "factor" .. .. ..- attr(*, "names")= chr [1:3] "status" "x[[3]]" "(cluster)" $ info : int 0 $ call : language glmmML(formula = status ~ x[[3]], family = binomial, data = x, cluster = Columns) - attr(*, "class")= chr "glmmML"
sh (2018-07-18 (水) 14:57:23)
テーブルのなかで「指定の文字を含むレコードを除く」のやり方が分からず、
(1)dbGetQueryで得られたテーブル1
(2)テーブル1をfilter,greplで絞り込んだテーブル2(部分一致用)
に対し、テーブル1とテーブル2の差集合をとる、
という手法で実現しようとしていますがやり方がわかりません…
setdiffでは目的の挙動をしてくれないのですが、
関数のチョイスが間違っているのでしょうか。
また、「指定の文字を含むレコードを除く」を一発で実現する方法があるのでしょうか。
ご教授いただければ幸いです。
> a = data.frame(x = c("abc", "fab", "xyz", "cabs", "xw"), y = c(2, 1, 4, 3, 5))
> a[!grepl("ab", a$x),]
x y
3 xyz 4
5 xw 5ワイン (2018-07-18 (水) 06:19:51)
お世話になります。poisson.test()で検定をするためヘルプファイルを確認したところ、以下のような引数になっていました。
poisson.test(x, T = 1, r = 1,alternative = c("two.sided", "less", "greater"), conf.level = 0.95)
x number of events. A vector of length one or two. T time base for event count. A vector of length one or two. r hypothesized rate or rate ratio
イベント発生数x=5の場合の検定結果は以下のとおりです。
> poisson.test(x=5) Exact Poisson test data: 5 time base: 1 number of events = 5, time base = 1, p-value = 0.00366 alternative hypothesis: true event rate is not equal to 1 95 percent confidence interval: 1.623486 11.668332 sample estimates: event rate 5
質問1:引数rやTの意味が分からないのですが、たとえばx=5,r=2とした場合、真のイベント発生数は10であるという帰無仮説で検定をするのでしょうか
質問2:ポアソン分布で検定をするならば、平均値λを定義しないと分布の形状が決定できないと思うのですが、引数で指定しなのはなぜでしょうか。
お手数ですが、ご回答宜しくお願い致します。
nkrit (2018-07-08 (日) 16:07:20)
> class(data) [1] "numeric" > cat(data) 2 2 4 6 4 5 2 3 1 2 0 4このような数値型のベクトルを、あとで他人が同じデータで試せるように、たとえば、
data <- c(2,2,4,6,4,5,2,3,1,2,0,4)のような形でスクリプトの一部として保存しておきたいのですが、それぞれの数値の要素の後ろにカンマをつける良い方法が思いつきません。
for (i in 1:50){ cat(paste0(data[i],sep=",")) }のようなことをして出力されたものをペーストしてみましたが、きっともっとよい方法があるのではないかと思いまして投稿しました。簡単な方法があれば教えてください。save(), load()で変数自体を保存するのではなく、ベタに保存しておきたいという希望です。初級者コースと聞いて質問致します。
nona (2018-07-08 (日) 14:44:41)
Rでプロット領域を分割して、かつフォントサイズをpsで指定してプロットさせると、
分割領域が2つまでは指定した通りのフォントサイズになるようなのですが、それ以上になるとフォントが小さくなって困っています。
領域が狭いのが原因と思い、マウスでウインドウを広げた後に再度プロットしたり、par(mar=)で余白を広げてもダメでした。
どなたか、複数のグラフを1枚にプロットするときに、フォントサイズが指定通りの大きさになる方法をお教えください。
以下、試した設定です。
- フォントサイズの指定がうまくいく
jpeg("myplot.jpg", height=1000, width=1000)
par(mfrow=c(1,2), ps=16)
plot(iris[,1],iris[,2])
plot(iris[,3],iris[,2])
dev.off()- フォントサイズを指定しても、上と比べて字が小さくなる
jpeg("myplot2.jpg", height=1000, width=1000)
par(mfrow=c(1,3), ps=16)
plot(iris[,1],iris[,2])
plot(iris[,1],iris[,2])
plot(iris[,1],iris[,2])
dev.off()- 軸周辺の余白を広げても変化なし
jpeg("myplot3.jpg", height=1000, width=1000)
par(ps = 16, mfrow=c(1,3), mar = c(6, 6, 4, 2))
plot(iris[,1],iris[,2])
plot(iris[,1],iris[,2])
plot(iris[,1],iris[,2])
dev.off()- プロット自体を小さくしても変化なし
jpeg("myplot4.jpg", height=1000, width=1000)
par(ps = 14, mfrow=c(1,3), pin = c(1,2))
plot(iris[,1],iris[,2])
plot(iris[,1],iris[,2])
plot(iris[,1],iris[,2])
dev.off()
kx (2018-07-08 (日) 12:52:20)
valores <- c(1,-1,0,-2,-3,-4,-2,0,1,1,4,0,1,-2,-3,0,4,2,-1,0,-2,0,3,2,2,4,0,-2,0,2,1,-1,2,3,2,1)
supplier <- factor(rep(1:3, each=12))
batches <- factor(rep(rep(1:4, each=3), 3))
のように入力し、
purity <- data.frame(valores,supplier,batches)
としてデータフレームを行い、
このpurityをsummaryでしたいのですが、この時にerrorの要素も入れるためには、
purity <- data.frame(valores,supplier,batches)だけではダメで、どのような操作が必要か分からずに困っています。
どなたかお教えくださいませんでしょうか。
(2018-06-25 (月) 23:56:24)
c(1,2,2,3,4,2,2,1,1,1)
のようなベクトルがあった時に
下記のように連続する値をもつ要素を削除する方法はありますでしょうか?
c(1,2,3,4,2,1)
実際のベクトルは非常に長いのでループ処理は避けたいです。
> x = c(1,2,2,3,4,2,2,1,1,1) > rle(x)$values [1] 1 2 3 4 2 1
ぐれいぴ (2018-06-22 (金) 09:29:57)
いろいろ調べたのですが、分からなかったのでお教え願います。
以下のようなデータがあった時に、要因(A、B、・・・)ごとの数を数えて集計したいと思いました。a
factor rate1 A a
2 A a
3 A a
4 A a
5 A a
6 A b
7 A b
8 A b
9 A c
10 A c
11 B a
12 B a
13 B b
14 B b
15 B b
16 B b
17 B b
18 B c
19 B c
20 B d
21 C a
22 C b
23 C b
24 C b
25 C c
26 C c
27 C c
28 C c
29 C d
30 C d
手入力でAについてaは5個、bは3個、・・・と求めることはできるのですが、このデータを読み込ませたときに自動的(A、B、Cとは全く指定しないで)に、
a b c dA 5 3 2 0
B 2 5 2 1
C 1 3 4 2
のように出力させることは可能でしょうか? よろしくお願いします。
> a <- data.frame(
+ factor = rep(LETTERS[1:3], each = 10),
+ rate = unlist(lapply(list(c(5, 3, 2, 0), c(2, 5, 2, 1), c(1, 3, 4, 2)), function (z) { rep(letters[1:4], z) })))
> table(a)
rate
factor a b c d
A 5 3 2 0
B 2 5 2 1
C 1 3 4 2jj (2018-06-08 (金) 01:40:16)
いつも勉強させていただいております。
一年死亡と血圧の関係をrestricted cubic splineを用いて
coxph(Surv(死亡までの時間,死亡イベントありなし) ~ rcs(PP,変曲点), data=dat)として表しました。
rcs関数だと変曲点の個数は最小3個までなのですが、変曲点1つ、2つの場合はどのように処理すればよいでしょうか?他の関数がありますでしょうか?
宜しくお願い致します。
さくらんぼ (2018-06-06 (水) 17:02:13)
宜しくお願い致します。
Mclustで繰り返し計算した際の再現性を得たいと考えています。
RdocumentationのMclustの項目に、
initialization
A list containing zero or more of the following components:
subset:
A logical or numeric vector specifying a subset of the data to be used in the initial hierarchical clustering phase. By default no subset is used unless the number of observations exceeds the value specified by mclust.options("subset"). Note that to guarantee exact reproducibility of results a seed must be specified (see set.seed).
とあることは分かったのですが、「再現性を得るためにはシードを設定する」とは具体的にどのようなことでしょうか?
ジョージ (2018-06-05 (火) 17:20:05)
最近Rを使用し始めたばかりの初心者です。
よろしくお願いいたします。
多重比較検定を行う必要があり、NSM3 というパッケージの pSDCFglig というコマンドを用いて Steel-Dwass 検定を行おうと考えています。そこで簡単な例を作ってテストしてみました。
すると、計算が異常に重く、Rが応答しないようになりました。
この原因がわからず困っています。解決法のご教授をお願いいたします。
- - - - - - - -
まずは,上の方にも書いてあるように,「投稿における注意事項」を読んで,それに従ってください。
これに従わない場合には,回答が得られにくい場合があります。
> system.time(pSDCFlig(dat,grp,method="Exact"))
ユーザ システム 経過
31.463 0.056 31.523シロツメクサ (2018-05-30 (水) 15:30:05)
よろしくお願いいたします。
Rの最新バージョンであるR3.5.0にbiOpsをインストールしたいです。
色々調べましたが、どの情報が新しいのかもよくわかりません。
どのページから行えばいいのかなど詳しい方法を教えていただけたらありがたいです。
よろしくお願いいたします。
ムーミン (2018-05-26 (土) 17:15:31)
よろしくお願いします。
文字列 "1月",~"12月"から月を除いた文字列を作りたく下記のプログラム書きました。
結果は 1月",~"9月","10","11","12"となりました。
どこが間違っているのかわかりません。
どなたか教えてください。
hkan1 <- C("1月",~"12月")
k <- 0
for (r in hkan1) {k <- k + 1 if ( k < 9 ) t1 <- substring(hkan1, 1, 1)
else t1 <- substring(hkan1, 1, 2)}
河童の屁は,河童にあらず,屁である。 (2018-05-25 (金) 21:41:41)
もうね
今後
こんな
礼儀知らずの
質問者には
回答しないことに
します
シント (2018-05-22 (火) 13:16:55)
複数カラムを持つデータセットを、そのうち1つのカラムデータを用いて絞り込みをしたいです。column01 column02 column03 column04 x1 y1 z1 a1 x2 y2 z2 a2 x3 y3 z3 a3 x3 y3 z2 a4 x1 y4 z4 a1 ...というデータセットに対して column01 に着目し、
[x1] x2 [x3] [x4]の選択ボックス的なものを設けて、対象としたいものをクリックして複数条件(or条件)で絞り込むというイメージです。
DT パッケージの Row 選択に近いのですが、あれは data frame や matrix でしか出力できないと読みました。
チェックボックスやラジオボタンで実現するしか無いのでしょうか。。
ご教授頂ければ幸いです。どうぞよろしくお願いします。
d[column01 == "x1" | column01 == "x3",] # ] の前のカンマを忘れないように!または
subset(d, column01 == "x1" | column01 == "x3")とするので十分でしょう。
イルカ (2018-05-11 (金) 14:12:07)
train.csvとtest.csvはそれぞれ一列目に画像の判定、二列目以降は30*30の画像データを900列(gray scale)で記したものになっています。
DNNでのrbayesianoptimizationパッケージはそこそこ資料もあるので何とかなるのですが、CNNにするとデータの構造が問題になるようで、どう解決すればいいのかわかりません。
もし、分かる方がいらっしゃいましたら教えてくださると助かります。
以下にコードとエラー、traceback結果を載せています。
長くて申し訳ありません。> options(max.print=2147483647) > library(mxnet) > train <- read.csv('C:/Users/User/R/train/getimage_1/train.csv', header = TRUE) > test <- read.csv('C:/Users/User/R/train/getimage_1/test.csv', header = TRUE) > train <- data.matrix(train) > test <- data.matrix(test) > train.x <- t(train[, -1]) > train.y <- train[, 1] > test.x <- t(test[, -1]) > test.y <- test[, 1] > dim <- 30 > channels <- 1 > test_array <- test.x > dim(test_array) <- c(dim, dim, channels, ncol(test.x)) > train_array <- train.x > dim(train_array) <- c(dim, dim, channels, ncol(train.x)) > > mxnet_bayes <- function(unit1, unit2, unit3, unit4) { + # Model + data <- mx.symbol.Variable('data') + # 1st convolutional layer 5x5 kernel + conv_1 <- mx.symbol.Convolution(data= data, kernel = c(5, 5), num_filter = unit1) + tanh_1 <- mx.symbol.Activation(data= conv_1, act_type = "relu") + pool_1 <- mx.symbol.Pooling(data = tanh_1, pool_type = "max", kernel = c(2, 2), stride = c(2, 2)) + # 2nd convolutional layer 5x5 kernel + conv_2 <- mx.symbol.Convolution(data = pool_1, kernel = c(5, 5), num_filter = unit2) + tanh_2 <- mx.symbol.Activation(data = conv_2, act_type = "relu") + pool_2 <- mx.symbol.Pooling(data = tanh_2, pool_type = "max", kernel = c(2, 2), stride = c(2, 2)) + # 1st fully connected layer + + fcl_1 <- mx.symbol.FullyConnected(data = pool_2, num_hidden = unit3) + tanh_3 <- mx.symbol.Activation(data = fcl_1, act_type = "tanh") + # 2nd fully connected layer + fcl_2 <- mx.symbol.FullyConnected(data = tanh_3, num_hidden = unit4) + # Output + model <- mx.symbol.SoftmaxOutput(data = fcl_2) + # Set seed for reproducibility + mx.set.seed(5000) + # Device used. Sadly not the GPU :-( + device <- mx.cpu() + model <- mx.model.FeedForward.create(model, X = train_array, y = train.y, + ctx = device, + num.round = 20, + array.batch.size = 100, + learning.rate = 0.07, + momentum = 0.99, + wd = 0.0000001, + eval.metric = mx.metric.rmse, + epoch.end.callback = mx.callback.log.train.metric(100), verbose=TRUE) + preds <- predict(model, test.x) + holdout_score <- rmse(preds, test.y) + list(Score=-holdout_score, Pred=-holdout_score) + } > library(Metrics) > library(rBayesianOptimization) > opt_res <- BayesianOptimization(mxnet_bayes, + bounds=list(unit1=c(200L, 300L), + unit2=c(10L, 50L), + unit3=c(100L, 150L), + unit4=c(80L, 150L)), + init_points=20, n_iter=1, acq='ei', + kappa=2.576, eps=0.0, verbose=TRUE) Start training with 1 devices [1] Train-rmse=0.694788357881959 [2] Train-rmse=0.698895939954201 [3] Train-rmse=0.702352686365657 [4] Train-rmse=0.703651129075772 [5] Train-rmse=0.704188237229376 [6] Train-rmse=0.704072243436174 [7] Train-rmse=0.704307457989986 [8] Train-rmse=0.704044050930675 [9] Train-rmse=0.704189340228431 [10] Train-rmse=0.704024917141773 [11] Train-rmse=0.704088730139514 [12] Train-rmse=0.704510854642202 [13] Train-rmse=0.704353378706923 [14] Train-rmse=0.704272895208268 [15] Train-rmse=0.704260412182352 [16] Train-rmse=0.704688375203385 [17] Train-rmse=0.704331001542547 [18] Train-rmse=0.704090463144038 [19] Train-rmse=0.704570122472829 [20] Train-rmse=0.704810586026211 symbol$infer.shape(list(...)) でエラー: Error in operator convolution10: [08:26:02] d:\program files (x86)\jenkins\workspace\mxnet\mxnet\src\operator\./convolution-inl.h:377: Check failed: dshp.ndim() == 4U (2 vs. 4) Input data should be 4D in batch-num_filter-y-x Timing stopped at: 721.7 39.89 440.3 > traceback() 16: stop(list(message = "Error in operator convolution10: [08:26:02] d:\\program files (x86)\\jenkins\\workspace\\mxnet\\mxnet\\src\\operator\\./convolution-inl.h:377: Check failed: dshp.ndim() == 4U (2 vs. 4) Input data should be 4D in batch-num_filter-y-x", call = symbol$infer.shape(list(...)), cppstack = list(file = "", line = -1L, stack = "C++ stack not available on this system"))) 15: .External(list(name = "CppMethod__invoke_notvoid", address = <pointer: 0x000000000858fda0>, dll = list(name = "Rcpp", path = "C:/Users/User/Documents/R/win-library/3.4/Rcpp/libs/x64/Rcpp.dll", dynamicLookup = TRUE, handle = <pointer: 0x000000006abc0000>, info = <pointer: 0x000000000652a390>), numParameters = -1L), <pointer: 0x0000000006522b90>, <pointer: 0x0000000008791920>, .pointer, ...) 14: symbol$infer.shape(list(...)) 13: mx.symbol.infer.shape(model$symbol, data = c(dimX[[2]], 1)) 12: mx.model.select.layout.predict(X, model) 11: predict.MXFeedForwardModel(model, test.x) 10: predict(model, test.x) at #34 9: (function (unit1, unit2, unit3, unit4) { data <- mx.symbol.Variable("data") conv_1 <- mx.symbol.Convolution(data = data, kernel = c(5, 5), num_filter = unit1) tanh_1 <- mx.symbol.Activation(data = conv_1, act_type = "relu") pool_1 <- mx.symbol.Pooling(data = tanh_1, pool_type = "max", kernel = c(2, 2), stride = c(2, 2)) conv_2 <- mx.symbol.Convolution(data = pool_1, kernel = c(5, 5), num_filter = unit2) tanh_2 <- mx.symbol.Activation(data = conv_2, act_type = "relu") pool_2 <- mx.symbol.Pooling(data = tanh_2, pool_type = "max", kernel = c(2, 2), stride = c(2, 2)) fcl_1 <- mx.symbol.FullyConnected(data = pool_2, num_hidden = unit3) tanh_3 <- mx.symbol.Activation(data = fcl_1, act_type = "tanh") fcl_2 <- mx.symbol.FullyConnected(data = tanh_3, num_hidden = unit4) model <- mx.symbol.SoftmaxOutput(data = fcl_2) mx.set.seed(5000) device <- mx.cpu() model <- mx.model.FeedForward.create(model, X = train_array, y = train.y, ctx = device, num.round = 20, array.batch.size = 100, learning.rate = 0.07, momentum = 0.99, wd = 1e-07, eval.metric = mx.metric.rmse, epoch.end.callback = mx.callback.log.train.metric(100), verbose = TRUE) preds <- predict(model, test.x) holdout_score <- rmse(preds, test.y) list(Score = -holdout_score, Pred = -holdout_score) })(unit1 = 296, unit2 = 37, unit3 = 118, unit4 = 95) 8: do.call(what = FUN, args = as.list(This_Par)) 7: system.time({ This_Score_Pred <- do.call(what = FUN, args = as.list(This_Par)) }) 6: eval(expr, pf) 5: eval(expr, pf) 4: withVisible(eval(expr, pf)) 3: evalVis(expr) 2: utils::capture.output({ This_Time <- system.time({ This_Score_Pred <- do.call(what = FUN, args = as.list(This_Par)) }) }) 1: BayesianOptimization(mxnet_bayes, bounds = list(unit1 = c(200L, 300L), unit2 = c(10L, 50L), unit3 = c(100L, 150L), unit4 = c(80L, 150L)), init_points = 20, n_iter = 1, acq = "ei", kappa = 2.576, eps = 0, verbose = TRUE)
代理転載人 (2018-05-10 (木) 13:02:56)
[下記はトップページへのコメントに間違って記入された書き込み(2018-05-08 (火) 20:45:31)の転載です]
これからRを使い始めようかと思っている程度の初心者です。RStudioを使いたいのですが私の持っている教科書は初心者向けのはずなのに、あまり親切ではなくインストールがわかりません。私の理解では初めに日本語のRを入れておけばRStudioでは言語を選ぶ必要はないように思うのですが間違っていますか?
ムーミン (2018-05-08 (火) 18:44:48)
よろしくお願いします
下記プログラムをRコマンダーで実行するとエラー: <text>となります、どうしてなのかわかりません教えていただけませんか?
ar <- c(1,3,5,9,11,17,8,7,21,13) for (x in ar) { if (x %% 2 == 0) next cat(x," ") }
> ar <- c(1,3,5,9,11,17,8,7,21,13)
> for (x in ar) {
+ if (x %% 2 == 0) next
+ cat(x," ")
+ }
1 3 5 9 11 17 7 21 13
この通りの結果が出たとすれば,R コマンダーの使い方がまずいんでしょうね。ar <- c(1,3,5,9,11,17,8,7,21,13)
for (x in ar) {
if (x %% 2 == 0) next
cat(x," ")
}
って入れて,「実行」ボタンをクリックしたら,出力欄(下の欄)に,
> ar <- c(1,3,5,9,11,17,8,7,21,13)
> for (x in ar) {
+ if (x %% 2 == 0) next
+ cat(x," ")
+ }
1 3 5 9 11 17 7 21 13 NULL
って表示されましたけど??(2018-05-02 (水) 16:08:07)
3次元マトリックスmat[100,100,10]は
100×100の行列で各セル毎に10個の属性値を保有しています。
属性値が最も小さい添え字(1~10)を持つ、
100×100の行列を作成するにはどのようなコードを書けばよいでしょうか?
> set.seed(123)
> mat = array(sample(10:15, 48, replace=TRUE), dim=c(4, 4, 3))
> mat
, , 1 # 第 3 次元の添え字が 1 の行列
[,1] [,2] [,3] [,4]
[1,] 11@ 15 13 14* *, @ が何を意味するかは後述
[2,] 14 10 12 13
[3,] 12 13 15 10
[4,] 15 15 12 15
, , 2 # 第 3 次元の添え字が 2 の行列
[,1] [,2] [,3] [,4]
[1,] 11@ 15 13 11*
[2,] 10 14 14 10
[3,] 11 13 13 15
[4,] 15 15 13 15
, , 3 # 第 3 次元の添え字が 3 の行列
[,1] [,2] [,3] [,4]
[1,] 14@ 14 10 10*
[2,] 14 11 12 10
[3,] 10 11 12 11
[4,] 12 11 12 12
あなたが望むのは,以下のような行列ですか?> apply(mat, 1:2, which.min)
[,1] [,2] [,3] [,4]
[1,] 1 3 3 3
[2,] 2 1 1 2
[3,] 3 3 3 1
[4,] 3 3 1 3
なお,最小値が同値の場合は最小の添え字が要素となる。たとえば,第 1 次元が 1,第 2 次元が 1 のとき,第 3 次元が 1,2,3 のときの値は @ で印をつけた (11, 11, 14) なので,最小値 11 は 2 つあり,その添え字は 1 と 2 であるが,小さい方の 1 が要素となる。(2018-04-30 (月) 01:24:12)
三次元空間からある形状の特徴を抽出するため
四分木もしくは八分木を作成することを試行したいのですが
パッケージはありますでしょうか?
ムーミン (2018-04-21 (土) 18:25:20)
よろしくお願いします
エクセルからのインポートはできかしたが、そのデーターからグラフを作成しょうとWEBをみて回りましたがさっぱりわかりません。(途中が省かれたり解説なかったり、例がインポートしたものではなかったりなど)”Rによるデーターサイエンス”の本図書館から借りてみましたが上級者向けで理解できませんでした。ホントの初心者です、エクセルからインポートしたデーターをグラフ化することが詳しく解説されている書籍を教えてください。
めげそうです。
ムーミン (2018-04-18 (水) 20:14:01)
どこに書き込んだらよいかわからなかったので新規投稿に書きました
よろしくお願いします。
手順は
1)data4=read.table("clipboard",header=TRUE)を記述、
2)データーをコーピー
3) 1)をエンターキーを押しました。
又は
1)、2)を逆にし実行
下記が結果です。何も起こりません
R version 3.4.4 (2018-03-15) -- "Someone to Lean On"
Copyright (C) 2018 The R Foundation for Statistical Computing
Platform: x86_64-w64-mingw32/x64 (64-bit)
R は、自由なソフトウェアであり、「完全に無保証」です。
一定の条件に従えば、自由にこれを再配布することができます。
配布条件の詳細に関しては、'license()' あるいは 'licence()' と入力してください。
R は多くの貢献者による共同プロジェクトです。
詳しくは 'contributors()' と入力してください。
また、R や R のパッケージを出版物で引用する際の形式については
'citation()' と入力してください。
'demo()' と入力すればデモをみることができます。
'help()' とすればオンラインヘルプが出ます。
'help.start()' で HTML ブラウザによるヘルプがみられます。
'q()' と入力すれば R を終了します。
data4=read.table("clipboard",header=TRUE)
AAA (2018-04-17 (火) 22:42:38)
お世話になります。
glm関数で症例対照研究のロジスティック回帰を実行しようとしたのですが、症例対照研究は症例群と対照群のサンプル数を任意に設定できるので、生存率やオッズの算出に意味はないという資料がありました。ロジスティック回帰式の左辺はlog(p/(1-p))で対数オッズを意味していますので、そもそも回帰することができないということになるでしょうか。
glmに目的変数と説明変数を与えると計算結果自体は出力されるのですが、根本的に前提が間違っているような気がしています。
宜しくお願いします。
ムーミン (2018-04-17 (火) 18:19:45)
よろしくお願いします
下記データーをRコンソール、にクリップボードを介してインポートする方法を教えてください。
R言語のインストールは下記のページを参考にして行いました
https://qiita.com/FukuharaYohei/items/8e0ddd0af11132031355
インポートは下記ページを参考にしましたがうまくいきません
http://monge.tec.fukuoka-u.ac.jp/r_analysis/input_clipbord00.html
月 電気量 電気料金
1月 306 8184
2月 335 9065
3月 341 9311
R言語のバージョンは3.4.4
ウィンドウズ10でエクセルのバージョンは1803です
kagakuma (2018-04-13 (金) 12:00:31)
両対数を取ると明らかに線形関数に乗るデータがあります。
このデータをlm()で線形回帰したところR2が0.95でフィッティングしました。
このデータ群は2つあり、線形回帰したのですが、いずれも違う直線に乗りました。 この結果から傾きと切片が2組でますが、両者の傾きと切片に有意差があるかどうかを確かめる方法はございませんでしょうか。
Rの機能で閉じていると助かりますが、手計算でもできるならやろうと思います。
単純には両者を引き算してそれの傾きと切片を求めるという方法も考えましたが、正当性がわかりません。 よろしくおねがいします。
ksaki (2018-04-12 (木) 14:42:13)
お世話になります。
井手剛先生著の
入門 機械学習による異常検知のp137の実行例5.1を真似て、以下のコマンドを入力しましたが、エラーが出ます。
何が間違っているか教えてください。
以下、入力したコマンド> data <- read.table("R_r1.csv",header=T) > data a.b.c.d.e.f.g.h.i.j.k 1 P2201A1,-4.78,11,1.44,-0.48,3.83,-10.05,15.79,4.31,1.44,18.66 2 P2201A2,-3.35,-21.53,-11,8.13,-6.22,18.66,-2.87,-0.96,2.39,-11.48 3 P2201A3,2.88,3.63,4.69,0.61,1.21,1.36,-1.67,1.06,1.06,1.67 4 P2201A4,-0.48,3.35,-5.26,-6.22,-3.35,-4.78,0.48,-0.48,1.91,3.35 5 P2201H1,-14.35,-5.26,0,-11.96,-7.66,14.35,-11.48,-5.26,14.35,-0.48 6 P2201H2,12.92,13.87,0.96,13.4,-0.96,1.91,-12.44,-13.87,-10.05,-14.35 7 P2201H3,7.18,3.83,3.35,0,-4.31,-8.61,-12.44,-3.35,1.44,5.74 8 P2201H4,-7.18,-16.27,-16.27,-5.26,2.39,8.61,12.92,12.92,6.7,-0.48 9 P2201V1,-6.7,-3.83,-12.92,2.39,2.39,2.87,12.44,2.87,1.91,-8.13 10 P2201V2,-3.03,-18.17,22.71,-27.25,27.25,-15.14,0,-10.6,-18.17,21.2 11 P2201V3,-4.31,-0.48,1.91,-1.44,-5.26,1.44,3.35,-8.61,-6.7,-2.87 12 P2201V4,0.48,2.39,1.91,3.35,-1.44,-7.18,-5.74,-1.91,7.66,7.66 13 P2202A1,-1.44,-0.96,3.35,1.91,-0.48,0.96,-3.35,-2.39,0.48,-6.7 14 P2202A2,-3.83,0,1.44,-6.22,-3.35,0.96,-1.44,0.48,-0.48,-1.91 15 P2202A3,0.61,-1.36,-0.15,0.61,0.76,-2.57,-2.12,-2.57,-0.91,0.76 16 P2202A4,0.45,2.27,-0.45,1.21,0.15,-0.15,0.76,-1.06,-1.21,1.06 17 P2202H1,5.26,-4.31,-0.48,-1.44,-3.83,0.48,0.48,5.74,-0.48,0.48 18 P2202H2,-0.96,-1.91,-0.96,-5.74,0,1.91,-5.74,1.44,-1.91,-6.7 19 P2202H3,-1.67,0.45,-0.45,2.57,2.27,-1.21,-0.61,-0.3,-0.15,0.45 > cc <- c("a","b","c","d","e","f","g","h","i","j","k") > mask <- is.element(colnames(data),cc) > Xc <- t(scale(data[,mask])) > colnames(Xc) <- t(data[,"a"]) Error in `[.data.frame`(data, , "a") : 未定義の列が選ばれましたコマンド、ここまで
〇環境設定> sessionInfo() R version 3.3.3 (2017-03-06) Platform: x86_64-w64-mingw32/x64 (64-bit) Running under: Windows 7 x64 (build 7601) Service Pack 1 locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base other attached packages: [1] MASS_7.3-45 loaded via a namespace (and not attached): [1] tools_3.3.3
> data = read.csv("R_r1.csv") # read.csv は header = TRUE はデフォルトなので,指定不要
> data
a b c d e f g h i j k
1 P2201A1 -4.78 11.00 1.44 -0.48 3.83 -10.05 15.79 4.31 1.44 18.66
2 P2201A2 -3.35 -21.53 -11.00 8.13 -6.22 18.66 -2.87 -0.96 2.39 -11.48
中略
18 P2202H2 -0.96 -1.91 -0.96 -5.74 0.00 1.91 -5.74 1.44 -1.91 -6.70
19 P2202H3 -1.67 0.45 -0.45 2.57 2.27 -1.21 -0.61 -0.30 -0.15 0.45
> cc = c("b","c","d","e","f","g","h","i","j","k") # 以下の意図はよく分からないけど,"a" は外さないとエラーが出るよ
> # cc = letters[2:11] とも書けるけど
> mask = is.element(colnames(data),cc)
> Xc = t(scale(data[,mask]))
> # Xc = t(scale(data[, 2:11])) で十分。mask や cc なんか使わなくても良いし
> colnames(Xc) = t(data[,"a"]) # t は不要