![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
![[RjpWiki] [RjpWiki]](image/../Rlogo.png)
初心者のための R および RjpWiki に関する質問コーナー
以下の 4 つの「●項目」のどれかをクリック!!
● 新規投稿欄: --- その前に,「投稿における注意事項」を読んでから!
● このページの目次 --- 質問への回答・コメントの参照
● 最新のスレッド --- 最も最近に書き込まれた「親」記事(最新のコメントが付いた記事ではありません)
● Q&A (初級者コース) 過去の目次 --- 今までの 17 個の書庫の参照
参照は,個々の項目をクリック(質問への回答・コメントの参照も,個々の項目をクリック)
●● 新規投稿 ●●
まずは,「投稿における注意事項」を読んで,それに従ってください。
これに従わない場合には,回答が得られにくい場合があります。
鈴木康弘 (2024-07-24 (水) 17:57:14)
[,1] [,2] [1,] 1 1 [2,] 2 NA [3,] 3 3 [4,] 4 NA
上のような行列があるとします。NAを1列目の値2,4で代用したく、
下記を試してみました。
> m[,2][is.na(dat[,2])] <- dat[,1] m[, 2][is.na(dat[, 2])] <- dat[, 1] でエラー: 置き換えるべき項目数が、置き換える数の倍数ではありませんでした
どうしたらいいのでしょう?
> dat <- matrix(c(1:4, 11, NA, 13, NA), ncol = 2)
> dat
[,1] [,2]
[1,] 1 11
[2,] 2 NA
[3,] 3 13
[4,] 4 NA
> i <- is.na(dat[, 2])
> dat[i, 2] <- dat[i, 1]
> dat
[,1] [,2]
[1,] 1 11
[2,] 2 2
[3,] 3 13
[4,] 4 4
もちろん、このくらいで速度やら最適化やらうるさくいうのもバカバカしいですし、ご自身でみつけられたforループを使う方法で全然構わないと思います。 -- 2024-07-25 (木) 17:25:25> dat <- matrix(c(1:4, 11, NA, 13, NA), ncol = 2)
> i <- is.na(dat[, 2])
> i
[1] FALSE TRUE FALSE TRUE
> j <- which(i)
> j
[1] 2 4
> dat[j, 2] <- dat[j, 1]
> dat
[,1] [,2]
[1,] 1 11
[2,] 2 2
[3,] 3 13
[4,] 4 4
また余計かもしれませんが、行列はRにおいてはベクトルの一種です。なので以下のようなこともできます。1列目にNAがないこと前提ですし、可読性が下がるのでまったくオススメしませんが、勉強のタネとして。 -- 2024-07-26 (金) 05:48:19
> dat <- matrix(c(1:4, 11, NA, 13, NA), ncol = 2)
> k <- which(is.na(dat))
> k
[1] 6 8
> dat[k] <- dat[k - nrow(dat)]
> dat
[,1] [,2]
[1,] 1 11
[2,] 2 2
[3,] 3 13
[4,] 4 4カイ (2024-04-14 (日) 14:17:43)
よろしくお願いいたします。
カイ二乗検定を行い、P値を取り出すとき以下のよう、マトリックスにして計算しております。
d <- c(10, 5, 8, 7)
d <- matrix(d,nrow=2,byrow=T)
d <-chisq.test(d)
d$p.value
しかし、
data <- matrix(c(10, 5, 8, 7, 16, 23, 21, 8, 27, 44, 61, 22, 10, 15, 20, 24, 38, 76, 23, 77), nrow=5, byrow=T)
colnames(data)<-c("a","b","c","d")
のようなデータがある場合、一気にカイ二乗検定を行い、d列の横にp値を入れていきたいのですが、マトリックスが出来なく困っております。
どの様にすればよいのかご教示いただけると幸いです。
(R version 4.2.2, win11)
> print(data)
a b c d
[1,] 10 5 8 7
[2,] 16 23 21 8
[3,] 27 44 61 22
[4,] 10 15 20 24
[5,] 38 76 23 77
> p <- apply(data, 1, function(x) { chisq.test(matrix(x, nrow = 2))$p.value })
> cbind(data, pval = p)
a b c d pval
[1,] 10 5 8 7 7.093881e-01
[2,] 16 23 21 8 2.012252e-02
[3,] 27 44 61 22 1.955183e-05
[4,] 10 15 20 24 8.518854e-01
[5,] 38 76 23 77 1.287840e-01
前述のとおり、2x2のデータ群と、そのそれぞれに対する検定結果のp値を、無理やりこのような5列の行列にすること自体の是非は正直あります。(一覧性の都合など、質問者様の固有の理由があるのだと思いますが。)> data2 <- array(t(data), dim = c(2, 2, nrow(data)))
> print(data2)
, , 1
[,1] [,2]
[1,] 10 8
[2,] 5 7
, , 2
[,1] [,2]
[1,] 16 21
[2,] 23 8
, , 3
[,1] [,2]
[1,] 27 61
[2,] 44 22
, , 4
[,1] [,2]
[1,] 10 20
[2,] 15 24
, , 5
[,1] [,2]
[1,] 38 23
[2,] 76 77
> apply(data2, 3, function(x) { chisq.test(x)$p.value })
[1] 7.093881e-01 2.012252e-02 1.955183e-05 8.518854e-01 1.287840e-01たたた (2023-11-20 (月) 13:23:54)
お世話になっております。表記のとおり2つのパッケージ間で画像をやりとりしたいです。画像をimagerで用いるcimgで読み込み、magickへの読みこむことは以下の方法でできるのですが、逆にmagickの画像からcimgにする方法をどなたか教えていただければ幸いです。image1 <- imager::load.image("image1.png") image1.mag <- magick::image_read(as.cimg(image1))一応、手元で、以下のようにすると、それっぽくはなるのですが、値が数値ではなくrawだし、次元数も変わってしまっており、どうしたらよいものかと思っております。
image_matrix <- magick::image_data(image1.mag) image_imager <- imager::cimg(image_matrix)どうぞよろしくお願いします。
徒弟 (2023-08-28 (月) 21:26:18)
皆様のお知恵をお貸しください
以下のコマンドの通りを試したところ、(3)でのみエラーが出ます
set.seed(1234)
tmp <- data.frame(Outcome=c(rep(1,10),rep(0,10)),X=rnorm(20))
tar_formula <- as.formula(paste0("Outcome","~X"))
#(1)
with(tmp,glm(Outcome~X,family=binomial))
#(2)
with(tmp,glm(as.formula(paste0("Outcome","~X")),family=binomial))
#(3)
with(tmp,glm(tar_formula,family=binomial))
エラーメッセージは" eval(predvars, data, env) でエラー:
オブジェクト 'Outcome' がありません"です
どういよう理由によってエラーが発生するのか教示頂ければ幸いです
> tar_formula <- as.formula("Outcome ~ X")
> str(tar_formula)
Class 'formula' language Outcome ~ X
..- attr(*, ".Environment")=<environment: R_GlobalEnv>
with内でas.formulaを実行した場合には、つくられたformulaはwithがつくった空間内を参照しにいっています。 -- 2023-08-29 (火) 06:39:21
> with(data.frame(), str(as.formula("Outcome ~ X")))
Class 'formula' language Outcome ~ X
..- attr(*, ".Environment")=<environment: 0xa6bb540>晴猫 (2023-08-27 (日) 19:36:31)
yokkunさんのページ
https://yokkuns.hatenadiary.org/entry/20110928/1317164851
にならってベイジアンネットワーク分析を試みたところ、ネットワークの矢印が正しく表示されません。
mybanlist <- matrix(c(2,1, 3,1, 4,1, 5,1, 6,1, 7,1, 3,2, 4,2, 5,2, 6,2, 7,2, 3,4, 4,3), ncol = 2, byrow = TRUE)banlist(ht.nw) <- mybanlist
で矢印を禁止するノードを指定します。その後
plot(ht.nw)
でネットワークを描いてみると、矢印がないと指定したノードの間に赤い破線で矢印が描かれます。しかし、その後の計算は正しくできるようです。ですからこれは矢印の表示の問題だと思います。通常は黒い実線でノード間の因果関係が表示されます。赤い破線は逆方向の因果関係を表しているようです。正方向の因果関係を表示させるにはどうしたらよいのでしょうか。
OSはWindows11, Rはversion 4.2.1を使用しています。よろしくお願いします
> names(ht.search) [1] "nw" "table" "trylist" > class(ht.search$nw) [1] "network"ですので、plot(ht.nw)ではなくplot(ht.search$nw)とすれば、autosearchし直さなくても解析結果を再表示できます。plot(ht.nw)は分析前のデータの描画、plot(ht.search$nw)が分析後の結果の描画です。その際、件の赤矢印が「事前のbanlistの内容」として一緒に描画されますが、それが邪魔ならplot時のshowban引数をFALSEにすれば描画しないことが可能です。
plot(ht.search$nw, showban = FALSE)networkクラスのオブジェクトの描画には、plot関数に用意されたnetworkメソッドが使われています。plot.networkのヘルプを参照していただければ、描画の仕様はわかるかと思います。 -- 2023-08-30 (水) 07:26:23
136s (2023-08-02 (水) 11:10:23)
質問: 掲題のバージョンが現時点で v4.1.3 ですが、linux-64 や osx-64 は v4.3.1 になっています。win-64 がアップデートされないのは、何か理由があるのでしょうか?
技術的不可能性があるのか、需要がないのか、、など。
参考: https://anaconda.org/conda-forge/r-base
経緯: Windows 10 マシンで、conda 環境内で R を管理して Python から rpy2 で R を使おうとしています。
通常、`conda install -c conda-forge r` コマンドで conda 環境内に R をインストールすると思います。
ただ、現在の最新の r パッケージ が noarch で v4.3 であり、r-base のバージョン不一致になってしまいます。
これ自体は r パッケージのインストール時に v4.1 を指定すれば解決するのですが、そもそも r-base が v4.1.3 からバージョンアップされないのか気になったので質問を投稿いたしました。
masa (2023-06-04 (日) 14:03:04)
①結果の入力方法(他に必要なパッケージなど) ②結果の解釈 を知りたいです。最低限①だけでも助かります。
島田泰夫 (2023-06-02 (金) 17:16:52)
教えてください
ggplot2で積み上げ棒グラフを作図したとき、棒の上に合計値を表示したいのです
tableのクロス集計表(re)で
0 1 2 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 402 0 3 2 1 2 37 0 0 0 0 0 1 10 15 20 0 0 1 0 2 1 0 0 404 0 0 0 0 0 0 1 2 2 12 1 5 3 2 0 1 9 0 0 0 0 0 0 406 0 2 0 0 15 1 19 21 3 0 3 8 0 0 0 0 0 2 0 0 0 0 0 411 0 0 0 20 5 6 10 11 12 8 9 0 0 0 2 6 10 3 2 0 2 1 1 416 16 11 18 1 0 0 1 0 2 0 0 0 0 0 0 8 0 0 14 16 1 1 0 418 0 0 0 0 0 0 0 20 13 5 2 2 10 18 62 94 60 11 3 4 4 27 0 422 0 0 0 0 0 0 0 0 9 6 5 10 12 14 8 30 26 22 14 9 3 4 3 428 0 0 15 4 9 1 12 2 3 2 2 15 1 10 2 4 0 0 0 0 9 16 6 429 10 6 0 0 0 21 7 2 7 0 0 1 17 16 9 4 3 0 3 1 4 0 3
が得られたので、データフレーム変換して
re2 <- as.data.frame(re)
Var1 Var2 Freq 1 402 0 0 2 404 0 0 3 406 0 0 ....
となりましたので
g <- ggplot(re2, aes(x = Var2, y = Freq, fill = Var1)) g <- g + geom_bar(stat = "identity") g <- g + geom_text(aes(label = Freq, y = Freq + 3), size = 2) plot(g)
とすると、積み上げる「各要素」の数値が入ってしまいます
これを避けて、積み上げた「合計値」を各棒の上部に記載したいのですが
どのようにすればよろしいでしょうか?
自己解決できそうです
re3 <- as_tibble(re2)
re4 <- re3 %>% group_by(Var2) %>% summarise( total= sum(Freq)) ggplot(data = re4, aes(x = Var2, y = total)) + geom_bar(stat = "identity", fill = "grey", colour = "black") + geom_text(aes(label = total, y = total + 3), size = 2) + geom_bar(data = re2, aes(x = Var2, y = Freq, fill = Var1), stat = "identity")
R初心者 (2023-05-08 (月) 09:58:23)
特徴量が1000個あるデータに対してPCAを実行しました.有効な特徴量を抽出するには,どの計算結果を参考にすればよろしいでしょうか...基本的な質問で申し訳ありません.
hs0519 (2023-03-14 (火) 23:18:04)
具体的な質問というより、一般的な考え方の質問です。RのWindows版をインストールした直後、Rを起動させて、install.packages("Rcmdr",dependencies=TRUE)などとやってインストールするのですが(ダウンロード先はTokyo)、エラーが出ます。インストール作業のプロセスが進行していることは動作をみていてわかりますね。メッセージなどが出て進行状況がわかります。そして最後にエラーが出るという図式です。
この流れの中で異質なこと、独特なところは何もありません。エラーが出ないときもあります。使用者側はここまでの流れで工夫するところがないので、提供側の問題としか思えません(ダウンロード中の欠損もありうる?)。そのためエラーメッセージを見ながらフィックスする努力は徒労でかつ、生産的でないように感じられます。やはりエラーメッセージをたよりにフィックスするのか、Rそのものの更新を待つべきか悩みが出ます。この部分は果報は寝て待てなんじゃないかと思うのですが、実際どうなのでしょうか。
 そんなところに原因が潜んでいる可能性があるものなのですね。 -- 2023-03-24 (金) 07:07:00
そんなところに原因が潜んでいる可能性があるものなのですね。 -- 2023-03-24 (金) 07:07:00まつ (2023-03-14 (火) 20:56:57)
mFDというパッケージでmFD::quality.fspacesを行った際に
Error in if (maxdim_pcoa < 1) { : the condition has length > 1
というエラーが出ました。教えて頂けると嬉しいです。ちなみにmaxdim_pcoaは2以上の色々な値で試していますが同じエラーが出ます。
(2023-03-13 (月) 14:40:09)
日付、性別、年代、検索行動1、検索行動2、検索行動3、収益額(この値は99%が0で、そのほかは1000円から50000円くらいの値が入っています。)
という情報があったときに収益をもたらすかどうかを示す指数を作りたいです。
指数の条件としては以下の2つです。
・値が大きいほど収益額1000円超の顧客の割合は大きくなる傾向にある
・値が大きいほど収益額が大きくなる傾向にある
lightgbmのregressionでやってみても全然だめでした。
お手数ですがお知恵をお貸しください。
hs0519 (2023-03-10 (金) 08:02:35)
saveVideo({for (i in 1:5){ plot(runif(10), ylim = c(0,1)) }}, interval = 1.0, video.name = "TEST.html")
というスクリプトだけで、ffmpegが呼び出せるとのことなので、実行してみると、
The command ""ffmpeg"" is not available in your system. Please install FFmpeg
というエラーメッセージが出ます。
ffmpegはインストールされており、パスも通っているのでシェルのコマンドラインから起動できます。
Rからffmpegを起動するにはどのようにしたらいいでしょうか。
バージョンにセンシティブな問題でしょうか。
Rは4.2.2, ffmpegは5.1.2が入っています。ただし、ffmpegはいろんなところ(irfanviewとか)から入る可能性があるので5.1.2でない可能性が大です。
パスが先になっている方だと思いますが。
たたた (2023-03-01 (水) 06:05:33)
エクセルからコピーしたタブ区切りの「データ0 データ1(ほげ) データ2? データ3? データ4」というデータがあり、 name <- read.table("clipboard",sep="\t")という処理を行いたいと思います。 手元のRの環境で、(や?があっても読めるものと読めない物があるのですが、読めないほうでも読むにはどのようにしたらよいかわかりますでしょうか。 読めるものは:R version 3.6.2 (2019-12-12)、LocaleがJapanese_Japan.932 。読めないほうは:R version 4.2.1 (2022-06-23 ucrt)。LocaleがJapanese_Japan.utf8 。いずれも、 Windows 10 x64 (build 19041)です。 なお、文字コードをShift-JIS系に指定しても以下のエラーが出てしまいます。read.table("clipboard",sep="\t",fileEncoding = "CP932") 警告メッセージ: 1: file(file, "rt", encoding = fileEncoding) で: 引数 'encoding' は無視されます 2: read.table("clipboard", sep = "\t", fileEncoding = "CP932") で: incomplete final line found by readTableHeader on 'clipboard'
OKD (2023-02-05 (日) 15:27:12)
こんにちは.
初歩的な質問で大変恐縮ですが,以下のご回答をよろしくお願い致します.
早速ですが, as.*() と as_*() の違いが理解できません.
"*" には,matrixやvector,factor などを想定しています.
"as"の部分は,”is”の場合でも違いがわかっていません.
どなたかご教示いただけますでしょうか?
> x <- correlate(iris)
Non-numeric variables removed from input: `Species`
Correlation computed with
• Method: 'pearson'
• Missing treated using: 'pairwise.complete.obs'
> class(x) # cor_dfという独自のクラスとなる
[1] "cor_df" "tbl_df" "tbl" "data.frame"
> print(x) # printするとmatrixぽく表示されるが…
# A tibble: 4 × 5
term Sepal.Length Sepal.Width Petal.Length Petal.Width
<chr> <dbl> <dbl> <dbl> <dbl>
1 Sepal.Length NA -0.118 0.872 0.818
2 Sepal.Width -0.118 NA -0.428 -0.366
3 Petal.Length 0.872 -0.428 NA 0.963
4 Petal.Width 0.818 -0.366 0.963 NA
> str(x) # 実際はtermという文字列要素をもつdata.frame
cor_df [4 × 5] (S3: cor_df/tbl_df/tbl/data.frame)
$ term : chr [1:4] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width"
$ Sepal.Length: num [1:4] NA -0.118 0.872 0.818
$ Sepal.Width : num [1:4] -0.118 NA -0.428 -0.366
$ Petal.Length: num [1:4] 0.872 -0.428 NA 0.963
$ Petal.Width : num [1:4] 0.818 -0.366 0.963 NA
> as_matrix(x) # 数値matrixに変換
Sepal.Length Sepal.Width Petal.Length Petal.Width
Sepal.Length NA -0.1175698 0.8717538 0.8179411
Sepal.Width -0.1175698 NA -0.4284401 -0.3661259
Petal.Length 0.8717538 -0.4284401 NA 0.9628654
Petal.Width 0.8179411 -0.3661259 0.9628654 NA
このcor_dfクラスというオブジェクトは、基本的にはdata.frameなのですが、変量名にあたる情報をtermという要素としてもっています。なので、単にビルトインのas.matrixを適用すると、文字列型のmatrixに強制変換されてしまうようです。
> as.matrix(x) # 4x5の文字列matrixになってしまった
term Sepal.Length Sepal.Width Petal.Length Petal.Width
[1,] "Sepal.Length" NA "-0.1175698" " 0.8717538" " 0.8179411"
[2,] "Sepal.Width" "-0.1175698" NA "-0.4284401" "-0.3661259"
[3,] "Petal.Length" " 0.8717538" "-0.4284401" NA " 0.9628654"
[4,] "Petal.Width" " 0.8179411" "-0.3661259" " 0.9628654" NA
それを防ぐため、「cor_dfオブジェクトをmatrixへと、数値データを数値のまま変換する」ための独自関数がas_matrixである、という感じでしょうか?> y <- data.frame(a = 1:3, b = 4:6)
> print(y)
a b
1 1 4
2 2 5
3 3 6
> as.matrix(y)
a b
[1,] 1 4
[2,] 2 5
[3,] 3 6
> as_matrix(y)
UseMethod("as_matrix") でエラー:
'as_matrix' をクラス "data.frame" のオブジェクトに適用できるようなメソッドがありません
混同して使ってしまうとまずいです。 -- 2023-02-07 (火) 07:38:32asao (2022-12-16 (金) 14:26:56)
こんにちは
素人質問で恐縮ですが時系列データの取り扱いについての質問です。
現在以下の形式のようなデータを用いて、時刻と使用率の関係を推定しようとしています。
ー-----データー------ー----------ー--
beginning_time ocp_rate inflow outflow
2020/6/1 23:58:00 0.023255858 1 0
2020/6/1 23:59:00 0.893255895 0 1
2020/6/2 0:00:00 0.023255826 1 2
2020/6/2 0:01:00 0.103255844 0 0
2020/6/2 0:02:00 0.045255836 0 1
2020/6/2 0:03:00 0.113255814 2 0
ー-----データー--------------------
そこで新たに
その日付の深夜0時(1行目でいうと2020/6/1 00:00:00)
からの経過時間(分)を示す行を追加したいと考えております。
どのように計算すればよいでしょうか。
今のところ、各日付の00:00:00を示す行を追加して、beginning_time列から引き算しようと試みておりますが、各日付の00:00:00の列を作ることにも苦戦しております。
ご教授いただけますと幸いです。
dat <- matrix(c(
"2020/6/1 23:58:00", "0.023255858", "1", "0",
"2020/6/1 23:59:00", "0.893255895", "0", "1",
"2020/6/2 0:00:00", "0.023255826", "1", "2",
"2020/6/2 0:01:00", "0.103255844", "0", "0",
"2020/6/2 0:02:00", "0.045255836", "0", "1",
"2020/6/2 0:03:00", "0.113255814", "2", "0"),
byrow = TRUE, ncol = 4)
colnames(dat) <- c("beginning_time", "ocp_rate", "inflow", "outflow")
time <- as.POSIXlt(dat[, "beginning_time"],
format = "%Y/%m/%d %H:%M:%S")
h <- as.numeric(format(time, "%H"))
m <- as.numeric(format(time, "%M"))
elapsed <- h * 60 + m
dat <- cbind(dat, elapsed = elapsed)
print(dat)
今回ご質問の作業「だけ」をするなら、sub関数などで時:分だけ取り出したりする方法もあります。が、時間データをもとに「何月何日の」とか「何曜日のデータだけ」とかアレコレいじる作業をしようと思うと、最初にPOSIXltなどにしてしまっておくのがラクかなと思います。 -- 2022-12-16 (金) 16:29:10okadasan (2022-12-13 (火) 15:32:33)
お世話になっております。
まだまだ、Rは初心者です。
http://okadasan.com/S_P.R
と言う関数を作りました。
p[r0,gamma,a]と言う関数が、
r0とgammaとaの変化で変わって行っているのがわかると思います。
ここで、r0をx軸にして、gammaをy軸にして、aをz軸にして、
3次元空間で、それら3つの係数の変化で決まる関数であるp[r0,gamma,a]の各点の大きさを、色の濃さで表したいのです。
そのような事は、Rで出来るのでしょうか?
plot3dで出来るのかなあ、とか、
別なグラフ関数を持って来るのかなあ、とか、
あるいは、その所作をコードの何処に入れるのかも
わかりません。
どなたか、お教えいただけるとありがたいのですが。
library(rgl)
r0 <- 1:20
gamma <- 1:36
a <- 1:100
f <- function(x1, x2, x3) {
# just for example
# not the true "Sound Pressure" function
return((x1 - 10)^2 * abs(x2 - 30) + sin(x3 / 10) * 1000)
}
xs <- expand.grid(r0, gamma, a)
p <- f(xs[, 1], xs[, 2], xs[, 3])
n_col <- 100
i <- round((p - min(p)) / diff(range(p)) * (n_col - 1)) + 1
col <- cm.colors(n_col)[i]
plot3d(xs[, 1], xs[, 2], xs[, 3],
xlab = "r0", ylab = "gamma", zlab = "a",
col = col, aspect = FALSE)
関数(f)の定義の部分は、前述のとおりわたしは理解していないので、適当な算術処理に置き換えました。単に「3次元配列の値を空間内にプロットする例」です。やっぱり空間を密に埋めるので、遮蔽の問題をどうにかしないと実用性がなさそうな気がしてしまいます。ざっと傾向を眺めるだけ、なら何かの役に立つものなのでしょうか。。。 -- 2022-12-13 (火) 17:31:28noname (2022-09-04 (日) 17:59:16)
lmer()関数でoffsetを指定した混合モデルにlmerTest::difflsmeans()関数を適用すると,offsetで指定した変数名のオブジェクトがないとエラーが出ます.
以下で再現すると思います.
library(lmerTest) data("cake", package="lme4") cake2 <- data.frame(cake,os = round(cake$temp/cake$angle,0)) model <- lmer(angle ~ recipe * temp + (1|recipe:replicate) + os, cake2) model2 <- lmer(angle ~ recipe * temp + (1|recipe:replicate) + offset(os), cake2) anova(model) anova(model2) difflsmeans(model) difflsmeans(model2) # オブジェクト 'os'がありませんというエラー
sessionInfo() R version 3.6.3 (2020-02-29) Platform: x86_64-apple-darwin15.6.0 (64-bit) Running under: macOS 10.16 Matrix products: default BLAS: /Library/Frameworks/R.framework/Versions/3.6/Resources/lib/libRblas.0.dylib LAPACK: /Library/Frameworks/R.framework/Versions/3.6/Resources/lib/libRlapack.dylib locale: [1] ja_JP.UTF-8/ja_JP.UTF-8/ja_JP.UTF-8/C/ja_JP.UTF-8/ja_JP.UTF-8 attached base packages: [1] stats graphics grDevices utils datasets methods base other attached packages: [1] lmerTest_3.1-3 lme4_1.1-27.1 Matrix_1.2-18 dbscan_1.1-8 colorout_1.2-2 pacman_0.5.1 loaded via a namespace (and not attached): [1] Rcpp_1.0.9 compiler_3.6.3 pillar_1.8.1 nloptr_1.2.2.2 tools_3.6.3 boot_1.3-24 lifecycle_1.0.1 [8] tibble_3.1.8 nlme_3.1-144 gtable_0.3.0 lattice_0.20-38 pkgconfig_2.0.3 rlang_1.0.4 cli_3.3.0 [15] DBI_1.1.1 dplyr_1.0.9 generics_0.1.3 vctrs_0.4.1 grid_3.6.3 tidyselect_1.1.2 glue_1.6.2 [22] R6_2.5.1 fansi_1.0.3 minqa_1.2.4 ggplot2_3.3.6 purrr_0.3.4 magrittr_2.0.3 scales_1.2.1 [29] MASS_7.3-51.5 splines_3.6.3 assertthat_0.2.1 colorspace_2.0-3 numDeriv_2016.8-1.1 utf8_1.2.2 munsell_0.5.0
stackexchangeやマニュアルを調べたものの,そもそもlmer()関数で実際にoffsetを使っている例*すら見つからず,皆様のお知恵を拝借出来れば幸いです. 一番近い例:https://stats.stackexchange.com/questions/88960/lme4-glmer-problems-with-offset [#pd4b1c0b]
※投稿における注意事項に記載されているユーザ名の求め方が間違っているようです.
http://www.okadajp.org/RWiki/?%E6%8A%95%E7%A8%BF%E3%81%AB%E3%81%8A%E3%81%91%E3%82%8B%E6%B3%A8%E6%84%8F%E4%BA%8B%E9%A0%85
修正しました.
offsetはformuraの中に入れるのではなく,別の引数として指定するものでした.
lmerTestパッケージのマニュアルにlmerでのoffset使用例がないことと,lme4::glmerの書き方を流用したこと,formuraのなかに入れてもエラーが出ないこと,何よりも私の確認不足・能力不足,があわさってお騒がせをしてしまいました. 誠に申し訳ございません.
library(lmerTest)
data("cake", package="lme4")
cake2 <- data.frame(cake,os = round(cake$temp/cake$angle,0))
model2 <- lmer(angle ~ recipe * temp + (1|recipe:replicate), offset=os, cake2)
anova(model2)
difflsmeans(model2) # エラーは出ない
花椒報告 (2022-09-02 (金) 13:10:19)
result<- brm(formula = sales ~ weather + temperature, family = gaussian(), data = d, seed = 1)
を実行すると、
Error in compileCode(f, code, language = language, verbose = verbose) :C:\rtools42\x86_64-w64-mingw32.static.posix\bin/ld.exe:
(以下、10行くらいにわたる長いエラー文)
が出てしまいます。
library(rstan)
library(brms)
は無事できています。
環境は
R version 4.2.1 (2022-06-23 ucrt)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 22000)
です。
どのように対処すればよいでしょうか?
てくてく (2022-08-23 (火) 16:18:12)
R version 4.2.1 (2022-06-23 ucrt)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 19044)
DEMの描画や、切り取りについて勉強中です。試しに、rasterパッケージ内のcropというオブジェクトのヘルプに記載の内容(下記)、
r <- raster(nrow=45, ncol=90)
values(r) <- 1:ncell(r)
e <- extent(-160, 10, 30, 60)
rc <- crop(r, e)
を実行してみたのですが、rcのextentはeで指定した値ではなく、-160, 8, 30, 58になってしまいます。
crop後のextentの値が、cropの範囲(上記のe)で指定した値にならないのはどうしてでしょうか。ご教授いただけたらと思います。よろしくお願いいたします。
ん (2022-08-04 (木) 14:18:29)
Mac book air (2018, Intel) OS MontereyでRを使用しています。
EZRが必要になり、頒布元が提示してくださっている手順に従って、R起動時に自動でEZRも起動する設定を行いました。
options(Rcmdr=list(plugins='RcmdrPlugin.EZR'))
library(Rcmdr)
local({
old <- getOption('defaultPackages')
options(defaultPackages = c(old, 'Rcmdr'))
})
しかしその後Rstudioを使用するようになり、Rstudio起動→Rにコネクト→EZR自動起動のためxquartzが起動→EZR起動→Rstudioがやっと立ち上がる、という状態になってしまいました。
options関数について調べてみましたが情報が少なく、
options(defaultPackages = c(getOption('defaultPackages')))
と入れてみましたが、自動起動設定は変わりませんでした。
起動時設定を初期化する方法はありますでしょうか?
ご教授いただければ幸いです。
はえ (2022-07-28 (木) 23:27:37)
各⾃に割り当てられた、個体の区画ごとの個体数のデータを使い、そのデータが5%⽔準でランダム分布と有意な差があるといえるかを、R のプログラムを作成して使⽤し、モンテカルロ検定する。以下の3つは必ずつける:(1)その検定結果、(2)モンテカルロ検定を⾏う R の⾃作プログラム、(3)モンテカルロ・サンプルの検定統計量のヒストグラム。
データ
2,0,0,5,0,0,0,3,4,18,0,3,2,6,0,4,0,2,0,3,3,9,5,0,0,15,0,6,0,4,0,0,5,0,0,0,0,2,0,9,0,0,0,2,0,0,0,0,0,0,0,5,0,0,5,6,3,0,0,0,0,0,5,0,3,12,5
総個体数=156 区画数=67
shima (2022-06-02 (木) 10:57:46)
患者毎の薬剤投薬状況の一覧を、見やすい形で提示したいのですが、データ加工方法がわからず悩んでいます。
データの加工方法についてアドバイス頂けませんでしょうか?ネットや本Wikiで検索してもわからなくて・・・
元のデータ
患者ID, 薬剤名, 投与量, 診断日, 投与日(診断日からの経過日数)
1111, drugA, 3, 2022/1/1, day2
1111, drugA, 2, 2022/1/1, day4
1111, drugA, 5, 2022/1/1, day8
1111, drugB, 1, 2022/1/1, day1
1111, drugB, 2, 2022/1/1, day4
1111, drugB, 1, 2022/1/1, day5
1111, drugB, 3, 2022/1/1, day6
1112, drugA, 7, 2022/1/7, day5
1112, drugA, 5, 2022/1/7, day8
1112, drugA, 2, 2022/1/7, day32
・
・
・
最終的に提示したい形
患者ID, 薬剤名, 診断日, (投与日毎の投与量)day0,day1,day2,day3,day4,day5,day6,day7,day8,day9,day10,day11,day12,day13・・・
1111, drugA, 2022/1/1, 0,0,3,0,2,0,0,0,5,0,0,0,0,・・・・
1111, drugB, 2022/1/1, 0,1,0,0,2,1,3,0,0,0,0,0,0,・・・・
1112, drugA, 2022/1/7, 0,0,0,0,0,7,0,0,5,0,0,0,0,・・・・
上記の最終的に提示したい形は、エクセルで見る場合に見やすいかな、と考えているものです。上記以外にも見やすい形式等のアドバイスをいただければ幸いです。
山本 (2022-05-18 (水) 12:10:24)
R-Tipsのウェブサイト(http://cse.naro.affrc.go.jp/takezawa/r-tips/r.html)が消えてしまったようですが、もしどこかに引っ越したのであれば、新しいURLをご存知の方、いらっしゃいませんか?
かず (2022-04-03 (日) 08:56:09)
Rjpwiki内で特定の語句を検索しても文字化けで見れません(?ばかりが並びます)
対処方法をご教授いただけると助かります
Microsoft EdgeやSafariいずれもです。
ずっと初心者 (2021-12-28 (火) 11:36:13)
Windows10 で Rを使っています。
openxlsx パッケージを使って Excelファイル の読み書きをしたところ,
修正後のファイルでは 修正前に入力してあった日本語の文字列が化けてしまいました。
対処法をご存知の方がいらっしゃいましたら ご教示ください。options(encoding="UTF-8") wb <- openxlsx::loadWorkbook("sample1.xlsx") openxlsx::addWorksheet(wb, "A new worksheet") openxlsx::writeData(wb = wb, sheet = 1, x = "え", startCol = 8, startRow = 1) openxlsx::saveWorkbook(wb, "sample1_mod.xlsx", overwrite = TRUE)
使用環境は下のとおりです。
> sessionInfo() R version 4.1.2 (2021-11-01) Platform: x86_64-w64-mingw32/x64 (64-bit) Running under: Windows 10 x64 (build 19043) Matrix products: default locale: [1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 [3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C [5] LC_TIME=Japanese_Japan.932 attached base packages: [1] stats graphics grDevices utils datasets methods base loaded via a namespace (and not attached): [1] compiler_4.1.2 tools_4.1.2 Rcpp_1.0.7 stringi_1.7.6 [5] zip_2.2.0 openxlsx_4.2.5
> strsplit(Sys.getlocale(), ";")[[1]] [1] "LC_CTYPE=ja_JP.UTF-8" "LC_NUMERIC=C" [3] "LC_TIME=ja_JP.UTF-8" "LC_COLLATE=ja_JP.UTF-8" [5] "LC_MONETARY=ja_JP.UTF-8" "LC_MESSAGES=ja_JP.UTF-8" [7] "LC_PAPER=ja_JP.UTF-8" "LC_NAME=C" [9] "LC_ADDRESS=C" "LC_TELEPHONE=C" [11] "LC_MEASUREMENT=ja_JP.UTF-8" "LC_IDENTIFICATION=C"取り急ぎの参考情報まで。 -- 2021-12-28 (火) 19:58:33
> options(encoding="UTF-8")
> wb <- openxlsx::loadWorkbook("sample1.xlsx")
警告メッセージ:
readLines(x, warn = FALSE, encoding = "UTF-8") で:
入力コネクション 'C:/Users/xxx/AppData/Local/Temp/RtmpXXXXXX/fileXXXXXXXXXXXX/xl/theme/theme1.xml' に不正な入力がありました
ちなみにoptionsでUTF-8を指定しなかった場合でも、エラーは違いますが本質的には同様の理由でコケる感じですね。 -- 2022-01-04 (火) 12:22:56
> wb <- openxlsx::loadWorkbook("sample1.xlsx")
substring(x[ind], so, eo) でエラー:
'<83><e3><82>ッ"/>' に不正なマルチバイト文字があります> Sys.setlocale("LC_ALL", "English")
[1] "LC_COLLATE=English_United States.1252;LC_CTYPE=English_United States.1252;LC_MONETARY=English_United States.1252;LC_NUMERIC=C;LC_TIME=English_United States.1252"
> strsplit(Sys.getlocale(), ";")[[1]]
[1] "LC_COLLATE=English_United States.1252"
[2] "LC_CTYPE=English_United States.1252"
[3] "LC_MONETARY=English_United States.1252"
[4] "LC_NUMERIC=C"
[5] "LC_TIME=English_United States.1252"
> wb <- openxlsx::loadWorkbook("sample1.xlsx")
> openxlsx::addWorksheet(wb, "A new worksheet")
> openxlsx::writeData(wb = wb, sheet = 1, x = "え", startCol = 8, startRow = 1)
> openxlsx::saveWorkbook(wb, "sample1_mod.xlsx", overwrite = TRUE)
結果、Linuxで出力されたのと同様の、文字化けのないsample1_mod.xlsxファイルができました。ただし注意として、その場合でもoptionsでUTF-8を指定すると失敗するようになります。(よくわかりませんw) Sys.setlocale("LC_ALL", "English")
wb <- openxlsx::loadWorkbook("sample1.xlsx")
openxlsx::addWorksheet(wb, "A new worksheet")
openxlsx::writeData(wb = wb, sheet = 1, x = "え", startCol = 8, startRow = 1)
openxlsx::saveWorkbook(wb, "sample1_mod.xlsx", overwrite = TRUE)ともちゃん (2021-12-04 (土) 15:43:31)
Rで反イメージ相関係数を計算したいのですがわかりません。
SPSSでの反イメージ相関係数の出力結果のような感じなのをRでもできるのでしょうか?ご教示いただけましたらありがたいです。
もいくんとぅ (2021-08-21 (土) 21:38:06)
Rコマンダー、EZR、その他の必要なパッケージがインストールできないので、インストールできる方法をお教えして頂ければ有り難いです。
使用OSは、Mac Book Pro 15inch Mid 2009 Mac 、Mac OS X Lion 10.7.5
Rのバージョンは、R-3.2.1-snowleopard.pkg
①CRANのホームページからインストーラーをダウンロードしインストール済みです。
②XQuartzプロジェクトからX11システムをインストール済みです。
③Rというアプリケーションを開き、install.packages("RcmdrPlugin.EZR", dependencies=TRUE)と入力、returnキーを押すが、下記エラーメッセージが表示されてしまいます。
【警告: リポジトリー https://ism//cran.ism.ac.jp/src/contrib に対する牽引にアクセスできません。警告: リポジトリーhttps://ism//cran.ism.jp/bin/ Macosx/contrib/3.2に対する牽引にアクセスできません。警告メッセージ:package ’Rcmdrplugin.EZR'is Not available(for R version3.2.1】
CRANミラーをJapan(Tokyo),Japan(Yonezawa)に変更して試してみましたが、上記同様のメッセージが表示されてしまします。
解決方法をお願い致します。
Rビギナー (2021-08-20 (金) 01:35:36)
コマンダーにおいて、パッケージ内のデーター→アタッチされたパッケージからデータセットを読み込む を行っても、CarDataとsandwichしかなく、datasetsが表示されません。どうしたらいいでしょうか、お教え願います。
よしの (2021-07-07 (水) 15:59:48)
多変量正規分布の積分を行いたいのですが,使用できる関数がわかりません.
正規分布(1変量)の場合,f_n <- function(x) dnorm(x, myu, sigma) integrate(f_n, -Inf, Inf)で計算でき,このような例はいくつかのサイトに示されていました.
この1変量の場合と同様に,多変量でも積分を行いたいです.
実際に試したいのは,3変量の場合で
f_mv <- function(x1, x2, x3) dmvnorm(cbind(x1, x2, x3), mean=Mu1, sigma=Si1)
と,関数を定義して
これをx1, x2, x3について積分したいのですが,integrateを用いた場合の設定がわかりません.
他の方法でも良いので,なにか情報いただければと思います.
よろしくお願いします.
RStudio ver 1.3.1093
library(cubature)
library(mvtnorm)
Mu <- c(0, 1, 2)
Sigma <- diag(length(Mu))
f_mv <- function(x) { dmvnorm(x, mean = Mu, sigma = Sigma) }
hcubature(f_mv, c(-3, -2, -1), c(1, 2, 3))
パッケージのヘルプとVignetteを読んで使ってみてください。(わたしは斜め読みしただけなので。)どの程度の精度が必要かにもよるかもしれません。hcubatureはC言語で書かれた関数実体へのラッパーとのことなので、必要があればそちらのコードを確認するべきかと思います。 -- しろうと 2021-07-08 (木) 08:12:13まるせん (2021-06-29 (火) 18:13:37)
被説明変数に0,1の指標、説明変数に4項目程度の連続データでプロビット分析を行っています。データ数は400程度となります。
Rでglmを使って無事モデルは構築できたのですが、右辺の被説明変数(標準正規分布の引数の部分)が標準正規分布に従っていません。(分布の形も正規分布ではなく、平均、分散もそれぞれ0,1ではありません。)
そもそも右辺の被説明変数には標準正規分布の仮定を置いていないと思っていますが、私の考えがあっているか教えてください。前提は誤差項の正規性ですよね。
これ読んどけ、というレポートを教えていただけるだけでもうれしいです。
お願いします。
つぐ太郎 (2021-06-19 (土) 15:47:10)
年齢と死亡率が普通メモリグラフでは曲線ですが、両対数目盛グラフでは直線になることを示したいのですが、その時の回帰直線がうまく書けません。試したプログラムは下記です。
age <- c(20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85)
mortality <- c(2, 5, 10, 20, 25, 37, 60, 80, 133, 190, 280, 410, 420, 380)
plot(mortality ~ age, log = "xy")
If <- glm(mortality ~ 1 + age)
cf <- If$coefficients
curve(cf[1] + cf[2] * x, from = 20, to = 80, col = "blue", cex=1.5, lwd=2, add = T)
rslt <- lm(log10(mortality) ~ log10(age)) summary(rslt)としてやればよいわけです。
cf <- coef(rslt)
par(mfrow = c(1, 2))
for (l in c("", "xy")) {
plot(mortality ~ age, log = l)
lines(age, 10 ^ (cf[1] + cf[2] * log10(age)))
}KF (2021-04-10 (土) 20:44:50)
以下を参考にrstanをダウンロードしようとしています。
http://www.eeso.ges.kyoto-u.ac.jp/emm/materials/bayesian/stan_install
rstan自体はダウンロード出来たのですが、rstanを使用するためにインストールしたrtoolsのパスを開こうとした際以下のエラーが出ました。
コード
writeLines('PATH="${RTOOLS40_HOME}\\mingw64\\bin;${RTOOLS40_HOME}\\usr\\bin;${PATH}"', con = "~/.Renviron")
警告メッセージ:file(con, "w") でエラー: コネクションを開くことができません 追加情報: 警告メッセージ: file(con, "w") で: ファイル 'C:/Users/*****/OneDrive/??????/.Renviron' を開くことができません: Invalid argument (*****=ユーザー名)
上のリンクでは「RでHomeとして使っているディレクトリ(デフォでgetwd()で表示されるパス)に,.Renviron というファイルが出来ているはず.」と書かれていましたが、そのようなファイルは作成されていないようでした。そのためエラーメッセージが発生したとみられます。
Rtoolsの設定がうまくいっていないせいか、rstanを起動しても以下のエラーメッセージが出ます。
プロシージャエントリポイント EXTPTR_PTRがダイナミックリンクライブラリC:\Users\*****\R\win-library\4.0\rstan\libs\x64\rstan.dllから見つかりませんでした(*****=ユーザー名)
操作環境は以下の通りです。
R version 4.0.0 (2020-04-24)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 19042)
解決方法をご教示いただけると幸いです。
よろしくお願いいたします。
Gorou (2021-04-07 (水) 20:41:39)
質問です。"UTF-8"ファイルの読み込み時に文字化けしてしまい困っています。
(Wikiの記法に不慣れな点はお許し願います。)
read.table()での文字化けは非常にありがちな質問ですが、
"read.table()"で読み込んだデータが"「部分的に」文字化け"してしまい、
困っています。
具体的には次のような症状です。
以下のデータファイルを読み込みたいのですが、[data.tsv] (TSV形式、UTF-8、BOMなし) Age Group Class DBH_mean DBH_sd 31 すべて 全立木 17.9 4.34 31 すべて 生立木 17.9 4.34 31 すべて 残存木 17.9 4.34 36 すべて 全立木 20.0 4.92 36 すべて 生立木 20.0 4.92 36 すべて 残存木 20.0 4.92 41 すべて 全立木 21.9 5.59 41 すべて 生立木 21.9 5.56 41 すべて 残存木 21.9 5.56
>df <- read.table("test.tsv",sep="\t",header=TRUE,skip=0, fileEncoding="UTF-8") >head(df)
とすると、
Age Group Class DBH_mean DBH_sd X 1 31 すべて \u5168\u7acb\u6728 17.9 4.34 NA 2 31 すべて \u751f\u7acb\u6728 17.9 4.34 NA 3 31 すべて \u6b8b\u5b58\u6728 17.9 4.34 NA 4 36 すべて \u5168\u7acb\u6728 20.0 4.92 NA 5 36 すべて \u751f\u7acb\u6728 20.0 4.92 NA 6 36 すべて \u6b8b\u5b58\u6728 20.0 4.92 NA ("\"はバックスラッシュ)
となり、「Group」のカラムは文字化けしないのに、「Class」のカラムは文字化けしてしまいます。完全に文字化けしているのならば文字コードの間違いだと納得できるのですが、
このような中途半端な文字化けは、いろいろググって見たのですがさっぱり分かりません。
また、DBH_sd の右に "X"のカラムができてしまうのも不可解です。DBH_sdのカラム、4.34, 4.34, 4.34, ... は数値の直後で改行されています(つまり元のファイルにはヘッダだけのカラムは無い)。
TSVをCSVにしたり、ファイル形式をShift-JISにして fileEncoding='Shift_JIS'としてみても、結果はほぼ同じで途方に暮れております。
おまじないにと,stringAsFactorsをTRUEにしたりFALSEにしても変わりません(変わるとも思えませんが)。
環境はWindows10 64bit Pro、Rのバージョンは64bitの4.0.4、Rstudioは1.4.1106。症状はRGuiでもRStudioでも同じです。
どなたか解決方法をご教示いただけましたら幸いです。何卒よろしくお願いします。
> df <- read.table("test_utf8.tsv", sep = "\t", header = TRUE, skip = 0, fileEncoding = "UTF-8")
> head(df)
Age Group Class D_mean H_mean N G V
1 31 すべて 1)間伐前 17.9 12.3 1535 40.95 260.0
2 31 すべて 2)間伐後 17.9 12.3 1535 40.95 260.0
3 36 すべて 1)間伐前 20.0 14.9 1535 51.33 393.1
4 36 すべて 2)間伐後 20.0 14.9 1535 51.33 393.1
5 41 すべて 1)間伐前 21.9 16.6 1535 61.40 517.8
6 41 すべて 2)間伐後 21.9 16.6 1530 61.36 517.6
なおこちらは、R version 4.0.4 on Ubuntu 16.04 LTSをbash上で起動しています。 -- 2021-04-09 (金) 17:09:52はい、その通りです。データを確認していただき、ありがとうございます。ちょっと書き換えましたがほぼ同様のデータです。
理由はさっぱり分からないのですが、Rを4.0.4から4.0.5にアップデートしたら文字化けしなくなりました(RStudioもRGuiも)。
違う点としては4.0.5は管理者として、4.0.4は一般ユーザとしてインストールしたことです。 (管理者だとC:\Program Files\R\R-4.0.5に、一般ユーザだとC:\Users\hogehoge\Documents\R\R-4.0.4にインストールされていました。)
それにしても、元のデータは
すべて 全立木
であるのに「文字化け」状態だと
すべて \u5168\u7acb\u6728
と部分的に化けてしまうと言うのはさっぱり訳が分かりません。 いずれにしても、これまでのデータやスクリプトが問題なく使えるので安心しました。 助言をくださったみなさん、どうもありがとうございます。
(ちなみにtidyverseのread_csvも、再確認したところ4.0.5にアップデートする前にはなぜか文字化けしてました。こっちも何が何やら。)
HO0217 (2020-12-29 (火) 21:28:39)
「赤札と青札がある。確率1/2でどちらかを引く。
これを100回繰り返し、赤札を55回以上引く確率を求めよ。」
という問題を解くため、正規分布に近似して確率を求めると、
15.87%でした。
(これは私の計算結果で、答えは知りません。あってるとは思いますが、間違っているかもしれません)
近似ではなく、二項分布で計算してみようと思い、
以下のコードで確率を計算すると、約18.41%(出力は0.1841008)で、
近似とはやや違う結果となりました。
tmp_nu<-0
n_nu<-100
k_nu<-55
for(i in k_nu:n_nu){tmp_nu<-( choose(n_nu,i)*(1/2)^(i+(100-i)) )+tmp_nu}
tmp_nu
次に、シミュレーションで確率を見積もるために以下のコードを実行しました。
出力は0.13554となりました。
simu_0_nu<-100 #100枚引く。
simu_1_nu<-1000000 # 何セット試すか。
result_0_ve<-rep(NA,simu_0_nu)
result_1_ve<-rep(NA,simu_0_nu)
result_2_ve<-rep(F,simu_1_nu)
set.seed(123)
for(i in 1:simu_1_nu){for(j in 1:simu_0_nu){ result_0_ve<-rnorm(simu_0_nu,0,1) #平均0の正規分布は確率1/2で0を越える数値を出す result_1_ve<-result_0_ve>0 #0を越えたら真 } result_2_ve[[i]]<-sum(result_1_ve)#真の数を数える}
sum(result_2_ve>55)
sum(result_2_ve>55)/simu_1_nu
ちなみに、set.seed(123)を
set.seed(777)にすると0.135447
set.seed(789)にすると0.136265
set.seed(111)にすると0.136006
set.seed(222)にすると0.135414
となりました。
どうやら偶然ではなく、私がシミュレーションに使ったコードでは、
他の計算よりも小さい値になっているようです。
何が原因でシミュレーションと二項分布の計算でこんなにも違ってしまうのでしょうか?
わかる方がいらっしゃいましたら、教えてくださると助かります。
よろしくお願いいたします。
> set.seed(1)
> N <- 1000000
> n <- replicate(N, {sum(rnorm(100) > 0)})
> sum(n > 55) / N
[1] 0.135948
> 1 - pbinom(55, 100, 0.5)
[1] 0.1356265
> sum(n >= 55) / N
[1] 0.184649
> 1 - pbinom(54, 100, 0.5)
[1] 0.1841008元Rユーザー (2020-12-27 (日) 22:33:22)
全盛期とは比較にならないほどの過疎ぶりですね・・・時代は完全にpythonになったのだなと思います。
R初心者 (2020-11-08 (日) 22:39:38)
パネルデータでplm分析を行ってますが、下記のメッセージが出て対応に苦慮しております。
警告メッセージ:
1: pdata.frame(data, index) で:duplicate couples (id-time) in resulting pdata.frame to find out which, use e.g. table(index(your_pdataframe), useNA = "ifany")2: is.pbalanced.default(index1, index2) で:
duplicate couples (id-time)
恐らく重複エラーなのかもしれませんが、下記を見ると年次などの下線部がエラーの様ですが(?)、削除はしたくなく対応方法を知りたいです。宜しくお願いいたします。
コード PBR 業種コントロール 年次 `1人当たり単体人件費`… 連結子会社数 売上高研究開発費 <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> 1 T1301 0.915 0.630 2019 5.87 26 0.0013 2 T1301 0.908 0.618 2018 5.88 24 0.00120 3 T1301 0.872 0.644 2017 6.18 24 0.0011 4 T1301 0.867 0.678 2016 6.15 25 0.00120
Rビギナー (2020-10-28 (水) 01:22:23)
SVM実装の際に、package(kernlab)を使用しています。ksvm関数を使用すると以下のようなエラーが出ます。これはどういう意味でしょうか?
SVM <- ksvm( data_Mic.dummy ~AveMaxTemp7+TP1yp+TN1yp+AveWind7, #モデル式data=data_Mic, kernel ="rbfdot", #カーネル関数の設定 kpar=list(sigma=0.2), #カーネル関数のパラメータ C=1, cross=10).local(x, ...) でエラー:
No Support Vectors found. You may want to change your parameters 追加情報: 警告メッセージ: .local(x, ...) で: 強制変換により NA が生成されました
R初心者 (2020-10-20 (火) 12:55:09)
R初心者で色々わかっていないので、質問の仕方が間違ってたらすみません。
Rで関数の処理を行いたいのですが、
解析結果のデータは
dds <- 解析結果
に格納しました。
その大量のデータの中で図示したい5つだけをリストアップし
dataという下記のようなデータフレームを作りました。
name ID
1 aaaaa 001
2 bbbbb 002
3 ccccc 003
4 ddddd 004
5 eeeee 005
処理したい関数が下記のように3つでpng→plotCount→dev.offでワンセットです。
図示したいのが1つであれば
png("aaaaa.png")
plotCounts(dds, gene="001", intgroup="condition" )
dev.off
でデータフレームの一行目のものが図として保存されます。
ですが連続してdataの二行目、三行目...と自動でループ処理させたくて、
forやlapplyなどを試したのですが、引数がnameとIDの二つということもあり、うまくプログラムを回せませんでした。
どなたかお詳しい方に上記の関数でpng→plotCount→dev.off→png→plotCount→dev.off→...を自動的に回す方法をご教授頂きたいです。
よろしくお願いします。
for(i in 1:nrow(dds)){
name_ <- dds$name[i];
ID_ <- dds$ID[i]
}
ループの中でpng, plotCount, dev.offを順次呼び出せば動くと思います。
うみねこ (2020-07-24 (金) 22:13:58)
R version 3.6.1 (2019-07-05)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 18363)
回帰直線の描き方と、その統計情報の出し方について、教えて頂けないでしょうか。
9列×6行の表があり、その2列目(length)と6列目(weight)にlog変換をして、RMA法を使って回帰直線を描きたいです。gx.rmaというファンクションを見つけましたが、以下のコードではエラーが出て動きません。
出来たら、95%予測区間や95%信頼区間等についても出し方を知りたいです。
どうぞご教授よろしくお願いいたします。
data<- read.table('body.txt', header=T)
row.names(data)=data[,1]
length <- setNames(log(data[,2]),data[,1])
weight <- setNames(log(data[,6]),data[,1])
model <-gx.rma(length,weight)
model
> set.seed = 1234567
> data = as.data.frame(matrix(runif(54), 6, 9))
> data[,1] = letters[1:6]
> row.names(data)=data[,1]
> length <- setNames(log(data[,2]),data[,1])
> weight <- setNames(log(data[,6]),data[,1])
> model <-gx.rma(length,weight)
Reduced Major Axis for length and weight
Means = -1.05 -0.679
SDs = 0.7428 0.8641
Corr = 0.5295
N = 6
Fit = 28%
SE 95% CLs
Slope = 1.163 0.4028 0.1277 <-> 2.199
Intercept = 0.5422 0.518067 -0.7895 <-> 1.874
Accept hypothesis that RMA is (0,1)
> model
$n
[1] 6
$mean
[,1] [,2]
[1,] -1.049833 -0.6789889
$sd
[,1] [,2]
[1,] 0.7428316 0.8640773
$corr
[1] 0.5295494
$a0
[1] 0.5421989
$a1
[1] 1.163221
$sea0
[1] 0.5180675
$sea1
[1] 0.4028333
他の人が追試できるように質問すべし。 -- 2020-07-26 (日) 17:41:28ifplot if a x-y plot of the independent variables is required set ifplot = TRUE. The plot is equi-scaled and the 1:1 line is added. If ifrma = TRUE ifplot will be set to TRUE.よくわからないまま,適当にプログラムを書いてもだめです。オンラインヘルプくらい読みましょう。それと,エラーメッセージを良く読めば,どこが間違えているか対処法もわかります(わかりやすく書かれているとはいえないでしょうが,それが問題解決の唯一の鍵ですからね) -- 2020-07-27 (月) 14:41:47
Usage
gx.rma(xx1, xx2, x1lab = NULL, x2lab = NULL, log = FALSE, ifplot = FALSE,
ifrma = FALSE, ifcoeffs = FALSE, ifform = FALSE, iftest = FALSE, ...)
あなたの例の場合だと,
gx.rma(length, weight, x1lab="Length(cm)", x2lab="Weight(g)", ifplot=TRUE, ifrma=TRUE)のようになる。 -- 2020-07-27 (月) 16:27:48
catdogcat (2020-07-24 (金) 18:20:51)
マッチングを試みましたが下記のエラーが出ました
Treatment indicator ('Tr') must be a logical variable---i.e., TRUE (1) or FALSE (0)
比較する群の変数は因子としてあり 0.1 となっているのですが
解決策がわかりません。
ご教授お願い致します。
(2020-07-22 (水) 23:44:38)
下記コードをpgm.cppに保存後、Rcpp::sourceCpp("pgm.cpp")でコンパイルし
test(matrix(c(1,2,3,4),2,2))を実行したのですが1行1列の値が返ってこずエラーでRが落ちます。もし良ければご助言いただけないでしょうか。
#include<Rcpp.h> #define MAX 100 using namespace std; // [[Rcpp::export]] int test(Rcpp::IntegerMatrix mtx){ int matrix[MAX][MAX]; for(int i=0; i < mtx.rows(); ++i){ for(int j=0; j < mtx.cols(); ++j){ matrix[i][j] = mtx(i,j); }} return matrix[0][0]; }
がんば (2020-07-22 (水) 08:35:12)
昨年はおせわになりました。実験結果をグラフにすると直線状ではなく「くの字」状に曲がった結果となる場合もあるのですがこの時の特異点をRで一発算出するような手段がないでしょうか?「データをソートして下限側から徐々に近似曲線を構築するのと上限側から構築するのをそれぞれ行っていって次の点がそれまでの近似曲線のバラツキ範囲外になる点を求める」ような操作をする(人の目視確認の場合はこんな手順だと思います)手段を意図してます。
動作環境:sessionInfo()
R version 4.0.2 (2020-06-22)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows 10 x64 (build 18363)
Matrix products: default
locale:
[1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932 LC_MONETARY=Japanese_Japan.932
[4] LC_NUMERIC=C LC_TIME=Japanese_Japan.932
attached base packages:
[1] stats graphics grDevices utils datasets methods base
loaded via a namespace (and not attached):
[1] compiler_4.0.2 tools_4.0.2
りょう (2020-06-30 (火) 21:38:04)
Rによる多変量解析と言う本でRを動かしているのですが、グラフ化すると日本語の部分が□□□のようになります。
よくわからないのが、"plot", "plotmeans"などでは日本語表示されるのですが、xyplot, histogramだと文字化けすることです。以下がその時のコードです。
jhk <- read.csv('人事評価結果.csv')
head(jhk, 3)
|ID|性別|部署|年代|協調性|技能|知識|ストレス|
|1|M|A部|中堅|70|65|71|53|
|2|F|B部|熟練|45|62|70|46|
・・・・・・
par(family = "HiraKakuProN-W3") #一応これ試してみました
library(lattice, gplots)
histogram(~ストレス, data = jhk, breaks = 20, type='count') #文字化けする
xyplot(知識~技能|年代+部署, data=jhk) #文字化けする
plotmeans(協調性~性別, data=jhk, p=0.95, ylim=c(49, 54)) #文字化けしない
・macOS Catalina バージョン10.15.5
・R studio version 4.0.2 です。この前に使用していたバージョン3.6.3でも同様の状態でした。
・CSVファイルはUTF-8で保存されています。
・par(family = "HiraKakuProN-W3") なども試しました。
GoogleやCRAN, マニュアルなどで既に調べたのですがわからず、困っているのでご教授いただけると幸いです。
pdf("test.pdf") # ひつようなら他のパラメータの設定も
histogram(~ストレス, data = jhk, breaks = 20, type='count') #文字化しない
xyplot(知識~技能|年代+部署, data=jhk) #文字化けしない
plotmeans(協調性~性別, data=jhk, p=0.95, ylim=c(49, 54)) #文字化けしない
dev.off()
どんなもんだ? -- 2020-07-14 (火) 14:04:24ぼう (2020-05-14 (木) 12:25:32)
R4.00でggplotを使おうと思っていますが、エラーが出てしまいます。
次のコマンドを実行すると、下のような結果が出てきてしまいます。> install.packages("ggplot2", dependencies = TRUE) > library(ggplot2) > str(iris) 'data.frame': 150 obs. of 5 variables:~ $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ... $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ... $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ... $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ... $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ... > ggplot(data=iris,aes(x=Sepal.Length,y=Sepal.Width))~ ggplot(data = iris, aes(x = Sepal.Length, y = Sepal.Width)) でエラー: 関数 "ggplot" を見つけることができませんでした
show (2020-03-10 (火) 12:58:28)
ccf(rw,rw2)と入力した場合、どちらを基準にしてlagを取っているのでしょうか?
> set.seed(12345) > n <- 10 > x <- rnorm(n) > y <- rnorm(n)ラグ lag = 2,で ccf してみる
> print(ccf(x, y, lag, plot=FALSE), digits=7)
Autocorrelations of series ‘X’, by lag
-2 -1 0 1 2
0.1416173 0.5327757 -0.2716881 0.2595251 -0.1681542
自分で相互相関係数を計算するときに必要なもの。
> mean.x <- mean(x) # x の平均(全部使う) > mean.y <- mean(y) # y の平均(全部使う) > ss.x <- var(x)*(n-1) # x の変動(全部使う) > ss.y <- var(y)*(n-1) # y の変動(全部使う)相互相関係数を計算する。lag = 2
> sum((x[(lag+1):n]-mean.x) * (y[1:(n-lag)]-mean.y) / sqrt(ss.x * ss.y) [1] -0.1681542上の ccf の lag = 2 の下に書かれている数値 -0.1681542 と一致しているでしょ。
私は誰でしょう? (2020-03-01 (日) 21:20:52)
今まで,最新の R にするために(新しもの好きなので)
あ,見ての通り,Mac ユーザです。
curl https://mac.r-project.org/el-capitan/R-3.6-branch/R-3.6-branch-el-capitan-sa-x86_64.tar.gz | sudo tar fvxz - -C /
をやっていたのですが,2 ヶ月ほど前から,実行結果が思わしくなくなっています。
つまるところ,件の URL はアクセス不可ということのようで,アップデートは出来ません。
念の為
http://mac.r-project.org/el-capitan/R-3.6-branch/R-3.6-branch-el-capitan-signed.pkg
を使えばアップデートは出来ます。
curl -O http://mac.r-project.org/el-capitan/R-3.6-branch/R-3.6-branch-el-capitan-signed.pkg sudo installer -pkg ./R-3.6-branch-el-capitan-signed.pkg -target /とかでお茶を濁しては... -- 何処の誰かは知らないけれど 2020-03-02 (月) 16:03:50
Rビギナー (2020-02-26 (水) 10:12:45)
お世話になります.
下記のように1~3まで連番のファイルを読み込みたいのですが,例えば2番のファイルが無い場合にそのまま1,3のファイルを読み込んでプログラムが止まらないようにするにはどうしたらいいでしょうか.
try関数で可能かと思いやってみたのですが,だめでした.
ご教示ください.
よろしくお願いいたします.
==ここからpoint <- 1:3 # for文を回す数を設定 for (i in point) { mypath <- file.path(getwd(), paste0(i,"t2-1.csv")) df <- assign(paste0("data",i), read.csv(mypath, header = T)) df <- data.frame(df) df head(df) df$X <- strptime(df$X,"%Y/%m/%d %H:%M'%S") head(df) df$X <- as.POSIXlt(df$X,"%Y/%m/%d %H:%M:%S",tz="GMT0") head(df) #https://qiita.com/tktz/items/733c37b1d6102ae52120 library(xts) df$X <- align.time(df$X-10*60/2,10*60) head(df) date = paste0("date",i) Temp = paste0("Temp",i) RH = paste0("RH",i) colnames(df) <- c(date,Temp,RH) head(df) mypath <- file.path(getwd(), paste0(i,"t1.csv")) write.csv(df,mypath) }
mypath <- file.path(getwd(), paste0(i,"t2-1.csv"))
if (! file.exists(mypath)) nextfiles = list.files(getwd(), pattern="[0-9]*t2-1.csv")
for (f in files) {
# describe what you want to do
}マリン (2020-02-23 (日) 10:13:04)
2つの時系列のデータ(約80個)の相関(どのくらい類似性があるか)を
見たいのですが、方法を教えてください。
(2020-02-14 (金) 14:34:30)
Book1というcsvファイルのデータを用いてGLMMによる解析を行ったのですが、固定効果の値を用いた回帰線がうまく引けません。
csvファイルおよび解析を行ったスクリプトをBook1GLMMというファイル名でアップロードしましたので、ご教授いただけますと幸いです。
よろしくお願いいたします。
data <- read.csv("Book1.csv")
data
# update.packages
# install.packages("lme4")
library(lme4)
par(family = "HiraKakuProN-W3")
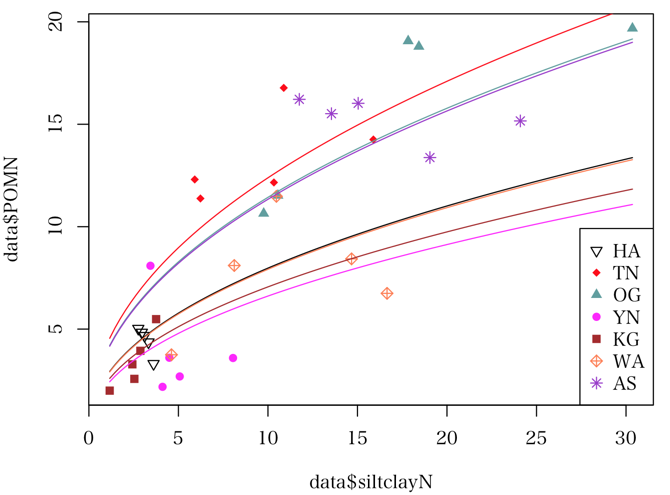
model1 <- glmer(POMN ~ log(siltclayN) + (1 | Place), family = Gamma(link = "log"), data = data)
summary(model1)
place = c("HA", "TN", "OG", "YN", "KG", "WA", "AS") # データフレームではこちらを使っているのでこれでなきゃだめ
place2 = c("半田山", "田野", "小川", "与那", "上賀茂", "西粟倉", "足寄") # 凡例用には別のものを用意する
pch = c(25, 18, 17, 16, 15, 9, 8)
col = c("black", "red", "cadetblue", "magenta", "brown", "coral", "darkorchid")
plot(data$siltclayN, data$POMN, pch = rep(pch, each = 5), col = rep(col, each = 5), cex = 3, cex.axis = 2)
legend("bottomright", legend = place2, pch = pch, col = col, cex = 2)
xx <- seq(min(data$siltclayN), max(data$siltclayN), length = 100)
# yy <- exp(1.1136 + 0.4651 * xx) # そもそも間違い
# lines(xx, yy, lwd = 2)
for (i in seq_along(place)) { # 7 本の曲線を引くんでしょ?
newdata = data.frame(siltclayN=xx, Place=rep(place[i], 100))
yy = predict(model1, newdata=newdata, type="response")
lines(xx, yy, col=col[i])
}(2020-02-06 (木) 21:14:35)
GLMMなどで、目的変数に用いるデータが連続値で0以上であればガンマ分布を用いると統計の教科書等には書いてあるのですが、連続値で0以上(データの最小値は0)のデータでガンマ分布を用いて解析しようとすると
「non-positive values not allowed for the 'gamma' family」というエラーが出ます。
また、同じデータで線形混合効果モデルなら結果は出るのですが、「boundary (singular) fit: see ?isSingular」という警告?が出るせいなのかランダム効果を考慮した回帰線が一本しか出ません。
解決策などありましたら、お教えいただけますと幸いです。
> library(lme4)
> d = read.csv("data.csv")
> model1 <- glmer(A ~ log(B) + (1|Place),f amily=Gamma(link="log"),data=d)
eval(family$initialize, rho) でエラー:
non-positive values not allowed for the 'gamma' family
using d2
> d2 = d[d$A > 0, ] > model1-2 <- glmer(A~log(B) + (1|Place), family=Gamma(link="log"), data=d2) > model1-2no error happend.
model2<-lmer(NitMAOMrecover~MAOMCN+(1|Place),data=example)
summary(model2)
place=c("HA","TN","OG","YN","KG","WA","AS")
pch=c(25,18,17,16,15,9,8)
col=c("black","red","cadetblue","magenta","brown","coral","darkorchid")
plot(example$B,example$A,pch=rep(pch,each=5),col=rep(col,each=5),cex=3,cex.axis=2)
legend("bottomright",legend=place,pch=pch,col=col,cex=1.5)
xx<-seq(min(example$B),max(example$B),length=1000)
for(i in seq_along(place)){
yy<-predict(model2,newdata=data.frame(B=xx,Place=rep(place[i],1000)),type="response")
lines(xx,yy,col=col[i],lwd=2)}![[heart]](image/face/heart.png) 投稿文書の書式
投稿文書の書式 ![[heart]](image/face/heart.png) を読んでください" -- 2020-02-07 (金) 08:20:14
を読んでください" -- 2020-02-07 (金) 08:20:14ra (2020-01-27 (月) 11:58:45)
R初心者です。
GLMMで解析を行った後、散布図に回帰線を引きたいのですが、predict関数を読み込ませようとすると(オブジェクト"Place"がありません)というエラーが出ます(Placeはランダム効果です)。このエラーを解消し、回帰線を引くにはどうすれば良いでしょうか?
入力したコマンドを載せておきます。
よろしくお願いいたします。
model1<-glmer(POMN~log(siltclayN)+(1|Place),family=Gamma(link="log"),data=data)
plot(data$siltclayN,data$POMN,pch=c(25,18,17,16,15,9,8),cex=3,cex.axis=2)
legend("bottomright",legend=c("HA","TN","OG","YN","KG","WA","AS"),pch=c(25,18,17,16,15,9,8),cex=2)
xx<-seq(min(data$siltclayN),max(data$siltclayN),length=100)
yy<-predict(model1,newdata=data.frame(siltclayN=xx),type="response")
lines(xx,yy,lwd=2)
POMN <- c(data$POMN)
siltclayN <- c(data$siltclayN + Place)
dataframe <- data.frame(POMN, siltclayN)
model1 <- glmer(POMN~log(siltclayN) + (1|Place1), family=Gamma(link="log"), data=dataframe)
plot(dataframe$siltclayN, dataframe$POMN, pch=c(25,18,17,16,15,9,8), cex=3, cex.axis=2)
legend("bottomright", legend=c("HA","TN","OG","YN","KG","WA","AS"), pch=c(25,18,17,16,15,9,8),cex=2)
xx <- seq(min(dataframe$siltclayN), max(dataframe$siltclayN), length=100)
yy <- predict(model1, newdata=data.frame(siltclayN=xx), type="response")
-- 2020-01-27 (月) 23:14:23> df = data.frame(x=c(3,2,1,3,5), f=factor(c(1,2,2,3,3)), y=c(2,4,7,5,6)) > fit = lm(y ~ x + f, data=df) # f may not be a factor > predict(fit) # this prediction uses data-frame df 1 2 3 4 5 2.0 5.4 5.6 5.7 5.3if you predict with another data-frame, the data-frame must contain 'x' and 'f' following newdata contains 'x' and 'f'. first 5 elements are same in df(df$x, df$f), last 3 elements is exactly new data
> newdata = data.frame(x=c(3,2,1,3,5, 2,3,5), f=factor(c(1,2,2,3,3, 1,2,2))) > predict(fit, newdata) 1 2 3 4 5 6 7 8 2.0 5.4 5.6 5.7 5.3 2.2 5.2 4.8 # first five values are identical to previous prediction. last 3 values are new prediction.do you understand?? -- 2020-01-28 (火) 16:28:06
siltclayN <- c(data$siltclayN + Place) dataframe <- data.frame(POMN, siltclayN)if Place is a factor variable you cant add to data$siltclayN. 'data$siltclayN + Place' cause an error. -- 2020-01-29 (水) 14:47:36
library(lme4)
data = read.csv("Book1.csv")
model1 <- glmer(POMN ~ log(siltclayN) + (1 | Place),
family = Gamma(link = "log"), data = data)
summary(model1)
place = c("HA", "TN", "OG", "YN", "KG", "WA", "AS")
pch = c(25, 18, 17, 16, 15, 9, 8)
col = c("black", "red", "cadetblue", "magenta", "brown", "coral", "darkorchid")
plot(data$siltclayN, data$POMN, pch = rep(pch, each = 5),
col = rep(col, each = 5)) # , cex=3, cex.axis=2)
legend("bottomright", legend = place, pch = pch, col = col) # ,cex=2)
xx <- seq(min(data$siltclayN), max(data$siltclayN), length = 100)
for (i in seq_along(place)) {
yy <- predict(model1,
newdata = data.frame(siltclayN = xx, Place = rep(place[i], 100)),
type = "response")
lines(xx, yy, col = col[i])
}
(2020-01-26 (日) 22:45:01)
半角・全角が混在する文字列からバイト数を指定して、部分文字列を抽出したいのですが、どのような関数がありますでしょうか。
例えば
x <- "あaいbうc"
という文字列から3-5byte目を抽出したいです。
("aい"という部分文字列を抽出したいです。)
substringは文字数を指定する関数のため、byte単位で抽出が不可能なようです。
お手数ですがご教示お願いいたします。
> x <- "あaいbうc" > charToRaw(x) [1] e3 81 82 61 e3 81 84 62 e3 81 86 63「あ」は3バイト文字なので途中で切られて、3-5byte目は「82 61 e3」です。これは「あ」の3バイト目、1バイト文字の「a」、「い」の1バイト目です。
> x <- "あaいbうc" # UTF-8: あ == \xe3\x81\x82 > xx = iconv(x, "utf-8", "euc-jp") > xx [1] "\\xa4\\xa2a\\xa4\\xa4b\\xa4\\xa6c" # EUC-JP: あ == \xa4\xa2 > Encoding(xx) = "bytes" > iconv(substr(xx, 3, 5), "euc-jp", "utf-8") [1] "aい"cp932 uses 2 bytes for 1 japanese character, too
> x <- "あaいbうc" > yy = iconv(x, "utf-8", "cp932") > yy [1] "\x82\xa0a\x82\xa2b\x82\xa4c" # CP932: あ == \x82\xa0 > Encoding(yy) = "bytes" > iconv(substr(yy, 3, 5), "cp932", "utf-8") [1] "aい"do you understand? -- 2020-01-28 (火) 11:38:51
TY (2020-01-07 (火) 15:59:04)
シミュレーションで同じ関数を繰り返し(10^5~10^6回程度)実行する場面を想定しています.
例えば,以下のsample_dataでREP毎にlm(y~x)を実行したいです.
for文で実行することはできるのですが,一般にapply系関数の方が実行が速いと聞き,
探しています.apply系関数に関する知識は,tapply関数にmean関数を適用できる,
tapply(sample_data$y, sample_data$REP,mean)
程度のレベルです.
【sample_data】y x REP1 133 3.9 1
2 81 -2.3 1
3 104 -1.2 1
4 89 0.7 1
5 142 4.0 1
6 74 -2.9 1
7 140 3.9 1
8 170 4.4 1
9 128 1.6 1
10 107 1.2 1
11 49 -4.3 2
12 79 -2.9 2
13 77 -3.2 2
14 125 1.8 2
15 82 -1.1 2
16 132 2.6 2
17 99 -0.0 2
18 140 2.1 2
19 140 4.9 2
20 88 -1.1 2